| |
| Med Sci (Paris). 2014 October; 30(10): 851–853. Published online 2014 October 14. doi: 10.1051/medsci/20143010011.Potentiel pandémique des virus influenza aviaires circulant actuellement et ayant des caractéristiques proches du virus de 1918 Bruno Lina1,2* 1Laboratoire de virologie Est des Hospices Civils de Lyon et centre national des virus influenzae, institut de microbiologie, Centre de biologie et de pathologie Est, groupement hospitalier Est, 59, boulevard Pinel, 69772Bron Cedex, France 2Laboratoire VIRPATH, EA4610, faculté de médecine Lyon Est, UCBL, 7, rue Guillaume Paradin, 69372Lyon Cedex 08, France MeSH keywords: Animaux, Oiseaux, Maladies transmissibles émergentes, épidémiologie, virologie, Histoire du 20ème siècle, Histoire du 21ème siècle, Humains, Sous-type H5N1 du virus de la grippe A, physiologie, Grippe chez les oiseaux, Grippe humaine, histoire, Pandémies, Zoonoses |
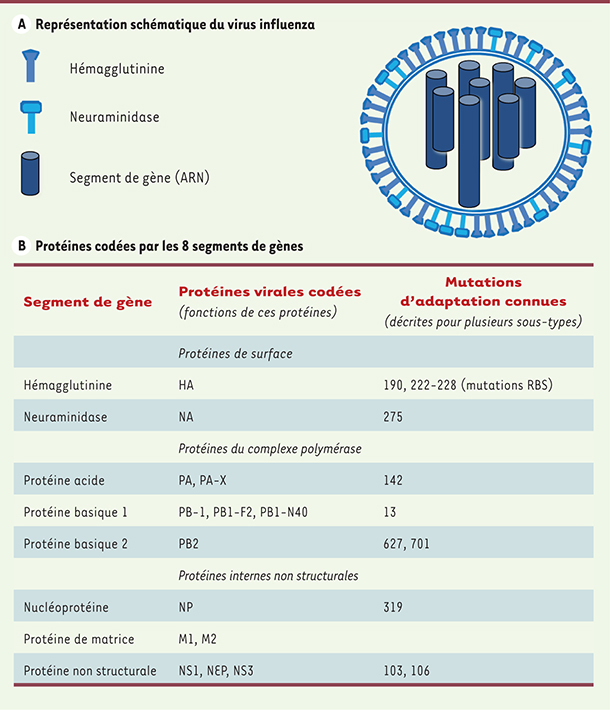
Les virus influenza de type A (Figure 1) sont des virus zoonotiques dont le réservoir est l’oiseau aquatique sauvage. Ces virus ont la caractéristique d’être potentiellement transmissibles à de nombreux hôtes mammifères chez lesquels ils peuvent s’adapter et persister. Chez l’homme, c’est cette introduction d’un virus influenza A à partir du réservoir animal qui provoque des pandémies [
1–
3]. L’adaptation de ces virus zoonotiques à l’homme semble suivre un chemin relativement stéréotypé, mais qui se fait sur une échelle de temps inconnue jalonnée de nombreux échecs. En effet, si on regarde le nombre d’infections humaines dues à des virus zoonotiques, on voit que dans l’immense majorité des cas, ces virus provoquent des cas sporadiques, parfois des cas groupés, mais pas de chaîne de transmission interhumaine (voir [
4] pour la liste des infections humaines à virus zoonotiques). Or, seule la mise en place d’un virus transmissible permet sa diffusion entraînant une pandémie.
 | Figure 1.
Présentation schématique d’un virus influenza. A. le virus comporte un génome segmenté avec 8 segments d’ARN simple brin de polarité négative capables d’être échangés entre virus par un mécanisme appelé réassortiment génétique. B. Tableau synthétique présentant les 8 segments de gène dont certains codent pour 1, 2 ou 3 protéines ; et, parmi ces protéines, certaines présentent des déterminants connus d’adaptation de l’oiseau vers le mammifère (numérotation des mutations connues indiquées sur la ligne correspondant à la protéine concernée, liste non exhaustive). |
Les mécanismes épidémiologiques, virologiques, moléculaires et immunologiques qui sous-tendent l’apparition des virus influenza pandémiques sont de mieux en mieux connus [
5]. Par ailleurs, le souvenir du cataclysme sanitaire qu’a représenté la pandémie H1N1 de 1918 (environ 40 millions de morts en 1 an, c’est-à-dire une moyenne de 100 000 décès par jour !) reste dans l’esprit des virologues qui travaillent sur la grippe [
6]. En miroir de la pandémie terrible de 1918, nous avons eu la pandémie modérée de 2009 (environ 450 000 décès, essentiellement en Afrique). Pour ces deux pandémies à virus H1N1, le virus sortait du réservoir animal et s’est parfaitement adapté à l’homme [1,
7]. Or, si en 1918 on s’accorde à dire que le virus venait du réservoir aviaire, il est clair que le virus pandémique de 2009 venait du réservoir porcin. En effet, même si les virus zoonotiques sont tous issus du réservoir primaire aviaire (oiseaux sauvages), il existe aussi des réservoirs secondaires (notamment les oiseaux de basse-cour et les cochons), qui jouent le rôle d’hôtes intermédiaires et de facilitateurs pour l’adaptation des virus. Au total, il existe donc une gigantesque librairie de virus chez les oiseaux sauvages, dont seule une petite partie est transmise aux hôtes intermédiaires qui sont les sources de contamination pour l’homme. Ce sont les virus qui transitent puis circulent dans ce réservoir intermédiaire qui sont à l’origine des pandémies. |
Quelles caractéristiques moléculaires pour un virus pandémique ? Les leçons du virus influenza 1918 Un pan très important de la recherche sur les virus influenza est de comprendre comment des virus zoonotiques peuvent, par des mécanismes combinant à la fois des mutations d’adaptation et des phénomènes de réassortiment génétique, devenir des nouveaux virus adaptés à l’homme. Ces travaux combinent des études de « mécanistique » (comprenant en particulier les études controversées sur les gains de fonction ou GOF pour gain of function), et des études plus « observationnelles » [
8,
9]. Dans ce domaine, un article récent met plus particulièrement en lumière la richesse des stocks de virus capables de fabriquer de nouveaux virus pandémiques pouvant ressembler terriblement au virus de 1918 [9]. Dans ce travail réalisé conjointement par plusieurs équipes japonaises, américaines et anglaises, deux objectifs était annoncés. Le premier consistait à démontrer que les constituants du virus de 1918 (soit les huit segments de gène) existent toujours dans le réservoir aviaire sauvage. Le second était de montrer que si on rassemblait ces constituants épars pour refaire un virus H1N1, il était possible d’obtenir un virus proche du H1N1 de 1918 présentant des caractéristiques de pathogénicité et de transmission d’un virus à haut risque pandémique. L’approche expérimentale a été rendue possible par plusieurs travaux antérieurs. Premièrement, en 2005, le virus de 1918 a été identifié puis reconstitué à partir de fragments d’ARN viraux retrouvés dans des nécropsies de patients décédés en 1918. Cette reconstitution a permis de tester en laboratoire la pathogénicité du virus, et de comprendre les mécanismes ayant conduit à son apparition, son adaptation et sa diffusion [1]. Deuxièmement, la surveillance moléculaire des virus retrouvés dans la faune aviaire sauvage a été renforcée ces dernières années, avec une volonté de partage d’informations à l’échelle planétaire. Cela a conduit à la mise en commun des bases de données moléculaires pour ces virus, et un accès simple à une vaste librairie de génomes viraux [
10]. Troisièmement, les déterminants d’adaptation des virus influenza permettant le franchissement de la barrière d’espèce oiseau vers l’homme ont été étudiés de façon intensive depuis la première alerte A (H5N1) en 1997. Ces études ont permis d’identifier les mutations d’adaptation indispensables pour permettre la réplication du virus et sa transmission de l’homme à l’homme. Les données des expériences de GOF ont conforté et étayé ces résultats [8]. Et quatrièmement, dès 1997, les techniques de génétique inverse permettent de reconstruire in vitro des virus influenza comportant les séquences génétiques choisies par l’expérimentateur. Cela permet de faire des virus influenza « à façon », puis de les tester dans les modèles expérimentaux choisis [5, 8,
11]. |
Construction d’un virus expérimental A/1918- like
Forts de toutes ces données et compétences, Wattanabe et collaborateurs [9] ont criblé les bases de données « influenza aviaires » pour retrouver des segments de gènes issus de virus de tous les sous-types connus dont les séquences codent des protéines proches de celles du virus de 1918. Pour les six segments « internes », ils ont identifié des virus récents qui comportaient des segments quasi identiques à ceux du virus de 1918 (de 0 à 8 acides aminés différents par segment). Pour les deux gènes codant pour les protéines de surface H1 et N1, il est aussi possible de trouver des virus proches, avec des différences un peu plus importantes (portant sur 31 et 33 acides aminés). Les chercheurs ont alors sélectionné les huit segments de gènes les plus proches du virus H1N1 historique (appelé dans l’étude A/1918 virus), et produit par génétique inverse un virus expérimental qu’ils ont appelé A/1918-like avian virus. Puis, en utilisant deux modèles animaux (souris et furet), ils ont analysé le pouvoir pathogène de ce virus, le comparant à deux références, le virus de 1918 (A/1918) hautement pathogène et un virus A H1N1 de canard faiblement pathogène (A/duck/Alb/35/76 ou A-duck). Les résultats obtenus ont montré que le virus A/1918-like présentait un pouvoir pathogène « intermédiaire », inférieur à celui du virus A/1918, mais nettement supérieur à celui de A-duck. De plus, comme A/1918, A/1918-like était capable de répliquer dans les voies aériennes inférieures des furets. Il apparaît donc que le potentiel de pathogénicité et de transmissibilité du virus A/1918-like est supérieur à celui des virus aviaires H1N1 classiques. Il reste toutefois moins pathogène que le virus pandémique A/1918. Pour déterminer les facteurs viraux qui pourraient expliquer la différence de pathogénicité entre A/1918 et A/1918-like, les auteurs ont alors testé plusieurs constructions réalisées à partir du virus A/1918-like. Ils ont préparé des virus A/1918-like réassortants qui comportaient soit le gène HA (hémagglutinine) de A/1918 (1918 HA/avian), soit le gène PB2 (polymerase basic 2 protein) de A/1918 (1918 PB2/avian), soit les deux gènes HA et PB2 de A/1918 (1918 PB2:HA/avian), soit les trois gènes HA, PB2 et NA de 1918 (1918 PB2:HA:NA/avian), soit les trois gènes du complexe polymérase, plus la NP (nucléoprotéine) et la HA de A/1918 (1918 [3P+NP]:HA/avian). Des furets ont été infectés par voie intranasale avec ces virus, puis surveillés. Rapidement, les expérimentateurs ont vu que tous les furets infectés ont été symptomatiques. Tous ont survécu à l’infection sauf ceux qui étaient infectés par les virus 1918 HA/avian, 1918 PB2:HA/avian et 1918 (3P+NP):HA/avian, pour lesquels une mortalité des deux-tiers était observée. Pour les auteurs, ces résultats confirment le rôle important de la HA et de PB2 dans la pathogenèse du virus A/1918. |
Facteurs responsables de la transmissibilité Afin d’étudier la transmissibilité du virus A/1918-like, les auteurs ont de nouveau comparé la transmissibilité des trois virus A/1918, A/1918-like et A-duck dans le modèle furet selon un protocole classique [8]. Seul le virus A/1918 était transmissible, aucune transmission n’a été observée avec les deux virus A/1918-like et A-duck. Pour explorer le rôle des différentes protéines virales dans cette transmission, des épreuves de transmissibilité entre furets ont été réalisées avec les virus expérimentaux (avec des réassortiments génétiques) de l’expérience précédente. Une transmission a été observée pour les virus 1918 PB2:HA :NA/avian et 1918 (3P+NP):HA/avian, confirmant que la transmission est dépendante des protéines de surface et de la polymérase. Ce résultat est cohérent avec des données publiées par d’autres équipes [8, 11]. Pour explorer plus avant, les auteurs ont construit des virus recombinants intégrant d’autres mutations d’adaptation décrites dans la littérature [8, 11] et ont analysé l’impact de ces mutations additionnelles sur la transmissibilité des virus par une approche in vitro en glycan-array et in vivo chez le furet. Ils ont confirmé que les mutations ponctuelles de PB2 (en 627 et 684) et PA (253) pour la polymérase combinées avec des mutations d’adaptation sur la HA (en 89, 113, et 190, 225 et 265 pour le site d’attachement du récepteur) permettaient aux virus mutants d’être transmissibles, et de coloniser les voies aériennes inférieures et supérieures des animaux. |
Quel risque d’émergence d’un virus pandémique avec un fort pouvoir pathogène ? Au total, les auteurs concluent que le vaste réservoir de virus aviaires permet sans difficulté de trouver les éléments viraux capables de faire l’assemblage de virus H1N1 à potentiel pandémique présentant des facteurs de pathogénicité presque équivalents à ceux du virus pandémique de 1918. Ils estiment aussi que leurs travaux soutiennent l’hypothèse d’une entrée directe du virus H1N1 de 1918 depuis le réservoir aviaire, sans passage par une phase d’adaptation chez le cochon [1,
2]. Ils confirment aussi le rôle clé des deux protéines PB2 et HA dans l’adaptation à l’homme des virus aviaires, en expliquant que cette adaptation peut se faire par accumulation progressive de mutations ponctuelles dont l’effet va s’additionner jusqu’à ce que le seuil de transmissibilité puisse être atteint par le virus émergent. Devant l’importance du réservoir chez l’animal, les capacités d’adaptation des virus influenza et la pathogénicité potentielle de ces virus, ils recommandent de rester vigilants car le risque de voir apparaître un virus pandémique avec un fort pouvoir pathogène est réel. La surveillance étroite du réservoir chez les oiseaux est indispensable, comme l’est la surveillance chez les vecteurs secondaires proches de l’homme. Ces surveillances ont pour objectif de dépister en temps réel les virus ayant des facteurs connus d’adaptation à l’homme et donc un potentiel pandémique. |
L’auteur déclare participer à des travaux scientifiques pour les entreprises Roche et GSK.
|
1.
Reid
AH
,
Taubenberger
JK
,
Fanning
TG
. Evidence of an absence: the genetic origins of the 1918 pandemic influenza virus . Nat Rev Microbiol.
2004; ; 2 : :909.–914. 2.
Scholtissek
C
,
Rohde
W
,
Von Hoyningen
V
,
Rott
R
. On the origin of the human influenza virus subtypes H2N2 and H3N2 . Virology.
1978; ; 87 : :13.–20. 3.
Smith
GJ
,
Vijaykrishna
D
,
Bahl
J
, et al.
Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic . Nature.
2009; ; 459 : :1122.–1125. 4.
Casalegno
JS
,
Bouscambert
M
,
Lina
B
. Vaccin antigrippal en situation d’émergence . Virologie.
2014; ; 18 : :151.–169. 5.
Essere
B
,
Yver
M
,
Gavazzi
C
, et al.
Critical role of segment-specific packaging signals in genetic reassortment of influenza A viruses . Proc Natl Acad Sci USA.
2013; ; 110 : :E3840.–E3848. 6.
Taubenberger
JK
,
Morens
DM
. 1918 influenza : the mother of all pandemics . Emerg Infect Dis.
2006; ; 12 : :15.–22. 7.
Tumpey
TM
,
Basler
CF
,
Aguilar
PV
, et al.
Characterization of the reconstructed 1918 Spanish influenza pandemic virus . Science.
2005; ; 310 : :77.–80. 8.
Herfst
S
,
Schrauwen
EJ
,
Linster
M
, et al.
Airborne transmission of influenza A/H5N1 virus between ferrets . Science.
2012; ; 336 : :1534.–1541. 9.
Watanabe
T
,
Zhong
G
,
Russell
CA
, et al.
Circulating avian Influenza viruses closely related to the 1918 virus have pandemic potential.
Cell Host Microbe.
2014; ; 15 : :692.–705. 10.
Liu
D
,
Shi
W
,
Shi
Y
, et al.
Origin and diversity of novel avian influenza A H7N9 viruses causing human infection: phylogenetic, structural, and coalescent analyses . Lancet.
2013; ; 381 : :1926.–1932. 11.
Mok
CK
,
Lee
HH
,
Lestra
M
, et al.
Amino acid substitutions in polymerase basic protein 2 gene contribute to the pathogenicity of the novel A/H7N9 influenza virus in mammalian hosts . J Virol.
2014; ; 88 : :3568.–3576. |