| |
| Med Sci (Paris). 2015 January; 31(1): 28–31. Published online 2015 February 6. doi: 10.1051/medsci/20153101009.L’hétérogénéité intraclonale des tumeurs et son impact sur la médecine de précision Les promesses de l’imagerie de cellules vivantes couplée à la modélisation mathématique Jérémie Roux,1* Jean-Luc Gouzé,2 and Paul Hofman1 1Institut de recherche sur le cancer et le vieillissement, Nice (IRCAN), Inserm U1081, CNRS UMR 7284, équipe 3, 33, avenue de Valombrose, 06107Nice, France 2BIOCORE, INRIA, 2004, route des lucioles, BP 93, 06902Sophia Antipolis, France MeSH keywords: Évolution clonale, Biologie informatique, Études d'associations génétiques, Hétérogénéité génétique, Génomique, Humains, Thérapie moléculaire ciblée, Tumeurs, Médecine individualisée, méthodes, tendances, génétique, anatomopathologie |
En oncologie, l’enjeu de la médecine personnalisée (encore appelée médecine stratifiée ou de précision) est de prédire la réponse d’une tumeur à une série de thérapies « ciblées » disponibles, et ainsi trouver la combinaison thérapeutique pour laquelle le patient ne développera pas de « résistance » primaire voire secondaire. Cette problématique n’est pas nouvelle en cancérologie (classification histologique des cancers du poumon non à petites cellules ou à petites cellules par exemple pour déterminer la chimiothérapie la plus efficace), mais l’émergence des résistances aux traitements ciblés a mis en évidence la grande variabilité qui existe entre chaque individu en termes de réponse aux traitements anticancéreux [1]. Celle-ci se manifeste par un spectre variable allant de l’absence de réponse au traitement jusqu’à une réponse partielle ou complète, réponse à laquelle il faut intégrer une toxicité plus ou moins importante selon les individus. Ce phénotype de résistance aux thérapies est irrémédiablement lié au décès du patient, et à 13 % de la mortalité mondiale due à un cancer [2]. |
Hétérogénéité intratumorale et hétérogénéité intraclonale La résistance aux médicaments est le mécanisme qui limite l’efficacité des thérapies anticancer : elle peut provenir d’une anomalie indépendante de la tumeur (distribution/métabolisme du médicament, environnement tumoral), ou d’une anomalie de la cellule tumorale (blocage de l’apoptose, autophagie, sénescence) [3]. Les techniques génomiques et protéomiques modernes ont pu identifier de nombreux gènes et mécanismes cellulaires impliqués dans la résistance aux thérapeutiques anticancéreuses. En analysant des cellules tumorales individuellement, ces études ont alors permis de mettre en évidence l’hétérogénéité cellulaire intratumorale (génétique et spatiale, présence de cellules souches cancéreuses) (Figure 1) [4, 13, 14]. L’ensemble de ces études a été déterminant pour associer des signatures d’expressions géniques ou protéiques aux données cliniques obtenues chez des patients, et permettre la découverte de certains marqueurs prédictifs de réponse et de progression du cancer. Cependant, bien que réalisées à l’échelon de la cellule individuelle, ces études restent encore essentiellement corrélatives, joignant les signatures génomiques/protéomiques des cellules d’un échantillon à la valeur moyenne d’une mesure phénotypique (diagnostic histologique, taille de la tumeur, pourcentage de cellules apoptotiques, croissance cellulaire, etc.).
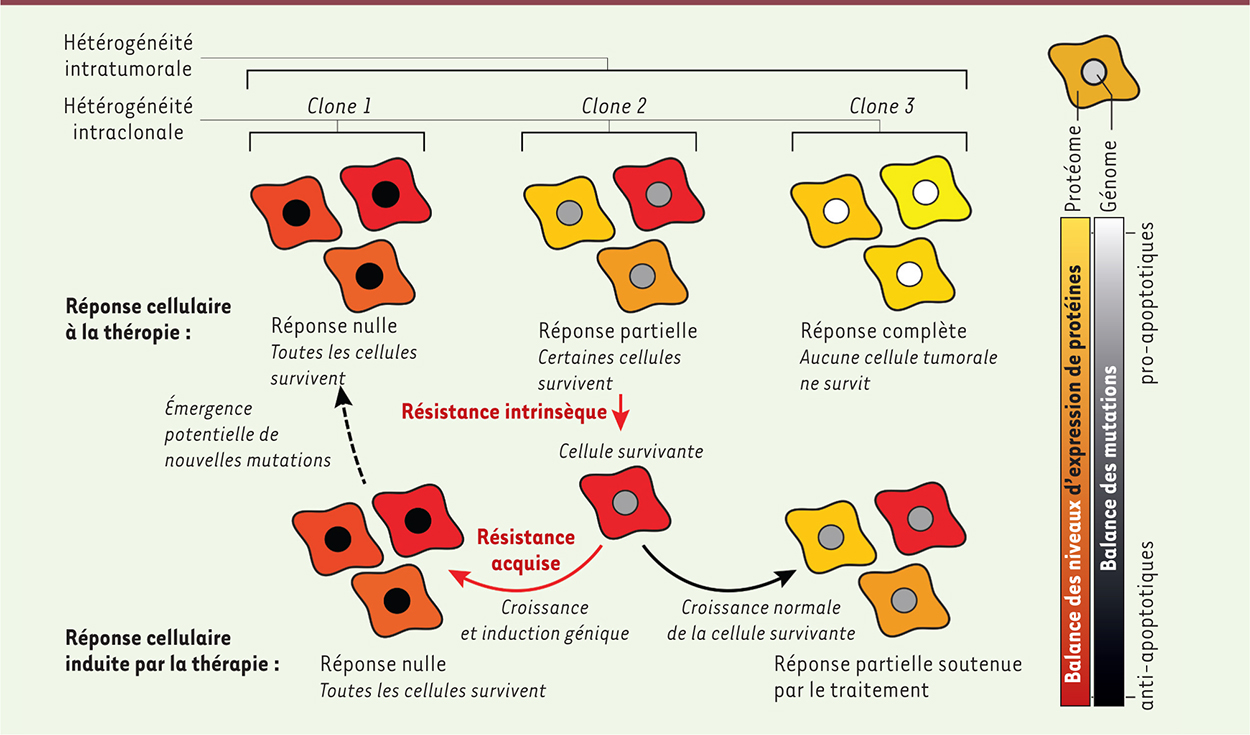
 | Figure 1.
Mécanismes de résistance aux thérapies anticancer dus à l’hétérogénéité intratumorale et intraclonale dans l’expression de gènes/protéines de la voie apoptotique. Au sein d’un clone exprimant une « réponse partielle », les fluctuations du protéome (représentées par les couleurs rouge, orange et jaune du cytoplasme du clone 2) peuvent faire émerger des contextes cellulaires où la quantité des protéines anti-apoptotiques dépasse celle des protéines pro-apoptotiques, empêchant la propagation du signal de l’apoptose. La cellule survivante peut donner naissance à une nouvelle tumeur d’hétérogénéité identique (réponse partielle maintenue par le traitement [ 12]), ou éventuellement activer un programme génétique favorisant la résistance de nouveaux clones (réponse adaptative). |
Plus récemment, les techniques d’imagerie de cellules vivantes ont montré que des cellules issues d’une même population monoclonale, peuvent générer une diversité phénotypique extraordinaire, indépendamment du cycle cellulaire : ainsi, des deux cellules sœurs issues d’une seule division, l’une répondra à une molécule thérapeutique et l’autre non (hétérogénéité intraclonale) (Figure 1) [5]. La cellule survivante pourra participer à la récidive tumorale. Il a été montré in vitro que c’est l’état du protéome au moment du traitement [6], et notamment les niveaux d’expression relatifs de protéines pro et anti-apoptotiques, qui expliquent cette hétérogénéité au sein d’une lignée cellulaire monoclonale (Figure 1, démontrée in silico [7]). Bien que ces études aient permis de lier directement une réponse cellulaire intégrative à une mesure phénotypique, telle que la mort ou la survie de la cellule, en réponse au traitement, elles ne permettent pas d’obtenir une mesure complète du protéome (Figure 2) et donc d’identifier une cible ou un groupe de cibles moléculaires.
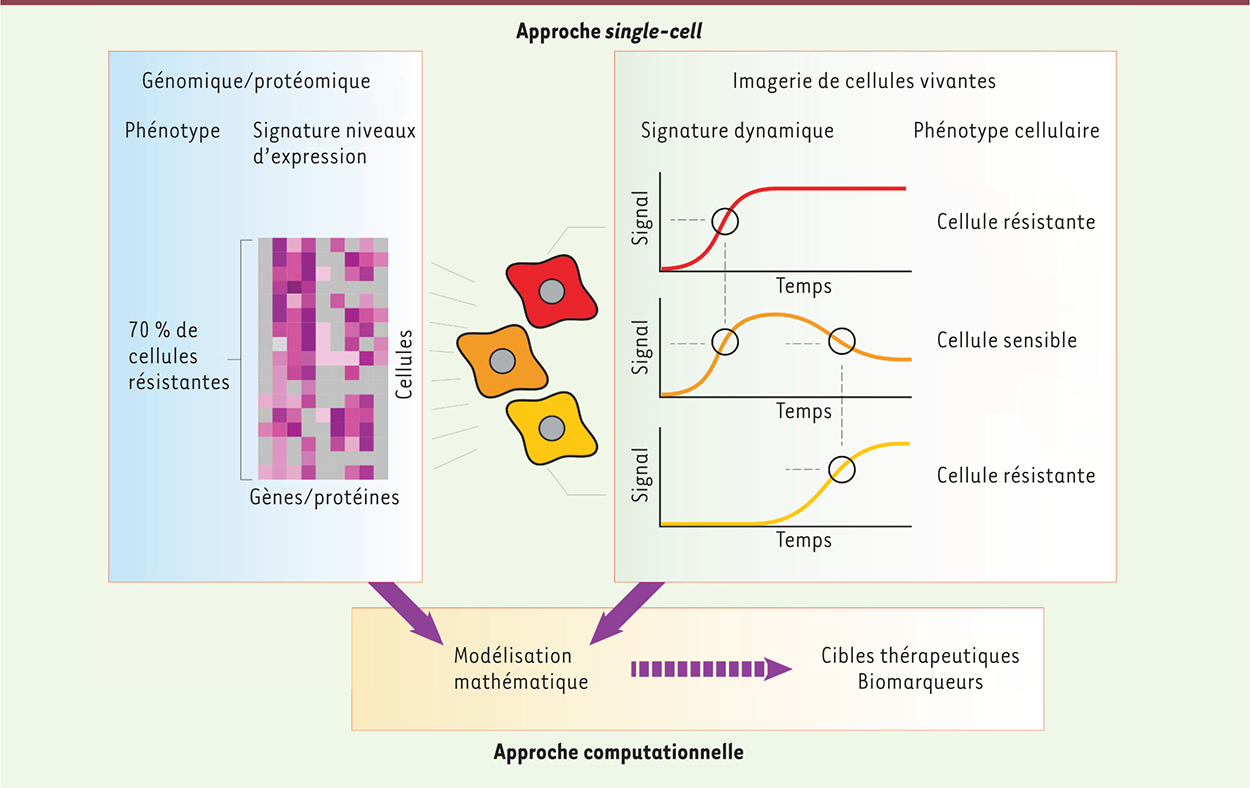
 | Figure 2.
Comparaison des approches génomique/protéomique avec l’imagerie de cellules vivantes et intégration de ces approches single-cell grâce à la biologie computationnelle. La génomique/protéomique permet de collecter une grande quantité de signaux dans une large population de cellules pour capturer l’hétérogénéité intratumorale et intraclonale. L’intégrité cellulaire étant perdue lors de la préparation des échantillons, ni la signature dynamique (cercles), ni la réponse cellulaire de la cellule analysée (phénotype) ne sont observables. L’approche d’imagerie de cellules vivantes permet de mesurer la signature dynamique d’une réponse cellulaire (signal) ainsi que le phénotype associé à cette dynamique. L’approche computationnelle permet d’intégrer ces 2 types d’ensembles de données par des modèles mathématiques dont l’analyse peut isoler des paramètres « sensibles ». Ces candidats peuvent être testés expérimentalement par la suite pour mettre en évidence de nouvelles cibles thérapeutiques ou des biomarqueurs potentiels. |
|
Comment lier un phénotype aux analyses « omiques » multiplex ? À l’heure actuelle, seule l’imagerie de cellules vivantes permet d’établir un lien direct entre un évènement biochimique cellulaire et le devenir de la cellule dans laquelle cet évènement est mesuré (Figure 2). Ce lien est particulièrement important dans la problématique de résistance aux thérapies anticancer où l’enjeu est de minimiser l’hétérogénéité intraclonale afin d’obtenir une réponse complète de l’ensemble de la population cellulaire. Il existe aujourd’hui de nombreuses sondes biochimiques fluorescentes qui peuvent mesurer un grand nombre de processus cellulaires, mais la plupart nécessitent la création de cellules génétiquement modifiées, et le nombre de signaux mesurés simultanément reste encore aujourd’hui limité [8]. La génomique et la protéomique, quant à elles, offrent des possibilités d’analyses simultanées (multiplex) très importantes qui peuvent être effectuées sur des cellules non modifiées, directement issues d’échantillons de patient. Mais ces techniques ne permettent pas le suivi de cellules individuelles dans le temps (signature dynamique), ce qui peut masquer l’effet de la thérapie et empêche l’évaluation de l’efficacité antitumorale du médicament pour chaque cellule (Figure 2). L’intégration de ces ensembles de données, fondamentalement différents (statiques et dynamiques), est envisageable aujourd’hui grâce à l’utilisation d’outils biostatistiques et à la création de modèles mathématiques. Les modèles mathématiques sont utilisés en biologie pour décrire de façon quantitative les régulations de voies de signalisations cellulaires, en codant chaque réaction biochimique sous la forme d’une équation mathématique. Ces modèles sont écrits sur la base des connaissances fondamentales en biologie cellulaire. L’utilisation de modèles mathématiques a déjà permis d’illustrer la nature multifactorielle de la résistance aux médicaments anticancéreux et de déterminer les contextes cellulaires (ensemble de paramètres) conduisant à la mort de la cellule tumorale [15]. La détermination de ces paramètres (paramétrisation des modèles) par des méthodes mathématiques est donc l’interface où peuvent être intégrées les données biologiques issues de l’imagerie de cellules vivantes et des analyses « omiques ». Les analyses « omiques » de cellules uniques renseignent sur la variabilité des niveaux d’expression de gènes ou de protéines trouvés dans une population, ceux-ci représentent les valeurs de paramètres et les conditions initiales des modèles mathématiques. L’imagerie de cellules vivantes, quant à elle, informe sur les cinétiques des réactions biochimiques servant ainsi à calibrer les modèles, et permet de s’assurer que ceux-ci capturent correctement les mécanismes cellulaires sous-jacents [9]. La puissance de cette méthode réside dans l’analyse « computationnelle » de sensibilité, afin de trouver des paramètres « sensibles » à des petites perturbations (associées à des protéines du réseau cellulaire étudié). Ces études permettent ensuite de suggérer des cibles thérapeutiques ou même des biomarqueurs potentiels [10]. Ces modèles peuvent aussi proposer des relations mathématiques entre les niveaux d’expressions de plusieurs protéines, qui individuellement ne représentent pas des biomarqueurs, mais qui, ensemble, acquièrent une valeur prédictive. |
Perspectives et enjeux de la biologie computationnelle pour la médicine personnalisée Prédire la réponse des cellules tumorales pour vaincre la résistance à une thérapie est l’enjeu de la médecine personnalisée ou de précision. Les résistances cellulaires intrinsèques et acquises lors du traitement peuvent coexister dans une même tumeur (hétérogénéité intratumorale), ce qui rend difficiles les prédictions thérapeutiques [1]. Les prédictions basées sur les données « omiques », même à l’échelon de cellules individuelles, restent encore aujourd’hui malheureusement très imprécises en partie à cause du caractère rapidement évolutif des résistances tumorales [11], mais aussi parce que ces prédictions sont faites en corrélant une signature génomique ou protéomique à une réponse globale de la population tumorale. Cependant, deux cellules génétiquement identiques peuvent avoir un protéome progressivement différent (changements dynamiques) et générer deux réponses opposées : résistance ou mort cellulaire (hétérogénéité intraclonale) [5]. Cette différence fondamentale de réponse cellulaire n’est observable qu’en suivant le devenir de la cellule par imagerie de cellules vivantes. Faire correspondre la réponse d’une cellule à son protéome et à son génome permettra de répondre à la question suivante : quelle signature moléculaire conduit à la résistance thérapeutique de la cellule qui va relancer la croissance tumorale ? Le défi de la biologie computationnelle est aujourd’hui de lier les analyses multidimensionnelles à haut débit (analyses statiques : génome, épigénome, protéome, et métabolome) et les analyses dynamiques des voies de signalisation cellulaire (analyses dynamiques : microscopie de cellules vivantes) par l’utilisation de modèles mathématiques interprétables en termes moléculaires. Ces outils permettront de compléter la médecine génomique afin de mieux prédire quel patient pourra bénéficier d’une thérapie spécifique, et de comprendre les raisons quantitatives de l’efficacité variable d’une thérapie. ‡ |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Les auteurs remercient les actions Marie Curie (FP7-PEOPLE-2013-IIF Marie Curie International Incoming Fellowship within the 7th European Community Framework Programme) pour le support du projet SysBioDRez N° 626190, la fondation ARC pour la recherche sur le cancer (contrat n° 12ARC001MRAR), l’Agence nationale de la recherche « Investissements d’avenir » (ANR-11-LABX-0028-0, LABEX SIGNALIFE), ainsi que le Canceropôle PACA.
|
1.
Bedard PL, Hansen AR, Ratain MJ, Siu LL. Tumour heterogeneity in the clinic . Nature. 2013; ; 501 : :355.–364. 2.
Bray F, Jemal A, Grey N, et al. Global cancer transitions according to the human development index (2008–2030): a population-based study . Lancet Oncol. 2012; ; 13 : :790.–801. 3.
Holohan C, Van Schaeybroeck S, Longley DB, Johnston PG. Cancer drug resistance: an evolving paradigm . Nat Rev Cancer. 2013; ; 13 : :714.–726. 4.
Burrell RA, McGranahan N, Bartek J, Swanton C. The causes and consequences of genetic heterogeneity in cancer evolution . Nature. 2013; ; 501 : :338.–345. 5.
Roux J, Hafner M, Bandara S, et al. Fractional killing in response to TRAIL and therapeutic antibody agonists arises from cell-to-cell variability in overcoming a caspase-8 activity threshold . FEBS J. 2014; ; 281 : :526.. 6.
Spencer SL, Gaudet S, Albeck JG, et al. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis . Nature. 2009; ; 459 : :428.–432. 7.
Gaudet S, Spencer SL, Chen WW, Sorger PK. Exploring the contextual sensitivity of factors that determine cell-to-cell variability in receptor-mediated apoptosis . PLoS Comput Biol. 2012; ; 8 : :e1002482.. 8.
Newman RH, Fosbrink MD, Zhang J. Genetically encodable fluorescent biosensors for tracking signaling dynamics in living cells . Chemical Reviews. 2011; ; 111 : :3614.–3666. 9.
Chen WW, Niepel M, Sorger PK. Classic and contemporary approaches to modeling biochemical reactions . Genes Dev. 2010; ; 24 : :1861.–1875. 10.
Luan D, Zai M, Varner JD. Computationally derived points of fragility of a human cascade are consistent with current therapeutic strategies . PLoS Comput Biol. 2007; ; 3 : :e142.. 11.
Beckman RA, Schemmann GS, Yeang CH. Impact of genetic dynamics and single-cell heterogeneity on development of nonstandard personalized medicine strategies for cancer . Proc Natl Acad Sci USA. 2012; ; 109 : :14586.–14591. 12.
Flusberg DA, Roux J, Spencer SL, Sorger PK. Cells surviving fractional killing by TRAIL exhibit transient but sustainable resistance and inflammatory phenotypes . Mol Biol Cell. 2013; ; 24 : :2186.–2200. 13.
Billaud M.. L’hétérogénéité intratumorale : un obstacle darwinien à la médecine personnalisée ? Med Sci (Paris). 2012; ; 28 : :1116.–1119. 14.
Jordan B.. Hétérogénéité des tumeurs : l’apport du séquençage sur cellules individuelles . Med Sci (Paris). 2014; ; 30 : :1184.–1186. 15.
Xia X, Owen MS, Lee RE, Gaudet S. Cell-to-cell variability in cell death: can systems biology help us make sense of it all ? Cell Death Dis. 2014; ; 5 : :e1261.. |