| |
| Med Sci (Paris). 2015 January; 31(1): 84–92. Published online 2015 February 6. doi: 10.1051/medsci/20153101017.PCR digitale en micro-compartiments I. Détection sensible de séquences d’acides nucléiques rares Karla Perez-Toralla,1* Deniz Pekin,1,2* Jean-François Bartolo,1 Fanny Garlan,1 Philippe Nizard,1 Pierre Laurent-Puig,1 Jean-Christophe Baret,2,3 and Valérie Taly1a 1Université Paris Sorbonne Cité, Inserm UMR-S1147, 45, rue des Saints-Pères, 75270Paris, France 2Droplets membranes and interfaces, Max Planck institute for dynamics and self-organization, Am Fassberg 17, D-37077Göttingen, Allemagne 3Soft micro systems, CNRS, université de Bordeaux, CRPP, UPR 8641, 115, avenue Schweitzer, 33600Pessac, France |

L’identification de mutations ou de variations génétiques est un élément clé de la recherche clinique. Ainsi, la recherche d’altérations génétiques spécifiques des cellules tumorales fait partie de la prise en charge des patients atteints de cancer. Les cellules cancéreuses présentes dans la tumeur d’un patient ont un profil génétique distinct des autres cellules de l’organisme. En effet, les cellules tumorales sont caractérisées par des modifications dans la structure et/ou la fonction de certains gènes clés du cycle cellulaire. Ces altérations consistent en des modifications localisées (substitution, délétion ou insertion de quelques nucléotides), mais également en d’importantes aberrations chromosomiques (translocation ou amplification de gènes, gain ou perte de chromosomes). Il est aujourd’hui possible d’utiliser ces altérations en tant que marqueurs tumoraux pour mieux prédire la progression de la maladie et mettre en place de nouvelles stratégies thérapeutiques [1]. |
De la PCR quantitative à la PCR digitale De nombreuses techniques ont été appliquées à la détection d’altérations génétiques dans les tumeurs. Le choix de la technique employée repose sur un compromis entre divers critères : le coût, la sensibilité, la disponibilité et l’adéquation avec le type d’échantillon analysé (Figure 1).
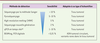 | Figure 1.
Méthodes utilisées en recherche clinique pour la détection d’altérations génétiques dans les tissus tumoraux. Les sensibilités des différentes techniques ont été décrites dans [ 40– 42]. *La sensibilité de la qPCR en temps réel est dépendante du type de test utilisé (modifiée d’après Diaz et al. [ 33]). |
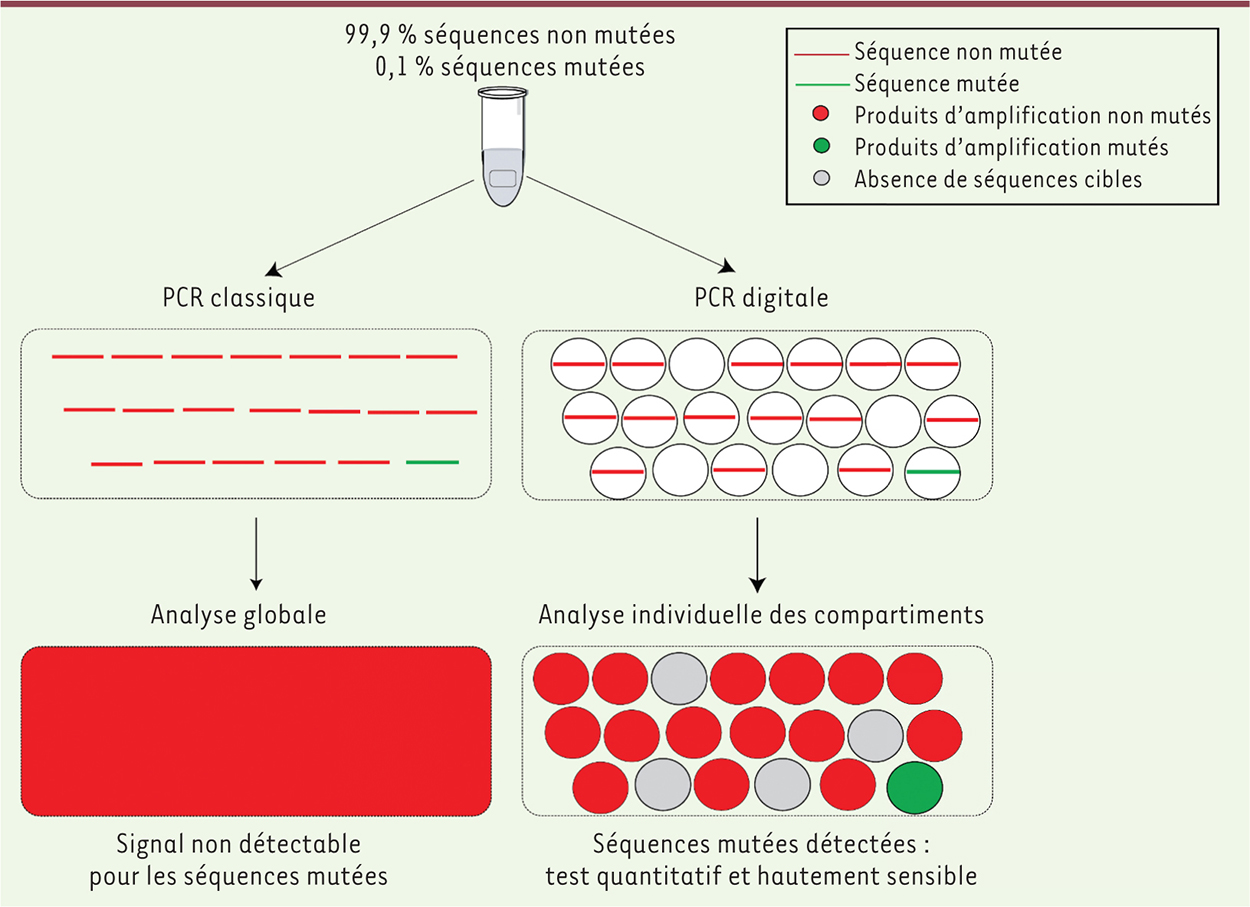
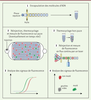
De nos jours, une des méthodes les plus couramment utilisées en recherche clinique pour la détection de mutations est la discrimination allélique par PCR quantitative (qPCR). Cette méthode permet l’amplification de molécules d’ADN présentes dans un échantillon et le suivi de cette amplification en temps réel [2, 3] avec une sensibilité entre 1-10 %. Cette sensibilité autorise l’analyse de tissus tumoraux, dans lesquels la proportion de clones mutants est très élevée, mais reste insuffisante pour la détection de variants rares ou celle d’ADN tumoral circulant extrait d’effluents biologiques, incluant le plasma. Ces applications nécessitent en effet de nombreuses optimisations de la méthode conventionnelle de qPCR afin d’augmenter sa sensibilité et de détecter efficacement les séquences cibles [4]. Ainsi, la détection et la caractérisation d’altérations génétiques sous représentées au sein d’échantillons biologiques complexes (sang, urine, selles, etc.) représentent un défi particulier pour la qPCR. En effet, ces séquences spécifiques de tumeurs sont très diluées au sein de séquences très proches (elles ne différent dans certains cas que d’un seul nucléotide) provenant des cellules saines. La PCR classique présente de nombreuses limitations (amplification préférentielle de petits fragments, production de séquences chimériques, difficulté de détecter des ADN peu représentés) en particulier pour l’amplification de mélanges complexes d’ADN (Figure 2, à gauche).
 | Figure 2.
Comparaison des procédures de la PCR classique et de la PCR digitale. Exemple de l’analyse d’un échantillon contenant 0,1 % de séquences mutées. L’expérience de PCR est réalisée avec un mélange de sondes spécifiques de la séquence mutée (en vert) et de la séquence non mutée (en rouge). La qPCR conventionnelle (gauche) amplifiera toutes les molécules d’ADN cibles présentes. Le signal obtenu représentera donc un signal moyen englobant l’ensemble des signaux correspondant aux différents ADN présents, et le signal provenant des séquences sous-représentées pourra être masqué par celui des séquences abondantes. La PCR digitale (droite) permet d’amplifier chaque ADN cible dans un compartiment indépendant. L’analyse de la fluorescence de ces compartiments indépendants permet de détecter ceux qui contiennent un ADN muté. La procédure est quantitative et sa sensibilité dépend du nombre de compartiments analysés (modifiée d’après Taly et al. [ 23].) |
Pour contourner ces limitations, Sykes et al. ont proposé en 1992 « une procédure générale pour la quantification du nombre initial de molécules cibles dans un échantillon, utilisant la dilution limite, la PCR et la loi de Poisson » [5]. Cette approche consiste à diluer en série un échantillon afin d’obtenir une dilution limite, où chaque partition contient, au plus, une molécule d’ADN cible. La distribution statistique de l’ADN dans les compartiments est alors donnée par la loi de Poisson, qui régit les statistiques d’événements rares. Les partitions sont ensuite soumis à une amplification par PCR et analysés individuellement. Le signal issu de la détection des séquences minoritaires de l’échantillon est alors mesurable. Ce concept a été étendu par Vogelstein et Kinzler [6] en 1999 et appliqué au développement d’une nouvelle procédure appelée PCR digitale ou dPCR au format microplaque et intégrant une étape de détection avec des sondes fluorescentes (molecular beacons). Une sonde reconnaît les produits d’amplification (mutés et non mutés), et une deuxième sonde émettant une couleur différente s’hybride aux séquences non mutées. À la fin de l’analyse, seuls les puits de la plaque émettant un signal de fluorescence sont analysés. Chaque compartiment produit ainsi, en fonction de la présence ou non de la molécule d’intérêt, un signal positif ou négatif, d’où le terme de « PCR digitale » (Figure 2, à droite). En calculant le rapport du nombre de puits émettant chaque couleur, il devient possible de quantifier la fraction d’ADN muté contenue dans l’échantillon sans avoir besoin de se référer à une courbe de calibration. Grâce à cette discrimination binaire, on peut détecter des évènements rares, à l’échelle de la molécule unique. Cette méthode a été optimisée et appliquée à la détection et la quantification de mutations de l’oncogène KRAS chez des patients atteints de cancer colorectal [6]. La sensibilité de la dPCR dépend principalement du volume d’échantillon et du nombre total de compartiments que l’on peut analyser. En effet, tout comme la résolution d’une image digitale dépend du nombre de pixels, la résolution d’une réaction de dPCR dépend du nombre de compartiments analysés. Ainsi, pour quantifier de faibles fractions de mutants, il suffirait donc de multiplier le nombre de compartiments jusqu’à obtenir des décomptes statistiquement fiables [7]. De plus, en raison de cette compartimentation, la dPCR permet d’augmenter le seuil de tolérance aux inhibiteurs classiques (par exemple : SDS, EDTA et héparine) et d’augmenter ainsi l’efficacité et la reproductibilité de la réaction de PCR [8]. Cependant, ces procédures, pourtant simples en théorie, demandent une implémentation plutôt complexe. De plus, l’utilisation des microplaques 384-puits contenant 5 µL par réaction nécessite des volumes importants de réactifs [9], expliquant que ces procédures ne soient pas économiquement viables si l’on souhaite des sensibilités élevées. Les contraintes techniques liées à l’utilisation de microplaques contenant des volumes inférieurs au microlitre (telles que l’évaporation ou les effets capillaires) limitent l’utilisation de cette technologie [10]. La dPCR a donc dû attendre le développement de nouvelles technologies de miniaturisation et de nanofabrication pour exprimer tout son potentiel pour l’analyse d’échantillons complexes. Les stratégies qui ont alors émergé sont basées sur le contrôle de ces volumes, soit dans des microchambres, soit dans des microgouttelettes. |
PCR digitale en microchambres Les méthodes de dPCR en micropuces permettent de réaliser des réactions dans des microchambres dont les volumes sont généralement de l’ordre du nanolitre [11]. Le principal avantage de ces plate-formes est la facilité d’utilisation et la possibilité d’automatiser le système de l’injection de l’échantillon jusqu’à l’analyse de la réaction. Cependant, afin de minimiser les dimensions de la puce, le nombre de compartiments dans les différents systèmes est souvent limité à quelques milliers [9]. Plusieurs systèmes de dPCR en microchambres ont été développés, parmi lesquels on distingue les systèmes utilisant des microvannes et ceux utilisant des micropuits (Figure 3).
 | Figure 3.
Les plates-formes de PCR digitale commercialisées. Plusieurs systèmes dPCR en microchambres ont été développés, parmi lesquels nous pouvons différencier les systèmes utilisant des microvannes (Fluidigm Corporation), des plaques perforées (Life Technologies) et les systèmes utilisant des micropuits (Slip Chip, pas encore disponible sur le marché) (modifié d’après Baker et al. [ 9]) |
Les pompes et les vannes constituent des éléments de contrôle des débits fluidiques à l’intérieur de la puce. Ces éléments sont complexes à fabriquer à partir des technologies utilisant le silicium, et souvent difficilement intégrables sur des puces dans les laboratoires. Grâce à l’utilisation de la technologie de lithographie « douce », fondée sur des techniques de moulage de polymère, une solution élégante utilisant des vannes flexibles en matériau élastomère a été proposée [12]. Ces vannes peuvent être activées en parallèle ou de manière indépendante, afin de contrôler le contenu de chaque chambre et faciliter la manipulation des fluides sur l’ensemble de la puce. L’intégration à grande échelle de microvannes flexibles a donné lieu à de nombreuses applications, en particulier en dPCR [13]. Ce système permet en effet de compartimenter un échantillon dilué ainsi que des réactifs de PCR de manière simple et efficace. Un exemple des systèmes actuellement disponibles est le Digital Array Chip 12.765® (Fluidigm) qui peut analyser 12 échantillons différents en parallèle, chacun étant réparti en 765 compartiments fermés d’un volume de 6 nL. Avec cette plate-forme, il est donc possible de réaliser 9 180 réactions de PCR simultanées [14]. D’autres puces avec un plus grand nombre de compartiments sont également disponibles (36 960 réactions en parallèle dans des volumes de 850 pL), permettant d’atteindre une sensibilité supérieure. Le thermocyclage et la lecture des signaux de fluorescence se font directement dans la même plate-forme, ce qui diminue le temps de l’expérience et limite le nombre de manipulations de l’échantillon. Ceci permet également d’implémenter des protocoles de qPCR pour détecter de possibles faux positifs et améliorer la sensibilité de l’analyse. Un autre système, qui permet la mesure du signal d’amplification en temps réel, développé par Morrison et al. [11], se présente sous la forme d’une plaque métallique perforée de la taille d’une lame de microscope. Chaque plaque comprend 3 072 orifices (48 zones de 64 micropuits) de 300 µm de diamètre et de 33 nL de volume interne. L’intérieur de chaque orifice est traité avec un revêtement hydrophile alors que l’extérieur de la plaque est hydrophobe, confinant ainsi l’échantillon. Cette configuration permet également d’isoler les différents compartiments afin d’éviter d’éventuelles contaminations. Différentes applications de ces systèmes ont été démontrées à l’aide du système commercial OpenArray® (LifeTechnologies), notamment pour la détection en temps réel de mutations génétiques ponctuelles [13, 15] ou la détection d’ARN non codants [16]. Plus récemment, la même entreprise a commercialisé une nouvelle station miniaturisée moins onéreuse et pouvant effectuer jusqu’à 20 000 réactions simultanément ; ceci au détriment de la mesure de fluorescence en temps réel. Enfin, le système « SlipChip » est un système microfluidique constitué de deux plaques microstructurées mises en contact et séparées par l’intermédiaire d’une fine couche d’huile. Chacune des plaques contient des micropuits qui peuvent être alignés et superposés pour créer un réseau de microcanaux permettant le remplissage du système. Par le simple glissement des plaques entre elles, les puits peuvent être isolés afin de compartimenter les molécules d’ADN et de les mélanger avec les réactifs nécessaires à la PCR (Figure 3). Le nombre de compartiments et la séquence des réactions à effectuer sont ainsi prédéfinis lors de la conception des plaques. Ce système a été optimisé pour la détection quantitative de molécules uniques d’ADN bactérien (gène nuc, Staphylococcus aureus) par dPCR [17]. Avec 1 280 puits de 2,6 nL (400 µm de longueur, 200 µm de largeur et 50 µm de profondeur), ces plaques ont permis l’analyse d’échantillons avec une concentration de 1 fg/µL d’ADN génomique. Les avantages principaux d’une telle plate-forme sont la simplicité d’utilisation et le faible coût, rendant son utilisation possible dans des milieux aux ressources limitées. |
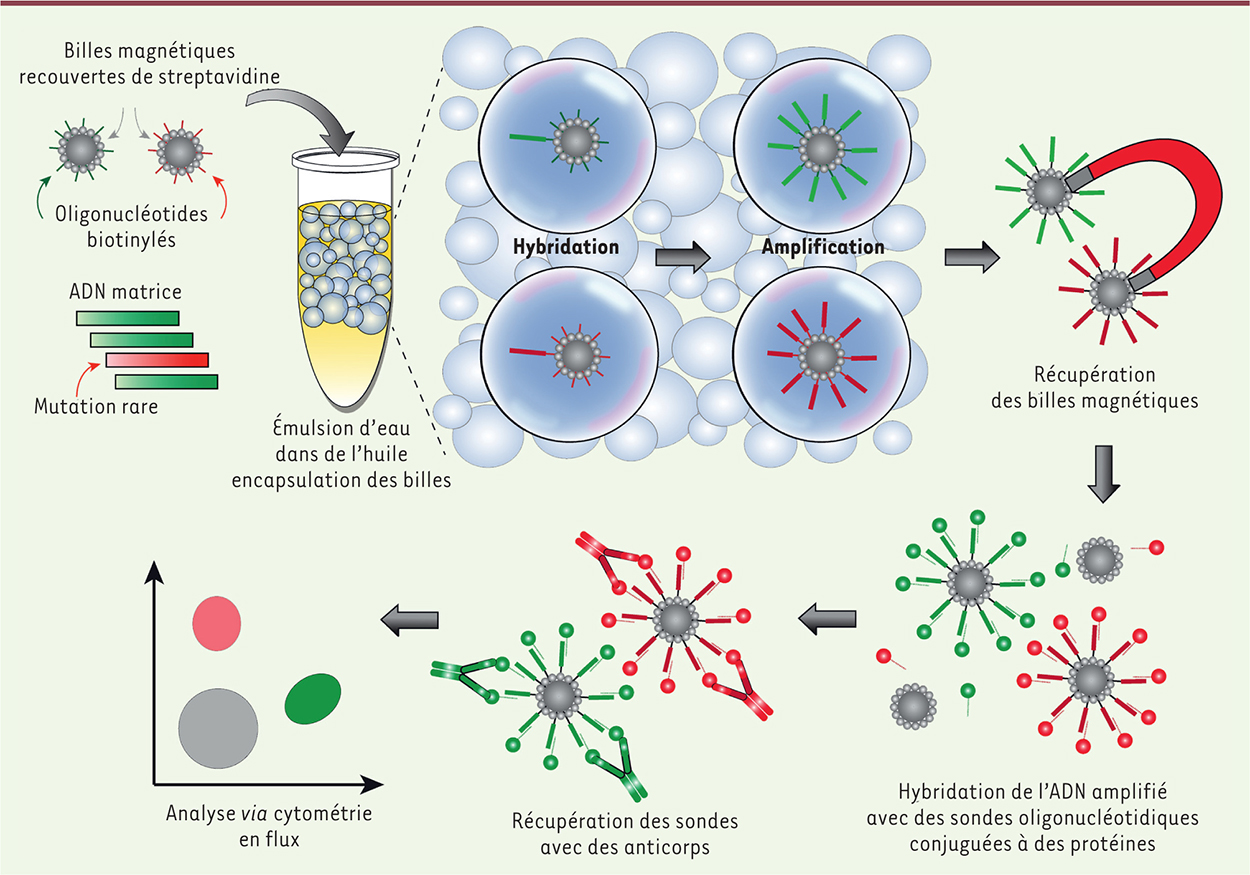
Alors que les systèmes décrits ci-dessus représentent une extension logique de la technologie des plaques de microtitration, un changement significatif dans l’approche expérimentale a été récemment proposé. Il consiste en l’utilisation de gouttelettes aqueuses dispersées dans de l’huile pour la compartimentation des réactions de PCR, ce qui permet d’envisager un nombre de compartiments théoriquement illimité (qui dépend du volume des gouttelettes et de celui de l’échantillon disponible) [18]. La phase aqueuse contient l’ADN cible et le mélange réactionnel nécessaire à la PCR. Cette stratégie, appelée PCR en émulsion (ePCR) permet de réaliser des tests très sensibles et théoriquement moins coûteux en comparaison des plate-formes utilisant des microchambres [9, 19]. Les premières procédures d’ePCR créaient des gouttes par agitation mécanique, ce qui entraînait la production de gouttes de tailles très variables [18]. Cette polydispersité des volumes réactionnels limitait l’obtention de résultats quantitatifs et augmentait le risque d’encapsuler plus d’une molécule cible par goutte. De plus, les gouttelettes générées par ces procédures sont difficilement manipulables [20]. Pour pallier ces limites, différentes stratégies utilisent des billes magnétiques afin de capturer les ADN amplifiés et de pouvoir les manipuler après la réaction de PCR. Ainsi, le BEAMing (beads, emulsion, amplification and magnetics) utilise des billes magnétiques fonctionnalisées avec des amorces complémentaires de l’ADN cible qui, une fois « recouvertes » de multiples copies de l’ADN cible initialement encapsulé, sont marquées par des fluorochromes et analysées en cytométrie en flux [21] (Figure 4). La sensibilité de cette technique (commercialisée par la société Sysmex Inostics) dépend principalement du taux d’erreurs générées par la polymérase utilisée lors de la préamplification des séquences d’intérêt. La possibilité de détecter une molécule d’ADN mutée parmi 10 000 molécules non mutées a ainsi été décrite [22]. Cette technique est très attractive pour la détection de séquences rares, mais sa mise en œuvre complexe la rend peu compatible avec une utilisation de routine en clinique.
 | Figure 4.
La technique BEAMing : chaque goutte contient en moyenne moins d’un ADN matrice fixé sur une bille. Après une étape de PCR, chaque bille comporte à sa surface plusieurs milliers de copies d’ADN. Les billes sont ensuite extraites de l’émulsion, purifiées, incubées dans une solution contenant des marqueurs fluorescents spécifiques des séquences d’ADN amplifiés. Elles sont analysées par cytométrie en flux (hormis les billes portant des séquences multiples, qui sont éliminées de l’analyse). De cette façon, la sensibilité du BEAMing n’est limitée que par le taux d’erreur de la polymérase utilisée [ 23]. |
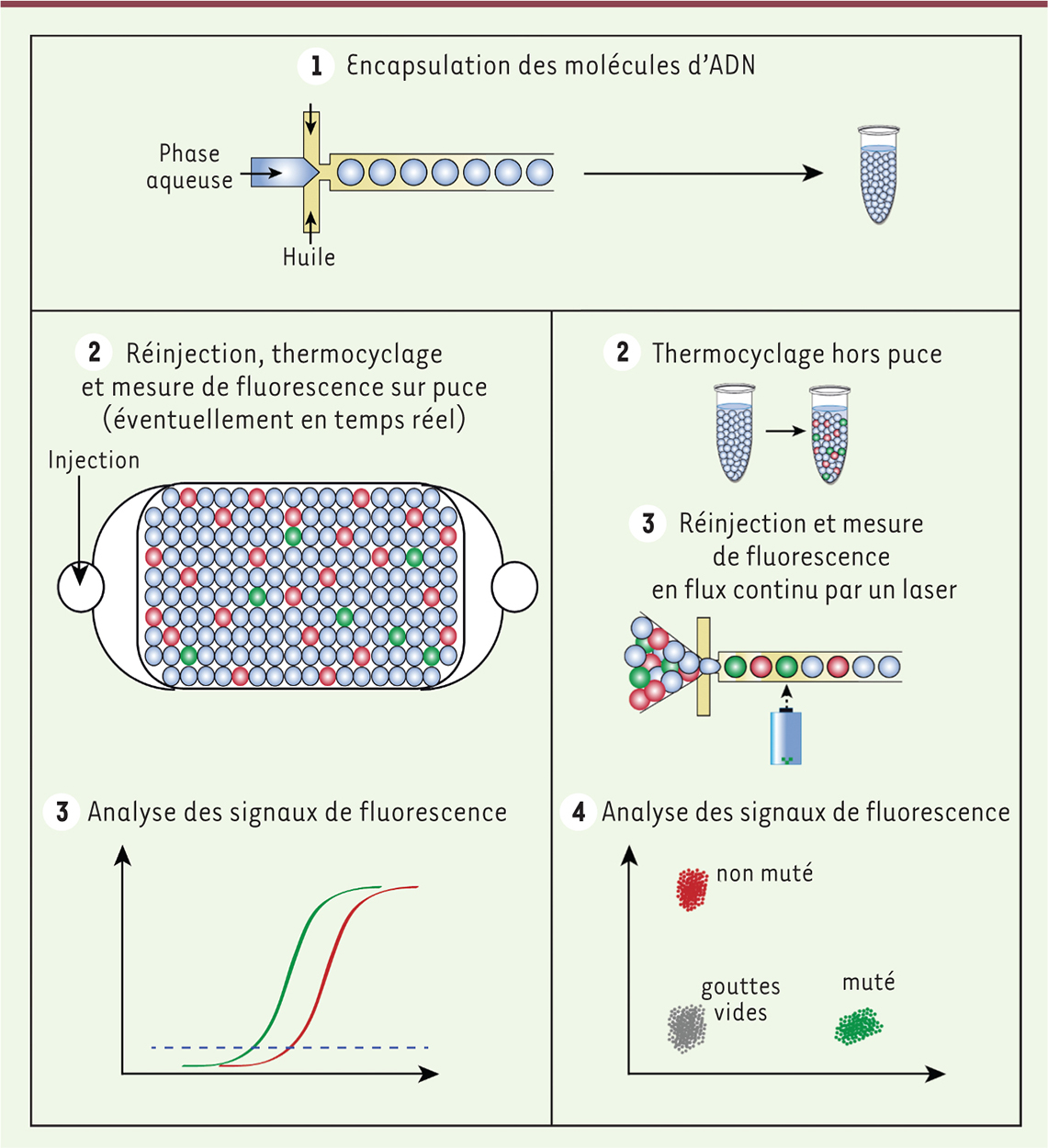
Afin de mieux contrôler le volume des émulsions, des systèmes microfluidiques ont également été développés pour la réalisation de réactions de dPCR en microgouttelettes [23]. La microfluidique est une technologie fondée sur la manipulation de très faibles quantités de fluides (de 10-9 à 10-18 L) dans des microcanaux [24]. En utilisant des écoulements diphasiques (combinaison d’une phase aqueuse et d’une phase d’huile), les systèmes microfluidiques permettent de produire, à haute fréquence, des gouttes micrométriques monodispersées [25]. L’utilisation de tensioactifs stabilise les gouttes en évitant leur coalescence lorsqu’elles entrent en contact [26]. Ces « microréacteurs » peuvent ensuite être manipulés dans les microcanaux grâce à l’implémentation de différents modules de fusion, de division, d’incubation ou encore de tri [10] . Le choix de la méthode de détection est également un élément clé de la miniaturisation. L’implémentation des techniques de détection utilisées en qPCR (sondes fluorescentes ou molécules s’intercalant dans l’ADN par exemple) permet une détection par fluorescence, ainsi qu’en multiplex, avec une grande sensibilité [2, 3]. Les systèmes microfluidiques fabriqués à partir de matériaux compatibles avec une détection par fluorescence sont particulièrement adaptés à ce type de détection [27]. Un exemple de système intégré combinant la production, le thermocyclage et l’analyse de gouttelettes dans une puce microfluidique a été développé par Beer et al. [28]. Des gouttelettes (d’environ 70 pL) sont immobilisées le long d’un canal par arrêt de l’écoulement, l’ensemble de la puce est soumis aux cycles de températures permettant l’amplification de l’ADN. La fluorescence des produits d’amplification est mesurée en continu. Un inconvénient de cette configuration « statique » est le nombre limité de gouttes analysables (environ 1000). Une évolution de cette technique, introduisant une modification de l’architecture des canaux et du système de détection, a permis à Hatch et al. de réaliser une expérience de dPCR comportant un million de gouttes [29]. Dans ce système, le signal de chaque goutte peut être quantifié en dépit de la forte densité de gouttes dans le réservoir, permettant ainsi d’atteindre une sensibilité de moins de dix copies d’ADN cible de Chlamydia (ADN matrice synthétique de 150-300 bp) par échantillon de 50 µL. Kiss et al. ont développé un système permettant le thermocyclage et l’analyse dynamique des gouttelettes en flux continu, assurant un suivi en temps réel des produits d’amplification [30]. Cette technique offre la possibilité de détecter une molécule cible à une concentration de 0,003 pg/µL (une molécule d’ADN viral provenant du vecteur pAdeasy-1 parmi 167 gouttelettes). Bien que ce système permette de faire une analyse complète en 35 minutes, l’étape de calibration reste délicate. De plus, le suivi d’une même goutte au cours du temps est impossible. Afin d’augmenter le débit d’analyse, les gouttes peuvent être collectées dans un réservoir extérieur et thermocyclées hors puce (Figure 5). Ce système a été appliqué à la détection de mutations rares [31]. Grâce à un module de réinjection, les gouttelettes défilent devant un faisceau laser qui permet de discriminer le signal émis par les différents variants alléliques. Une limite de détection d’une molécule mutée parmi 200 000 non mutées a été démontrée en utilisant de l’ADN génomique extrait d’une lignée cellulaire de cancer colorectal. En utilisant un système de discrimination allélique par sondes TaqMan®, plusieurs cibles peuvent être détectées simultanément [32]. D’autres travaux ont montré qu’il était possible d’atteindre des sensibilités équivalentes (1 séquence d’ADN génomique mutée parmi 100 000 séquences non mutées) [7]. Des plate-formes utilisant ces stratégies sont actuellement commercialisées (Figure 6), et sont particulièrement adaptées à l’analyse quantitative et sensible d’échantillons cliniques pour la détection de mutations rares.
 | Figure 5.
Encapsulation et analyse par dPCR en microgouttelettes. Comparaison des systèmes entièrement intégrés (gauche) et découplés (droite). |
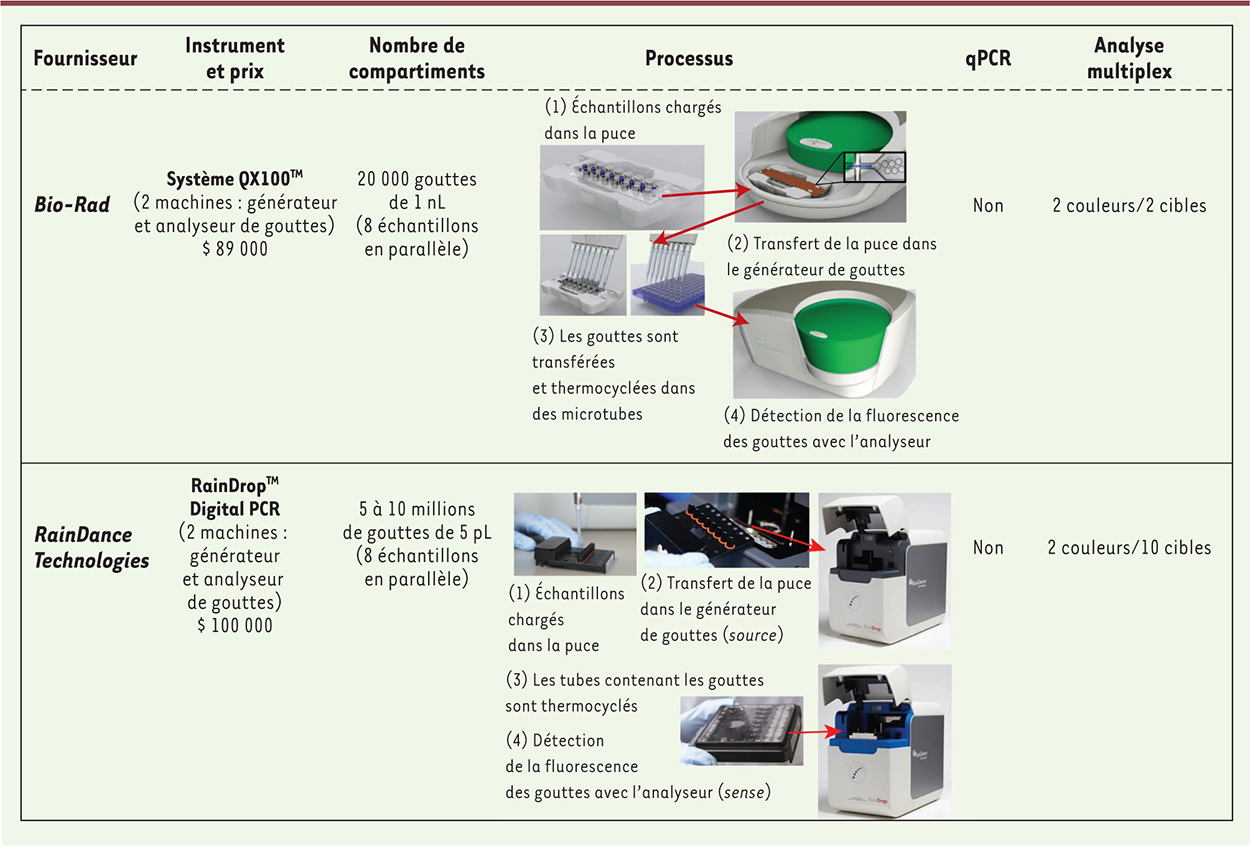
 | Figure 6.
Plates-formes microfluidiques de PCR digitale en microgouttelettes commerciales (modifiée d’après Baker et al. [ 9]). |
|
Applications biomédicales de la PCR digitale Ces dernières années, les applications de la PCR digitale se sont beaucoup développées, par exemple pour la détection de diverses cibles présentes dans la circulation sanguine, telles que l’ARN ou l’ADN provenant de virus, l’ADN fœtal ou l’ADN tumoral circulant [33]. De nombreuses équipes de recherche ont démontré la pertinence de ces tests pour la recherche clinique. La méthode conventionnelle utilisée en clinique pour la détection de virus (hépatites B et C, VIH, virus de l’herpès) est la qPCR. Comme nous l’avons vu plus haut, cette technique n’est cependant pas standardisée, et ses performances varient selon le laboratoire d’analyse, menant ainsi à des résultats discordants. La PCR digitale, capable de quantifier les acides nucléiques circulants sans courbe de calibration, permet de s’affranchir de ces inconvénients [34]. Dans ce cadre, une étude a récemment prouvé l’utilité clinique de détecter l’ADN de cytomégalovirus humain (hCMV) dans le sang de patients par la technique de dPCR en microgouttelettes [35]. Un suivi régulier de la charge virale est notamment requis pour évaluer la nécessité de prescrire des antiviraux aux patients ayant subi une greffe de cellules hématopoïétiques et qui présentent une hausse de la concentration sanguine en hCMV. En outre, la PCR digitale en microgouttelettes s’est avérée plus précise que la qPCR, la charge virale quantitative pouvant être mesurée sur une échelle de quatre décades de concentrations [35]. Une autre application de la PCR digitale est la réalisation de tests de diagnostic prénatal non invasifs. En effet, l’ADN fœtal est fortement dilué dans le sang maternel et seulement une faible fraction de cet ADN est détectable dans la circulation. Dans ce contexte, la PCR digitale permet une analyse de l’ADN circulant avec une variabilité plus faible (coefficient de variation de 16 %) qu’en qPCR (49 %) [36]. Se basant sur ces résultats, d’autres travaux ont également démontré l’intérêt de la dPCR pour la détection d’aneuploïdie fœtale, telle que la trisomie 21 [37], ou de mutations, notamment du gène bêta-globine [38]. Ces applications pourraient ainsi contribuer à une orientation thérapeutique précoce et une meilleure prise en charge. Finalement, la dPCR s’est également montrée très efficace pour la détection et l’analyse de l’ADN tumoral circulant [33] qui semble être un bon marqueur pronostic et prédictif de l’évolution de la maladie. Nous traiterons de cette application dans un second article (voir note 1) [43]. |
La miniaturisation et l’utilisation d’outils microfluidiques ont permis le développement de systèmes robustes pour la quantification par dPCR de séquences rares d’ADN muté. Diverses approches ont été explorées pour l’encapsulation de molécules uniques à partir d’échantillons soumis à une dilution limite. L’emploi de microgouttelettes ou de microchambres permet d’atteindre des volumes de compartimentation de l’ordre du picolitre et ce, de manière reproductible. De cette façon, il est possible de générer des millions de compartiments afin d’augmenter la sensibilité du test. Une analyse binaire à l’échelle de la molécule unique confère à la dPCR une très forte sensibilité par comparaison avec les procédures conventionnelles. Un autre élément clé de cette technologie est la possibilité de réaliser des expériences réellement quantitatives, permettant de déterminer avec précision la fraction des séquences cibles dans les échantillons. Bien que les systèmes microfluidiques restent coûteux pour une utilisation routinière, de nouveaux matériaux sont en cours de développement qui réduiront les coûts de fabrication. De plus, l’analyse de centaines d’échantillons en parallèle permet de rentabiliser le prix de chaque puce. Le champ de la microfluidique promet de nombreux avantages tels que l’automatisation de processus complexes successifs [39]. La commercialisation de machines automatisées pour la dPCR a permis son intégration dans de nombreux projets de recherche. Ces récents développements ont été suivis par la définition de directives d’informations minimales pour la publication d’expériences quantitatives de PCR en temps réel (MIQE, minimum information for publication of quantitative real-time PCR experiments) [19] qui contribueront encore à la démocratisation de ces technologies. Les prochains développements devraient impliquer des plates-formes hautement intégrées utilisant la dPCR comme un élément parmi une chaîne d’opérations miniaturisées et automatisées. |
V. Taly et P. Laurent-Puig déclarent participer à des rapports d’expertise pour l’entreprise Raindance Technologies.
Les autres auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Les auteurs remercient, pour leur soutien financier, le ministère de l’Enseignement supérieur et de la Recherche, l’université Paris-Descartes, l’université de Strasbourg, le Centre national de la recherche scientifique (CNRS), l’Institut national de la santé et de la recherche médicale (Inserm), l’Institut national du cancer (Inca, n° 2009-1-RT-03-US-1 et 2009-RT-03-UP5-1), l’Association pour la recherche contre le cancer (ARC, no SL220100601375), l’Agence nationale de la recherche (ANR Nanobiotechnologies), la région Alsace, et le SIRIC CARPEM. J.-C. Baret remercie la Société Max Planck et l’Initiative d’Excellence de l’université de Bordeaux pour leur soutien financier.
|
1.
Sawyers CL. The cancer biomarker problem . Nature. 2008; ; 452 : :548.–552. 2.
Morrison TB, Weis JJ, Wittwer CT. Quantification of low-copy transcripts by continuous SYBR Green I monitoring during amplification . Biotechniques. 1998; ; 24 : :954.–962. 3.
Holland PM, Abramson RD, Watson R, Gelfand DH. Detection of specific polymerase chain reaction product by utilizing the 5’-3’ exonuclease activity of Thermus aquaticus DNA polymerase . Proc Natl Acad Sci USA. 1991; ; 88 : :7276.–7280. 4.
Thierry AR, Mouliere F, El Messaoudi S, et al. Clinical validation of the detection of KRAS and BRAF mutations from circulating tumor DNA . Nat Med. 2014; ; 20 : :430.–435. 5.
Sykes PJ, Neoh SH, Brisco MJ, et al. Quantitation of targets for PCR by use of limiting dilution . Biotechniques. 1992; ; 13 : :444.–449. 6.
Vogelstein B, Kinzler KW, Digital PCR . Proc Natl Acad Sci USA. 1999; ; 96 : :9236.–9241. 7.
Hindson BJ, Ness KD, Masquelier DA, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number . Anal Chem. 2011; ; 83 : :8604.–8610. 8.
Dingle TC, Sedlak RH, Cook L, Jerome KR. Tolerance of droplet-digital PCR versus real-time quantitative PCR to inhibitory substances . Clin Chem. 2013; ; 59 : :1670.–1672. 9.
Baker M.. Digital PCR hits its stride . Nat Methods. 2012; ; 9 : :541.–544. 10.
Baret JC, Taly V, Ryckelynck M, et al. Gouttes et émulsions : criblage à très haut débit en biologie . Med Sci (Paris). 2009; ; 25 : :627.–632. 11.
Morrison T, Hurley J, Garcia J, et al. Nanoliter high throughput quantitative PCR . Nucleic Acids Res. 2006; ; 34 : :e123.. 12.
Thorsen T, Maerkl SJ, Quake SR. Microfluidic large-scale integration . Science. 2002; ; 298 : :580.–584. 13.
Warren L, Bryder D, Weissman IL, Quake SR. Transcription factor profiling in individual hematopoietic progenitors by digital RT-PCR . Proc Natl Acad Sci USA. 2006; ; 103 : :17807.–17812. 14.
Yung TKF, Chan KCA, Mok TSK, et al. Single-molecule detection of epidermal growth factor receptor mutations in plasma by microfluidics digital PCR in non-small cell lung cancer patients . Clin Cancer Res. 2009; ; 15 : :2076.–2084. 15.
Henríquez-Hernández LA, Valenciano A, Herrera-Ramos E, et al. High-throughput genotyping system as a robust and useful tool in oncology: experience from a single institution . Biologicals. 2013; ; 41 : :424.–429. 16.
Hudson J, Duncavage E, Tamburrino A, et al. Overexpression of miR-10a and miR-375 and downregulation of YAP1 in medullary thyroid carcinoma . Exp Mol Pathol. 2013; ; 95 : :62.–67. 17.
Shen F, Du W, JE Kreutz JE, et al. Digital PCR on a SlipChip . Lab Chip. 2010; ; 10 : :2666.–2672. 18.
Williams R, Peisajovich SG, Miller OJ, et al. Amplification of complex gene libraries by emulsion PCR . Nat Methods. 2006; ; 3 : :545.–550. 19.
Huggett JF, Foy CA, Benes V, et al. The digital MIQE guidelines: minimum information for publication of quantitative digital PCR experiments . Clin Chem. 2013; ; 59 : :892.–902. 20.
Theberge AB, Courtois F, Schaerli Y, et al. Microdroplets in microfluidics: an evolving platform for discoveries in chemistry and biology . Angew Chemie. 2010; ; 49 : :5846.–5868. 21.
Dressman D, Yan H, Traverso G, et al. Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations . Proc Natl Acad Sci USA. 2003; ; 100 : :8817.–8822. 22.
Diehl F, Li M, Dressman D, et al. Detection and quantification of mutations in the plasma of patients with colorectal tumors . Proc Natl Acad Sci USA. 2005; ; 102 : :16368.–16373. 23.
Taly V, Pekin D, El Abed A, Laurent-Puig P. Detecting biomarkers with microdroplet technology . Trends Mol Med. 2012; ; 18 : :405.–416. 24.
Whitesides GM. The origins and the future of microfluidics . Nature. 2006; ; 442 : :368.–373. 25.
The SY, Lin R, Hung LH, Lee AP. Droplet microfluidics . Lab Chip. 2008; ; 8 : :198.–220. 26.
Baret JC. Surfactants in droplet-based microfluidics . Lab Chip. 2012; ; 12 : :422.–433. 27.
Baret JC, Beck Y, Billas-Massobrio I, et al. Quantitative cell-based reporter gene assays using droplet-based microfluidics . Chem Biol. 2010; ; 17 : :528.–536. 28.
Beer NR, Wheeler EK, Lee-houghton L, et al. On-chip single-copy real-time reverse-transcription PCR in isolated picoliter droplets . Anal Chem. 2008; ; 80 : :1854.–1858. 29.
Hatch AC, Fisher JS, Tovar AR, et al. 1-Million droplet array with wide-field fluorescence imaging for digital PCR . Lab Chip. 2011; ; 11 : :3838.–3845. 30.
Kiss MM, Ortoleva-Donnelly L, Beer NR, et al. High-throughput quantitative polymerase chain reaction in picoliter droplets . Anal Chem. 2008; ; 80 : :8975.–8981. 31.
Pekin D, Skhiri Y, Baret JC, et al. Quantitative and sensitive detection of rare mutations using droplet-based microfluidics . Lab Chip. 2011; ; 11 : :2156.–2166. 32.
Taly V, Pekin D, Benhaim L, et al. Multiplex picodroplet digital PCR to detect KRAS mutations in circulating DNA from the plasma of colorectal cancer patients . Clin Chem. 2013; ; 59 : :1722.–1731. 33.
Diaz LA, Bardelli A. Liquid biopsies: genotyping circulating tumor DNA . J Clin Oncol. 2014; ; 32 : :579.–586. 34.
Nixon G, Garson JA, Grant P, et al. Comparative study of sensitivity, linearity, and resistance to inhibition of digital and nondigital polymerase chain reaction and loop mediated isothermal amplification assays for quantification of human cytomegalovirus . Anal Chem. 2014; ; 86 : :438794.. 35.
Sedlak RH, Cook L, Cheng A, et al. Evaluation of the clinical utility of droplet digital PCR for human cytomegalovirus . J Clin Microbiol. 2014; ; 52 : :6.–11. 36.
Lun FMF, Chiu RWK, Allen Chan KC, et al. Microfluidics digital PCR reveals a higher than expected fraction of fetal DNA in maternal plasma . Clin Chem. 2008; ; 54 : :1664.–1672. 37.
Fan HC, Quake SR. Detection of aneuploidy with digital polymerase chain reaction . Anal Chem. 2007; ; 79 : :7576.–7579. 38.
Lun FMF, Tsui NBY, Chan KCA, et al. Noninvasive prenatal diagnosis of monogenic diseases by digital size selection and relative mutation dosage on DNA in maternal plasma . Proc Natl Acad Sci USA. 2008; ; 105 : :19920.–19925. 39.
Fallah-Araghi A, Baret JC, Ryckelynck M, Griffiths AD. A completely in vitro ultrahigh-throughput droplet-based microfluidic screening system for protein engineering and directed evolution . Lab Chip. 2012; ; 12 : :882.–891. 40.
Simi L, Pratesi N, Vignoli M, et al. High-resolution melting analysis for rapid detection of KRAS, BRAF, and PIK3CA gene mutations in colorectal cancer . Am J Clin Pathol. 2008; ; 130 : :247.–253. 41.
Weichert W, Schewe C, Lehmann A, et al. KRAS genotyping of paraffin-embedded colorectal cancer tissue in routine diagnostics . J Mol Diagnostics. 2010; ; 12 : :35.–42. 42.
Lièvre A, Bachet J-B, Boige V, et al. KRAS mutations as an independent prognostic factor in patients with advanced colorectal cancer treated with cetuximab . J Clin Oncol. 2008; ; 26 : :374.–379. 43.
Caen O, Nizard P, Garrigou S, et al. Apport de la PCR digitale pour la détection quantitative d’ADN tumoral circulant . Med Sci (Paris). 2015; ; 31 (sous presse). |