| |
| Med Sci (Paris). 2015 March; 31(3): 291–303. Published online 2015 April 8. doi: 10.1051/medsci/20153103015.Principes et applications de l’optogénétique en neuroscience Guillaume P. Dugué*1 and Ludovic Tricoire2 1CNRS UMR 8197, Inserm U1024, IBENS S4.9, 46, rue d’Ulm, 75005Paris, France 2CNRS UMR 8246, Inserm U1130, université Pierre et Marie Curie UM CR119, 9, quai Saint Bernard, 75005Paris, France |
Le fonctionnement du cerveau repose sur l’interaction de multiples structures nerveuses remplissant des tâches complémentaires. Cette division du travail existe aussi au niveau cellulaire puisqu’un même microvolume de tissu nerveux rassemble le plus souvent plusieurs types neuronaux aux fonctions distinctes. L’imbrication de ces différentes populations neuronales pose un défi méthodologique fondamental : comment évaluer séparément le(s) rôle(s) de chaque type neuronal dans l’émergence de fonctions cérébrales intégrées et dans la genèse des comportements ? Nous avons pour cela besoin de méthodes d’observation et d’intervention qui soient à la fois sélectives et peu invasives. Francis Crick, codécouvreur de la structure de l’ADN avec James Watson en 1952, a consacré plusieurs articles spéculatifs à cette problématique dès la fin des années 1970 [1, 2]. Il fut l’un des premiers à entrevoir le potentiel d’une association entre manipulations génétiques et utilisation de la lumière. Crick imagina en particulier la possibilité d’utiliser des outils moléculaires capables d’émettre de la lumière en lien avec l’activité neuronale, ou capables de modifier l’activité neuronale une fois photoactivés, et dont l’expression pourrait être ciblée dans des types neuronaux spécifiques. Il anticipa ainsi la naissance d’une nouvelle discipline connue aujourd’hui sous le nom d’optogénétique. Le terme « optogénétique », inventé en 2006 par l’un des principaux pionniers dans le domaine, Karl Deisseroth [3], rassemble dans un concept unique un ensemble d’approches apparues indépendamment depuis la fin des années 1990. Initialement cantonnée aux neurosciences, l’optogénétique s’est ensuite développée dans de nombreux domaines de la biologie. Le terme désigne aujourd’hui l’utilisation d’outils moléculaires qui peuvent être génétiquement ciblés pour l’observation et la manipulation de structures ou fonctions cellulaires spécifiques à l’aide de la lumière [4]. En neuroscience, l’optogénétique permet d’observer et de contrôler l’activité de groupes de neurones spécifiques par simple illumination du tissu nerveux, dans des préparations allant de la culture de neurones jusqu’à l’animal en situation comportementale. Cette nouvelle discipline est considérée par les neurobiologistes, de manière quasi unanime, comme une révolution technologique majeure (→).
(→) Voir l’éditorial d'A.R. Adamantidis, page 231 de ce numéro
|
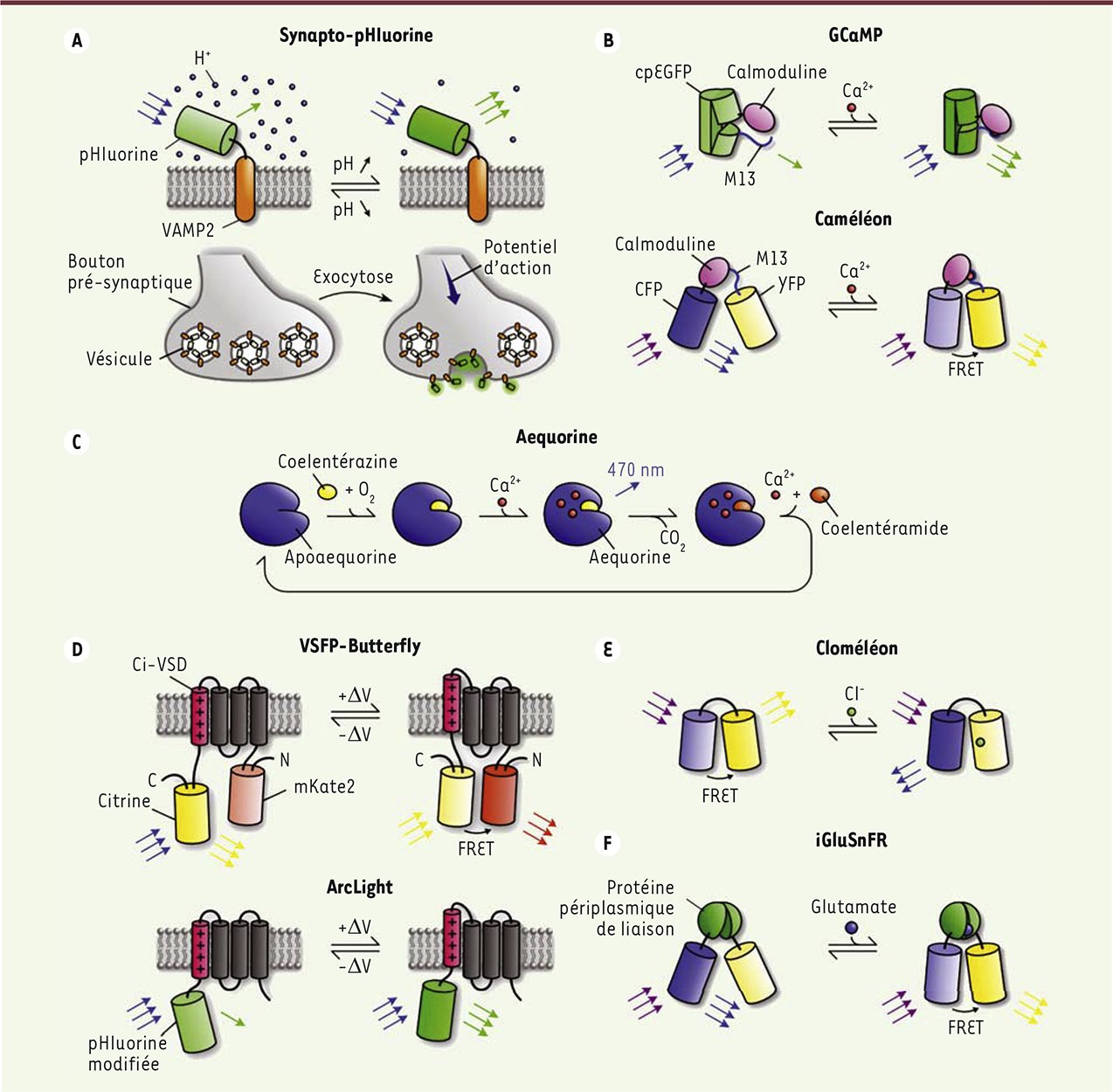
Imagerie optogénétique de l’activité neuronale L’activité électrique cérébrale est communément schématisée à l’attention du grand public par un crépitement de flashs lumineux parcourant des réseaux de neurones. Cette représentation, bien que totalement fantaisiste, correspond toutefois à un vieux rêve des neurobiologistes : disposer d’une méthode permettant une lecture optique de l’activité neuronale et ainsi s’affranchir des limitations inhérentes aux techniques d’électrophysiologie extracellulaire, qui ne permettent pas toujours d’identifier avec certitude les types neuronaux enregistrés. Plusieurs pistes furent successivement explorées, comme par exemple la mesure de changements dans les propriétés optiques des neurones en fonction de leur activité, jusqu’à la synthèse, dans les années 1980-1990, de molécules organiques dont la fluorescence est sensible, soit directement aux variations du potentiel transmembranaire [5], soit aux fluctuations de la concentration de calcium intracellulaire induites par l’activité neuronale [6]. Ces indicateurs, dont certains sont encore largement utilisés, représentaient un réel progrès mais avaient l’inconvénient de marquer indistinctement neurones et cellules gliales. Les efforts se sont tournés, dès le milieu des années 1990, vers la synthèse d’indicateurs protéiques, dont l’expression pouvait être ciblée dans des populations cellulaires spécifiques par les techniques alors émergentes de génie génétique. Les indicateurs d’activité codés génétiquement, en particulier les sondes sensibles au calcium et les sondes sensibles à la différence de potentiel transmembranaire, reçoivent une attention de plus en plus forte de la part de la communauté des neurobiologistes. Les grandes étapes de leur développement ont fait l’objet de nombreuses revues dont nous ne citerons ici que les plus récentes [7–9]. Indicateurs fluorescents de l’activité neuronale Le clonage en 1992 du gène codant la GFP ( green fluorescent protein), protéine fluorescente de la méduse Aequorea victoria, fut à la fois l’aboutissement d’un travail de longue haleine débuté 30 ans plus tôt par la découverte de cette protéine, et le point de départ d’une série de travaux qui allaient conduire à la synthèse des premiers indicateurs fluorescents codés génétiquement [ 10]. Au fil des années, des efforts concertés en biochimie et génétique moléculaire ont permis d’améliorer la stabilité et la brillance de protéines dérivées de la GFP, ainsi que de diversifier leurs propriétés spectrales. En parallèle, de nouvelles protéines fluorescentes issues d’autres espèces marines ont été isolées, la découverte la plus notable étant celle de la DsRed à partir du corail Discosoma sp. [ 11]. Son spectre d’émission à dominante rouge est venu enrichir la palette de couleurs des protéines fluorescentes utilisées en biologie [ 12, 13]. Les pHluorines Afin de transformer une protéine fluorescente en indicateur, une première méthode consiste à exacerber par mutagenèse sa sensibilité naturelle à certains paramètres physico-chimiques. Cette méthode fut utilisée par exemple dans le cas de la sensibilité de la GFP au pH, qui fut exploitée pour donner naissance aux pHluorines, une famille de protéines dont la fluorescence est fortement diminuée en milieu acide [ 14]. La fusion d’une pHluorine avec VAMP2 ( vesicle-associated membrane protein 2), une protéine associée aux vésicules synaptiques, a permis de générer un outil, la synaptopHluorine, capable de signaler l’activité présynaptique d’un neurone [ 14] : la fluorescence de la synaptopHluorine est inhibée par l’acidité du pH intravésiculaire et n’est révélée que lorsque la vésicule libère son contenu dans le milieu extracellulaire, dont le pH est neutre (Figure 1A).
 | Figure 1.
Principaux indicateurs optogénétiques de l’activité neuronale. A. Haut : les synaptopHluorines exposent une protéine fluorescente sensible au pH (pHluorine) dans la lumière des vésicules synaptiques (pH = 4-5). Bas : Les pHluorines recouvrent leur fluorescence au contact du milieu extracellulaire lors de la fusion des vésicules (pH = 7,4). B. Exemple de deux indicateurs calciques fluorescents codés génétiquement, basés sur le couple senseur calmoduline-M13 et utilisant soit une GFP permutée circulairement (cpEGFP) dans le cas des GCaMP (haut), soit une paire de protéines fluorescentes compatibles avec le phénomène de FRET dans le cas des Caméléons (bas). C. Cycle enzymatique de l’aequorine aboutissant à l’émission d’un photon consécutivement à l’oxydation de la coelentérazine en présence de calcium. D. Exemple de deux indicateurs de la différence de potentiel transmembranaire basés sur le domaine sensible au voltage d’une phosphatase de l’ascidie Ciona Intestinalis (Ci-VSD), l’un utilisant le FRET (VSFP-Butterfly, haut) et l’autre utilisant une pHluorine modifiée (ArcLight, bas). E. Dans les indicateurs d’ions chlorure de la famille Cloméléon, le phénomène de FRET a lieu à l’état basal. La liaison d’ions chlorure sur l’accepteur (YFP) induit une extinction de sa fluorescence et une diminution du ratio de fluorescence accepteur/donneur (YFP/CFP). Les sondes Cloméléon sont utilisées pour détecter les flux d’ions chlorures associés à l’ouverture de récepteurs canaux activés par les neurotransmetteurs inhibiteurs. F. Exemple de sonde FRET utilisée pour la détection de glutamate (le neurotransmetteur excitateur majoritaire du cerveau), basée sur une protéine périplasmique bactérienne liant le glutamate. Par convention, les membranes sont schématisées avec la face cytoplasmique vers le bas et la face extracellulaire ou luminale vers le haut. Les flèches de couleurs pointant vers ou partant des protéines fluorescentes symbolisent respectivement la lumière absorbée et la lumière émise (reproduit d’après [ 4]). |
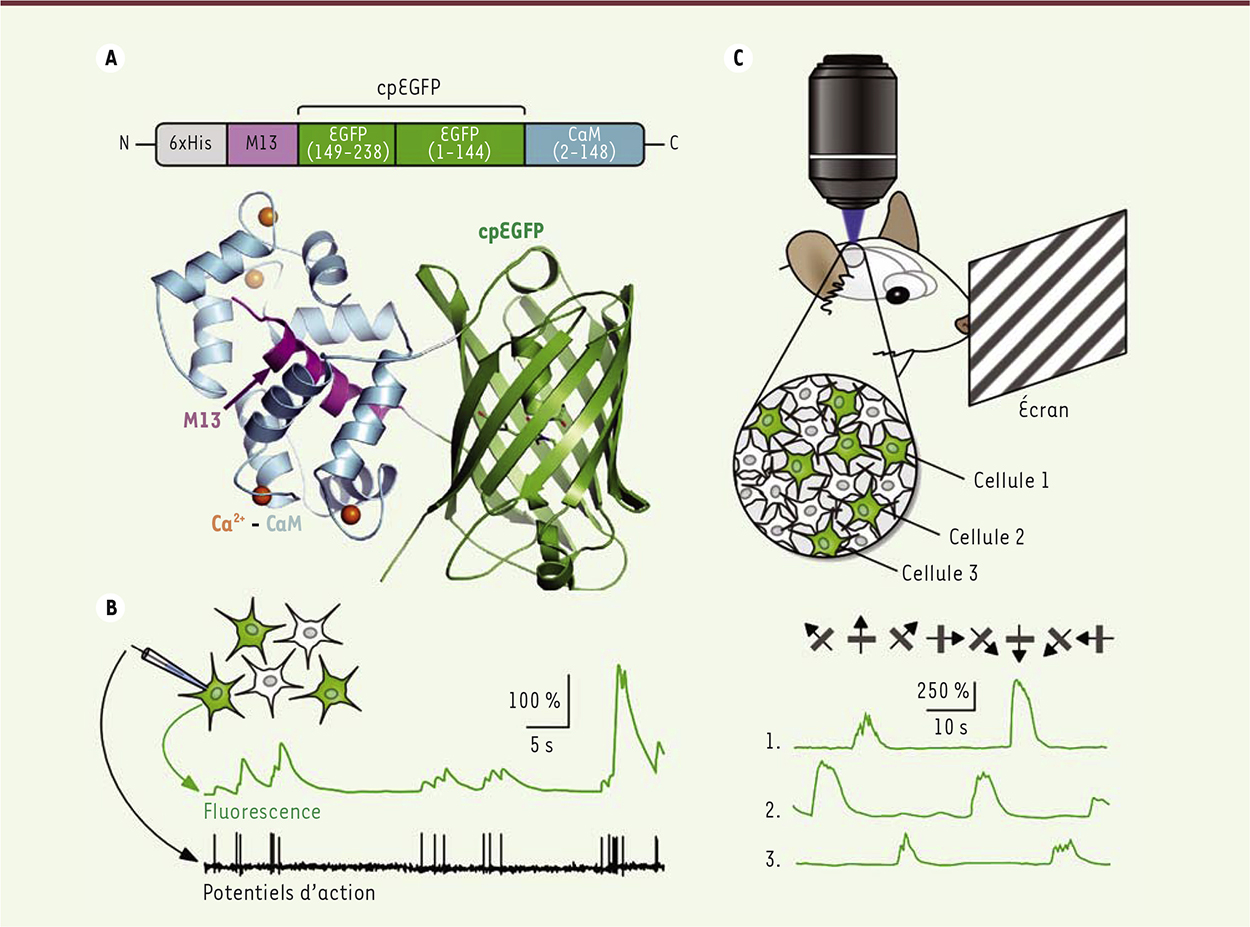
Indicateurs composés d’une protéine fluorescente unique Une deuxième approche, aux possibilités d’applications plus étendues, consiste à construire un indicateur en greffant un domaine protéique senseur sur une protéine fluorescente. En interagissant avec des ions, des biomolécules, ou en fonction de paramètres physico-chimiques spécifiques, le domaine senseur imprime un changement de conformation à la structure de la protéine fluorescente, ce qui a pour effet de faire varier sa fluorescence. Dans de tels indicateurs, la protéine fluorescente est souvent modifiée afin de rendre sa fluorescence plus sensible aux changements conformationnels induits par le senseur (par exemple par permutation circulaire). Les indicateurs d’activité neuronale construits selon ce principe regroupent des sondes sensibles au calcium (sondes calciques) et des sondes sensibles à la différence de potentiel transmembranaire. Parmi les sondes calciques, les indicateurs de la famille GCaMP (Figure 1B) ont fait l’objet d’un travail d’optimisation particulièrement poussé au cours des six dernières années, facilité en particulier par la connaissance de leur structure tridimensionnelle obtenue par cristallographie aux rayons X [ 15]. Les GCaMP sont constituées d’une GFP permutée, intercalée entre une protéine liant le calcium appelée calmoduline (CaM) et un peptide synthétique appelé M13 (identique au domaine de liaison à la CaM de la kinase de la chaîne légère de la myosine ;
Figure 2A
). En l’absence de calcium, la GFP permutée est faiblement fluorescente. En liant le calcium, le domaine CaM s’enroule autour du peptide M13 et provoque un changement conformationnel de la protéine qui restaure la fluorescence de la GFP. On dénombre aujourd’hui une vingtaine de variants dérivés du modèle GCaMP, dont les plus récents ont démontré leur capacité à détecter des fluctuations de calcium intracellulaire associées à des potentiels d’action uniques dans les corps cellulaires et terminaisons axonales de neurones corticaux observés in vivo (Figure 2B–C) [ 16]. Les GCaMP ont permis de démontrer la faisabilité d’enregistrements prolongés in vivo de l’activité de plusieurs centaines voire de plusieurs milliers de neurones ciblés génétiquement, dans des modèles aussi divers que le nématode, la drosophile, le poisson zèbre et la souris [ 17– 22]. Des progrès significatifs ont également été réalisés dans le domaine des sondes sensibles à la différence de potentiel transmembranaire, avec notamment les sondes ArcLight et ASAP1 (Figure 1D), capables de signaler des potentiels d’action uniques dans des préparations isolées (neurones en culture ou explants). Cependant, la taille des signaux obtenus reste de l’ordre de quatre fois inférieure à celle des meilleures sondes calciques [ 23, 24].
 | Figure 2.
Mesure de l’activité neuronale à l’aide des sondes calciques GCaMP. A. Haut : Schéma de la séquence peptidique d’une sonde GCaMP. L’EGFP ( enhanced green fluorescent protein) a été modifiée par permutation circulaire (cpEGFP). Les séquences peptidiques assurant la jonction entre les différents domaines ne sont pas représentées. Bas : structure tridimensionnelle de la GCaMP2 obtenue par cristallographie aux rayons X. Reproduit d’après [ 15]. B. Enregistrement simultané de la fluorescence d’un neurone unique du cortex visuel exprimant la GCaMP6s (tracé vert) et de son activité électrique (tracé noir) in vivo. Chaque trait vertical dans la trace électrophysiologique représente un potentiel d’action unique. Reproduit d’après [ 16]. C. Variations de la fluorescence de neurones du cortex visuel primaire de souris exprimant GCaMP6s, induites par le mouvement de rayures verticales projetées sur un écran (préparation : souris anesthésiée). On observe que chaque neurone s’active en réponse à une direction particulière du mouvement (les tracés verts représentent la réponse de trois neurones différents enregistrés simultanément). Reproduit d’après [ 16]. |
Indicateurs basés sur le phénomène de FRET Une troisième approche consiste à intercaler un domaine senseur entre deux protéines fluorescentes. Les indicateurs construits selon ce schéma tirent parti d’un phénomène de transfert d’énergie non radiatif appelé FRET ( Förster resonance energy transfer) entre les deux protéines fluorescentes, l’une jouant le rôle de donneur d’énergie et l’autre le rôle d’accepteur. Pour que ce phénomène ait lieu, il faut que le spectre d’émission du donneur chevauche au moins en partie le spectre d’absorption de l’accepteur. L’efficacité du FRET (mesurée par le ratio de fluorescence accepteur/donneur) est extrêmement sensible à la distance entre les deux protéines fluorescentes ainsi qu’à leur arrangement relatif. Dans un indicateur basé sur le FRET, le changement conformationnel du domaine senseur induit un changement de l’arrangement spatial des deux protéines fluorescentes qui se traduit par une variation de leur ratio de fluorescence (on parle d’indicateur « ratiométrique »). Ce principe a été très largement appliqué à la détection de nombreux ions et métabolites. Dans le cas spécifique des indicateurs d’activité neuronale, les sondes FRET incluent des senseurs de neurotransmetteurs tels que la glycine ou le glutamate (Figure 1E–F), des sondes calciques (Figure 1B) et des indicateurs sensibles à la différence de potentiel transmembranaire (Figure 1D). Dans le cas des indicateurs calciques Caméléon (Figure 1B), le couple peptide M13-CaM est fusionné à une paire de variants bleu et jaune de la GFP (CFP et YFP, respectivement). En présence de calcium, CFP et YFP se rapprochent, facilitant le transfert d’énergie entre ces deux protéines, ce qui se traduit par une augmentation du ratio de fluorescence YFP/CFP. Même si aucun des indicateurs basés sur le FRET n’a aujourd’hui la popularité des sondes GCaMP, les dernières versions des sondes calciques FRET (en particulier les sondes Twitch basées sur l’architecture TN-XXL, qui utilise la troponine comme senseur calcique) ainsi que la sonde sensible au voltage VSFP-Butterfly (Figure 1D), ont donné des résultats très encourageants in vivo [ 25– 27]. L’aequorine : un indicateur calcique luminescent L’aequorine est une enzyme luminescente issue de la méduse Aequorea victoria, qui fut découverte en même temps que la GFP. Il s’agit d’un complexe entre une protéine possédant trois sites de liaison au calcium, l’apoaequorine, et un groupe prosthétique luminophore (molécule organique non protéique qui peut émettre de la lumière une fois excitée) appelé coelentérazine. En réponse à une augmentation de calcium, l’aequorine émet rapidement (en environ 10 ms) de la lumière bleue suite à l’oxydation de la coelentérazine en coelentéramide (
Figure 1C
; chez Aequorea victoria, la lumière émise est décalée vers le vert en raison d’un transfert d’énergie non radiatif similaire au FRET entre l’aequorine et la GFP) [ 28]. D’autres enzymes fonctionnant comme l’aequorine ont été découvertes, comme l’obéline, la clytine et la mitrocomine. Ensemble, elles constituent la famille des photoprotéines. Historiquement, l’aequorine fut le premier indicateur calcique à être utilisé en biologie pour l’étude des influx calciques dans les fibres musculaires d’un crustacé marin, la bernacle [ 29], ou pour l’étude du rôle du calcium dans la libération des neurotransmetteurs chez le calmar [ 30]. La protéine devait alors être purifiée et injectée directement dans l’animal. Après le clonage de son gène en 1985 [ 31], l’aequorine put ensuite être exprimée par voie génétique et fut utilisée dans des tests de criblages pharmacologiques, mais aussi pour mesurer les variations de la concentration intracellulaire de calcium dans de multiples organismes, en particulier les plantes [ 32]. À l’instar de la GFP, l’aequorine a fait l’objet d’un travail d’optimisation en vue d’améliorer son niveau d’expression, de cibler son adressage vers différents compartiments subcellulaires comme la mitochondrie ou le réticulum endoplasmique, et de décaler son spectre d’émission, en particulier vers le rouge [ 33]. Lorsqu’elle est exprimée dans des neurones, l’apoaequorine n’est pas toxique et altère peu l’homéostasie calcique. Pour reconstituer l’aequorine, il suffit d’ajouter la coelentérazine dans le milieu extracellulaire (cette dernière traverse bien les membranes cellulaires). L’aequorine peut alors indiquer des augmentations de la concentration intracellulaire de calcium associées à l’activité neuronale par des augmentations de l’intensité de la lumière émise. L’imagerie basée sur l’aequorine n’est pas suffisamment sensible pour détecter des potentiels d’action uniques, mais elle génère des signaux de grande taille en réponse à des bouffées de potentiels d’actions [34]. Contrairement à l’imagerie basée sur l’utilisation d’indicateurs fluorescents, elle ne nécessite pas de source de lumière et ne souffre donc pas des inconvénients liés à l’illumination du tissu (potentielle phototoxicité de la lumière d’excitation, photoblanchiment des protéines fluorescentes et autofluorescence du tissu qui vient parasiter les mesures). Malgré ces avantages, l’utilisation de l’aequorine in vivo dans l’étude du système nerveux est limitée par la nécessité de supplémenter le milieu extracellulaire en coelentérazine. Cette molécule franchit mal la barrière hémato-encéphalique, probablement sous l’action d’un transporteur responsable du phénomène de multirésistance aux médicaments [35], ce qui réduit le rendement des injections de coelentérazine par voie systémique. L’aequorine est ainsi majoritairement utilisée dans des préparations isolées telles que les tranches de cerveau [36], dans des modèles permettant une pénétration facile de la coelenterazine dans le tissu nerveux tels que la larve de poisson zèbre [37] ou la drosophile [38], ou enfin pour l’imagerie des patrons de contractions musculaires chez la souris vigile libre de ses mouvements [39]. |
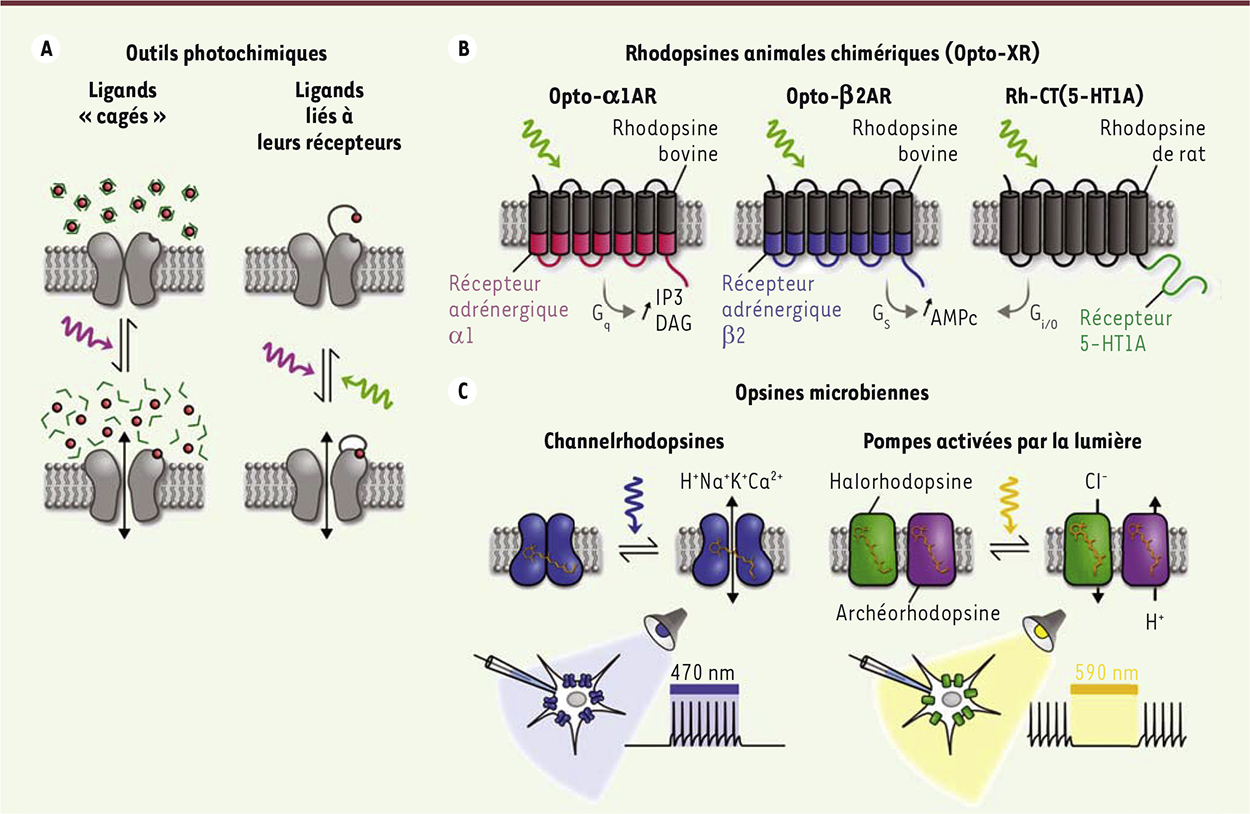
Contrôle optogénétique de l’activité neuronale Pour comprendre le fonctionnement du cerveau, une étape essentielle consiste à mettre en évidence les relations de causalité (nécessité et/ou suffisance) reliant l’activité de neurones spécifiques à des fonctions neurophysiologiques ou comportements particuliers. Les méthodes traditionnelles visant à manipuler l’activité neuronale reposent très majoritairement sur des stimulations électriques ou magnétiques, et sur l’utilisation d’agents pharmacologiques. Ces méthodes sont limitées par leur faible capacité de ciblage de populations neuronales spécifiques ainsi que par leur faible résolution spatiale et/ou temporelle (faible résolution spatiale des stimulations électriques et magnétiques, faible résolution temporelle des approches pharmacologiques). Des efforts récents dans les domaines de la photochimie et de l’ingénierie génétique ont fourni plusieurs familles d’outils optogénétiques permettant de contrôler l’activité de neurones ciblés génétiquement avec, pour certains d’entre eux, une précision spatiotemporelle jusque-là inégalée. La fréquence à laquelle un neurone émet des potentiels d’action (son taux de décharge) est sous le contrôle direct de sa différence de potentiel transmembranaire, qui, elle-même, est influencée par les flux d’ions à travers sa membrane. Ces flux sont contrôlés par des protéines transmembranaires jouant le rôle de pompes ou de canaux (assurant respectivement le transport actif et passif de différentes espèces ioniques). Les stratégies de contrôle optogénétique de l’activité neuronale visent à générer des flux d’ions sous l’effet de la lumière, soit à l’aide d’outils photosensibles capables d’influencer l’activité de ces protéines transmembranaires, soit en exprimant dans les neurones des canaux ou des pompes directement photosensibles. Outils issus de la photochimie L’activité de certains canaux est conditionnée par la présence d’un ligand : on parle alors de récepteur-canal ou récepteur « ionotropique ». Certaines approches photochimiques visent à synthétiser des couples ligand-récepteur dont l’interaction est contrôlée par la lumière [ 40]. L’activité du ligand peut par exemple être bloquée par un groupement photosensible et restaurée sous l’effet de la lumière par photolyse ou changement conformationnel de ce groupement (on parle alors respectivement de composé « cagé » et de ligand « photochromique » ;
Figure 3A
). Afin de contrôler plus finement le processus d’activation photochimique, le ligand peut être greffé de manière covalente sur son récepteur par l’intermédiaire d’un groupement photochromique. L’interaction du ligand avec son récepteur est alors rendue possible par l’isomérisation du photochrome sous l’effet de la lumière (Figure 3A).
 | Figure 3.
Principaux outils optogénétiques pour le contrôle de l’activité neuronale. A. Les outils photochimiques comprennent un élément codé génétiquement (le récepteur, en gris) ainsi qu’un ligand organique exogène (rouge) en solution (gauche) ou attaché de manière covalente à son récepteur (droite). La lumière est utilisée soit pour « décager » le ligand par photolyse (gauche), soit pour le présenter de manière réversible à sa poche de liaison par isomérisation (droite). B. Les rhodopsines animales sont aujourd’hui surtout utilisées pour photoactiver des voies de signalisation intracellulaires spécifiques à l’aide de protéines chimères. Les protéines « Opto-XR » en particulier ciblent des seconds messagers intracellulaires différents selon le récepteur métabotropique auquel elles empruntent des domaines cytoplasmiques. C. Les opsines microbiennes sont de loin les outils de contrôle les plus utilisés. Les channelrhodopsines permettent de dépolariser des neurones rapidement en faisant circuler des cations à travers la membrane en présence de lumière bleue. Les pompes activées par la lumière vert-jaune (halorhodopsine et archéorhodopsines) permettent de générer des flux d’ions hyperpolarisants. |
Parmi les outils photochimiques disponibles, n’entrent dans la catégorie des outils optogénétiques que ceux dont l’expression peut être ciblée génétiquement. Ce peut être le cas par exemple d’un récepteur natif (non modifié) exprimé de manière hétérologue, c’est-à-dire dans un organisme hôte qui ne possède pas naturellement ce récepteur. Le laboratoire de Gero Miesenbock est ainsi parvenu à contrôler spécifiquement l’activité des neurones du ganglion thoracique de drosophile par l’introduction ciblée d’un récepteur de l’adénosine triphosphate (ATP) de mammifère [41]. L’activité de ce récepteur a pu être contrôlée in vivo par la photolyse d’ATP cagé, ce qui a permis de démontrer que l’activité des neurones ciblés était suffisante pour déclencher le battement des ailes. Une approche plus ambitieuse consiste à synthétiser des récepteurs n’existant pas à l’état naturel, activables par un ligand photochromique artificiel. Citons par exemple le canal potassique « HyLighter », construit par fusion d’un récepteur au glutamate de mammifère et d’un canal potassique de bactérie, qui permet d’inhiber l’activité neuronale de manière réversible [42]. Enfin, une méthode plus facilement généralisable consiste à muter des récepteurs existants afin de pouvoir leur greffer un ligand photochromique in situ, tout en conservant leur sensibilité au ligand endogène. Une fois réintroduits dans le génome, ces récepteurs mutants s’expriment et se comportent comme leur version native, mais peuvent être activés ou inhibés par la lumière après addition d’un ligand photochromique artificiel. Cette méthode a été baptisée « pharmacologie optogénétique », car la lumière se substitue en quelque sorte aux agonistes et antagonistes pharmacologiques traditionnellement utilisés pour influencer l’activité des récepteurs [43]. Elle va même plus loin puisqu’elle offre théoriquement la possibilité de cibler des récepteurs pour lesquels il n’existe pas de pharmacologie spécifique connue. Les outils optogénétiques photochimiques basés sur des récepteurs ionotropiques agissent sur le potentiel transmembranaire à l’échelle de la milliseconde et permettent donc l’étude de phénomènes neuronaux rapides. Un désavantage majeur réside toutefois dans le fait que le ligand photochromique doit être fourni de manière exogène, ce qui complique l’utilisation de ces outils in vivo. Enfin, de manière générale, les longueurs d’onde utilisées par la photochimie sont relativement courtes (350-400 nm) et pénètrent peu dans le tissu nerveux, ce qui limite leur rayon d’action. Outils basés sur les opsines animales Il est possible de modifier le potentiel transmembranaire d’une cellule à l’aide de la lumière sans faire appel à la photochimie. De nombreux types de cellules photoréceptrices ont développé depuis des centaines de millions d’années des machineries protéiques de phototransduction couplées à des canaux ioniques. Chez les animaux ainsi que chez un certain nombre d’eucaryotes unicellulaires et de procaryotes, l’élément clé de ces assemblages moléculaires est une superfamille de protéines transmembranaires appelées opsines [ 44]. Après avoir incorporé un pigment photosensible, les opsines sont capables d’utiliser l’énergie lumineuse captée par ce pigment pour changer de conformation. Chez les animaux, ce changement conformationnel constitue le point de départ d’une cascade de signalisation intracellulaire impliquant des protéines G (en ce sens les opsines animales sont des récepteurs « métabotropiques »). La phototransduction rétinienne se base par exemple sur les rhodopsines, nom donné à l’association d’une opsine avec un pigment appelé rétinal (l’une des formes de la vitamine A). Un certain nombre d’études, dont certaines remontent à la fin des années 1980, ont montré que l’expression hétérologue de rhodopsines animales est suffisante pour rendre le potentiel transmembranaire de divers types cellulaires sensible à la lumière bleue (450-500 nm) [45, 46]. Ceci peut s’expliquer par le fait que les mécanismes intracellulaires de transduction en aval des rhodopsines sont présents dans de nombreux types cellulaires, où ils sont susceptibles de moduler l’activité de canaux ioniques endogènes. Selon le type cellulaire et la nature de la voie intracellulaire impliquée, l’effet de la lumière peut être dépolarisant ou hyperpolarisant. Il est par ailleurs possible de coupler artificiellement une rhodopsine à des cascades de signalisation spécifiques en remplaçant ses domaines intracellulaires par ceux d’autres récepteurs métabotropiques [47]. Des expériences menées au cours des dix dernières années ont montré que l’illumination de neurones exprimant différentes rhodopsines animales peut induire une modulation de leur taux de décharge [48–50]. Cependant, l’effet de la lumière est le plus souvent modeste et se manifeste avec une latence allant de quelques dixièmes à quelques dizaines de secondes, c’est-à-dire bien au-delà de l’échelle de temps du potentiel d’action (la milliseconde). L’intérêt principal des rhodopsines animales en tant qu’outil optogénétique est donc en premier lieu de pouvoir manipuler des voies de signalisation intracellulaires spécifiques au sein de types cellulaires identifiés (Figure 3B) [51, 52]. La révolution des opsines microbiennes Stimulation de l’activité neuronale à l’aide des channelrhodopsines Le temps de réponse des outils de contrôle de l’activité neuronale basés sur les rhodopsines animales est fortement limité par la mise en jeu de voies de signalisation intracellulaire complexes. C’est pourquoi certains laboratoires ont dirigé leurs efforts vers la recherche d’outils plus simples et donc potentiellement plus rapides, regroupant en une même protéine les fonctions de photorécepteur et d’effecteur. De telles protéines existent à l’état naturel au sein de la famille des opsines microbiennes. Isolées au début des années 2000 chez des algues vertes unicellulaires flagellées du genre Chlamydomonas, les channelrhodopsines sont des rhodopsines présentant les propriétés d’un canal cationique contrôlé par la lumière [ 53, 54]. Leur rôle physiologique est de générer des courants ioniques permettant d’adapter la phototaxie de ces algues photosynthétiques aux conditions lumineuses. Exprimées dans des neurones de mammifère, les channelrhodopsines produisent des courants dépolarisants en présence de lumière bleue. Certaines d’entre elles, en particulier la channelrhodopsine-2 de Chlamydomonas reinhardtii (ChR2), s’expriment en quantités suffisantes et produisent des photocourants suffisamment grands pour déclencher des potentiels d’action en réponse à des impulsions lumineuses, avec une latence proche de la milliseconde (Figure 3C). Depuis leur première utilisation en 2005 [ 49, 55], ces protéines sont devenues le centre d’intérêt de toute la communauté des neurosciences et ont fait l’objet d’un effort important d’optimisation, portant sur un certain nombre de propriétés biophysiques clés (conductance, sélectivité ionique, désactivation, sensibilité spectrale et désensibilisation). Plus d’une cinquantaine de variants de channelrhodopsines ont ainsi été créés en laboratoire à partir de seulement sept variants naturels. Les mutants ChETA, par exemple, possèdent une cinétique rapide d’activation et de désactivation qui permet d’imposer des régimes de décharge à haute fréquence [ 56]. À l’inverse, les mutants SFO ( step function opsins) présentent des temps d’ouverture pouvant atteindre la demi-heure. Ces mutants produisent des dépolarisations de faible amplitude, mais permettent de générer de l’activité persistante en augmentant durablement l’excitabilité de populations de neurones [ 57]. Enfin, mentionnons l’existence de channelrhodopsines telles que les protéines ReaChR et Chrimson [ 58, 59], aux propriétés spectrales décalées vers le rouge, permettant l’utilisation de longueurs d’ondes suffisamment pénétrantes pour la photostimulation transcrânienne et transcutanée de structures nerveuses de surface. Rhodopsines microbiennes utilisées pour l’inhibition de l’activité neuronale Peu de temps après la caractérisation des channelrhodopsines, une partie des efforts s’est portée vers la recherche d’outils similaires permettant non pas de stimuler l’activité neuronale par la lumière, mais de l’inhiber. Il est apparu que certaines catégories de rhodopsines microbiennes décrites depuis plusieurs décennies pouvaient remplir ce rôle. Ces protéines sont des pompes qui utilisent l’énergie lumineuse dans la gamme vert-jaune (550-590 nm) pour transloquer des ions de part et d’autre de la membrane cellulaire dans le sens d’une hyperpolarisation de la membrane (entrée d’anions dans la cellule ou sortie de cations). L’halorhodopsine (NpHR), isolée chez l’archée extrêmophile Natronomonas pharaonis, génère un flux entrant d’ions chlorure (Cl -) tandis que bactériorhodopsines (BR) et archéorhodopsines (Arch et ArchT), isolées chez des bactéries et des archées, génèrent un flux sortant de protons (H +). Contrairement aux channelrhodopsines, ces pompes s’expriment mal à l’état natif dans des cellules de mammifère. C’est pourquoi un certain nombre de modifications telles que l’ajout de séquences d’adressage intracellulaire ont été nécessaires avant d’obtenir des outils réellement fonctionnels [ 60]. Exprimées dans des neurones de mammifère, les versions les plus récentes de ces protéines, comme eNpHR3.0 et eArchT3.0, induisent une hyperpolarisation rapide de la membrane cytoplasmique en réponse à la lumière (Figure 3C). Cette hyperpolarisation est suffisamment robuste pour stopper la décharge de nombreux types neuronaux, y compris in vivo. Récemment, deux types d’outils sont venus enrichir la palette des rhodopsines microbiennes utilisées pour l’inhibition de l’activité neuronale. Le premier, baptisé Jaws, est une pompe de la famille des cruxhalorhodopsines isolée chez une archée halophile et présentant une forte homologie avec les halorhodopsines. Son spectre décalé vers le rouge permet d’inhiber efficacement l’activité neuronale in vivo jusqu’à 3 mm de profondeur, par simple illumination transcrânienne [61]. Le deuxième correspond à une nouvelle famille de channelrhodopsines converties en canaux sélectifs pour l’ion chlorure par mutagenèse dirigée [62, 63]. Ces channelrhodopsines inhibitrices, en particulier ChloC et iC1C2, permettent d’interrompre la décharge neuronale avec une précision temporelle supérieure à celle des pompes activées par la lumière citées plus haut (NpHR, Arch ou Jaws). Fait inattendu, l’archéorhodopsine Arch, utilisée initialement comme outil d’inhibition de l’activité neuronale, présente une fluorescence intrinsèque qui varie fortement en fonction de la différence de potentiel transmembranaire, ce qui en fait également un outil potentiellement intéressant pour l’imagerie optogénétique de l’activité neuronale. La protéine Arch native est bien sûr totalement inadaptée à la mesure du potentiel transmembranaire puisqu’elle génère un photocourant hyperpolarisant. Dans la pratique, seuls des mutants de Arch dont l’activité de pompe électrogénique a été supprimée peuvent être utilisés dans ce but. Certains de ces mutants (en particulier les mutants QuasAr) parviennent à détecter des potentiels d’action uniques, rivalisant ainsi avec les meilleurs indicateurs fluorescents sensibles à la différence de potentiel transmembranaire [64–66]. Les opsines microbiennes font maintenant partie de l’outillage standard de plusieurs centaines de laboratoires de neurobiologie à travers le monde. Elles sont utilisées à tous les niveaux d’étude dans des modèles animaux allant du nématode jusqu’au primate, en passant par la mouche et la souris. Jamais nouvel outil n’a été adopté de manière aussi fulgurante et massive par la communauté des neurosciences. Il est intéressant de noter que c’est dans ce contexte de relative frénésie que le terme « optogénétique » a été proposé par l’un des pionniers dans le domaine [3], ce qui explique pourquoi il est souvent utilisé de manière abusivement restrictive pour ne décrire que les stratégies de contrôle de l’activité neuronale par la lumière. |
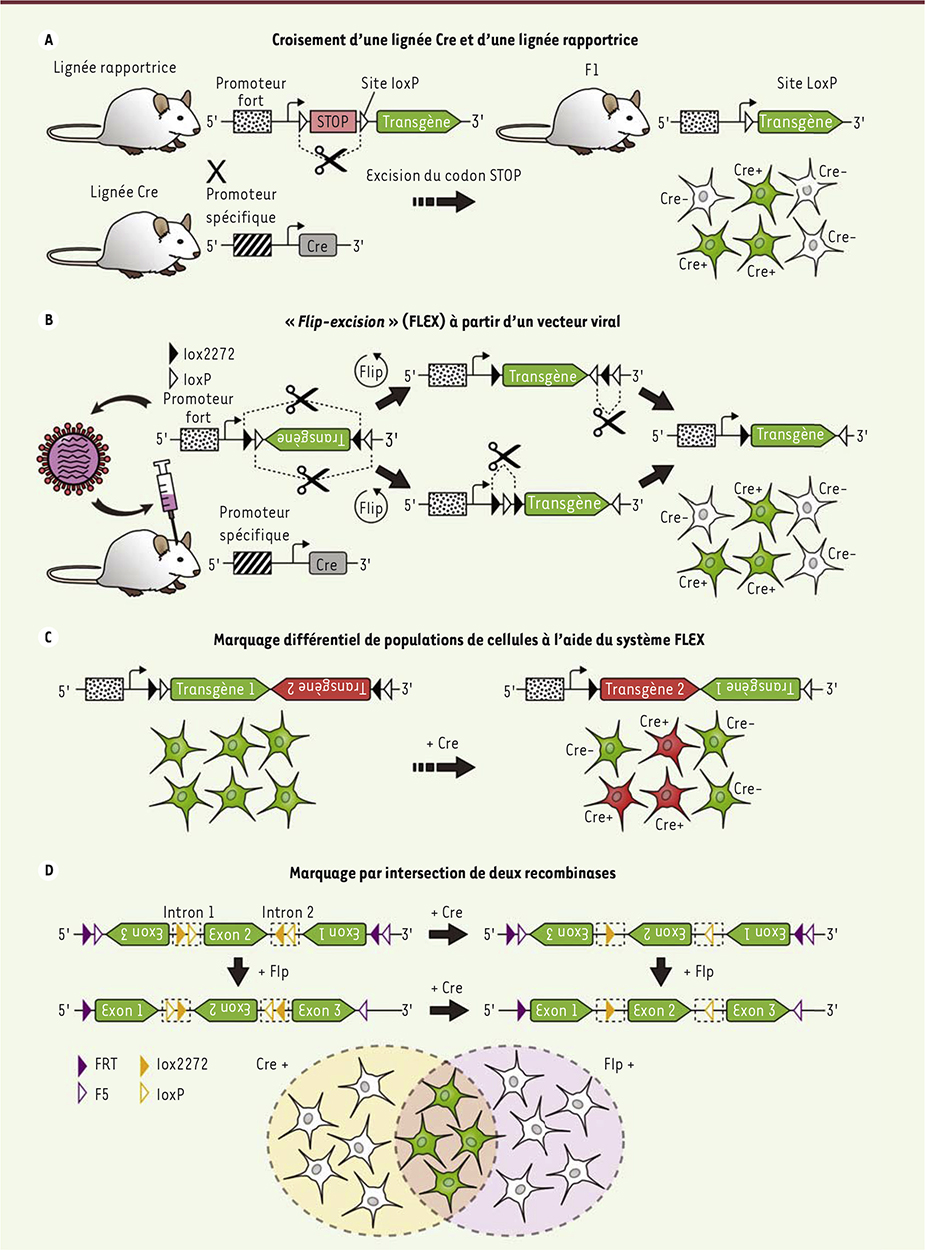
Anciennes techniques, nouveaux défis L’optogénétique ne s’est pas constituée ex nihilo, mais a émergé de la convergence de disciplines existantes. Les deux piliers de l’optogénétique sont les techniques de génie génétique et les techniques d’optique et de photonique. En créant de nouveaux besoins méthodologiques, la rencontre de ces deux disciplines a eu pour effet d’orienter l’innovation dans chacune d’entre elles. Évolution des techniques de transgenèse Bien que les techniques de ciblage génétique soient antérieures à l’optogénétique, l’utilisation de sondes codées génétiquement, et plus encore d’opsines microbiennes, a pesé sur leur évolution récente. On distingue généralement deux types de transgenèse : la transgenèse de la lignée germinale, qui produit des individus dont les modifications génétiques se transmettent de génération en génération (on parle alors de lignée transgénique), et la transgenèse somatique, qui vise à modifier le génome de cellules non reproductrices chez un individu unique. L’optogénétique a eu pour effet de généraliser l’utilisation de la transgenèse somatique par voie virale dans les laboratoires de neurobiologie. Cette méthode présente au moins quatre avantages décisifs. Les vecteurs viraux peuvent être modifiés rapidement pour incorporer de nouvelles versions des outils optogénétiques utilisés (la production d’une solution virale injectable à partir d’un nouveau transgène ne prend que quelques semaines, alors que le travail pour la fabrication d’une nouvelle lignée de souris transgéniques s’échelonne sur plusieurs mois). L’injection intracrânienne de vecteurs viraux permet de transduire les neurones de nombreuses espèces de mammifères, alors que la souris et, dans une moindre mesure, le rat restent les principaux modèles mammifères pour la transgenèse de la lignée germinale. Les approches virales assurent généralement des niveaux d’expression élevés, ce qui permet de contrebalancer le fait que les opsines microbiennes produisent des photocourants unitaires de petite taille. Enfin, les vecteurs viraux facilitent les études de traçage anatomo-fonctionnel en offrant la possibilité de transduire des populations localisées de neurones, voire (pour certains d’entre eux) en permettant des marquages rétrogrades et/ou trans-synaptiques [ 67, 68]. Quelle que soit la technique de transgenèse employée, la clé des stratégies d’expression réside dans l’utilisation de séquences d’ADN régulatrices appelées promoteurs. Les promoteurs sont sélectionnés soit pour leur pouvoir transcriptionnel, soit pour leur spécificité pour un type cellulaire particulier. De manière générale, il est rare que fort pouvoir transcriptionnel et forte spécificité aillent de pair. C’est pourquoi, pour obtenir une expression à la fois forte et spécifique, on fait souvent appel à des systèmes d’expression à deux composants tels que le système Cre-Lox (Figure 4), dans lequel l’expression d’un gène sous le contrôle d’un promoteur à forte activité transcriptionnelle est conditionnée par la présence d’une enzyme (une recombinase appelée Cre) exprimée sous le contrôle d’un promoteur spécifique. Dans la version classique du système Cre-Lox, l’expression du gène d’intérêt est activée dans les cellules exprimant Cre suite à l’excision d’un codon-stop par recombinaison. Alors que cette version donne des résultats satisfaisants dans le cas des lignées transgéniques (Figure 4A), elle s’est révélée insuffisante une fois transposée aux vecteurs viraux. Pour y remédier, une version alternative du système Cre-Lox a été développée : baptisée « FLEX switch » [69] et également désignée par l’appellation DIO (double-floxed inverted open reading frame), cette nouvelle version se décline aujourd’hui en plusieurs variantes (Figure 4B–D). L’évolution la plus récente de cette technique, baptisée INTRSECT (intronic recombinase sites enabling combinatorial targeting), permet de n’exprimer une protéine d’intérêt que dans les cellules coexprimant deux recombinases (Cre et Flp ;
Figure 4D
). Dans le futur, cette approche va permettre de cibler des populations neuronales définies par la coexpression de deux gènes, ce qui augmentera considérablement la finesse des stratégies d’imagerie et de contrôle optogénétique de l’activité neuronale.
 | Figure 4.
Stratégies d’expression conditionnelle utilisant le système Cre-lox. A. Une version classique du système Cre-lox consiste à croiser une souris exprimant Cre dans une population spécifique de cellules (lignée Cre) avec une souris dite « rapportrice », dans laquelle l’expression d’un transgène codant une protéine d’intérêt est sous le contrôle d’un promoteur à forte activité transcriptionnelle. En amont de la séquence codante se trouve un codon STOP flanqué de sites loxP (« floxé ») qui empêche la transcription du transgène. Dans les souris issues de ce croisement (la génération F1), le codon STOP est excisé uniquement dans les cellules exprimant Cre, ce qui permet la transcription du transgène. B. Cette version du système Cre-lox s’est révélée en partie inefficace lorsque le transgène d’intérêt est délivré par voie virale, ce qui a conduit à l’utilisation d’une de ses variantes récentes, le système « FLEX switch ». Dans ce système, la séquence codante du transgène est inversée, ce qui conduit à l’expression d’une protéine antisens non fonctionnelle en l’absence de Cre. La clé du système réside dans l’utilisation de deux paires de sites lox (loxP et lox2272) mutuellement incompatibles. En présence de Cre, le transgène est excisé par coupure au niveau de l’une de ces paires de sites lox. Il peut alors s’inverser avant de se recoller, retrouvant son orientation normale. Les sites lox se trouvent alors dans une configuration favorable à l’excision de l’un d’entre eux, ce qui rend la transformation irréversible. C. La même stratégie peut être utilisée pour obtenir l’expression non chevauchante de deux transgènes dans deux populations de cellules différentes (l’une des deux exprimant Cre). Il suffit pour cela d’insérer les deux transgènes dans des orientations opposées. D. Le système INTRSECT utilise simultanément deux versions de la stratégie FLEX switch, l’une fonctionnant avec la recombinase Cre, l’autre avec la recombinase Flp. Seule l’intervention des deux recombinases permet d’obtenir un transgène fonctionnel (dans lequel tous les exons sont dans la bonne orientation), ce qui permet de marquer sélectivement les cellules qui coexpriment Cre et Flp. |
Évolution des techniques de photonique Dans le domaine de l’imagerie optogénétique de l’activité neuronale, la possibilité de suivre l’activité de circuits neuronaux non plus sur quelques heures, mais sur plusieurs jours voire semaines, a poussé certains laboratoires à perfectionner leurs techniques d’imagerie optique chronique. Des protocoles rendus auparavant fastidieux par la nécessité d’injecter un indicateur d’activité in situ avant chaque séance de mesure, sont maintenant grandement facilités par l’utilisation d’indicateurs codés génétiquement. Dans le cas de régions cérébrales facilement accessibles, il est ainsi possible de suivre l’activité de neurones individuels chez un animal éveillé en train de réaliser des tâches comportementales, au cours de séances quotidiennes pouvant durer plusieurs dizaines de minutes [ 70]. L’utilisation de sondes codées génétiquement induit également un regain d’intérêt pour les mesures optiques d’activité en profondeur (au-delà du millimètre). Plusieurs études ont ainsi testé des dispositifs optiques permettant de suivre les changements de fluorescence d’une grande partie, voire de la totalité des neurones de petits organismes transparents (nématodes et larves de poisson zèbre), avec des fréquences d’acquisition allant de 1 à 20 Hz [ 19– 22]. Chez les rats et les souris, plusieurs prototypes d’endomicroscopes implantables permettent aujourd’hui d’envisager des mesures profondes en situation comportementale [ 71]. Ce type de mesure peut aussi être réalisé au moyen de simples fibres optiques. Dans ce cas, aucune image n’est restituée, mais l’intensité lumineuse fournit une mesure du niveau global d’activité de populations ou voies de projection neuronales spécifiques [ 72, 73]. Dans le domaine du contrôle optogénétique de l’activité neuronale, les efforts d’innovation se sont concentrés sur la possibilité d’illuminer de manière indépendante plusieurs points dans le cerveau. Les techniques d’imagerie digitale, en particulier l’holographie digitale, ont ainsi ouvert un nouveau champ d’application consistant à projeter des patrons lumineux sur des structures nerveuses de surface [74]. Par cette approche, il est possible de photostimuler des zones de tissu précisément délimitées et de réaliser des études de cartographie fonctionnelle à haute résolution. De nombreuses solutions ont également été imaginées pour la photostimulation de structures profondes. Nombre d’entre elles font appel à des techniques de microfabrication (micro-usinage ou lithographie) afin de créer des réseaux de guides de lumière microscopiques [75]. Ces réseaux miniatures permettent d’illuminer le tissu nerveux en de nombreux points de manière indépendante, et ainsi de générer des patrons spatio-temporels d’activité arbitraires. Une alternative récente aux guides de lumière consiste à implanter directement des sources de lumière dans le cerveau, sous la forme de diodes électroluminescentes microscopiques [76]. Enfin, une pratique maintenant courante consiste à coupler l’illumination du tissu à la mesure électrique d’activité neuronale, à l’aide d’assemblages de guides de lumière et de microélectrodes appelés « opto-électrodes » ou « optrodes ». Ces optrodes permettent d’évaluer directement l’impact du stimulus lumineux sur les neurones dont on souhaite contrôler l’activité, en vue notamment de calibrer les paramètres d’illumination. La puissance lumineuse est par exemple un paramètre qu’il faut ajuster avec soin, afin de réaliser les expériences dans une gamme d’intensités qui ne produisent pas d’effet délétère (décharge artéfactuelle par échauffement du tissu, voire même mort cellulaire) tout en fournissant l’effet désiré. Les optrodes permettent également d’identifier en temps réel les types neuronaux enregistrés in vivo, grâce à l’expression ciblée de la channelrhodopsine (un neurone exprimant la channelrhodopsine peut en effet être identifié par le fait qu’il répond à des impulsions lumineuses avec une latence très courte et relativement constante) [77] |
Évolutions futures de l’optogénétique en neuroscience L’optogénétique n’en est sans nul doute qu’à ses débuts. D’une part, il y a fort à parier que la diversité des génomes microbiens et végétaux recèle encore de nombreux gènes pouvant trouver leur utilité dans des applications optogénétiques, comme en témoignent les résultats récemment obtenus par criblage de plus d’une centaine d’espèces d’algues [59]. D’autre part, les efforts de bio-ingénierie vont se poursuivre pour continuer à améliorer et diversifier les outils optogénétiques existants, et en synthétiser de nouveaux. L’une des grandes évolutions futures de l’optogénétique consistera à combiner plusieurs outils dans une même expérience. La diversité des propriétés spectrales des outils disponibles sera la clé de ces approches « tout optique ». On pourra ainsi envisager de contrôler indépendamment des groupes de neurones distincts à l’aide d’outils sensibles à des longueurs d’onde différentes [59], de combiner activation et inhibition, voire même de combiner le contrôle et la mesure d’activité [65, 78]. Chacune de ces options a déjà démontré sa faisabilité expérimentale. En associant la combinatoire offerte par la diversité des outils optogénétiques à la flexibilité des méthodes d’expression et d’illumination actuelles (Figure 5), on atteint des possibilités de configurations expérimentales tellement vastes que l’imagination de l’expérimentateur sera susceptible de devenir le facteur limitant. Dans le domaine de l’expérimentation in vivo, l’optogénétique trouvera également son essor dans l’émergence d’un nouveau type d’instrumentation de pointe, qui visera à miniaturiser et densifier les interfaces optiques avec le tissu nerveux, et qui s’efforcera de les intégrer aux systèmes existants (injection ou prélèvement d’échantillons liquides, stimulation et mesures électriques). Enfin les techniques de contrôle optogénétique de l’activité neuronale commencent à inspirer de nouvelles pistes pour le traitement de certaines maladies du système nerveux, comme par exemple la dégénérescence rétinienne ou l’épilepsie. Ces premières perspectives thérapeutiques feront l’objet d’une revue dans un prochain numéro de médecine/sciences.
 | Figure 5.
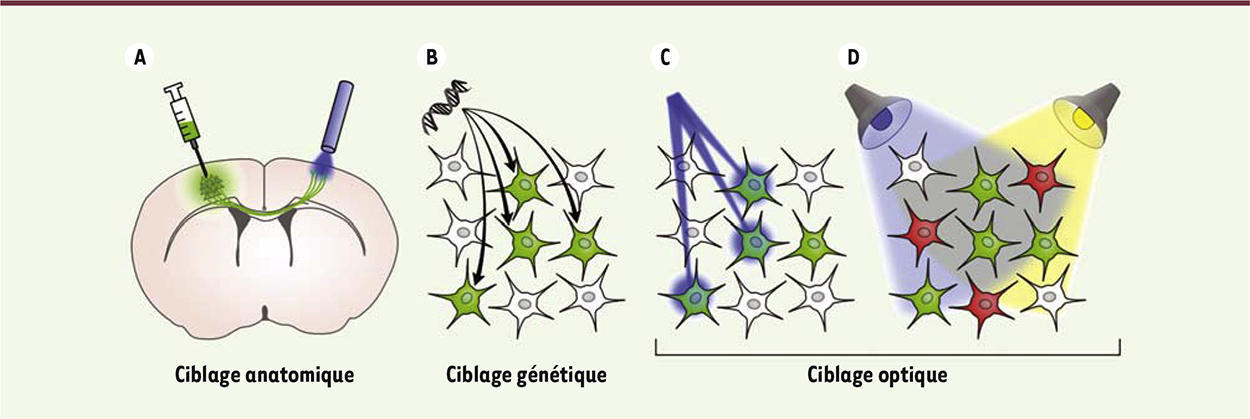
Les différents niveaux de spécificité des approches optogénétiques. A. Sur le plan anatomique, il est relativement aisé d’injecter des vecteurs viraux dans des régions de quelques millièmes de millimètres cubes. L’utilisation de fibres optiques permet également d’illuminer de petites portions de tissu nerveux. L’exemple schématisé ici consiste à marquer un groupe de neurones corticaux et à illuminer leurs projections contralatérales. B. Les techniques de ciblage génétique permettent d’exploiter la diversité des séquences régulatrices pour exprimer une protéine d’intérêt dans des types neuronaux spécifiques. C. L’excitation des outils optogénétiques peut faire appel à des techniques d’illumination ciblée atteignant une résolution micrométrique. D. Enfin des longueurs d’onde différentes peuvent être utilisées pour exciter sélectivement des outils optogénétiques présentant des spectres d’absorption suffisamment distincts. |
|
Outil optogénétique Molécule ou assemblage moléculaire génétiquement adressable (dont une partie au moins est codée par un gène) permettant l’observation ou la manipulation de structures ou fonctions cellulaires spécifiques à l’aide de la lumière.
Indicateur/sonde optogénétique Outil optogénétique (généralement formé d’au moins une protéine fluorescente ou luminescente) dont l’émission de lumière est sensible à des ions, biomolécules ou paramètres physicochimiques particuliers. La sensibilité d’un indicateur est souvent déterminée par le fait qu’il contient un domaine senseur (généralement dépourvu de capacités d’émission de lumière) à l’origine des changements structuraux conduisant au changement d’émission de lumière.
Imagerie optogénétique de l’activité neuronale Utilisation d’indicateurs optogénétiques sensibles à des paramètres cellulaires liés à l’activité neuronale, afin d’observer des changements d’activité de neurones identifiés.
Contrôle optogénétique de l’activité neuronale Contrôle de l’activité de neurones identifiés par l’utilisation d’outils optogénétiques photosensibles capables d’induire (directement ou indirectement) une modification du potentiel transmembranaire, une fois excités par une longueur d’onde appropriée.
Propriétés spectrales d’une molécule Elles sont définies par son spectre d’excitation (gamme de longueurs d’onde capables d’exciter cette molécule) et/ou par son spectre d’émission (gamme de longueurs d’onde pouvant être émises par cette molécule).
|
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Crick FH. Thinking about the brain . Sci Am. 1979; ; 241 : :219.–232. 2.
Crick FH. The impact of molecular biology on neuroscience . Philos Trans R Soc Lond B Biol Sci. 1999; ; 354 : :2021.–2025. 3.
Deisseroth K, Feng G, Majewska AK, et al. Next-generation optical technologies for illuminating genetically targeted brain circuits . J Neurosci. 2006; ; 26 : :10380.–10386. 4.
Dugue GP, Akemann W, Knopfel T. A comprehensive concept of optogenetics . Prog Brain Res. 2012; ; 196 : :1.–28. 5.
Peterka DS, Takahashi H, Yuste R. Imaging voltage in neurons . Neuron. 2011; d : :9.–21. 6.
Paredes RM, Etzler JC, Watts LT, Leichleiter JD. Chemical calcium indicators . Methods. 2008; ; 46 : :143.–151. 7.
Knopfel T. Genetically encoded optical indicators for the analysis of neuronal circuits . Nat Rev Neurosci. 2012; ; 13 : :687.–700. 8.
Perron A, Akemann W, Mutoh H, Knöpfel T. Genetically encoded probes for optical imaging of brain electrical activity . Prog Brain Res. 2012; ; 196 : :63.–77. 9.
Tian L, Hires SA, Looger LL. Imaging neuronal activity with genetically encoded calcium indicators . Cold Spring Harb Protoc. 2012 : :647.–656. 10.
Prasher DC, Eckenrode VK, Ward WW, et al. Primary structure of the Aequorea victoria green-fluorescent protein . Gene. 1992; ; 111 : :229.–233. 11.
Matz MV, Fradkov AF, Labas YA, et al. Fluorescent proteins from nonbioluminescent Anthozoa species . Nat Biotechnol. 1999; ; 17 : :969.–973. 12.
Shaner NC, Patterson GH, Davidson MW. Advances in fluorescent protein technology . J Cell Sci. 2007; ; 120 : :4247.–4260. 13.
Chudakov DM, Matz MV, Lukyanov S, Lukyanov KA. Fluorescent proteins and their applications in imaging living cells and tissues . Physiol Rev. 2010; ; 90 : :1103.–1163. 14.
Miesenbock G, De Angelis DA, Rothman JE. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins . Nature. 1998; ; 394 : :192.–195. 15.
Akerboom J, Rivera JD, Guilbe MM, et al. Crystal structures of the GCaMP calcium sensor reveal the mechanism of fluorescence signal change and aid rational design . J Biol Chem. 2009; ; 284 : :6455.–6464. 16.
Chen TW, Wardill TJ, Sun Y, et al. Ultrasensitive fluorescent proteins for imaging neuronal activity . Nature. 2013; ; 499 : :295.–300. 17.
Akerboom J, Chen TW, Wardill TJ, et al. Optimization of a GCaMP calcium indicator for neural activity imaging . J Neurosci. 2012; ; 32 : :13819.–13840. 18.
Tian L, Hires SA, Mao T, et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators . Nat Methods. 2009; ; 6 : :875.–881. 19.
Schrodel T, Prevedel R, Aumayr K, et al. Brain-wide 3D imaging of neuronal activity in Caenorhabditis elegans with sculpted light . Nat Methods. 2013; ; 10 : :1013.–1020. 20.
Ahrens MB, Orger MB, Robson DN, et al. Whole-brain functional imaging at cellular resolution using light-sheet microscopy . Nat Methods. 2013; ; 10 : :413.–420. 21.
Panier T, Romano SA, Olive R, et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy . Front Neural Circuits. 2013; ; 7 : :65.. 22.
Prevedel R, Yoon YG, Hoffmann M, et al. Simultaneous whole-animal 3D imaging of neuronal activity using light-field microscopy . Nat Methods. 2014; ; 11 : :727.–730. 23.
Cao G, Platisa J, Pieribone VA, et al. Genetically targeted optical electrophysiology in intact neural circuits . Cell. 2013; ; 154 : :904.–913. 24.
St-Pierre F, Marshall JD, Yang Y, et al. High-fidelity optical reporting of neuronal electrical activity with an ultrafast fluorescent voltage sensor . Nat Neurosci. 2014; ; 17 : :884.–889. 25.
Akemann W, Mutoh H, Perron A, et al. Imaging neural circuit dynamics with a voltage-sensitive fluorescent protein . J Neurophysiol. 2012; ; 108 : :2323.–2337. 26.
Horikawa K, Yamada Y, Matsuda T, et al. Spontaneous network activity visualized by ultrasensitive Ca2+ indicators, yellow Cameleon-Nano . Nat Methods. 2010; ; 7 : :729.–732. 27.
Thestrup T, Litzlbauer J, Bartholomäus I, et al. Optimized ratiometric calcium sensors for functional in vivo imaging of neurons and T lymphocytes . Nat Methods. 2014; ; 11 : :175.–182. 28.
Wilson T, Hastings JW. Bioluminescence . Annu Rev Cell Dev Biol. 1998; ; 14 : :197.–230. 29.
Ridgway EB, Ashley CC. Calcium transients in single muscle fibers . Biochem Biophys Res Commun. 1967; ; 29 : :229.–234. 30.
Llinas R, Blinks JR, Nicholson C. Calcium transient in presynaptic terminal of squid giant synapse: detection with aequorin . Science. 1972; ; 176 : :1127.–1129. 31.
Prasher D, McCann RO, Cormier MJ. Cloning and expression of the cDNA coding for aequorin, a bioluminescent calcium-binding protein . Biochem Biophys Res Commun. 1985; ; 126 : :1259.–1268. 32.
Knight H, Knight MR. Recombinant aequorin methods for intracellular calcium measurement in plants . Methods Cell Biol. 1995; ; 49 : :201.–216. 33.
Curie T, Rogers KL, Colasante C, Brûlet P. Red-shifted aequorin-based bioluminescent reporters for in vivo imaging of Ca2+ signaling . Mol Imaging. 2007; ; 6 : :30.–42. 34.
Drobac E, Tricoire L, Chaffotte AF, et al. Calcium imaging in single neurons from brain slices using bioluminescent reporters . J Neurosci Res. 2010; ; 88 : :695.–711. 35.
Pichler A, Prior JL, Piwnica-Worms D. Imaging reversal of multidrug resistance in living mice with bioluminescence: MDR1 P-glycoprotein transports coelenterazine . Proc Natl Acad Sci USA. 2004; ; 101 : :1702.–1707. 36.
Tricoire L, Lambolez B. Neuronal network imaging in acute slices using Ca2+ sensitive bioluminescent reporter . Methods Mol Biol. 2014; ; 1098 : :33.–45. 37.
Naumann EA, Kampff AR, Prober DA, et al. Monitoring neural activity with bioluminescence during natural behavior . Nat Neurosci. 2010; ; 13 : :513.–520. 38.
Martin JR, Rogers KL, Chagneau C, Brûlet P. In vivo bioluminescence imaging of Ca signalling in the brain of Drosophila . PLoS One. 2007; ; 2 : :e275.. 39.
Rogers KL, Picaud S, Roncali E, et al. Non-invasive in vivo imaging of calcium signaling in mice . PLoS One. 2007; ; 2 : :e974.. 40.
Szobota S, McKenzie C, Janovjak H. Optical control of ligand-gated ion channels . Methods Mol Biol. 2013; ; 998 : :417.–435. 41.
Lima SQ, Miesenbock G. Remote control of behavior through genetically targeted photostimulation of neurons . Cell. 2005; ; 121 : :141.–152. 42.
Janovjak H, Szobota S, Wyart C, et al. A light-gated, potassium-selective glutamate receptor for the optical inhibition of neuronal firing . Nat Neurosci. 2010; ; 13 : :1027.–1032. 43.
Kramer RH, Mourot A, Adesnik H. Optogenetic pharmacology for control of native neuronal signaling proteins . Nat Neurosci. 2013; ; 16 : :816.–823. 44.
Porter ML, Blasic JR, Bok MJ, et al. Shedding new light on opsin evolution . Proc Biol Sci. 2012; ; 279 : :3.–14. 45.
Khorana HG, Knox BE, Nasi E, et al. Expression of a bovine rhodopsin gene in Xenopus oocytes: demonstration of light-dependent ionic currents . Proc Natl Acad Sci USA. 1988; ; 85 : :7917.–7921. 46.
Melyan Z, Curran J, Hofmann K, et al. Addition of human melanopsin renders mammalian cells photoresponsive . Nature. 2005; ; 433 : :741.–745. 47.
Airan RD, Thompson KR, Fenno LE, et al. Temporally precise in vivo control of intracellular signalling . Nature. 2009; ; 458 : :1025.–1029. 48.
Zemelman BV, Lee GA, Ng M, Miesenböck G. Selective photostimulation of genetically chARGed neurons . Neuron. 2002; ; 33 : :15.–22. 49.
Li X, Gutierrez DV, Hanson MG, et al. Fast noninvasive activation and inhibition of neural and network activity by vertebrate rhodopsin and green algae channelrhodopsin . Proc Natl Acad Sci USA. 2005; ; 102 : :17816.–17821. 50.
Gutierrez DV, Mark MD, Masseck O, et al. Optogenetic control of motor coordination by Gi/o protein-coupled vertebrate rhodopsin in cerebellar Purkinje cells . J Biol Chem. 2011; ; 286 : :25848.–25858. 51.
Masseck OA, Rubelowski JM, Spoida K, Herlitze S. Light- and drug-activated G-protein-coupled receptors to control intracellular signalling . Exp Physiol. 2011; ; 96 : :51.–56. 52.
Koyanagi M, Terakita A. Diversity of animal opsin-based pigments and their optogenetic potential . Biochim Biophys Acta. 2014; ; 1837 : :710.–716. 53.
Nagel G, Ollig D, Fuhrmann M, et al. Channelrhodopsin-1: a light-gated proton channel in green algae . Science. 2002; ; 296 : :2395.–2398. 54.
Nagel G, Szellas T, Huhn W, et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel . Proc Natl Acad Sci USA. 2003; ; 100 : :13940.–13945. 55.
Boyden ES, Zhang F, Bamberg E, et al. Millisecond-timescale, genetically targeted optical control of neural activity . Nat Neurosci. 2005; ; 8 : :1263.–1268. 56.
Gunaydin LA, Yizhar O, Berndt A, et al. Ultrafast optogenetic control . Nat Neurosci. 2010; ; 13 : :387.–392. 57.
Yizhar O, Fenno LE, Prigge M, et al. Neocortical excitation/inhibition balance in information processing and social dysfunction . Nature. 2011; ; 477 : :171.–178. 58.
Lin JY, Knutsen PM, Muller A, et al. ReaChR: a red-shifted variant of channelrhodopsin enables deep transcranial optogenetic excitation . Nat Neurosci. 2013; ; 16 : :1499.–1508. 59.
Klapoetke NC, Murata Y, Kim SS, et al. Independent optical excitation of distinct neural populations . Nat Methods. 2014; ; 11 : :338.–346. 60.
Gradinaru V, Zhang F, Ramakrishnan C, et al. Molecular and cellular approaches for diversifying and extending optogenetics . Cell. 2010; ; 141 : :154.–165. 61.
Chuong AS, Miri ML, Busskamp V, et al. Noninvasive optical inhibition with a red-shifted microbial rhodopsin . Nat Neurosci. 2014; ; 17 : :1123.–1129. 62.
Wietek J, Wiegert JS, Adeishvili N, et al. Conversion of channelrhodopsin into a light-gated chloride channel . Science. 2014; ; 344 : :409.–412. 63.
Berndt A, Lee SY, Ramakrishnan C, Deisseroth K. Structure-guided transformation of channelrhodopsin into a light-activated chloride channel . Science. 2014;; 344 : :420.–424. 64.
Gong Y, Li JZ, Schnitzer MJ. Enhanced archaerhodopsin fluorescent protein voltage indicators . PLoS One. 2013; ; 8 : :e66959.. 65.
Hochbaum DR, Zhao Y, Farhi SL, et al. All-optical electrophysiology in mammalian neurons using engineered microbial rhodopsins . Nat Methods. 2014; ; 11 : :825.–833. 66.
Zou P, Zhao Y, Douglass AD, et al. Bright and fast multicoloured voltage reporters via electrochromic FRET . Nat Commun. 2014; ; 5 : :4625.. 67.
Lo L, Anderson DJ. A Cre-dependent, anterograde transsynaptic viral tracer for mapping output pathways of genetically marked neurons . Neuron. 2011; ; 72 : :938.–950. 68.
Osakada F, Mori T, Cetin AH, et al. New rabies virus variants for monitoring and manipulating activity and gene expression in defined neural circuits . Neuron. 2011; ; 71 : :617.–631. 69.
Atasoy D, Aponte Y, Su HH, Sternson SM. A FLEX switch targets Channelrhodopsin-2 to multiple cell types for imaging and long-range circuit mapping . J Neurosci. 2008; ; 28 : :7025.–7030. 70.
Petreanu L, Gutnisky DA, Huber D, et al. Activity in motor-sensory projections reveals distributed coding in somatosensation . Nature. 2012; ; 489 : :299.–303. 71.
Flusberg BA, Cocker ED, Piyawattanametha W, et al. Fiber-optic fluorescence imaging . Nat Methods. 2005; ; 2 : :941.–950. 72.
Cui G, Jun SB, Jin X, et al. Concurrent activation of striatal direct and indirect pathways during action initiation . Nature. 2013; ; 494 : :238.–242. 73.
Gunaydin LA, Grosenick L, Finkelstein JC, et al. Natural neural projection dynamics underlying social behavior . Cell. 2014; ; 157 : :1535.–1551. 74.
Vaziri A, Emiliani V. Reshaping the optical dimension in optogenetics . Curr Opin Neurobiol. 2012; ; 22 : :128.–137. 75.
Zorzos AN, Scholvin J, Boyden ES, Fonstad CG. Three-dimensional multiwaveguide probe array for light delivery to distributed brain circuits . Opt Lett. 2012; ; 37 : :4841.–4843. 76.
Kim TI, McCall JG, Jung YH, et al. Injectable, cellular-scale optoelectronics with applications for wireless optogenetics . Science. 2013; ; 340 : :211.–216. 77.
Lima SQ, Hromádka T, Znamenskiy P, Zador AM. PINP: a new method of tagging neuronal populations for identification during in vivo electrophysiological recording . PLoS One. 2009; ; 4 : :e6099.. 78.
Packer AM, Russell LE, Dalgleish HW, Häusser M. Simultaneous all-optical manipulation and recording of neural circuit activity with cellular resolution in vivo . Nat Methods. 2014 ; doi : 10.1038/nmeth.3217 |