| |
| Med Sci (Paris). 2015 April; 31(4): 377–382. Published online 2015 May 8. doi: 10.1051/medsci/20153104011.Les IFITM, un obstacle commun à de nombreux virus
Kevin Tartour1,2,3,4,5* and Andrea Cimarelli1,2,3,4,5 1CIRI (centre international de recherche en infectiologie), 69364Lyon, France 2Inserm U1111, 46, allée d’Italie, 69364Lyon, France 3École normale supérieure de Lyon, 46, allée d’Italie, 69364Lyon, France 4CNRS, UMR5308, 46, allée d’Italie, 69364Lyon, France 5Université de Lyon, Lyon I, UMS3444/US8 BioSciences Gerland, 69364Lyon, France |
Depuis la découverte du VIH (virus de l’immunodéficience humaine) en 1983, la compréhension de la réplication de ce virus et de ses interactions avec la cellule hôte n’a cessé de progresser. Si le VIH a besoin de la cellule pour se multiplier, en revanche, l’hôte est agressé et il a, au cours de l’évolution, mis en place différents mécanismes pour contrecarrer les infections, quel que soit le pathogène en cause (virus, bactérie ou parasite). Ainsi, dans le cas des virus, certaines protéines cellulaires sont nécessaires au virus (facteurs de dépendance), alors que d’autres en restreignent la réplication (facteurs de restriction). Le premier facteur de restriction de la réplication du VIH décrit fut l’apolipoprotein B mRNA editing enzyme, catalytic polypeptide-like 3G (APOBEC3G) [1]. Par la suite, Trim5 (tripartite motif 5a protein), la tétherine/Bst2 (bone marrow stromal antigen 2), SAMHD1 (SAM domain and HD domain-containing protein 1) et Mx2 ont été identifiés comme autant de facteurs pouvant inhiber le VIH [2–9, 41, 42]. Néanmoins, ces quelques facteurs de restriction ne permettent pas, à eux seuls, d’expliquer l’ensemble des phénotypes, permissif ou restrictif, observés in cellulo. Il est évident que d’autres facteurs participent à la restriction de la propagation du VIH dans certaines conditions, et de nombreuses études ont identifié de tels facteurs sans pour autant qu’ils atteignent l’efficacité des protéines précédemment citées. Ce domaine de recherche est très actif, et outre le VIH, concerne aussi le virus de la grippe, ainsi que les virus émergents. C’est lors d’un vaste crible d’ARNi (ARN interférents) qu’en 2009, l’équipe du Dr Elledge a identifié une nouvelle famille de facteurs de restriction, les IFITM (interferon induced transmembrane proteins), actifs sur le virus de la grippe, mais aussi celui de la dengue, du Nile occidental et de la fièvre jaune [10]. Par la suite, plusieurs équipes ont cherché à déterminer le spectre d’activité antivirale de ces protéines, et il est apparu qu’un très grand nombre de virus étaient inhibés par les IFITM (Tableau I) [10–19]. Ainsi, les IFITM sont capables d’inhiber plusieurs virus à ARN, indépendamment de la polarité de leur génome1,. De même, ils agissent sur des particules virales enveloppées ou non2, indépendamment du mécanisme utilisé par le virus pour entrer dans la cellule cible. Pour accéder à la cellule, les virus peuvent fusionner directement avec la membrane plasmique, ou bien ils sont internalisés dans des vésicules ou endosomes. Dans ce dernier cas, le faible H des endosomes entraîne alors un changement de conformation de l’enveloppe, qui est nécessaire à la fusion entre les membranes cellulaire et virale (l’entrée de ces virus est dans ce cas définie comme étant dépendante du pH).
Tableau I.
| Famille |
Virus |
Génome (polarité) |
Dépendance au pH |
Enveloppe |
Références |
|
Restreints
|
|
|
|
|
|
|
| Orthomyxoviridae |
Grippe A/B |
ARN (-) |
**
|
Oui |
[10] |
|
| Flaviviridae |
Nile occidental
Dengue
Hépatite C |
ARN (+) |
*
**
*
|
Oui |
[10] |
|
| Rhabdoviridae |
VSV
Rage |
ARN (-) |
*
**
|
Oui |
[12, 19] |
|
| Filoviridae |
Ébola, Marburg |
ARN (-) |
**
|
Oui |
[16] |
|
| Coronaviridae |
SARS |
ARN (+) |
**
|
Oui |
[16] |
|
| Retroviridae |
VIH-1
JSRV |
ARN (+) rétrotranscrit |
Non
*
|
Oui |
[11, 34] |
|
| Alphaviridae |
Virus de la forêt de Semliki |
ARN (+) |
*
|
Oui |
[34] |
|
| Bunyaviridae |
La Crosse, Hantaan, Andes, fièvre de la vallée du rift |
ARN (-) |
**
|
Oui |
[15] |
|
| Reoviridae |
Réovirus |
ARN double brin |
**
|
Non |
[13] |
|
|
Non restreints
|
|
|
|
|
|
|
| Retroviridae |
MLV |
ARN (+) rétrotranscrit |
Non |
Oui |
[10] |
|
| Arenavirus |
Lassa, Machupo, chorioméningite lymphocytaire |
ARN (-) |
*
|
Oui |
[10] |
|
| Bunyaviridae |
Fièvre hémorragique de Crimea-Congo |
ARN (-) |
**
|
Oui |
[15] |
|
| Papillomavirdae |
HPV16 |
ADN double brin |
**
|
Non |
[17] |
|
| Herpesviridae |
HCMV |
ADN double brin |
Dépendant du type cellulaire |
Oui |
[17] |
|
| Adenoviridae |
Adénovirus 5 |
ADN double brin |
**
|
Non |
[17] |
|
|
Favorisés
|
|
|
|
|
|
|
| Coronaviridae |
OC43 |
ARN (+) |
**
|
Oui |
[18] |
Résumé de l’activité antivirale des IFITM. HCMV : human cytomegalovirus ; HPV16 : virus du papillome humain ; JSRV : Jaagsiekte sheep retrovirus ; MLV : virus de la leucémie murine ; OC43 : coronavirus ; SARS : severe acute respiratory syndrome ; VSV : virus de la stomatite vésiculaire |
|
Les protéines IFITM sont codées chez l’homme par une famille de cinq gènes : IFITM1, IFITM2, IFITM3, IFITM5 et IFITM10, ainsi qu’un pseudogène IFITM4 ; tous sont situés sur le chromosome 11 [20]. Des études phylogénétiques ont mis en évidence trois gènes IFITM chez le dernier ancêtre commun des vertébrés, indiquant que des évènements de duplication indépendants (intra-espèce) ont contribué à élargir cette famille [20]. C’est ainsi que IFITM1, IFITM2 et IFITM3 ont émergé par duplication chez l’homme. Ces trois gènes IFITM étant apparus tardivement au cours de l’évolution, après la spéciation humaine, les protéines possèdent une très forte homologie de séquence : 91% entre IFITM2 et IFITM3 et 59 % entre IFITM2 et IFITM1, cette dernière étant amputée des 20 premiers acides aminés (Figure 1A). Seules IFITM1, IFITM2 et IFITM3 (que nous désignerons collectivement par IFITM dans la suite du texte) ont une activité antivirale et constituent donc le centre d’intérêt de cette revue. Les protéines IFITM sont présentes dans la plupart des cellules de l’organisme à un niveau basal, mais leur expression peut être fortement induite après stimulation par l’IFN (interféron) grâce à un élément ISRE (interferon stimulated responsive element) présent dans le promoteur des gènes correspondants.
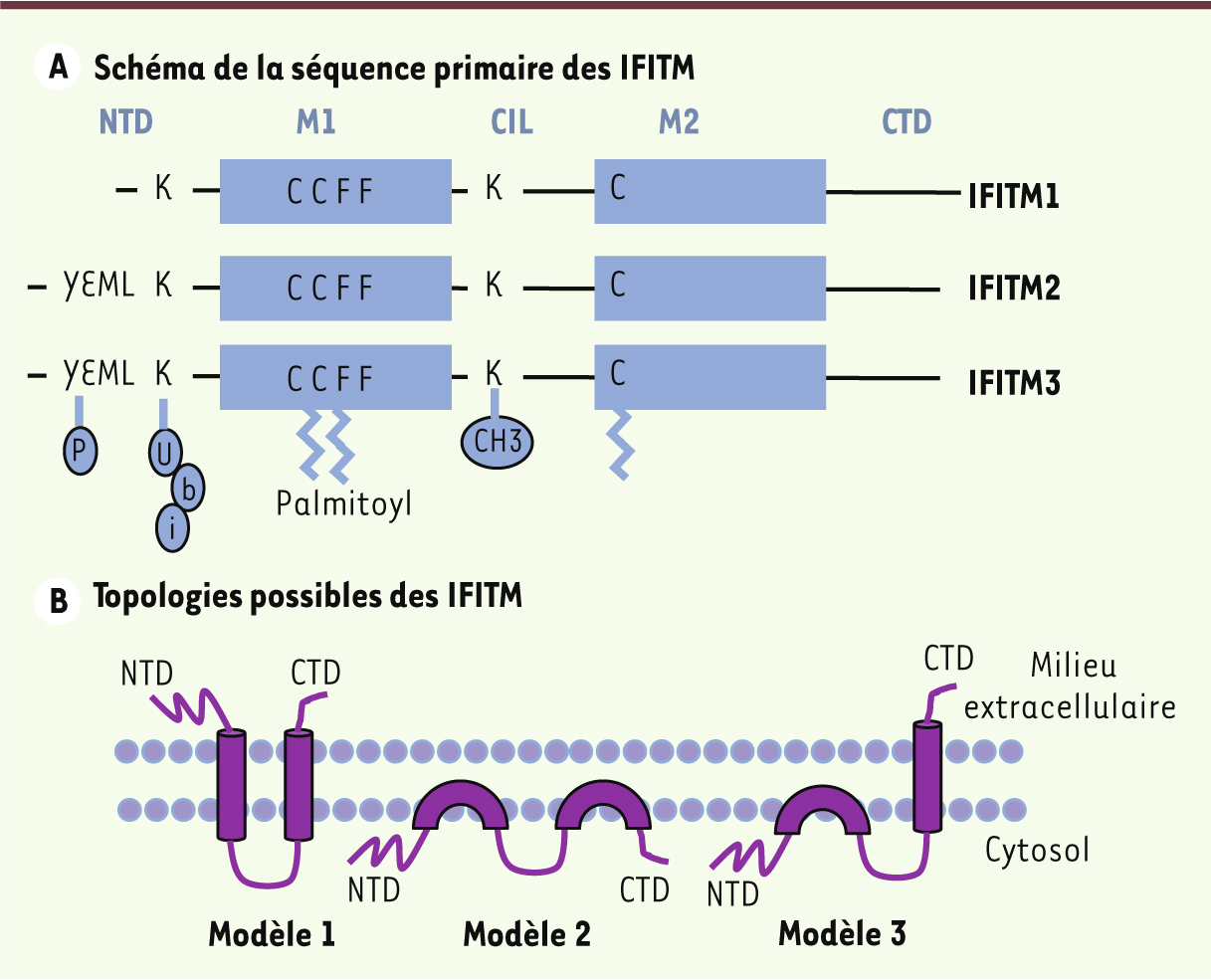
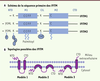 | Figure 1.
Organisation et topologie des IFITM. A. Représentation schématique des IFITM avec les acides aminés importants et leurs modifications. Seule IFITM3 a été étudiée en détails ; les acides aminés conservés sont représentés ; ils pourraient avoir un rôle équivalent dans les différentes IFITM. CIL : boucle cytosolique ; CTD : domaine carboxy-terminal ; NTD : domaine amino-terminal ; M1 : domaine hydrophobe 1 ; M2 : domaine hydrophobe 2. B. Représentation des trois topologies proposées pour les IFITM. Des résultats récents proposent que le modèle 3 est prédominant, pour IFITM1 et IFITM3. |
Le modèle représentant la structure des IFITM n’est pas encore figé. L’analyse de la séquence primaire prédit deux domaines hydrophobes. Initialement, les IFITM étaient représentées avec deux domaines transmembranaires [10], puis avec deux domaines intramembranaires [21]. Des données récentes suggèrent aujourd’hui qu’il existe un équilibre entre trois formes, avec la prédominance d’une topologie de type 3 : un premier domaine intramembranaire et un second transmembranaire exposant le domaine carboxy-terminal à l’extérieur de la cellule (Figure 1B) [22, 23]. Il ne s’agit encore que de données indirectes, et la représentation correcte de la structure péri- ou transmembranaire des IFITM ne sera établie qu’une fois les données structurales obtenues. Comme c’est le cas pour de nombreuses protéines, la séquence peptidique est en effet insuffisante pour correctement décrire les IFITM. Ces protéines peuvent former des oligomères (homo ou hétéro-oligomères), ce qui requiert la présence de deux phénylalanines (F75 et F78 pour IFITM3,
Figure 1
) [24]. De plus, elles subissent différentes modifications posttraductionnelles : phosphorylation de la tyrosine 20 [25], palmitoylation des cystéines 71, 72 et 104 [26], ubiquitination de la lysine 24 [21] et monométhylation de la lysine 88 [27]. Le rôle de toutes ces modifications n’est pas encore bien compris, et ne repose que sur des corrélations : la palmitoylation est importante pour la fonction antivirale [21, 26], tandis que la phosphorylation [25, 28] et l’ubiquitination [21] participent à la localisation cellulaire des IFITM. |
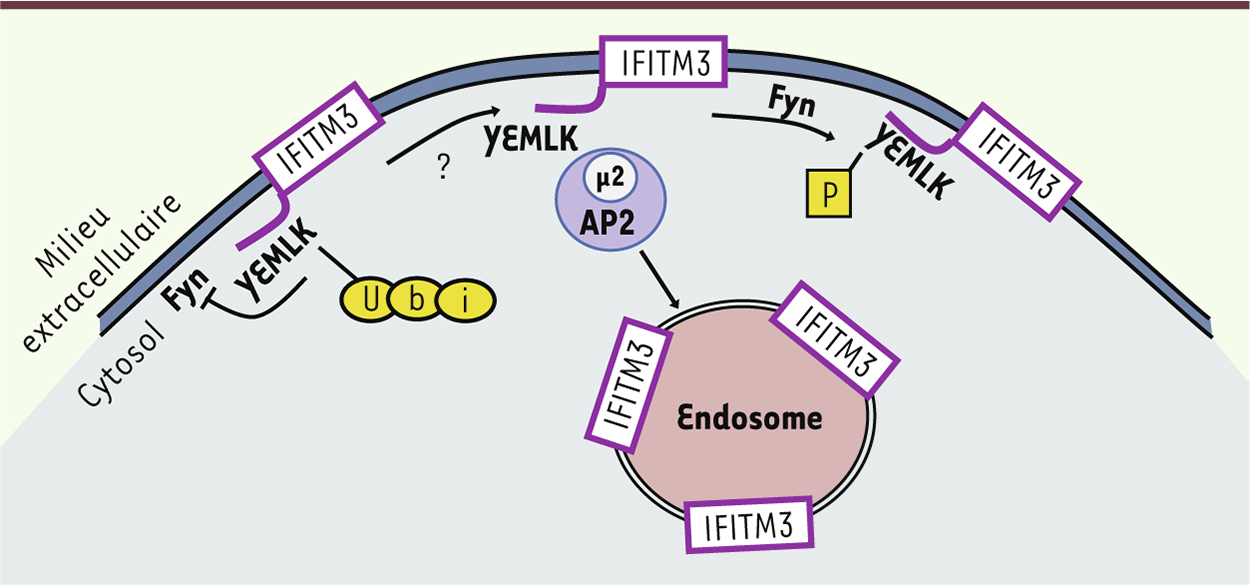
La localisation et le trafic intracellulaire des IFITM En raison de leurs deux domaines hydrophobes conservés, les IFITM sont des protéines essentiellement membranaires. Cependant, certaines différences existent entres elles : IFITM1 est plus enrichie à la membrane plasmique, tandis qu’IFITM2 et IFITM3 prédominent dans des compartiments intracellulaires [11], suggérant une possible diversification du rôle des trois IFITM. Des expériences d’immunofluorescence confocale ont montré qu’IFITM2 et IFITM3 colocalisent avec les endosomes tardifs, voire avec les lysosomes (colocalisation avec LAMP1 [lysosomal-associated membrane protein 1], Rab7 et LTRed [29], et avec CD63 [30]). De plus, en présence des IFITM, ces études de microscopie confocale ont observé un accroissement des compartiments endosomaux, une acidification excessive [29], ainsi que l’accumulation de cholestérol [30] dans ces compartiments. Les IFITM naissent dans le réticulum endoplasmique, puis transitent par l’appareil de Golgi pour atteindre la membrane plasmique. De là, la sous-unité m2 du complexe AP2 (adaptor complex 2) reconnaît le motif YxxF (YEML) présent dans la queue amino-terminale d’IFITM2 et IFITM3 (absente dans IFITM1) [31] (Figure 2). Le recrutement d’AP2 entraîne l’endocytose d’IFITM2 et IFITM3 (Figure 2). Afin de réguler finement la localisation des IFITM, l’enzyme Fyn peut phosphoryler la tyrosine 20 [28], ce qui empêche l’association du complexe AP2 à IFITM. De plus, l’ubiquitination de la lysine 24 semble empêcher la reconnaissance de la tyrosine 20 par Fyn, à moins que ce ne soit l’ubiquitination qui ne puisse se faire qu’en l’absence de phosphorylation ou après endocytose [28]. Des expériences supplémentaires seront nécessaires pour distinguer ces deux hypothèses. Pour l’instant, ces mécanismes ont été décrits pour IFITM3, mais comme les séquences et les localisations intracellulaires des IFITM2 et -3 sont similaires, il est probable qu’ils s’appliquent aussi à IFITM2. En revanche, IFITM1 possède un domaine amino-terminal raccourci, donc dépourvu du motif de signalisation YxxF, ce qui explique sa localisation essentiellement à la membrane plasmique [11]. Ces différences de localisation membranaire expliquent très probablement que les virus qui pénètrent dans la cellule cible en fusionnant directement avec la membrane plasmique ou en transitant dans les endosomes précoces (VIH, VSV [virus de la stomatite vésiculaire]), soient mieux bloqués par IFITM1, tandis que ceux qui entrent dans les endosomes tardifs ou les lysosomes sont préférentiellement inhibés par IFITM2 et IFITM3 (Tableau I) [32].
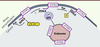 | Figure 2.
Localisation dynamique d’IFITM3. IFITM3 néosynthétisée est ciblée vers la membrane plasmique. Le domaine YEML s’associe à la sous-unité μ2 du complexe AP2. Le recrutement d’AP2 entraîne l’endocytose d’IFITM3 qui est alors localisée majoritairement à la membrane des endosomes. La phosphorylation de la tyrosine masque le motif YEML ; IFITM3 n’est plus internalisée et se trouve alors à la membrane plasmique. Le rôle de l’ubiquitination de la lysine 24 (K) est moins bien compris, mais cette étape pourrait empêcher la phosphorylation de la tyrosine et, donc, permettre la localisation endosomale d’IFITM3. Seule IFITM3 a été étudiée, mais les séquences importantes étant conservées avec IFITM2, ce dernier pourrait avoir la même localisation qu’IFITM3. En revanche, IFITM1, qui ne possède pas le domaine YEML, est localisée essentiellement à la membrane plasmique (non représenté ici). |
|
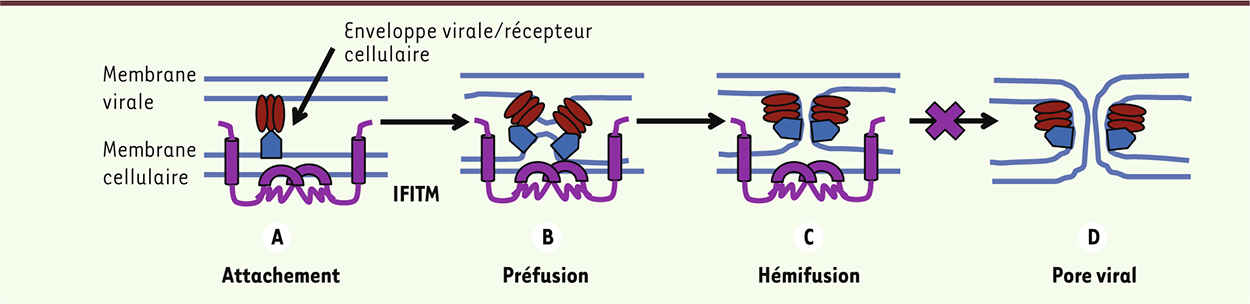
La plupart des facteurs de restriction décrits jusqu’à présent inhibent le virus après son entrée dans la cellule. La caractéristique des IFITM est de bloquer les virus avant même qu’ils n’aient commencé à envahir la première cellule. Pour autant, les récepteurs d’entrée sont toujours présents [10] et les virus peuvent s’attacher normalement à leur cellule cible [12, 29]. C’est lors du processus de fusion que les IFITM inhibent l’entrée du virus [11, 12, 29] ; le contenu des particules virales n’est en effet pas libéré dans le cytosol. Ceci a pu être déterminé grâce à l’utilisation de vecteurs lentiviraux portant la séquence codant l’enzyme b-lactamase fusionnée avec celle codant la protéine accessoire Vpr (viral protein R). Celle-ci est activement recrutée dans les virions et, lors de la fusion entre les membranes cellulaire et virale, l’enzyme est libérée dans le cytosol où elle peut être détectée grâce à un test colorimétrique [33]. Une preuve additionnelle de l’inhibition de la fusion des membranes virale et cellulaire par les IFITM est la diminution - en présence des IFITM - des syncytia formés entre les cellules porteuses de l’enveloppe virale et celles exprimant le (les) récepteur(s) [34]. L’étape de fusion se décompose elle-même en plusieurs processus (Figure 3). Dans un premier temps, l’enveloppe virale et son récepteur subissent des changements de conformation aboutissant à l’hémifusion ; les hémimembranes externes fusionnent, tandis que les hémimembranes internes restent intactes. Vient ensuite l’étape d’ouverture d’un pore viral très étroit qui s’élargit, aboutissant à la fusion complète. Deux études ont cherché à déterminer quelle était l’étape bloquée par les IFITM. La première suggère que le processus d’hémifusion n’a pas lieu ; elle se fonde sur le fait qu’une drogue (chlorpromazine, Cpz), qui favorise le passage de l’état d’hémifusion à l’ouverture du pore, ne lève pas la restriction imposée par IFITM3 [34]. La seconde suggère que l’ouverture du pore est inhibée ; elle utilise des fluorophores différents pour la membrane virale et l’intérieur du virion, et observe la fusion des membranes cellulaire et virale, mais l’absence de libération du contenu des virus dans le cytoplasme cellulaire [35]. Cette seconde étude, moins intrusive, propose que le processus d’hémifusion puisse avoir lieu et suggère que les IFITM bloquent probablement l’ouverture du pore, même en présence de Cpz (Figure 3). Une dernière hypothèse, potentiellement intéressante, n’a pas été explorée de manière exhaustive à ce jour. En effet, les IFITM sont capables d’interagir entre elles, et on peut imaginer que ces multimères puissent former un maillage sous-membranaire qui empêcherait la libération du contenu du virus dans le cytosol, tout en stabilisant le pore.
 | Figure 3.
Modèle de l’action antivirale des IFITM. A. Reconnaissance du récepteur par l’enveloppe virale. B. Déclenchement du processus de fusion. C. Hémifusion : les hémimembranes externes fusionnent et les hémimembranes internes restent intactes. Les IFITM forment un réseau dans l’hémimembrane interne ; celle-ci est rigidifiée et stabilisée, ce qui bloque le passage au stade pore viral. D. Ouverture du pore viral en l’absence des IFITM. |
|
Mécanisme de l’action antivirale des IFITM Le mécanisme bloquant le processus de fusion ne fait pas encore consensus et plusieurs équipes ont avancé différentes hypothèses fondées sur leurs résultats expérimentaux. D’une part, les IFITM participent à la rigidification des membranes et, en favorisant une courbure positive, conduiraient à une diminution du processus de fusion [34]. Une seconde équipe a mis en évidence une interaction entre VAPA (vesicle-associated membrane protein-associated protein A) et IFITM3, inhibant l’interaction VAPA-OSBP (oxysterol-binding protein) qui est primordiale pour l’homéostasie intracellulaire du cholestérol [30]. Cela entraîne une accumulation de cholestérol dans les endosomes portant IFITM3, bloquant par conséquent le processus de fusion. Néanmoins, ces deux modèles ont été récemment remis en cause. Contrairement à ce qui avait été observé précédemment, la modification de la courbure membranaire n’abroge pas la restriction imposée par IFITM3 [36], et l’augmentation importante de la quantité de cholestérol en présence d’IFITM3 n’est pas non plus observée [36]. De plus, la manipulation artificielle des taux de cholestérol est inefficace : son augmentation n’inhibe pas l’entrée des virus restreinte par IFITM3, et son extraction ne restaure pas l’infection des cellules exprimant IFITM3 [36]. Les travaux de cette équipe suggèrent que les IFITM, par leur capacité à former des multimères et en rigidifiant la membrane, bloquent physiquement la formation du pore après l’hémifusion, en stabilisant l’hémimembrane interne. Une modification concomitante de la composition membranaire, aussi bien lipidique que protéique, n’est toutefois pas exclue. L’essentiel des travaux ont été réalisés avec IFITM3 et le virus de la grippe ou le JRSV (Jaagsiekte sheep retrovirus) mais, étant donné les similitudes du mode d’inhibition des autres virus, il est probable que le mécanisme d’action soit le même pour tous. Néanmoins, les résultats portant sur l’inhibition du VIH amènent à penser qu’IFITM1 pourrait agir différemment. |
Bien que l’étude identifiant les IFITM comme des facteurs de restriction n’ait pu mettre en évidence une inhibition du VIH, l’équipe du Dr Liang a montré que les trois IFITM inhibent la réplication du VIH [11]. IFITM1 est la plus efficace. IFITM2 et IFITM3 restreignent la réplication dès le processus d’entrée, de façon similaire à ce qui a été montré pour les autres virus, mais IFITM1 n’a aucun effet sur cette étape et n’inhibe par ailleurs aucune étape précoce de l’infection. Le rôle d’IFITM1 est donc autre, et il a été montré que ce facteur diminue la production de particules virales infectieuses [11]. En accord avec cette observation, l’équipe du Dr. Suzu a montré que les IFITM diminuent la production de ces particules en inhibant l’expression de plusieurs protéines virales (Gag [group-specific antigen protein], Nef [negative regulatory factor] et Vif [viral infectivity factor]) [37]. Il apparaît ainsi que les IFITM agissent à plusieurs étapes au cours de la réplication, en inhibant l’entrée du virus, mais possiblement aussi la synthèse des protéines virales. Pour mieux comprendre l’action d’IFITM1 sur le VIH, le virus a été « adapté » à la surexpression d’IFITM1 dans des cellules lymphocytaires (SupT1) [38]. Deux virus mutants ont été obtenus qui sont capables de se répliquer de façon plus robuste en présence d’IFITM1. Ces virus contiennent des mutations dans l’enveloppe et la protéine Vpu, leur permettant de se répliquer plus efficacement. Puisque l’enveloppe et Vpu partagent une localisation membranaire avec les IFITM, ces données sont intéressantes. Cependant, dans ces études, l’augmentation de la réplication des virus mutants a lieu dans les cellules en absence ou en présence d’IFITM1, suggérant que ces mutations pourraient agir indépendamment des IFITM. |
Au cours des dernières années, les IFITM ont été caractérisés comme des facteurs de restriction dotés d’un très large spectre antiviral. Contrairement aux facteurs décrits jusqu’alors, ces protéines inhibent l’entrée même du virus et empêchent l’infection avant de la combattre. Le mode d’action de ces protéines n’est pas encore totalement élucidé et de prochaines études permettront certainement de mieux comprendre comment elles peuvent bloquer un si grand nombre de virus. Au cours des différentes études cherchant à déterminer le mode d’action des IFITM, il est apparu que ces facteurs modifient les propriétés de la membrane. La question qui reste en suspens est comment ? Trois hypothèses sont possibles : (1) les IFITM modifient la fluidité membranaire, (2) elles altèrent la courbure de la membrane ou (3) elles changent la composition de la membrane. Puisque ces trois possibilités sont intimement liées, il est difficile de déterminer laquelle est responsable de l’effet antiviral des IFITM. Les IFITM agissent-elles directement pour inhiber le virus, ou sont-elles à l’origine d’une cascade d’évènements qui aboutissent à l’action antivirale ? De plus, la fonction des IFITM ne semble pas restreinte à l’inhibition virale, et les premières fonctions qui leur ont été attribuées intervenaient dans la mise en place de centres germinatifs [39], ainsi que dans la cancérogenèse et l’inflammation du tube digestif [40]. Il est donc tout à fait possible que les différentes fonctions des IFITM s’exercent via une propriété particulière à ces protéines, mais si tel est le cas, cette dernière reste à déterminer. En conclusion, les IFITM offrent une grande diversité de fonctions, cellulaires comme de défense antivirale. Nul doute que dans un futur proche, des résultats excitants nous en apprendront plus à ce sujet. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Sheehy AM, Gaddis NC, Choi JD, Malim MH. Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein . Nature. 2002; ; 418 : :646.–650. 2.
Neil SJ, Zang T, Bieniasz PD. Tetherin inhibits retrovirus release and is antagonized by HIV-1 Vpu . Nature. 2008; ; 451 : :425.–430. 3.
Stremlau M, Owens CM, Perron MJ, et al. The cytoplasmic body component TRIM5alpha restricts HIV-1 infection in old world monkeys . Nature. 2004; ; 427 : :848.–853. 4.
Hrecka K, Hao C, Gierszewska M, et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein . Nature. 2011; ; 474 : :658.–661. 5.
Laguette N, Sobhian B, Casartelli N, et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx . Nature. 2011; ; 474 : :654.–657. 6.
Goujon C, Moncorge O, Bauby H, et al. Human MX2 is an interferon-induced post-entry inhibitor of HIV-1 infection . Nature. 2013; ; 502 : :559.–562. 7.
Kane M, Yadav SS, Bitzegeio J, et al. MX2 is an interferon-induced inhibitor of HIV-1 infection . Nature. 2013; ; 502 : :563.–566. 8.
Liu Z, Pan Q, Ding S, et al. The interferon-inducible MxB protein inhibits HIV-1 infection . Cell Host Microbe. 2013; ; 14 : :398.–410. 9.
La Goujon C. protéine MX2 humaine est l’un des acteurs de la réponse interféron contre le VIH-1 . Med Sci (Paris). 2014; ; 30 : :233.–235. 10.
Brass AL, Huang IC, Benita Y, et al. The IFITM proteins mediate cellular resistance to influenza A H1N1 virus, West Nile virus, and dengue virus . Cell. 2009; ; 139 : :1243.–1254. 11.
Lu J, Pan Q, Rong L, et al. The IFITM proteins inhibit HIV-1 infection . J Virol. 2011; ; 85 : :2126.–2137. 12.
Weidner JM, Jiang D, Pan XB, et al. Interferon-induced cell membrane proteins, IFITM3 and tetherin, inhibit vesicular stomatitis virus infection via distinct mechanisms . J Virol. 2010; ; 84 : :12646.–12657. 13.
Anafu AA, Bowen CH, Chin CR, et al. Interferon-inducible transmembrane protein 3 (IFITM3) restricts reovirus cell entry . J Biol Chem. 2013; ; 288 : :17261.–17271. 14.
Wilkins C, Woodward J, Lau DT, et al. IFITM1 is a tight junction protein that inhibits hepatitis C virus entry . Hepatology. 2013; ; 57 : :461.–469. 15.
Mudhasani R, Tran JP, Retterer C, et al. IFITM-2 and IFITM-3 but not IFITM-1 restrict Rift valley fever virus . J Virol. 2013; ; 87 : :8451.–8464. 16.
Huang IC, Bailey CC, Weyer JL, et al. Distinct patterns of IFITM-mediated restriction of filoviruses, SARS coronavirus, and influenza A virus . PLoS Pathog. 2011; ; 7 : :e1001258.. 17.
Warren CJ, Griffin LM, Little AS, et al. The antiviral restriction factors IFITM1, 2 and 3 do not inhibit infection of human papillomavirus, cytomegalovirus and adenovirus . PLoS One. 2014; ; 9 : :e96579.. 18.
Zhao X, Guo F, Liu F, et al. Interferon induction of IFITM proteins promotes infection by human coronavirus OC43 . Proc Natl Acad Sci USA. 2014; ; 111 : :6756.–6761. 19.
Smith SE, Gibson MS, Wash RS, et al. Chicken interferon-inducible transmembrane protein 3 restricts influenza viruses and lyssaviruses in vitro . J Virol. 2013; ; 87 : :12957.–12966. 20.
Hickford D, Frankenberg S, Shaw G, Renfree MB. Evolution of vertebrate interferon inducible transmembrane proteins . BMC Genomics. 2012; ; 13 : :155.. 21.
Yount JS, Karssemeijer RA, Hang HC. S-palmitoylation and ubiquitination differentially regulate interferon-induced transmembrane protein 3 (IFITM3)-mediated resistance to influenza virus . J Biol Chem. 2012; ; 287 : :19631.–19641. 22.
Bailey CC, Kondur HR, Huang IC, Farzan M. Interferon-induced transmembrane protein 3 is a type II transmembrane protein . J Biol Chem. 2013; ; 288 : :32184.–32193. 23.
Weston S, Czieso S, White IJ, et al. A membrane topology model for human interferon inducible transmembrane protein 1 . PLoS One. 2014; ; 9 : :e104341.. 24.
John SP, Chin CR, Perreira JM, et al. The CD225 domain of IFITM3 is required for both IFITM protein association and inhibition of influenza A virus and dengue virus replication . J Virol. 2013; ; 87 : :7837.–7852. 25.
Jia R, Pan Q, Ding S, et al. The N-terminal region of IFITM3 modulates its antiviral activity by regulating IFITM3 cellular localization . J Virol. 2012; ; 86 : :13697.–13707. 26.
Yount JS, Moltedo B, Yang YY, et al. Palmitoylome profiling reveals S-palmitoylation-dependent antiviral activity of IFITM3 . Nat Chem Biol. 2010; ; 6 : :610.–614. 27.
Shan Z, Han Q, Nie J, et al. Negative regulation of interferon-induced transmembrane protein 3 by SET7-mediated lysine monomethylation . J Biol Chem. 2013; ; 288 : :35093.–35103. 28.
Chesarino NM, McMichael TM, Hach JC, Yount JS. Phosphorylation of the antiviral protein interferon-inducible transmembrane protein 3 (IFITM3) dually regulates its endocytosis and ubiquitination . J Biol Chem. 2014; ; 289 : :11986.–11992. 29.
Feeley EM, Sims JS, John SP, et al. IFITM3 inhibits influenza A virus infection by preventing cytosolic entry . PLoS Pathog. 2011; ; 7 : :e1002337.. 30.
Amini-Bavil-Olyaee S, Choi YJ, Lee JH, et al. The antiviral effector IFITM3 disrupts intracellular cholesterol homeostasis to block viral entry . Cell Host Microbe. 2013; ; 13 : :452.–464. 31.
Jia R, Xu F, Qian J, et al. Identification of an endocytic signal essential for the antiviral action of IFITM3 . Cell Microbiol. 2014; ; 16 : :1080.–1093. 32.
Diamond MS, Farzan M. The broad-spectrum antiviral functions of IFIT and IFITM proteins . Nat Rev Immunol. 2013; ; 13 : :46.–57. 33.
Cavrois M, De Noronha C, Greene WC. A sensitive and specific enzyme-based assay detecting HIV-1 virion fusion in primary T lymphocytes . Nat Biotechnol. 2002; ; 20 : :1151.–1154. 34.
Li K, Markosyan RM, Zheng YM, et al. IFITM proteins restrict viral membrane hemifusion . PLoS Pathog. 2013; ; 9 : :e1003124.. 35.
Desai TM, Marin M, Chin CR, et al. IFITM3 restricts influenza A virus entry by blocking the formation of fusion pores following virus-endosome hemifusion . PLoS Pathog. 2014; ; 10 : :e1004048.. 36.
Lin TY, Chin CR, Everitt AR, et al. Amphotericin B increases influenza A virus infection by preventing IFITM3-mediated restriction . Cell Rep. 2013; ; 5 : :895.–908. 37.
Chutiwitoonchai N, Hiyoshi M, Hiyoshi-Yoshidomi Y, et al. Characteristics of IFITM, the newly identified IFN-inducible anti-HIV-1 family proteins . Microbes Infect. 2013; ; 15 : :280.–290. 38.
Ding S, Pan Q, Liu SL, Liang C. HIV-1 mutates to evade IFITM1 restriction . Virology. 2014; ; 454–5 : :11.–24. 39.
Saitou M, Barton SC, Surani MA. A molecular programme for the specification of germ cell fate in mice . Nature. 2002; ; 418 : :293.–300. 40.
Hisamatsu T, Watanabe M, Ogata H, et al. Interferon-inducible gene family 1–8U expression in colitis-associated colon cancer and severely inflamed mucosa in ulcerative colitis . Cancer Res. 1999; ; 59 : :5927.–5931. 41.
Marquet R, Guerreri S, Bernacchi S, et al. Un facteur de transcription se fait complice du VIH-1 pour détruire les défenses cellulaires . Med Sci (Paris). 2012;; 28 : :356.–358. 42.
Lahouassa H, Dragin L, Transy C, et al. SAMHD1 prive le VIH des nucléotides essentiels à la synthèse d’ADN viral . Med Sci (Paris). 2012; ; 28 : :909.–910. |