| |
| Med Sci (Paris). 2015 June; 31(6-7): 612–615. Published online 2015 July 7. doi: 10.1051/medsci/20153106013.miR-122 continue de nous surprendre Chloé Mengardi1,2,3,4,5** and Théophile Ohlmann1,2,3,4,5* 1CIRI, centre international de recherche en infectiologie, Université de Lyon, Lyon, France 2Inserm, U1111, Lyon, France 3École normale supérieure de Lyon, Lyon, France 4Université Lyon 1, centre international de recherche en infectiologie, Lyon, France 5CNRS, UMR5308, Lyon, France MeSH keywords: Carcinome hépatocellulaire, Évolution de la maladie, Régulation de l'expression des gènes viraux, Hepacivirus, Hépatite C, Humains, Tumeurs du foie, microARN, ARN viral, Cellules cancéreuses en culture, Réplication virale, génétique, anatomopathologie, virologie, complications, physiologie, métabolisme |
Le virus de l’hépatite C (VHC), de la famille des Flaviviridae, est un pathogène humain du foie qui infecte actuellement 185 millions de personnes dans le monde. Son génome est un ARN positif simple brin codant pour une polyprotéine, clivée par la suite en dix protéines essentielles à sa réplication. L’ARN génomique (ARNg) du VHC est naturellement non coiffé, non polyadénylé, et il a la particularité de posséder dans sa région 5’ non traduite (5’UTR) un site d’entrée interne du ribosome appelé IRES (internal ribosome entry site) [1]. La réplication du VHC est stimulée par un microARN (miARN) très exprimé dans le foie : le miR-122 [2]. De manière générale, les miARN servent de guide pour le complexe RISC (RNA-induced silencing complex) dont Argonaute 2 (Ago2) est un des composants majeurs [3]. La plupart du temps, la fixation d’un miARN associé au complexe RISC sur la 3’UTR d’ARN messagers cibles induit l’inhibition de la synthèse protéique du transcrit ciblé [4]. De manière surprenante, miR-122 se lie à deux séquences localisées dans la région 5’ du génome viral, dans le domaine I de l’IRES du VHC, ce qui induit une augmentation de la réplication du virus [5] en stimulant sa traduction [6] et en protégeant son extrémité 5’ de la dégradation par l’exonucléase XrnI [7, 8]. Dix ans après la découverte de l’implication du miARN miR-122 dans la réplication du virus de l’hépatite C (VHC) [2], deux publications récentes [9, 10] nous incitent à porter un nouveau regard sur le rôle de ce miARN. |
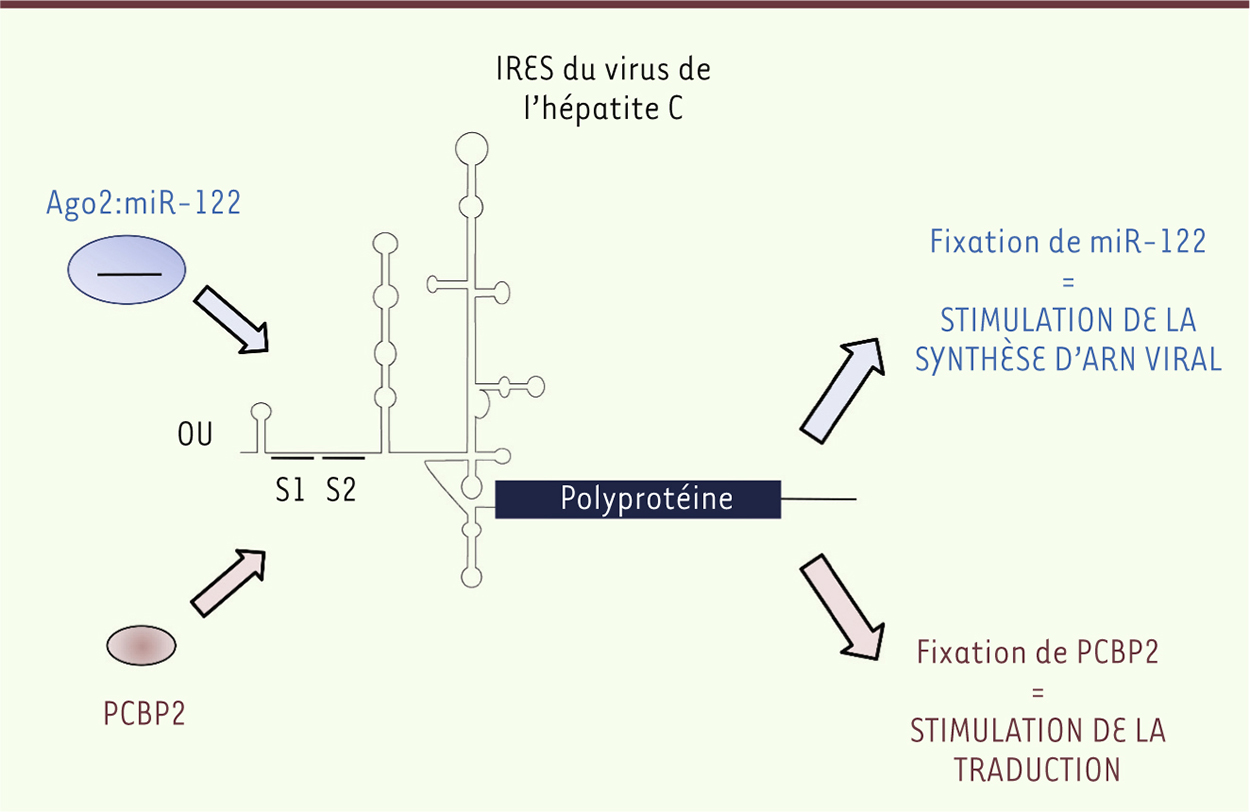
miR-122 réduit la circularisation de l’ARNg du VHC et stimule sa synthèse Dans l’étude publiée dans Cell Host & Microbe [9], l’équipe de Stanley Lemon démontre que miR-122 agit comme un élément régulateur de l’équilibre entre la synthèse de l’ARN viral et sa traduction. La surexpression de miR-122 dans une lignée de cellules hépatocytaires infectées par le VHC induit la stimulation de la synthèse de l’ARNg et la hausse de la production de protéines virales. Ce phénomène est observé aussi bien dans des cellules contrôles qu’après déplétion de la protéine Xrn11 (un membre de la famille d’exonucléase 5’-3’); ainsi, l’augmentation de la synthèse d’ARNg ne s’explique pas par une hausse de la stabilité du transcrit. En surexprimant miR-122 au cours de l’infection, les chercheurs ont mis en évidence que la hausse du taux d’ARNg néosynthétisé précède l’augmentation de la synthèse protéique, qui n’est que la conséquence logique de l’accroissement de la population d’ARN viral. Cette hypothèse est confirmée en montrant que miR-122 n’a plus aucun effet si la polymérase virale est préalablement bloquée avec des inhibiteurs spécifiques. En revanche, l’expérience inverse, qui consiste à bloquer la synthèse protéique en utilisant de la puromycine ou de la cycloheximide, a des conséquences plus étonnantes. En effet, l’addition de cycloheximide seule (sans surexpression de miR-122) bloque à la fois la traduction et la synthèse de l’ARN viral, alors que la puromycine inhibe la synthèse protéique mais provoque une augmentation de la production de l’ARNg du VHC. Ce phénomène peut s’expliquer par le fait que la puromycine libère l’ARNg des polysomes, alors que la cycloheximide fixe ces derniers sur le transcrit. De fait, en présence de puromycine, l’ARN est alors disponible pour la réplication par la polymérase. En effet, il est utile de rappeler que l’ARNg du VHC sert à la fois d’ARN messager (ARNm) pour la synthèse des protéines virales et de matrice pour la synthèse et la réplication du génome. Ces deux fonctions, nécessaires à la réplication virale, sont mutuellement exclusives avec un flux de ribosomes qui migrent dans le sens 5’ → 3’ sur l’ARNg, alors que la polymérase progresse en 3’ → 5’ pour réaliser la réplication. Toutefois, l’addition de miR-122 dans ces cellules traitées par les inhibiteurs de la traduction n’augmente pas le taux d’ARNg néosynthétisé, ce qui indique que la synthèse des protéines est néanmoins une étape nécessaire. À partir de ces données, les auteurs émettent l’hypothèse selon laquelle miR-122 pourrait jouer le même rôle que la puromycine, c’est-à-dire inhiber la synthèse protéique en libérant l’ARNg des polysomes, créant ainsi une situation favorable à sa réplication. Ceci est étayé par l’utilisation de gradients de polysomes qui montrent que l’addition de miR-122 provoque une réduction de la fraction polysomale au profit de l’émergence de monosomes 80S [16] (→). Afin de décortiquer le mécanisme moléculaire par lequel miR-122 induit une telle transition, les chercheurs se sont alors intéressés à la protéine PCBP2 (Poly(C) binding protein 2), qui a la particularité de se lier à l’extrémité 5’ de l’ARNg du VHC et de stimuler sa traduction tout en favorisant la « circularisation » du génome viral [11]. Or, le site de fixation de PCBP2 chevauche le second site de liaison de miR-122 sur le 5’ de l’ARN et entre donc en compétition avec ce dernier (Figure 1). De fait, miR-122 n’a plus d’effet ni sur la synthèse de l’ARN viral ni sur la production protéique dans des cellules dépourvues de PCBP2, confirmant ainsi l’implication de cette protéine. Les auteurs suggèrent que le déplacement de PCBP2 de l’ARNg réduirait la circularisation de ce dernier, ce qui libérerait la 3’UTR rendue disponible pour interagir avec la polymérase (Figure 1).
 | Figure 1.
Le complexe miR-122 : Argonaute 2 régule l’équilibre entre traduction et réplication de l’ARN génomique du virus de l’hépatite C (VHC). La protéine poly(C) binding protein 2 (PCBP2) a la particularité de « circulariser » l’ARN viral et de stimuler sa traduction. miR-122 entre en compétition avec PCBP2 pour se fixer sur l’IRES, car leurs sites de liaison respectifs se chevauchent (au niveau du site S2 de miR-122). Le complexe miR-122 associé à la protéine Argonaute 2 (Ago2) déplace PCBP2 de l’ARN viral, ce qui engendre une rupture de la circularisation de l’ARN génomique rendant la 3’UTR disponible à la polymérase ce qui favorise la synthèse de l’ARN. |
(→) Voir la Synthèse de L. Tafforeau, page 622 de ce numéro
|
Séquestration de miR-122 par l’ARN génomique du VHC La deuxième étude, publiée dans Cell en mars 2015 [10], est plus globale et vise à mesurer l’impact de l’infection des cellules par le VHC sur la relation entre miR-122 et ses cibles cellulaires. Pour cette étude, J.M. Luna et al. ont utilisé un éventail de nouvelles techniques combinant de multiples approches « -omiques ». Dans un premier temps, en conjuguant séquençage à haut débit et pontage covalent suivi d’une immunoprécipitation (HIT-CLIP, high-throughput sequencing of RNA isolated by crosslinking immunoprecipitation) de la protéine Ago2, les chercheurs ont établi la carte des interactions de cette dernière le long de l’ARNg du VHC. Ils ont ensuite démontré, en utilisant des cellules de la lignée hépatocytaire HuH7 dans lesquelles le gène Drosha - codant pour une ribonucléase de type III nécessaire à la maturation des précurseurs des miARN - a été éliminé par la technique CRISPR (→), mais dont l’expression de miR-122 a été restaurée, que la majorité des sites de liaison d’Ago2 sur le génome du VHC correspondaient aux sites de liaison de miR-122. En affinant cette approche par CIMS (crosslink-induced mutation site analysis) [12], ils ont cartographié dans l’IRES avec une résolution de l’ordre du nucléotide, 3 sites de liaison de miR-122 dont deux au niveau du domaine I qui correspond aux sites S1 et S2, et un autre à la jonction entre le pseudo-nœud et le codon AUG. Ces sites ont déjà été décrits comme étant les cibles préférentielles de la liaison de miR-122 [5].
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 12, décembre 2014, page 1066
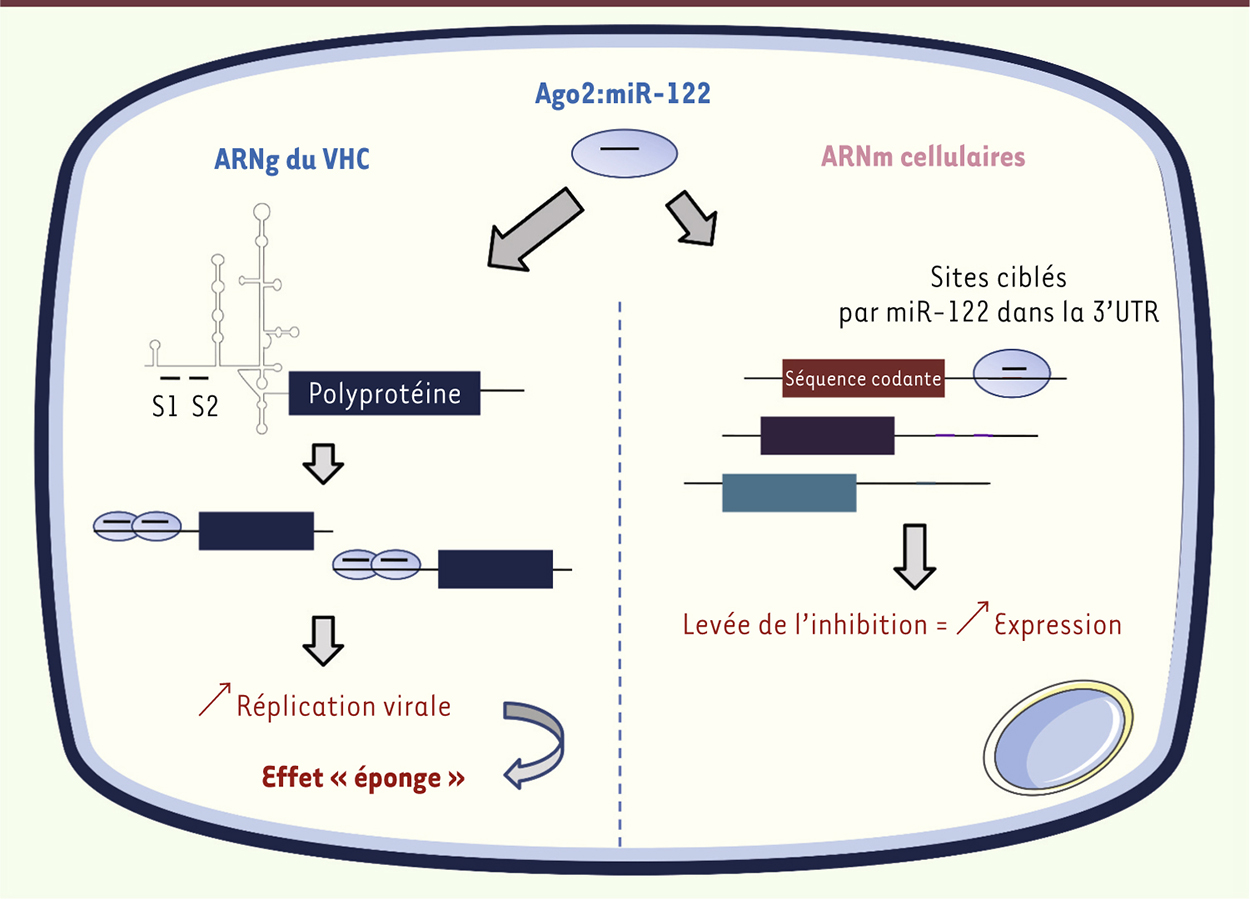
L’expérience de HIT-CLIP avec Ago2 a ensuite été reproduite sur les ARNm cellulaires de cellules infectées, ou non, par le VHC. En conditions d’infection, une forte réduction de la liaison d’Ago2 à l’ensemble des ARNm cellulaires contenant un, ou plusieurs, sites de liaison à miR-122 est observée. En transfectant dans des cellules des gènes rapporteurs qui possèdent dans leur région 3’UTR entre 1 et 6 sites ciblés par miR-122, les auteurs montrent que l’infection de ces cellules par le VHC entraîne une dérépression de l’expression de ces gènes. Celle-ci intervient de manière stœchiométrique et dépend à la fois du nombre de sites de liaison miR-122 et de la concentration intracellulaire du transcrit. Cela suggère que l’ARNg du VHC agit comme une « éponge » en séquestrant miR-122 pour l’empêcher de se fixer à ses cibles cellulaires (Figure 2).
 | Figure 2.
L’ARN génomique du virus de l’hépatite C agit comme une « éponge » pour miR-122. miR-122 se fixe préférentiellement sur deux sites (S1 et S2) en 5’ de l’ARN viral. Dans les cellules hépatocytaires, l’ARN du VHC va séquestrer miR-122, empêchant ce dernier de réguler ses cibles cellulaires. Comme miR-122 stimule également la réplication virale, et donc la production d’ARN génomique néosynthétisé, cela crée une boucle de régulation positive qui permet d’amplifier d’avantage le phénomène d’ARN « éponge ». |
Cette hypothèse a été confirmée en mutant dans le virus les sites de liaison S1 et S2 pour miR-122 afin de les transformer en sites de fixation pour le miR-15a/b. En effet, miR-15a/b est en concentration aussi abondante que miR-122 dans les cellules HuH7. Ce virus mutant est viable et se réplique désormais indépendamment de miR-122, mais requiert désormais la fixation de miR-15a/b. La réplication de ce virus mutant dans les cellules hôtes provoque la levée de l’inhibition de l’expression des gènes cellulaires ciblés par miR-15a/b sans toutefois affecter ceux qui sont sous la dépendance de miR-122. Cela confirme donc de manière élégante la théorie de l’ARN « éponge », décrite précédemment pour des ARN cellulaires appelés « ceARN » [13], mais aussi dans le cas de génomes viraux comme ceux du cytomégalovirus et des herpes virus [14, 15]. Cependant, le VHC ajoute un niveau de régulation encore plus subtil à ce mécanisme, puisqu’il utilise aussi miR-122 pour sa réplication, ce qui provoque une augmentation du nombre de copies de l’ARNg, qui ne font qu’augmenter sa capacité à séquestrer miR-122. |
Quelles conséquences de la séquestration de miR-122 ? Ces deux articles ouvrent des perspectives intéressantes pour notre compréhension du rôle des miARN au cours de la réplication virale. En effet, dans le cas du rôle de miR-122 dans la régulation de la balance entre traduction et réplication de l’ARN viral, il est très probable que d’autres virus à ARN positif puissent utiliser un mécanisme similaire. Dans le deuxième cas de figure, la séquestration de miR-122 au cours de l’infection chronique par le VHC pourrait avoir des conséquences importantes sur la régulation de gènes cellulaires, notamment ceux qui sont impliqués dans la progression vers l’hépatocarcinome. |
Footnotes |
1.
Tsukiyama-Kohara K, Iizuka N, Kohara M, Nomoto A. Internal ribosome entry site within hepatitis C virus RNA . J Virol. 1992; ; 66 : :1476.–1483. 2.
Jopling CL, Yi M, Lancaster AM, Lemon SM, Sarnow P. Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA . Science. 2005; ; 309 : :1577.–1581. 3.
Fabian MR, Sonenberg N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC . Nat Struct Mol Biol. 2012; ; 19 : :586.–593. 4.
Ricci EP, Limousin T, Soto-Rifo R, et al. miRNA repression of translation in vitro takes place during 43S ribosomal scanning . Nucleic Acids Res. 2013; ; 41 : :586.–598. 5.
Jopling CL, Schütz S, Sarnow P. Position-dependent function for a tandem microRNA miR-122-binding site located in the hepatitis C virus RNA genome . Cell Host Microbe. 2008; ; 4 : :77.–85. 6.
Roberts APE, Lewis AP, Jopling CL. miR-122 activates hepatitis C virus translation by a specialized mechanism requiring particular RNA components . Nucleic Acids Res. 2011; ; 39 : :7716.–7729. 7.
Li Y, Masaki T, Yamane D, McGivern DR, Lemon SM. Competing and noncompeting activities of miR-122 and the 5’ exonuclease Xrn1 in regulation of hepatitis C virus replication . Proc Natl Acad Sci USA. 2013; ; 110 : :1881.–1886. 8.
Sedano CD, Sarnow P. Hepatitis C virus subverts liver-specific miR-122 to protect the viral genome from exoribonuclease Xrn2 . Cell Host Microbe. 2014; ; 16 : :257.–264. 9.
Masaki T, Arend KC, Li Y, et al. miR-122 stimulates hepatitis C virus RNA synthesis by altering the balance of viral RNAs engaged in replication versus translation . Cell Host Microbe. 2015; ; 17 : :217.–228. 10.
Luna JM, Scheel TKH, Danino T, et al. Hepatitis C virus RNA functionally sequesters miR-122 . Cell. 2015; ; 160 : :1099.–1110. 11.
Wang L, Jeng KS, Lai MMC. Poly(C)-binding protein 2 interacts with sequences required for viral replication in the hepatitis C virus (HCV) 5’ untranslated region and directs HCV RNA replication through circularizing the viral genome . J Virol. 2011; ; 85 : :7954.–7964. 12.
Zhang C, Darnell RB. Mapping in vivo protein-RNA interactions at single-nucleotide resolution from HITS-CLIP data . Nat Biotechnol. 2011; ; 29 : :607.–614. 13.
Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language? Cell. 2011; ; 146 : :353.–358. 14.
Cazalla D, Yario T, Steitz JA, Steitz J. Down-regulation of a host microRNA by a Herpesvirus saimiri noncoding RNA . Science. 2010; ; 328 : :1563.–1566. 15.
Lee S, Song J, Kim S, et al. Selective degradation of host MicroRNAs by an intergenic HCMV noncoding RNA accelerates virus production . Cell Host Microbe. 2013; ; 13 : :678.–690. 16.
Tafforeau L. Qu’en est-il de la biogenèse des ribosomes chez l’homme ? Med Sci (Paris). 2015; ; 31 : :622.–628. |