| |
| Med Sci (Paris). 2015 June; 31(6-7): 680–686. Published online 2015 July 7. doi: 10.1051/medsci/20153106023.L’apophénie d’ENCODE ou Pangloss examine le génome humain Didier Casane,1,2** Julien Fumey,1*** and Patrick Laurenti1,2* 1Laboratoire Évolution, génomes, comportement, écologie, CNRS université Paris-Sud UMR 9191, IRD UMR 247, Avenue de la Terrasse, bâtiment 13, boîte postale 1, 91198Gif-sur-Yvette, France 2Université Paris-Diderot, Sorbonne Paris-Cité, Paris, France |

Le 7 septembre 2012, il était proclamé dans la revue Science : « ENCODE project writes eulogy for junk DNA » [1], ce qui peut être traduit par : « ENCODE écrit l’éloge funèbre de l’ADN poubelle ». L’ADN poubelle est mort, vive l’ADN fonctionnel ! L’invraisemblable nombre de fragments de transposons, de pseudogènes qui ne peuvent permettre la production d’ARN fonctionnels, tout ce fatras d’ADN qui ne ressemble à rien de nécessaire et qui apparaît et disparaît des génomes au fil du temps… Et bien, non, ce ne serait pas des déchets accumulés dans les génomes, mais de l’ADN fonctionnel ! Il ne restait plus alors qu’à identifier plus précisément ces fonctions [2]. Le consortium ENCODE annonçait avoir « renversé un paradigme », et il fallait se préparer à réécrire les manuels universitaires [1]. Mais, au fait, le projet ENCODE, c’est quoi au juste ? C’est l’Encyclopedia of DNA Elements. Il existe aussi modENCODE (model organism ENCODE). Le but de ces deux projets est d’identifier tous les éléments fonctionnels du génome humain et des génomes d’organismes modèles (drosophiles et nématodes). La stratégie retenue est de compiler, de la façon la plus exhaustive et précise possible, le niveau de transcription, l’association à des facteurs de transcription, l’organisation de la chromatine et les modifications des histones, à l’échelle de l’ensemble du génome, et ce pour différents types cellulaires et pour différentes espèces. C’est un travail colossal et une base de données de cette nature est potentiellement de première importance pour un très grand nombre de biologistes. Les investissements humains et financiers furent à l’échelle du projet : 442 chercheurs et 288 millions de dollars [1]. Mais peut-on aujourd’hui investir autant de moyens pour générer une base de données énorme sans obtenir un résultat biologique extraordinaire ? On peut facilement répondre oui en termes d’avancement des sciences, mais non en termes de publicité. Un fort investissement réclame un résultat fort. Ainsi, la NASA nous a accoutumés à des découvertes fracassantes, comme la démonstration de la présence de vie sur Mars ou de l’existence de molécules d’ADN contenant de l’arsenic… (→). Ces découvertes furent réfutées, mais la NASA se doit de justifier de temps en temps son colossal budget. L’objectif d’une stratégie très coûteuse est en effet de découvrir ce qui n’est pas à la portée des chercheurs ordinaires, c’est-à-dire passablement isolés et disposant de peu de moyens. De fait, ENCODE a produit des milliers de jeux de données de grande taille et publié un grand nombre d’articles en 2012. Malheureusement, l’analyse de ces masses étourdissantes de données ne fut pas guidée par des questions scientifiques précises. Les graphiques colorés abondaient, mais il ne ressortait rien de vraiment nouveau de tout ça. Et c’était tout à fait normal puisque ce n’était pas le but du projet. Il fallait toutefois, et à toute force, obtenir un résultat percutant et un paradigme renversé. Ce fut donc « 80 % de l’ADN humain est fonctionnel ». Sachant qu’auparavant, il n’avait été identifié qu’environ 10 % à 15 % de génome utile [3, 4], il y aurait donc 70 % de matière noire en attente de fonction et à explorer [5]. Fallait-il être bête pour croire que l’ADN humain, la molécule la plus parfaite de la plus parfaite des créations de la nature n’abritait pas quelques fonctions cachées ? La publicité faite à cette découverte sortit alors largement du cadre des spécialistes de la génomique. Certains dès lors pouvaient aussi « légitimement » s’interroger sur la nécessité du maintien de la « SMALL » science, comme la biologie évolutive ou la génétique, reposant sur peu de chercheurs qui disposent de peu de moyens [6]. Ces affirmations semèrent la consternation chez les spécialistes du domaine. Mais, accoutumés à ce que la biologie évolutive soit l’objet de spéculations hasardeuses, la plupart des évolutionnistes haussèrent les épaules, et retournèrent à leurs chères études sous financées. Cependant, quelques uns, et non des moindres, firent l’effort de publier des analyses critiques très argumentées [7–10]. Dan Graur s’est fait le champion des pourfendeurs des inepties propagées à grande échelle autour de cette découverte. Adepte d’un humour grinçant, auteur de chroniques hilarantes sur la « BIG » science en général et ENCODE en particulier sur son blog dont nous conseillons vivement la lecture1, il écrivit en 2013 un article très caustique [11] qui ramena la découverte faite par ENCODE à ce qu’elle est : un affichage publicitaire.
(→) Voir la Brève de Sophia Häfner, m/s n° 2, février 2011, page 145
En 2014, nouvelle livraison d’articles par le consortium ENCODE dans la prestigieuse revue Nature. Les articles sont toujours aussi ennuyeux et toujours aussi bien illustrés par des graphiques toujours aussi colorés. L’information mise en avant est que la masse de données croît très régulièrement [12]. La base de données a plus que doublé de volume. Bravo ! En revanche, les conclusions des articles de 2014 semblent bien insipides. Qu’on en juge par la platitude de la conclusion de la comparaison des transcriptomes humains / drosophiles / nématodes : « nos résultats soulignent l’importance de comparer des organismes modèles distants avec les humains afin de distinguer les principes biologiques [sic !] conservés des adaptations spécifiques aux lignées
2 » [13]. Finalement, l’information principale n’apparaît qu’en creux : l’utilité de 80 % du génome n’est plus évoquée nulle part. Au contraire, comme l’a fait remarquer Dan Graur dans une chronique récente3,, le News and Views associé aux cinq publications de Nature prend soin de souligner qu’aucun de ces articles ne permet de conclure quant à la fonctionnalité des séquences étudiées [12]. En somme, les interprétations panglossiennes font une sortie de scène aussi discrète que leur entrée avait été tonitruante. Pourtant, ce même News and Views rappelle en introduction : « Les projets Encyclopédie des éléments d’ADN (ENCODE) et ENCODE des organismes modèles (modENCODE) furent lancés dans le but d’identifier tous les éléments fonctionnels des génomes de l’espèce humaine, de la mouche Drosophila melanogaster et du ver Caenorhabditis elegans »4. Si le but est toujours d’identifier l’ensemble des éléments fonctionnels des génomes, la disparition de la « fonction » de l’ADN poubelle et la critique des interprétations de 2012 auraient dû faire les grands titres des commentaires. Cette étonnante omission nous fait craindre que le ver ne soit toujours dans le fruit. Aussi, il nous semble important d’enfoncer encore quelques clous dans le cercueil du Dr Pangloss afin qu’il ne revienne hanter les interprétations des analyses génomiques à venir. De façon plus générale, nous nous interrogeons sur les dérives de la communication des grands projets scientifiques de type « BIG science », et plaidons pour une plus grande reconnaissance des bases théoriques qu’apportent les « SMALL sciences » comme la génétique ou la biologie évolutive. |
L’apophénie d’ENCODE : ils voient des fonctions partout Revenons tout d’abord à l’affirmation de 2012. Le génome serait à 80 % fonctionnel ! Bon, pourquoi pas, mais quelle est la définition de « fonction » pour ENCODE ? Dans son acception courante, une fonction implique une chose « utile » à quelque chose. Pour la plupart des biologistes, un fragment d’ADN possède une fonction au niveau de l’organisme qui le contient lorsqu’il est impliqué dans quelque processus utile à la vie de cet organisme et à la transmission de son patrimoine génétique [14]. Mais ENCODE utilise une définition plus inattendue : le fragment d’ADN doit être transcrit et/ou impliqué dans des interactions avec des protéines connues pour leurs rôles dans la régulation de la transcription, et ce dans au moins un des types cellulaires testés. Donc, si un fragment d’ADN est transcrit ou s’il sert à transcrire un autre fragment d’ADN, c’est un fragment d’ADN fonctionnel. Il est depuis longtemps établi que l’essentiel du génome est transcrit [15] et que les séquences sur lesquelles se fixent les facteurs de transcription sont de très petite taille et donc présentes en très nombreuses copies dans les génomes [16]. La philosophie d’ENCODE, simpliste, est la suivante : si ça existe, c’est bien pour quelque chose. Pourquoi pas, mais encore faut-il le démontrer. Surtout dans le cas de l’ADN, pour lequel les preuves du contraire s’accumulent depuis trente ans [10]. L’erreur d’ENCODE fut de tenter de ressusciter des idées sur l’évolution qui furent progressivement abandonnées au cours de la fin du xxe siècle. En effet, dans la première moitié du siècle dernier, la sélection a parfois fait figure de mécanisme tout puissant qui optimise toutes les caractéristiques des organismes, génome compris, et maximise ainsi la valeur sélective. Il n’y a pas de but prédéfini, comme dans une conception créationniste, mais un état optimal vers lequel tend la population sous l’effet de la sélection. En somme, tout va pour le mieux, ou pour le moins tend vers le mieux, dans le meilleur des mondes possibles. C’est ce qu’on appelle le paradigme panglossien : tout caractère observé ne peut s’expliquer que par son utilité (voir Encadré). Cette vision pan-adaptationniste de l’évolution fut énergiquement remise en cause au cours de la deuxième moitié du xxe siècle, en particulier par S.J. Gould and R.C. Lewontin [17]. Une large part de la recherche en biologie évolutive depuis plus de quarante ans a donc consisté, et avec succès, à montrer les limites d’une vision « adaptationniste » du monde vivant. Tout d’abord, la plupart des mutations sont neutres : elles n’améliorent ni ne détériorent la valeur sélective des organismes. Ces modifications du génome disparaissent ou se fixent au gré du hasard. C’est donc essentiellement la dérive génétique, c’est-à-dire les fluctuations aléatoires des fréquences alléliques au cours des générations5 qui fait évoluer l’architecture des génomes [18]. Par ailleurs, même pour les locus soumis à sélection, la sélection naturelle n’est pas toute puissante : la dérive génétique peut contrecarrer la sélection et permettre la fixation d’une mutation délétère et la disparition d’une mutation avantageuse [19]. Enfin, la sélection agit à différents niveaux d’organisation, entre gènes dans un génome, entre organismes dans une population, entre populations dans une espèce, voire entre espèces. C’est l’origine de nombreux conflits génétiques qui entraînent la mise en place de compromis qui ne sont pas des solutions optimales à chaque niveau d’organisation. Ainsi, à un locus, un allèle peut se fixer au détriment d’un autre allèle tout en abaissant la valeur sélective de l’organisme qui le porte [20]. L’architecture et le fonctionnement des génomes ne sont donc pas le résultat de la maximisation d’un bien commun.
« Le melon a été divisé en tranches par la nature afin d’être mangé en famille ;
la citrouille, étant plus grosse, peut être mangée avec les voisins. »
Jacques-Henri Bernardin de Saint-Pierre, Études de la Nature, 1784.
Pangloss enseignait la métaphysico-théologo-cosmolo-nigologie. Il prouvait admirablement qu’il n’y a point d’effet sans cause […].
« Il est démontré, disait-il, que les choses ne peuvent être autrement : car, tout étant fait pour une fin, tout est nécessairement pour la meilleure fin. Remarquez bien que les nez ont été faits pour porter des lunettes ; aussi avons-nous des lunettes. Les jambes sont visiblement instituées pour être chaussées, et nous avons des chausses. Les pierres ont été formées pour être taillées et pour en faire des châteaux ; aussi monseigneur a un très beau château : le plus grand baron de la province doit être le mieux logé ; et les cochons étant faits pour être mangés, nous mangeons du porc toute l’année. Par conséquent, ceux qui ont avancé que tout est bien ont dit une sottise : il fallait dire que tout est au mieux. » […]
Et Pangloss disait quelquefois à Candide : « Tous les événements sont enchaînés dans le meilleur des mondes possibles ; car enfin, si vous n’aviez pas été chassé d’un beau château à grands coups de pied dans le derrière pour l’amour de Mlle Cunégonde, si vous n’aviez pas été mis à l’Inquisition, si vous n’aviez pas couru l’Amérique à pied, si vous n’aviez pas donné un bon coup d’épée au baron, si vous n’aviez pas perdu tous vos moutons du bon pays d’Eldorado, vous ne mangeriez pas ici des cédrats confits et des pistaches. – Cela est bien dit, répondit Candide, mais il faut cultiver notre jardin. »
Voltaire, Candide ou l’Optimisme, 1759.
|
|
L’ADN humain serait à 80 % fonctionnel, en utilisant la définition et les données d’ENCODE. Si c’est vrai, alors il l’est très vraisemblablement à 100 %, car tous les types cellulaires et tous les environnements cellulaires n’ont pas été testés. Par ailleurs, il fut souligné assez malicieusement que l’activité de réplication n’a pas été prise en compte, alors que 100 % du génome participe à sa propre réplication. Il n’y a donc rien à jeter dans notre génome. Encore plus malicieusement, Dan Graur s’interroge sur les raisons de cette retenue d’ENCODE. S’agit-il de donner un air plus scientifique à cette « découverte » ou de laisser un peu d’espace pour les concepts éculés de la biologie évolutive [11] ? |
La matière noire du génome humain Sachant que la biologie évolutive, en mesurant la pression de sélection sur les séquences, propose qu’il y a environ 10 % à 15 % d’ADN utile dans notre génome [3, 4], que contiennent les 85 % de matière noire aux supposées crypto-fonctions ? En fait, cet ADN n’est pas si noir que ça, car il est depuis longtemps établi qu’il est pour l’essentiel composé de fragments de transposons, plus ou moins anciens et donc parfois très difficiles à identifier [21], et de pseudogènes qui ne sont pas transcrits en ARN utiles au fonctionnement de l’organisme [22]. Il y a en outre une bonne dose de séquences de plus ou moins grandes tailles qui sont plus ou moins répétées, les microsatellites, minisatellites et autres satellites. L’origine de tous ces éléments est très bien identifiée.
-
Les transposons ont la propriété de faire des copies d’eux-mêmes qui s’insèrent dans le génome, mais souvent seuls des fragments sont dupliqués et, pour la plupart, ils perdent ce faisant la capacité à se propager dans le génome. C’est un des exemples les mieux analysés de conflit génétique : les transposons maximisent leur transmission en proliférant dans les génomes tandis que le reste du génome lutte contre cette prolifération. La multiplication des copies de transposons se fait au détriment des autres gènes du fait de l’effet délétère de certaines de ces insertions. Les cycles de propagation et de contrôle des transposons sont très bien étudiés tant d’un point de vue théorique qu’expérimental, et, pour la plupart de ces transpositions, il n’a été détecté aucune utilité pour l’organisme [23, 24].
-
La deuxième source massive d’ADN sans fonction est représentée par les pseudogènes qui se forment à la suite d’évènements de duplication de gènes. La formation de deux copies entraîne la présence de deux sources redondantes de la même fonction. Dans la plupart des très nombreux cas répertoriés, une des copies accumule des mutations qui la rendent non fonctionnelle, et ce sans perte de fitness au niveau de l’organisme [25].
-
De plus, des séquences répétées naissent régulièrement, car la réplication présente une fâcheuse tendance : elle bégaie ! En présence d’une répétition de quelques nucléotides, l’ADN polymérase ajoute ou enlève fréquemment des répétitions. La recombinaison a également tendance à produire des séquences répétées en tandem.
Une grande quantité d’ADN sans fonction à l’échelle de l’organisme peut donc être produite et se maintenir longtemps, à la condition qu’il n’y ait pas de mécanisme efficace pour l’éliminer, c’est-à-dire des délétions de fragments d’ADN qui donnent un avantage sélectif suffisant aux individus porteurs pour qu’elles se fixent dans les populations. Sans gain de valeur sélective, la perte d’ADN inutile ne s’effectue donc que du seul fait du hasard, la dérive génétique. Donc, si cet ADN sans fonction est neutre ou presque neutre du point de vue de la sélection, il peut être stocké longtemps dans le génome. C’est l’origine de l’ADN poubelle (junk DNA), un ADN qui ne contient aucune information utile à la vie ni à la transmission du génome d’un individu. Il est vrai que des fragments de transposons, des pseudogènes et des séquences non codantes répétées en tandem acquièrent quelquefois des fonctions. On parle alors d’« exaptation » [26]. Mais ces observations confirment, et n’infirment en rien, l’existence de l’ADN poubelle. La grande quantité d’ADN poubelle repose sur la multitude de mécanismes qui le génèrent, le peu de puissance des mécanismes qui l’éliminent, et le fait que son recyclage fonctionnel est très rare. Cette conception de la dynamique de l’architecture des génomes n’est donc en rien invalidée par les résultats d’ENCODE. La seule nouveauté en la matière est la découverte récente de la possibilité de l’émergence au sein de l’ADN poubelle de nouveaux gènes codant des protéines [27]. |
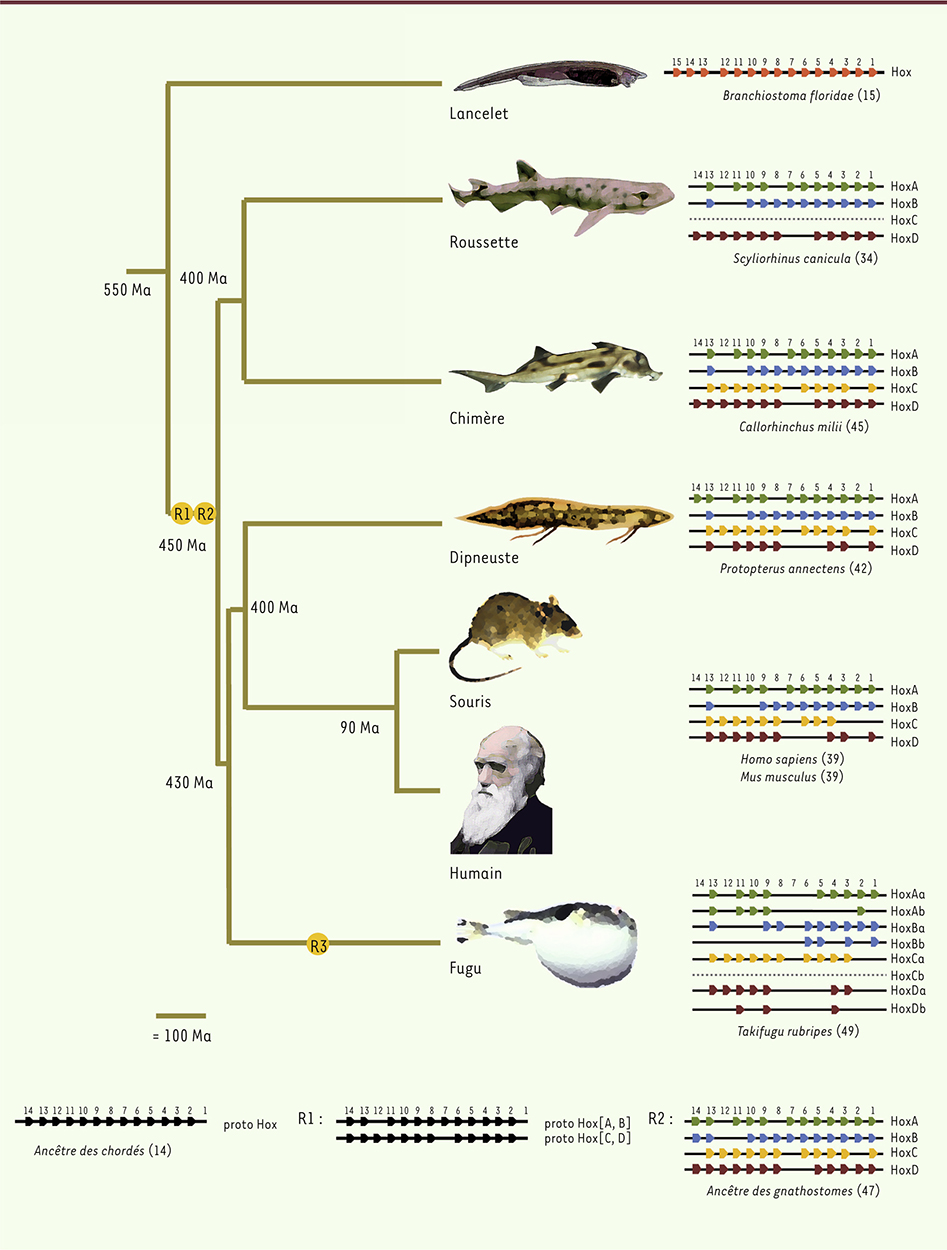
Crypto-fonction de la matière noire : tout s’obscurcit ! La seule façon de maintenir l’hypothèse d’une fonction portant sur l’ensemble du génome est de proposer que cette fonction ne dépende pas de sa séquence, mais de la quantité totale d’ADN. Pour une raison absolument inconnue à ce jour, nos cellules devraient contenir environ 7 pg d’ADN. Une telle fonction de la « masse noire » s’oppose à l’ensemble des connaissances de la dynamique des génomes. Les deux mécanismes principaux qui permettent de faire varier fortement la quantité d’ADN d’un génome sont la polyploïdisation et les pics de transpositions [18]. Notre génome a ainsi vu son poids doubler deux fois à l’occasion de deux évènements de tétraploïdisation (R1, R2) qui ont eu lieu il y a environ 500 millions d’années. Il a ensuite perdu beaucoup de poids. On peut retracer cette histoire en étudiant la structure des complexes de gènes Hox (Figure 1). Ainsi, bien que chaque gène doive être présent sous la forme de quatre copies, une par complexe, il ne reste en général que deux ou trois copies, rarement les quatre copies de départ. Les requins ont perdu un complexe entier, le complexe HoxC [28, 29]. Chez les poissons actinoptérygiens, une tétraploïdisation supplémentaire (R3) est à l’origine de huit complexes de gènes. Après cette duplication des complexes de gènes, il y a eu de nombreuses pertes de gènes, voire de complexes complets. Finalement, chez tous les vertébrés à mâchoires, le nombre de gènes Hox varie dans une gamme assez étroite, à l’exception des espèces chez lesquelles une nouvelle duplication du génome s’est produite récemment, ne laissant pas le temps à la perte de gènes. Ainsi, on compte 105 gènes Hox chez le saumon [30]. Les génomes ont donc tendance à revenir à des tailles intermédiaires après les évènements d’amplification massive, mais on observe chez certaines espèces des génomes extraordinairement grands et, chez d’autres, des génomes extraordinairement petits. Parmi les vertébrés, le génome humain a une taille intermédiaire (7 pg/cellule diploïde). Le fugu possède un des plus petits génomes (0,8 pg) et un dipneuste africain possède un des plus grands génomes (266 pg) (Figure 2). La découverte que la taille du génome du fugu est huit fois inférieure à celle du génome humain pour un nombre de gènes équivalent fut d’ailleurs saluée, il y a plus de vingt ans par Bertrand Jordan dans ces mêmes colonnes, comme un argument définitif en faveur de l’absence de fonction de l’ADN poubelle [31]. Aucune explication fonctionnelle n’a pu être proposée, à ce jour, pour expliquer ces différences de taille de génomes. Il est probable que la taille de ces génomes dépende essentiellement du ratio prolifération/élimination de l’ADN poubelle [32, 33].
 | Figure 1.
Événements de tétraploïdisation et évolution des complexes Hox chez les vertébrés.
|
 | Figure 2.
Classement de trois vertébrés en fonction de la taille de leur génome.
|
|
Le dessin inintelligent de notre génome Reconsidérons maintenant les observations d’ENCODE en prenant en compte que notre génome contient une multitude de séquences sur lesquelles des facteurs de transcription peuvent se fixer. Il n’est pas étonnant qu’il soit entièrement transcrit, au moins à faible niveau. Le très fort taux de turnover de ces sites de fixation et de ces transcrits au cours de l’évolution, ainsi que la difficulté à leur trouver quelque fonction biologique, montrent qu’ils ne sont très vraisemblablement pas indispensables [34]. La petite taille des motifs reconnus par les facteurs de transcription explique très simplement la forte probabilité d’apparition d’un tel site dans un quelconque fragment d’ADN [16]. La transcription semble être un processus biochimique qui peut démarrer assez facilement un peu n’importe où dans un génome [15]. Clairement, notre génome n’est pas optimisé, ni au niveau de son organisation, ni au niveau de son fonctionnement. Mais, c’est aussi vrai au niveau d’autres processus biochimiques et jusqu’à celui de la morpho-anatomie. Le mieux n’est pas nécessairement accessible, parce que tout simplement il n’apparaît pas (toutes les mutations possibles ne se réalisent pas, et loin de là), ou parce que lorsque la (ou les) mutation(s) nécessaire(s) se produise(nt) chez un individu, elle(s) ne peut (peuvent) pas se fixer dans la population, du simple fait du hasard ou à cause de forces sélectives antagonistes. L’ADN poubelle n’est donc probablement pas maintenu pour son utilité aujourd’hui, et encore moins, bien sûr, pour une éventuelle utilité dans un avenir indéterminé. Les organismes ne peuvent prévoir de quoi sera fait leur futur. En revanche, cet ADN constituerait une source d’adaptabilité, ou de façon plus générale une source d’« évolvabilité », qui se serait formée fortuitement, comme un produit secondaire de l’impuissance des mécanismes moléculaires à évacuer l’ADN sans fonction. Par bien des égards, il y a une forte similitude avec le maintien de la reproduction sexuée. Alors qu’il y a un avantage pour une femelle à éliminer la production de mâle et à ne produire que des femelles, chez les eucaryotes la reproduction sexuée est la norme et la reproduction asexuée l’exception [35]. La distribution phylogénétique des organismes asexués indique que ceux-ci apparaissent régulièrement, mais qu’ils ne se maintiennent pas sur le long terme, même si quelques rares contre-exemples existent [36, 37]. Des arguments théoriques et expérimentaux suggèrent que les organismes asexués ont une adaptabilité réduite du fait de la perte de la recombinaison qui permet la combinaison d’allèles favorables et l’élimination d’allèles délétères. La sélection entre lignées éliminerait les lignées asexuées, avantagées sur le court terme, mais désavantagées sur le long terme [35]. De façon analogue, un génome réduit à sa partie utile serait un avantage mineur à court terme et un désavantage à long terme, et la sélection de groupe entre lignées évolutives pourrait alors expliquer le maintien de l’ADN poubelle dans la plupart des génomes. Cette hypothèse peut être testée en étudiant l’âge des génomes exceptionnellement petits chez les plantes et les animaux. Ils devraient être pour la plupart relativement récents, à l’échelle de l’évolution de ces taxons. |
Plaidoyer en faveur de la « petite » science Rien en biologie n’a de sens, si ce n’est à la lumière de l’évolution. Cet aphorisme de Theodosius Dobzhansky est plus que jamais d’actualité. À l’heure des grands projets et lorsque les exploits technologiques et la quantité de données produites servent de mesure de la qualité, il est devenu difficile de défendre les approches reposant sur l’élaboration d’hypothèses, leurs développements formels et leurs tests expérimentaux. Force est de constater que des énormités sont régulièrement proférées pour justifier l’argent investi dans les projets « BIG » science. Seule une bonne connaissance des acquis théoriques et expérimentaux en biologie évolutive pourrait limiter ces errements répétés d’une techno-science irréfléchie, notamment les raisonnements panglossiens. Malheureusement, les universités françaises ne recrutent que peu (voire pas du tout !) d’enseignants-chercheurs ayant une solide formation en biologie évolutive. De plus, la principale agence de financement de la recherche en France n’a plus de programme spécifique pour soutenir les chercheurs travaillant dans ce domaine. Pourrons-nous encore longtemps contenir les errements des Dr Pangloss et, en bon Candide, cultiver notre jardin ? |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous tenons à exprimer ici toute notre gratitude à nos collègues Mélanie Debiais-Thibaud et Alice Michel-Salzat pour leur relecture attentive et critique de notre manuscrit, ainsi qu’à notre collègue Cushla Metcalfe, pour l’amélioration des titres et du résumé en anglais.
|
Footnotes |
1.
Pennisi E. ENCODE project writes eulogy for junk DNA . Science. 2012; ; 337 : :1159.–1161. 2.
Dunham I, Kundaje A, Aldred SF, et al. An integrated encyclopedia of DNA elements in the human genome . Nature. 2012; ; 489 : :57.–74. 3.
Rands CM, Meader S, Ponting CP, Lunter G.. 8.2% of the Human genome is constrained: variation in rates of turnover across functional element classes in the Human lineage . PLoS Genet. 2014; ; 10 : :e1004525.. 4.
Ponting CP, Hardison RC. What fraction of the human genome is functional? Genome Res. 2011; ; 21 : :1769.–1776. 5.
Ecker JR. Forum: Genomics ENCODE explained . Nature. 2012; ; 489 : :52.–53. 6.
The Alberts B.. End of small science? Science. 2012; ; 337 : :1583.. 7.
Doolittle WF. Is junk DNA bunk? A critique of ENCODE . Proc Natl Acad Sci USA. 2013; ; 110 : :5294.–5300. 8.
Eddy SR. The ENCODE project: missteps overshadowing a success . Curr Biol. 2013; ; 23 : :R259.–R261. 9.
Niu DK, Jiang L. Can ENCODE tell us how much junk DNA we carry in our genome? Biochem Biophys Res Commun. 2013; ; 430 : :1340.–1343. 10.
Palazzo AF, Gregory TR., The case for junk DNA . PLoS Genet. 2014; ; 10 : :e1004351.. 11.
Graur D, Zheng YC, Price N, et al. On the immortality of television sets: function in the Human genome according to the evolution-free gospel of ENCODE . Genome Biol Evol. 2013; ; 5 : :578.–590. 12.
Muerdter F, Stark A. Genomics: hiding in plain sight . Nature. 2014; ; 512 : :374.–375. 13.
Gerstein MB, Rozowsky J, Yan KK, et al. Comparative analysis of the transcriptome across distant species . Nature. 2014; ; 512 : :445.–448. 14.
Doolittle WF, Brunet TDP, Linquist S, Gregory TR. Distinguishing between function and effect in genome biology . Genome Biol Evol. 2014; ; 6 : :1234.–1237. 15.
Struhl K. Transcriptional noise and the fidelity of initiation by RNA polymerase II . Nat Struct Mol Biol. 2007; ; 14 : :103.–105. 16.
Ruths T, Nakhleh L.. ncDNA, drift drive binding site accumulation . BMC Evol Biol. 2012; ; 12 : :159.. 17.
Gould SJ, Lewontin RC. The spandrels of San Marco and the panglossian paradigm: a critique of the adaptationist programme . Proc R Soc Lond B. 1979; ; 205 : :581.–598. 18.
Lynch M.. The origins of genome architecture . Sunderland, Massachusetts: : Sinauer; , 2007. 19.
Kimura M.. The neutral theory of molecular evolution . New York: : Cambridge University Press; , 1983. 20.
Rice WR. Nothing in genetics makes sense except in light of genomic conflict . Annu Rev Ecol Evol Syst. 2013; ; 44 : :217.–237. 21.
De Koning APJ, Gu WJ, Castoe TA, et al. Repetitive elements may comprise over two-thirds of the Human genome . Plos Genet. 2011; ; 7 : :e1002384.. 22.
Zhang ZL, Harrison PM, Liu Y, Gerstein M. Millions of years of evolution preserved: a comprehensive catalog of the processed pseudogenes in the human genome . Genome Res. 2003; ; 13 : :2541.–2558. 23.
Jacobs FMJ, Greenberg D, Nguyen N, et al. An evolutionary arms race between KRAB zinc-finger genes ZNF91/93 and SVA/L1 retrotransposons . Nature. 2014; ; 516 : :242.–245. 24.
Hua-Van A, Le Rouzic A, Boutin TS, et al. The struggle for life of the genome’s selfish architects . Biol Direct. 2011; ; 6 : :19.. 25.
Lynch M, Conery JS. The evolutionary fate and consequences of duplicate genes . Science. 2000; ; 290 : :1151.–1155. 26.
De Souza FSJ, Franchini LF, Rubinstein M. Exaptation of transposable elements into novel cis-regulatory elements: is the evidence always strong? Mol Biol Evol. 2013; ; 30 : :1239.–1251. 27.
Casane D, Laurenti P. Syllogomanie moléculaire : l’ADN non codant enrichit le jeu des possibles . Med Sci (Paris). 2014; ; 30 : :1177.–1183. 28.
Oulion S, Debiais-Thibaud M, d’Aubenton-Carafa Y, et al. Evolution of Hox gene clusters in gnathostomes: insights from a survey of a shark (Scyliorhinus canicula) transcriptome . Mol Biol Evol. 2010; ; 27 : :2829.–2838. 29.
Oulion S, Laurenti P, Casane D. Organisation des gènes Hox : l’étude de vertébrés non-modèles mène à un nouveau paradigme . Med Sci (Paris). 2012; ; 28 : :350.–353. 30.
Pascual-Anaya J, D’Aniello S, Kuratani S, Garcia-Fernandez J.. Evolution of Hox gene clusters in deuterostomesBMC Dev Biol. 2013; ; 13 : :26.. 31.
Jordan B. Fugu story . Med Sci (Paris). 1994; ; 10 : :1154.–1156. 32.
Metcalfe CJ, Casane D.. Accommodating the load: the transposable element content of very large genomes . Mob Genet Elements. 2013; ; 3 : :e24775.. 33.
Metcalfe CJ, Filee J, Germon I, et al. Evolution of the Australian lungfish (Neoceratodus forsteri) genome: a major role for CR1 and L2 LINE elements . Mol Biol Evol. 2012; ; 29 : :3529.–3539. 34.
Kapusta A, Feschotte C. Volatile evolution of long noncoding RNA repertoires: mechanisms and biological implications . Trends Genet. 2014; ; 30 : :439.–452. 35.
De Vienne DM, Giraud T, Gouyon PH.. Lineage selection and the maintenance of sex . PLoS One. 2013; ; 8 : :e66906.. 36.
Boschetti C, Carr A, Crisp A, et al. Biochemical diversification through foreign gene expression in bdelloid rotifers . PLoS Genet. 2012; ; 8 : :e1003035.. 37.
Flot JF, Hespeels B, Li X, et al. Genomic evidence for ameiotic evolution in the bdelloid rotifer Adineta vaga . Nature. 2013; ; 500 : :453.–457. |