| |
| Med Sci (Paris). 2015 October; 31(10): 929–932. Published online 2015 October 19. doi: 10.1051/medsci/20153110020.Chroniques génomiques - Séquencer les bébés ? Bertrand Jordan1,2* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS, Espace éthique méditerranéen, hôpital d’adultes la Timone, 264, rue Saint-Pierre, 13385Marseille Cedex 05, France 2CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Analyse de mutations d'ADN, Dépistage sur goutte de sang séché, Europe, Dépistage génétique, Étude d'association pangénomique, Séquençage nucléotidique à haut débit, Humains, Nouveau-né, Dépistage néonatal, Guides de bonnes pratiques cliniques comme sujet, États-Unis d'Amérique, éthique, méthodes, normes |

Depuis que s’est confirmée la chute vertigineuse du coût du séquençage d’ADN, une idée refait périodiquement surface, celle d’une lecture intégrale du génome de chaque enfant à la naissance, afin de disposer d’un panorama complet de son patrimoine génétique. On pourrait ainsi détecter les mutations délétères dont il est éventuellement porteur et prendre les mesures indiquées, qu’il s’agisse de prévention ou de thérapie. Il s’agirait en somme d’une extension du dépistage néonatal systématique qui est actuellement pratiqué mais se limite à quelques affections. De plus, cette séquence pourrait suivre l’individu tout au long de son existence (être inscrite sur sa carte Vitale) et être utilisée à de multiples reprises, notamment pour vérifier le bien-fondé de l’emploi de certains médicaments. Comme on le verra dans cette chronique, cette éventualité parfois présentée comme toute proche soulève, en fait, un ensemble de questions techniques, organisationnelles et éthiques qui devront être explorées avant qu’elle ne devienne éventuellement une réalité. |
L’état actuel du dépistage néonatal Commençons par un panorama de la pratique actuelle du dépistage néonatal. Il est effectué, dans tous les pays développés, à partir de quelques gouttes de sang obtenues par piqûre sur le talon du bébé et soumises ensuite à un ensemble d’analyses biochimiques (souvent par spectrométrie de masse couplée ou tandem MS). La liste des affections recherchées est étonnamment variable d’une nation à l’autre. En France, elles sont au nombre de quatre : la phénylcétonurie, bien sûr, l’hypothyroïdie congénitale, l’hyperplasie des surrénales, et la mucoviscidose, plus éventuellement la drépanocytose « si l’enfant est considéré « à risque » du fait de son origine ethnique », selon les termes de l’Assurance maladie1. Mais l’étendue du dépistage est très variable en Europe puisque le nombre de maladies rares dépistées varie de un (pour le Monténégro) à 29 (pour l’Autriche) [1]. Aux États-Unis, le nombre de maladies dépistées est de 57 [2]. Les tests effectivement pratiqués varient selon les états2 : ils sont par exemple au nombre de 57 en Californie et de 47 dans le Dakota du Nord – la variation est donc minime entre ces deux états dont l’un peut être considéré comme très sophistiqué, l’autre plutôt rural et traditionnel. Les informations pour les États-Unis (données par le site indiqué plus haut) sont très complètes ; au-delà de la liste des affections (qui inclut bien sûr les quatre examinées en France et dans la plupart des états européens), on y trouve aussi le coût de ces analyses qui est modéré : 102,75 dollars pour l’ensemble en Californie et 60 pour le Dakota du Nord. Notons que le test, considéré comme une mesure de santé publique faisant partie intégrante des soins périnataux, n’est pas soumis au consentement des parents même si ceux-ci peuvent en principe le refuser ; quant au coût, il est très généralement pris en charge par l’hôpital ou l’Assurance maladie et, en pratique, plus de 99 % des enfants sont effectivement dépistés. Bien entendu, les affections inclues dans la liste (pour la plupart très rares) sont susceptibles d’un traitement efficace ou tout au moins d’une prise en charge dont l’effet est positif. Il est tout de même étonnant de constater une telle diversité dans l’étendue du dépistage, et de voir que, contrairement à ce que l’on aurait pu supposer, notre nation et son système de santé ne sont pas en pôle position sur ce point. Même si l’on peut douter de l’utilité réelle de certaines analyses, notre panoplie semble un peu restreinte : elle n’inclut pas, par exemple, les déficits immunitaires (SCID, severe combined immune disorders) qui sont recherchés en Californie (mais pas dans le Dakota du Nord). |
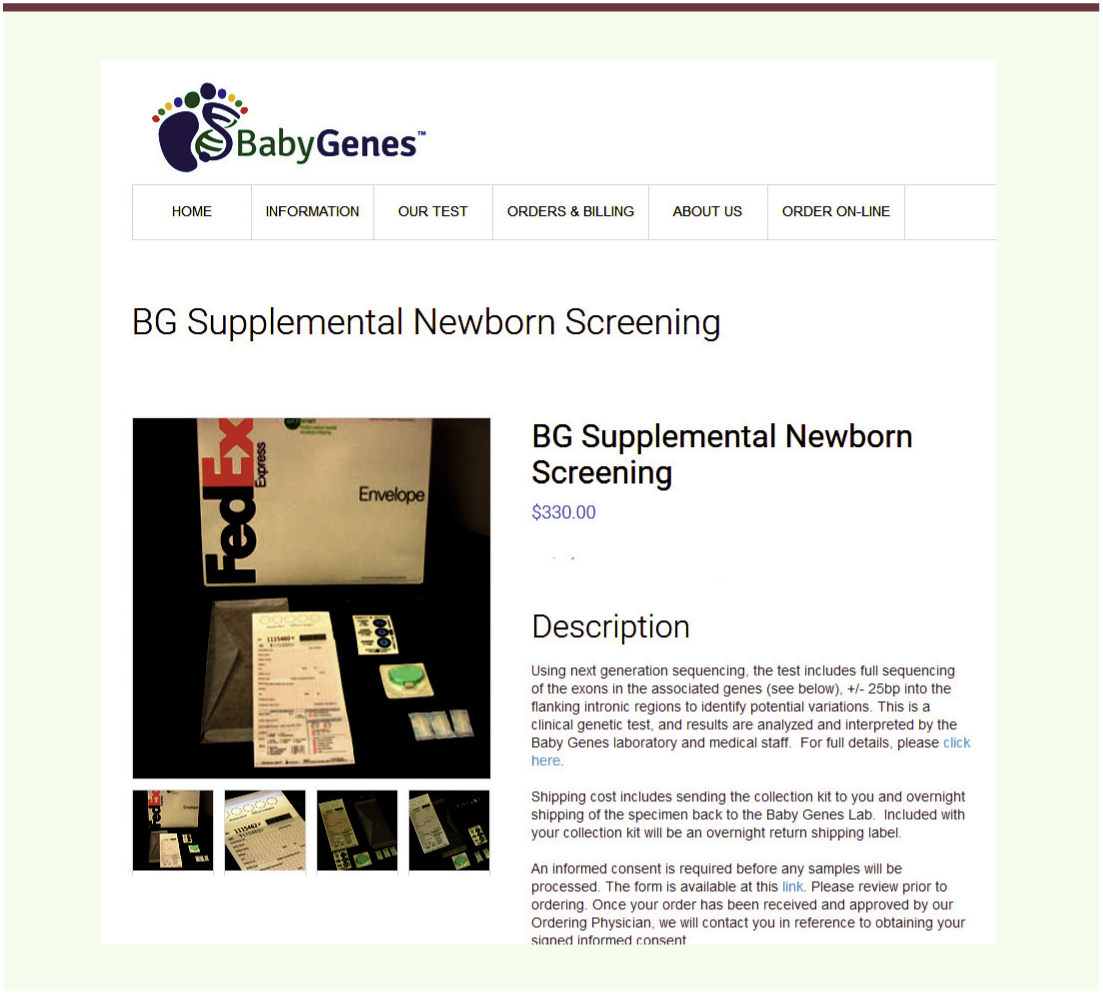
Le passage à la séquence : modalités et arguments Techniquement, il est tout à fait envisageable d’effectuer un séquençage de l’ADN du bébé à partir du sang prélevé à la naissance. Comme pour le séquençage des tumeurs, souvent commenté dans ces Chroniques [3], on a le choix entre différentes modalités : séquençage intégral (WGS, whole genome sequencing), séquençage de l’exome ou encore d’un panel plus ou moins large de gènes. On pourrait aussi procéder à un séquençage large ou même exhaustif, mais limiter l’analyse aux gènes susceptibles de porter des mutations dont l’effet pathogène justifie une intervention précoce. La séquence obtenue devrait logiquement être conservée, afin que l’on puisse la consulter au cours de la vie de la personne, par exemple pour y déceler des incompatibilités avec certains médicaments, ou des vulnérabilités à telle ou telle maladie – certaines restant à découvrir : nos connaissances vont s’accroître, mais l’ADN du nouveau-né ne changera (quasiment) pas durant son existence. Cette analyse pratiquée et interprétée à la naissance permettrait donc un dépistage plus complet, et fournirait une « carte d’identité génétique » utile tout au long de la vie et facilitant la transition vers une médecine personnalisée. Faut-il donc se lancer sans plus attendre ? Il reste en fait différents obstacles. Le coût, pour commencer : le « génome à 1 000 dollars » n’est toujours pas une réalité3 alors que le test pratiqué en Californie (57 affections) ne coûte qu’une centaine de dollars. Même un exome à visée médicale ne descend guère actuellement, avec son interprétation, au-dessous de 1 000 dollars. Et, comme pour les autres séquençages cliniques, il va se poser le problème des résultats imprévus, de leur interprétation et de leur communication, ou non, aux parents. Le consentement éclairé des parents, oublié dans le système actuel, va devenir nécessaire – et quid du consentement de l’enfant, que celui-ci n’est évidemment pas en mesure de donner ? La révélation d’une affection non apparente mais grave à terme ne risque-t-elle pas d’interférer avec l’investissement affectif des parents ? Et comment garantir la confidentialité d’une séquence d’ADN qui a vocation à être consultée à de multiples reprises ? Ces questions ne sont pas seulement théoriques : des entreprises proposent déjà de tels tests. Baby Genes, par exemple4,, propose d’analyser 89 gènes correspondant à 60 affections traitables, à partir d’une tache de sang séchée, en 96 heures et pour 330 dollars (Figure 1). Jusqu’ici, les patients paient de leur poche, et comme il s’agit d’un test pratiqué par l’entreprise elle-même (LDT, laboratory developed test), la seule exigence de la FDA (food and drug administration)5 est que le laboratoire soit agréé selon la norme CLIA (clinical laboratory improvement amendments). Il semble donc urgent de baliser le terrain avant que ces analyses ne se généralisent.…
 | Figure 1.
Le test commercialisé par Baby Genes. Noter qu’il y a intervention d’un médecin (fin du texte),… mais que ce dernier appartient à l’entreprise (extrait du site de Baby Genes). |
|
En Europe, un examen des objectifs et des modalités Un examen approfondi de ces questions a été effectué par la société européenne de génétique humaine (ESHG, european society of human genetics) en collaboration avec le comité éthique de HUGO (human genome organisation) et deux fondations (PHG foundation et p3g international paediatric platform) ; il a été publié début 2015 [4]. Cet article présente de façon très claire l’état actuel de la question, et formule un ensemble de recommandations résumées ci-dessous pour la mise en place d’un dépistage néonatal par séquençage d’ADN :
-
l’objectif doit être d’identifier des variants entraînant un risque élevé pour une affection à traiter précocement – ce qui suppose une approche ciblée (au niveau du séquençage ou de l’analyse) ;
-
la preuve de l’effet de ces variants doit être solide et appuyée par un large ensemble de données ;
-
les coûts doivent être évalués de manière réaliste en incluant les différentes étapes en amont et en aval du séquençage proprement dit ;
-
l’implication des différents acteurs (y compris les patients et leurs organisations) doit être effective pour définir les modalités du dépistage ;
-
l’information des parents doit être développée tout en cherchant à maximiser le taux de participation ;
-
l’éducation des professionnels de santé doit être développée ;
-
la conservation de la séquence en vue de son utilisation future est prématurée et doit faire l’objet d’études (utilité, risques d’identification) avant toute mise en place ;
-
les résultats non recherchés doivent être communiqués aux parents s’ils impliquent, pour l’enfant, des risques sérieux d’affections pour lesquelles un traitement existe.
Les recommandations sont bien sûr plus détaillées et nuancées que le résumé ci-dessus, et témoignent des discussions approfondies qui ont été menées tout au long de leur élaboration. Dans l’esprit du document, elles sont à envisager avant de lancer un programme de dépistage néonatal par séquençage, qui doit se justifier d’un point de vue de santé publique et privilégier l’intérêt de l’enfant. Il reste bien sûr à voir comment elles seront prises en compte dans les décisions qui interviendront au niveau national. |
Aux États-Unis, des essais grandeur nature Aux États-Unis, une approche plus pragmatique a été lancée dès 2012 par les NIH (national institutes of health), avec un appel à propositions doté de 25 millions de dollars (sur 5 ans) pour « explorer les implications, défis et opportunités d’une possible utilisation de la séquence du génome en situation néonatale »6. Quatre projets ont été retenus. Il s’agit d’études pilotes qui se proposent, sur quelques centaines de cas, de voir comment ce séquençage peut être réalisé en pratique, ce qu’en tirent les cliniciens, quelle influence cela a sur leur pratique, et comment réagissent les parents à ces informations [5]. La mise en route des projets a été longue, car il s’est avéré que la FDA imposait sa réglementation7, et leur lancement effectif n’a lieu que cette année (2015). Je vais donner quelques indications, à titre d’exemple, sur le projet « BabySeq » dirigé par Robert Green (Brigham and women’s hospital) et Alan Beggs (Boston children’s hospital), dont le démarrage est maintenant acquis [6, 7]. C’est un essai randomisé qui doit porter sur 240 nourrissons en bonne santé nés dans le premier hôpital (Brigham and women’s) et 240 nourrissons suivis en soins intensifs dans le deuxième ; dans chaque groupe, la moitié des sujets feront l’objet d’un séquençage ciblé sur 1 700 gènes associés à des affections apparaissant dans l’enfance. Ces modalités pourront évoluer au cours du projet et aller vers le séquençage de l’exome ou même de l’ensemble du génome, en fonction de l’évolution de la technologie. L’objectif est de comparer, dans chacun des deux groupes, le suivi clinique et les coûts, ainsi que les réactions des parents et la qualité de la relation parent-enfant selon qu’il y a eu ou non séquençage. Le consentement des parents est requis ; un généticien et un conseiller en génétique leur décriront les résultats des analyses de séquence, qui seront également transmis à leur pédiatre. Les gènes impliqués dans des affections apparaissant à l’âge adulte (cancer du sein, Alzheimer, etc.) ne seront pas analysés. Enfin, les parents pourront obtenir les données de séquence brute s’ils les réclament après discussion – une possibilité offerte par l’équipe avec réticence (en raison du risque d’interprétation erronée) mais au nom du droit à l’information. Ce projet est financé sur cinq ans, à hauteur de 6 millions de dollars US [6, 7]. Les trois autres projets, soutenus à un niveau équivalent, sont centrés sur différents aspects de la même question [5] : l’un étudiera exclusivement des nourrissons en soins intensifs, pour évaluer l’apport à la prise en charge d’un séquençage très rapidement effectué, le deuxième étendra l’analyse aux gènes impactant la réponse à différents médicaments et s’attachera aux aspects juridiques de ce séquençage, le troisième enfin s’efforcera de rassembler un échantillon multiculturel et s’intéressera principalement à la manière la plus efficace de communiquer les résultats aux parents et aux médecins. Au total, on a là un ensemble d’études qui vont être très utiles pour préciser l’impact réel d’un éventuel séquençage systématique des nouveau-nés, ses avantages, ses inconvénients et les meilleures manières de le mettre en œuvre s’il s’avère réellement bénéfique. |
Finalement, et malgré les progrès fulgurants de la technologie, on voit que l’introduction d’un séquençage systématique à la naissance soulève de nombreuses interrogations. Il est certes techniquement possible, et à peu près accessible financièrement ; mais de nombreuses questions subsistent sur son utilité clinique réelle, et sur de possibles effets négatifs souvent liés à la croyance erronée, mais très répandue, en un strict déterminisme génétique. Les comportements face à cette situation sont assez caractéristiques : des entreprises se lancent sur ce marché, en considérant que le consommateur tranchera et qu’il a toujours raison ; des organisation professionnelles (notamment en Europe) étudient la question et produisent des recommandations dont on peut se demander si elles seront suivies d’effet ; et les NIH lancent une série d’expérimentations très intéressantes mais qui démarrent seulement maintenant et dont les résultats risquent d’arriver un peu tard. N’oublions pas qu’entretemps la technologie va continuer à évoluer et qu’une nouvelle baisse significative des coûts reste possible, ce qui rendrait sans doute irrésistibles, pour une bonne partie du public, les offres commerciales en « libre accès »… |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Loeber JG, Burgard P, Cornel MC, et al. Newborn screening programmes in Europe: arguments and efforts regarding harmonization. Part 1. From blood spot to screening result . J Inherit Metab Dis. 2012; ; 35 : :603.–611. 2.
Kemper AR, Green NS, Calonge N, et al. Decision-making process for conditions nominated to the recommended uniform screening panel: statement of the US Department of health and human services secretary’s advisory committee on heritable disorders in newborns and children . Genet Med. 2014; ; 16 : :183.–187. 3.
Jordan B. Les retombées cliniques du séquençage de nouvelle génération . Med Sci (Paris). 2014; ; 30 : :589.–593. 4.
Howard HC, Knoppers BM, Cornel MC, et al. Whole-genome sequencing in newborn screening? A statement on the continued importance of targeted approaches in newborn screening programmes . Eur J Hum Genet. 2015; 28 janvier. doi: 10.1038/ejhg.2014.289. |