| |
| Med Sci (Paris). 32(5): 433–435. doi: 10.1051/medsci/20163205002.Étude des récepteurs membranaires par microscopie à force atomique David Alsteens1* 1Université catholique de Louvain, institut des sciences de la vie, Place croix du sud 4-5, 1348Louvain-La-Neuve, Belgique MeSH keywords: Animaux, Sites de fixation, Membrane cellulaire, Métabolisme énergétique, Humains, Microscopie à force atomique, Liaison aux protéines, Récepteurs de surface cellulaire, composition chimique, métabolisme, ultrastructure |
La microscopie à force atomique La microscopie à force atomique (AFM, atomic force microscopy) est une technique qui a fortement évolué ces trois dernières décennies [9–11] (→).
(→) Voir le Dossier technique de M.C. Giocondi et al., m/s n° 1, janvier 2003, page 92, la Synthèse de J. Bernaud et al., m/s n° 5, mai 2015, page 522 et la Nouvelle de S. Kasas et al., m/s n° 5, mai 2015, page 369
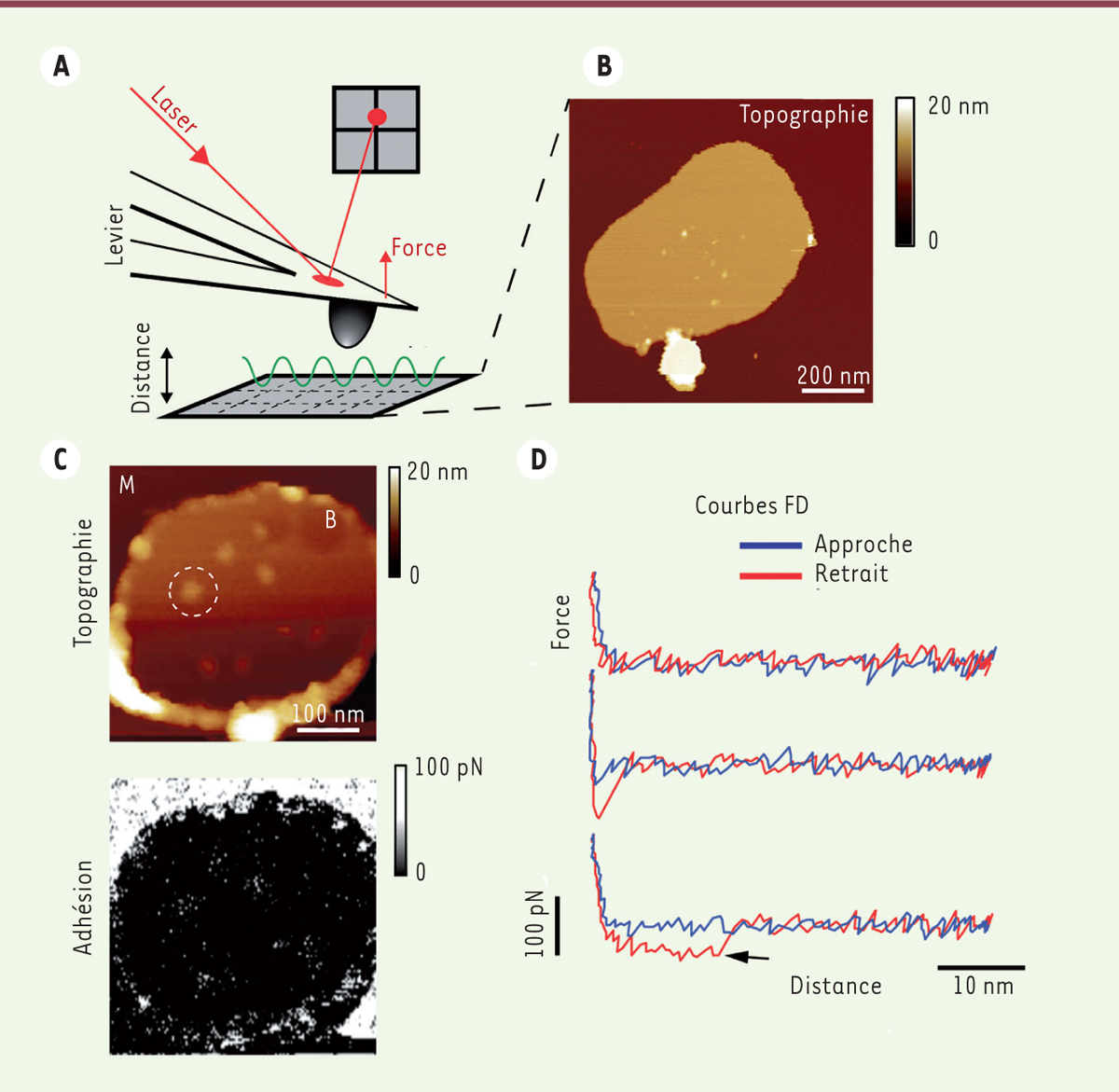
Inventé en 1986, par Binnig, Quate et Gerber, cet outil repose sur l’utilisation d’une sonde nanoscopique souple qui balaye un échantillon [1]. Les déflexions d’un levier, sur lequel est fixée une pointe, sont détectées de manière très précise via un système optique utilisant un laser se réfléchissant à l’extrémité du levier dans une photodiode (Figure 1A). La déflexion du levier est directement proportionnelle aux forces d’interactions entrant en jeu entre la pointe et l’échantillon. En balayant sa surface et en minimisant la force appliquée, le microscope dresse une cartographie point par point de cet échantillon. La résolution latérale dépend en grande partie du rayon de courbure de la pointe, qui mesure généralement entre 2 et 10 nm. Le grand avantage de ce microscope est sa capacité à mesurer des forces très faibles, avec une sensibilité de l’ordre du piconewton (pN). Combiné à l’utilisation d’un scanner piézo-électrique, permettant de déplacer l’échantillon, ou la pointe, de manière très précise, il est possible d’acquérir des images avec une résolution verticale sub-nanométrique.
 | Figure 1.
Cartographie des sites d’interactions de PAR1 à l’aide de l’AFM et de l’imagerie FD. (A) Principe de l’AFM. Une pointe fine est placée à l’extrémité d’un levier souple dont la déflexion est mesurée à l’aide d’un laser focalisé à son extrémité et réfléchi dans un photodétecteur. (B) Image topographique d’une bicouche lipidique déposée sur du mica contenant des récepteurs PAR1 isolés. (C) Image topographique et carte d’adhésion enregistrées simultanément montrant que les récepteurs PAR1 interagissent avec la pointe fonctionnalisée, portant à son apex un peptide SFLLRN. (D) Exemple de courbes FD, avec la courbe d’approche en bleu et le retrait en rouge. Au retrait de la pointe, soit la courbe ne montre pas d’adhésion (courbes du haut), soit une adhésion non-spécifique (pas d’extension du linker, courbes du milieu), ou une adhésion spécifique (courbes du bas). AFM : microscopie à force atomique ; FD : force distance ; PAR1 : protease activated receptor 1 ; SFLLRN : thrombin receptor activating peptide. |
Cette technique a fortement évolué. Elle permet maintenant d’imager des échantillons biologiques dans des conditions physiologiques (avec contrôle du milieu, du pH, de la température, de l’atmosphère, etc.) et de sonder simultanément des propriétés physiques, chimiques ou biologiques avec une résolution moléculaire, grâce à l’enregistrement concomitant de courbes « force distance » (courbes FD), aussi appelées imagerie FD. En outre, la pointe peut être modifiée à son apex et ainsi exposer des groupements chimiques bien définis ou des ligands biologiques individuels. Ceci, combiné aux récents développements liés aux mesures de courbes FD, a permis d’atteindre une nouvelle étape dans l’utilisation de l’AFM. Elle permet désormais d’imager l’architecture de systèmes biologiques plus complexes, tels que des cellules vivantes ou des membranes cellulaires, et de quantifier simultanément leurs propriétés biophysiques en conditions natives [2, 3]. Au cours des dernières années, la vitesse d’imagerie a fortement augmenté. Alors que, jusqu’à présent, l’imagerie FD était assez lente, nécessitant environ 30 min pour établir une carte de 32 pixels de côté, il est maintenant possible d’acquérir, avec la même sensibilité, une carte de 512 pixels de côté en quelques minutes. Une telle amélioration permet dès à présent d’envisager la détection et la localisation d’interactions moléculaires à très haute résolution [4, 5]. |
Imagerie de PAR1 et détection de liaisons spécifiques Dans une étude récente, nous avons montré l’intérêt de l’utilisation de l’imagerie FD pour imager à haute résolution des récepteurs individuels couplés aux protéines G (GPCR, G protein-coupled receptors) reconstitués dans des protéoliposomes1 [6, 7]. En collaboration avec le Prof. Brian Kobilka (Stanford University School of Medecine, États-Unis), nous avons imagé à haute résolution le récepteur PAR1 (protease activated receptor 1) impliqué dans l’hémostase [8] (→).
(→) Voir la Nouvelle de M. Jandrot-Perrus, m/s n° 1, janvier 2002, page 19
À la suite de sa liaison à ce récepteur, sur un site spécifique proche de l’extrémité aminoterminale, la thrombine, un facteur de coagulation, le clive, provoquant le démasquage sur PAR1 d’une nouvelle extrémité constituée du peptide SFLLRN (thrombin receptor activating peptide) qui agit comme un agoniste intramoléculaire. Afin de mimer l’activation du récepteur, nous avons greffé sur la pointe AFM un peptide orienté dont la séquence et la structure simule ce peptide actif du récepteur. Lors de la réalisation de courbes FD, le peptide est progressivement approché du récepteur. Il peut, dès lors, se lier sur son site de liaison situé sur le récepteur PAR1. Lors du retrait de la pointe, le peptide est extrait de son site et la force d’interaction est ainsi mesurée. À l’aide de l’imagerie FD, nous avons enregistré la topographie de bicouches lipidiques adsorbées sur du mica (Figure 1B). L’image hauteur, représentant la topographie de l’échantillon, permet d’observer une bicouche régulière d’une épaisseur d’environ 4,5 nm au sein de laquelle des protrusions, correspondant à des récepteurs PAR1 individuels, sont parsemées. Par l’imagerie FD, il est possible d’extraire simultanément les forces d’interactions et d’en dresser une cartographie, c’est-à-dire une carte d’adhésion (Figure 1C). Grâce à l’enregistrement simultané de la topographie et de l’adhésion, nous pouvons observer que la grande majorité des événements adhésifs sont corrélés à la présence d’un récepteur. En visualisant les courbes FD individuelles (Figure 1D), il est aussi possible de confirmer le caractère spécifique de l’interaction en observant l’extension d’un lien flexible entre la pointe et le peptide, servant ainsi de contrôle interne. En effet, on peut observer que le pic d’adhésion dû à l’extension du lien flexible entre la pointe et le peptide (
Figure 1D
; voir flèche) se situe à une distance de séparation supérieure à 5 nm. |
Analyse du paysage énergétique d’interaction L’analyse plus approfondie des courbes FD permet d’extraire des informations supplémentaires sur le paysage énergétique de la liaison. En analysant la relation entre la force de liaison et le taux de charge (c’est-à-dire l’augmentation de la force par unité de temps) appliqué sur le lien, il est possible d’extraire des paramètres décrivant le potentiel d’interaction (la distance de l’état de transition, la constante cinétique de rupture de la liaison ou l’énergie de liaison). Avec la méthode que nous proposons [6], il suffit d’une image de l’échantillon pour accéder à toutes ces données. En appliquant un mouvement sinusoïdal à la pointe, une large gamme de taux de charge peut-être explorée en un seul passage. Nous avons ainsi pu mesurer que le peptide natif interagissait avec une grande affinité pour le récepteur PAR1. En analysant la séquence du peptide et en remplaçant un par un les acides aminés le constituant, nous avons démontré que les résidus phénylalanine (F) et arginine (R) étaient très importants pour la stabilité de la liaison, leur remplacement réduisant significativement l’affinité [6]. Nous avons également étudié l’action d’un médicament, le vorapaxar2,, récemment approuvé par l’agence américaine des produits alimentaires et médicamenteux (FDA). Difficilement soluble dans l’eau, celui-ci a été incorporé dans le récepteur PAR1 lors de sa reconstitution en protéoliposome. De manière surprenante, bien que bloqué par le vorapaxar, le récepteur est toujours capable de former un lien avec le peptide agoniste mais l’affinité est cependant fortement réduite. Ce résultat démontre que le récepteur PAR1 possède au moins deux sites d’interactions pour le peptide, un de faible affinité et l’autre de haute affinité. Le vorapaxar bloque uniquement le site de liaison à haute affinité. Nous avons aussi démontré que le site à faible affinité est moins spécifique (liant de manière similaire des peptides de séquences différentes). D’autres études nous laissent penser que ce second site pourrait être situé sur la boucle extracellulaire 23 du récepteur PAR1, servant de site « d’attente » avant de pénétrer dans le site de liaison central à haute affinité. Cette diffusion nécessiterait un changement conformationnel du récepteur. Lors de cette étude, nous avons démontré que l’imagerie FD par microscopie à force atomique est un outil puissant qui permet l’imagerie à haute résolution d’un récepteur individuel dans des conditions natives et la caractérisation du paysage énergétique de sa liaison avec son agoniste. Nous avons montré qu’il était possible d’investiguer à la fois le régime cinétique et thermodynamique de liaison. Par la suite, nous souhaitons développer la technique afin de pouvoir examiner ce type d’interaction directement sur des cellules vivantes. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Binnig G, Quate CF, Gerber C. Atomic force microscope . Phys Rev Lett. 1986; ; 56 : :930.–933. 2.
Alsteens D, Dupres V, Yunus S, et al. High-resolution imaging of chemical and biological sites on living cells using peak force tapping atomic force microscopy . Langmuir. 2012; ; 28 : :16738.–16744. 3.
Alsteens D, Trabelsi H, Soumillion P, Dufrene YF. Multiparametric atomic force microscopy imaging of single bacteriophages extruding from living bacteria . Nat Commun. 2013; ; 4 : :2926.. 4.
Dufrêne YF, Martinez-Martin D, Medalsy I, et al. Multiparametric imaging of biological systems by force-distance curve-based AFM . Nat Methods. 2013; ; 10 : :847.–854. 5.
Pfreundschuh M, Alsteens D, Hilbert M, et al. Localizing chemical groups while imaging single native proteins by high-resolution atomic force microscopy . Nano Letters. 2014; ; 14 : :2957.–2964. 6.
Alsteens D, Pfreundschuh M, Zhang C, et al. Imaging G protein-coupled receptors while quantifying their ligand-binding free-energy landscape . Nat Methods. 2015; ; 12 : :845.–851. 7.
Pfreundschuh M, Alsteens D, Wieneke R, et al. Identifying and quantifying two ligand-binding sites while imaging native human membrane receptors by AFM . Nat Commun. 2015; ; 6 : :8857.. 8.
La Jandrot-Perrus M. thrombine et ses récepteurs : implications dans l’hémostase et le développement embryonnaire . Med Sci (Paris). 2002; ; 18 : :19.–22. 9.
Giocondi MC, Milhiet PE, Lesniewska E, Le Grimellec C. Microscopie à force atomique : de l’imagerie cellulaire à la manipulation moléculaire . Med Sci (Paris). 2003; ; 19 : :92.–99. 10.
Bernaud J, Castelnovo M, Muriaux D, Faivre-Moskalenko C. Microscopie à force atomique pour l’étude du cycle viral . Med Sci (Paris). 2015; ; 31 : :522.–528. 11.
Kasas S. Petar Stupar P, Giovanni Longo G, Dietler G. Détecter la vie grâce à la microscopie à force atomique . Med Sci (Paris). 2015; ; 31 : :369.–371. |