| |
| Med Sci (Paris). 32(5): 519–522. doi: 10.1051/medsci/20163205022.Chroniques génomiques - 23andMe et le mal des transports Bertrand Jordan1,2* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS, Espace éthique méditerranéen, hôpital d’adultesla Timone, 264, rue Saint-Pierre, 13385Marseille Cedex 05, France 2CoReBio PACA, case901, parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Commerce, Industrie pharmaceutique, Dépistage génétique, Étude d'association pangénomique, Humains, Modèles économiques, Mal des transports, Participation du patient, Polymorphisme de nucléotide simple, Autorapport, méthodes, tendances, épidémiologie, génétique |
Un remède basique contre le mal des transports1.
Lequel d’entre nous n’a pas souffert, à un moment ou un autre – et notamment durant l’enfance – de ce mal des transports, de ces nausées qui ont gâché bien des promenades en voiture et encore plus de traversées ? Nous connaissons souvent des personnes particulièrement sensibles à ce désagrément, qui pâlissent dès qu’elles mettent le pied sur un bateau, et, en effet, il a été démontré que la sensibilité au mal des transports présente une composante génétique notable. Une étude de jumeaux, publiée il y a une dizaine d’années [1], montrait une concordance2 de 57 % pour les jumeaux homozygotes et de 17 % seulement pour les hétérozygotes, ce qui aboutit à une héritabilité estimée à 53 %. Un article émanant de l’entreprise 23andMe [2] présente maintenant les résultats d’une étude GWAS (genome-wide association study) qui cherche à découvrir les déterminants génétiques de cette sensibilité – et qui identifie trente-cinq snip (single nucleotide polymorphisms) associés à ce phénotype. L’objectif de la présente chronique est d’analyser cet article et, à travers cet exemple, la stratégie mise au point par cette firme pour utiliser son large pool de clients « consentants » (Opt-in customers) à des fins d’études génétiques. Rappelons que 23andMe, entreprise fondée en 2006, vendait directement aux consommateurs et pour 999 dollars (au début) des analyses génétiques portant sur 500 000 snip, interprétées en termes d’ascendance et de facteurs de risque pour une centaine d’affections. Fin 2013 (le prix du test était alors descendu à 99 dollars), la FDA (food and drug administration) lui interdisait de commercialiser ces informations médicales, considérées comme non validées et potentiellement dangereuses [3, 4]. 23andMe a néanmoins poursuivi son activité en commercialisant uniquement les résultats en termes d’ascendance (et en perdant la moitié de son chiffre d’affaires…). Récemment, la FDA a autorisé l’inclusion de certaines données médicales (essentiellement les mutations trouvées à l’état hétérozygote dans les gènes d’affections mendéliennes), et le prix du test est remonté à 199 dollars.

|
Une analyse GWAS assez spéciale mais convaincante L’étude récemment publiée [2] cherche donc à identifier les composantes génétiques de la propension au mal des transports. 23andMe, au fil des années, a analysé le profil génétique de plus d’un million de clients, et a constamment eu le souci de les associer à d’éventuelles études génétiques, en leur proposant un formulaire de consentement éclairé avec la possibilité de retrait à tout moment et pour chaque étude, et en les tenant systématiquement au courant des résultats obtenus dans le cadre de ses programmes de recherche. Bien entendu les données sont agrégées et dé-identifiées de manière à préserver – autant que faire se peut – l’anonymat de chacun3, : du coup, 80 % environ des clients acceptent de fournir les informations phénotypiques demandées par l’entreprise. L’étude sur le mal de transports a porté sur un peu plus de 80 000 personnes (toutes d’ascendance européenne, et non apparentées), qui ont répondu à un questionnaire détaillé et dont les profils génétiques avaient déjà été établis grâce à des microarrays interrogeant de 500 000 à 1 000 000 snip. Le Manhattan plot
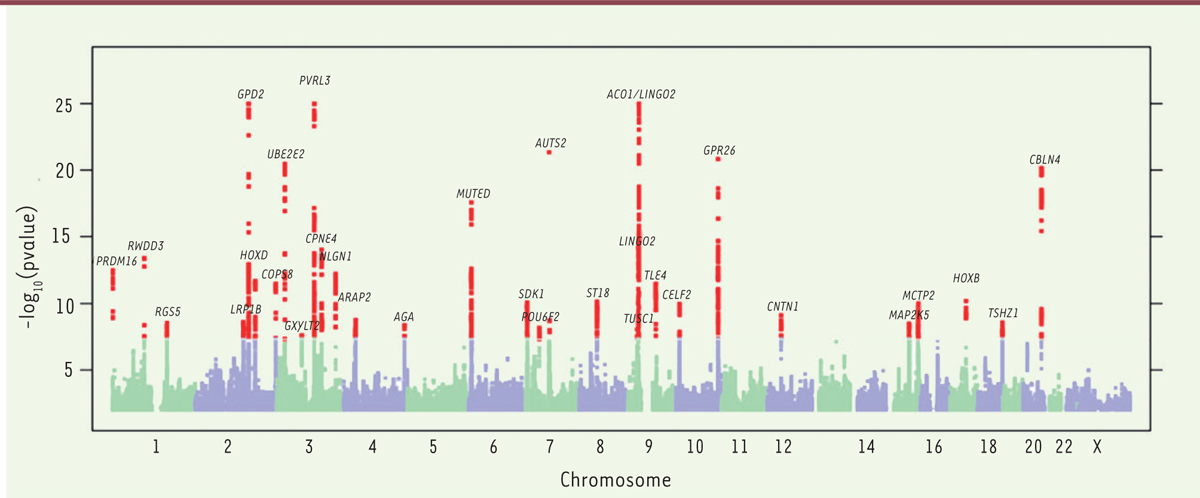
4 obtenu (Figure 1) est assez clair et la plupart des 35 pics correspondant aux signaux d’association se détachent nettement du bruit de fond.
 | Figure 1.
Manhattan plot des signaux d’association observés. Les pics significatifs (probabilité de faux positif inférieure à 5x10 −8) sont indiqués en rouge. Les gènes situés à proximité des snip (polymorphismes) en cause sont indiqués en noir ; leur implication est vraisemblable mais non prouvée (figure 1 de [ 2], Open Access). |
On peut alors examiner les gènes situés à proximité des snip ainsi repérés. Il n’est bien sûr pas prouvé qu’ils sont impliqués dans le phénotype – il faudrait pour cela de longues études complémentaires – mais cela reste relativement probable. Le signal le plus fort correspond à un snip situé à une mégabase du gène PVRL3
5, dont la perte d’expression provoque des défauts oculaires ; deux des suivants (MUTED et TSHZ1) sont impliqués dans l’équilibre et dans le développement de l’oreille interne. D’autres snip sont proches de gènes (HOXD, TLE4) intervenant dans le développement précoce, d’autres encore sont proches de gènes impliqués dans la mise en place du système nerveux et notamment dans la formation des synapses (CBLN4, NLGN1, SDK1, etc.). On considère généralement que le mal des transports est lié à un conflit d’informations entre le système vestibulaire (qui indique que l’on est en mouvement) et le système visuel (qui montre que la personne est immobile par rapport au véhicule) : il est donc vraisemblable que des variants de gènes liés à l’oreille interne, au système visuel et au cerveau puissent jouer sur la sensibilité au mal des transports. Quelques autres snip significativement associés se trouvent au voisinage de gènes intervenant dans la régulation du glucose et de l’insuline (GPD2, GPR23, etc.). Ces associations, ainsi que le reste des 35 observées, sont plus difficiles à interpréter, mais on voit que globalement les signaux ressortant de cette étude d’association sont nets et que beaucoup d’entre eux pointent vers des gènes pour lesquels une interprétation biologique est assez vraisemblable. Disposant des réponses détaillées des participants, les auteurs ont pu également étudier les associations entre différents caractères phénotypiques et montrer une forte corrélation positive entre la propension au mal des transports et la tendance aux étourdissements, au mal d’altitude et aux maux de tête après consommation de vin, et des corrélations négatives avec la qualité de sommeil (les bons dormeurs sont moins malades), le fait d’être fumeur ou même la ponctualité.… Bien sûr, cela confine à la génétique récréative, mais on imagine facilement des recherches (sur des maladies mentales par exemple) pour lesquelles de telles indications s’avéreraient précieuses. |
Le modèle participatif de 23andMeEn réalité l’entreprise développe déjà cette approche depuis quelque temps. J’avais exprimé mon scepticisme sur la qualité de données recueillies par simple questionnaire auprès des clients [5], mais les résultats acquis aujourd’hui (au-delà de l’article analysé dans cette Chronique) valident assez largement la démarche. Il y a quelque temps déjà, un article démontrait la réplication de plus de 180 associations génétiques déjà connues à partir des profils génétiques et phénotypiques de 20 000 clients, pour des caractères binaires allant de la couleur des yeux au psoriasis et au diabète de type 1 [6]. Des publications récentes montrent des résultats convaincants pour trois types d’allergie [7] et pour le risque de thrombose [8] : à chaque fois, plusieurs associations déjà connues sont retrouvées et de nouvelles sont découvertes. Il faut noter que ces travaux peuvent être réalisés très rapidement et à peu de frais, contrairement aux études GWAS classiques : les participants (cas et contrôles) sont déjà présents dans la base de données de l’entreprise, avec leurs échantillons d’ADN ainsi que leurs profils génétiques détaillés ; leurs données médicales, souvent déjà répertoriées, peuvent être précisées lors de la demande de participation que leur envoie 23andMe (et qu’ils acceptent à 80 %) ; il ne reste plus qu’à faire tourner les programmes d’analyse et qu’à écrire l’article. On note d’ailleurs que les articles publiés par 23andMe ne comportent qu’une ou deux dizaines d’auteurs, alors que ce nombre frise parfois la centaine pour les études GWAS classiques. Aujourd’hui, avec la puissance des outils de la génomique, c’est l’assemblage de milliers de participants de phénotype connu qui représente le principal goulet d’étranglement pour de telles études, et l’entreprise a réussi à construire une base de plus d’un million de clients participants, réactifs, génotypés et ayant déjà fourni des informations phénotypiques. Ces participants qu’elle peut recontacter presque en temps réel (bien sûr, tous les échanges se font sur Internet) lui donnent une incontestable force de frappe. Selon sa directrice, Anne Wojcicki6, 23andMe a dans sa base 240 000 porteurs de l’allèle APOE e4 (facteur de risque pour la maladie d’Alzheimer), 75 000 malades du cancer, 188 000 malades cardiovasculaires …et 350 millions de données phénotypiques. Chaque client fournit en moyenne plus de 80 indications lorsqu’il accède à son compte. Anne Wojcicki ambitionne de passer à des dizaines, peut-être des centaines de millions de personnes, et de se lancer dans la mise au point de médicaments, tout au moins au niveau de l’identification de cibles. Rappelons que c’est pour la même raison (l’existence d’un « stock » de nombreux clients déjà génotypés, dont l’ADN et le phénotype sont accessibles) que 23andMe a pu conclure de juteux contrats avec des entreprises pharmaceutiques. Elle les autorise, pour quelques dizaines de millions de dollars, à « piocher » dans sa base de données et à sélectionner (sur la base de leur profil de snip et de leur phénotype) quelques milliers de patients et de témoins dont l’ADN sera séquencé dans le cadre d’une recherche de cible pour des médicaments en développement [5]. L’entreprise n’a apparemment pas eu trop de difficulté à obtenir l’accord de ses clients pour de telles études ; a fortiori, leur consentement pour des travaux plus désintéressés comme celui que je rapporte ici semble largement acquis. On imagine qu’en France les clients réclameraient sans doute une part du gâteau7, cela manifeste sans doute une nette différence de mentalité mais montre aussi que 23andMe a su établir un rapport de confiance avec ses clients (en les prévenant de tout usage de leurs données, en leur donnant toute possibilité de refus, et en les tenant au courant des résultats obtenus), ce qui aujourd’hui fait sa force. Bien entendu l’approche n’est pas sans défauts : pour certains phénotypes requérant une analyse biochimique précise ou un diagnostic clinique standardisé, les souvenirs et les impressions du client risquent d’être insuffisants ; la population concernée est assez particulière, majoritairement de nationalité états-unienne, « caucasienne » et appartenant à la classe moyenne-supérieure, donc pas très représentative de la diversité humaine. |
Une génétique participative ? Selon Anne Wojcicki, 23andMe instaure une nouvelle manière de faire de la recherche en génomique, mettant en pratique l’un des quatre « P » (Participative) de la « Médecine 4P » vantée par Lee Hood [9] : une implication poussée du public qui, non content de payer pour une analyse génétique, accepte de fournir « à la demande » des informations sur son état de santé, avec pour seule contrepartie l’impression de participer à la recherche et, éventuellement, l’obtention d’informations nouvelles sur son risque de développer telle ou telle affection. Le fait qu’au centre du système se trouve une entreprise qui en retire d’importants bénéfices – souvenons-nous que le contrat avec Genentech était évalué à 60 millions de dollars [5] – ne doit pas nous faire oublier que cette modalité contourne, et même résout, une très importante difficulté : la constitution des grands ensembles de patients et témoins nécessaires à l’obtention de données fiables pour les affections complexes. Rappelons qu’il a fallu presque 40 000 cas et plus de 110 000 témoins pour avoir enfin des résultats fiables en GWAS pour la schizophrénie [10]. On aurait certes souhaité que cette contribution du public à l’avancement des recherches se fasse dans un cadre associatif plutôt que commercial – mais il faut reconnaître l’intelligence des responsables de 23andMe qui ont probablement conçu dès le départ leur business model dans cette optique [11] et ont réussi à en montrer la pertinence. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Reavley CM, Golding JF, Cherkas LF, et al. Genetic influences on motion sickness susceptibility in adult women: a classical twin study . Aviat Space Environ Med. 2006; ; 77 : :1148.–1152. 2.
Hromatka BS, Tung JY, Kiefer AK, et al. Genetic variants associated with motion sickness point to roles for inner ear development, neurological processes and glucose homeostasis . Hum Mol Genet. 2015; ; 24 : :2700.–2708. 3.
Jordan B. Le réveil de la Food and Drug Administration . Med Sci (Paris). 2014; ; 30 : :43.. 4.
Jordan B. Grandes manœuvres autour des profils génétiques en libre accès . Med Sci (Paris). 2014; ; 30 : :227.–228. 5.
Jordan B. 23andMe ou comment (très bien) valoriser ses clients . Med Sci (Paris). 2015; ; 31 : :447.–449. 6.
Tung JY, Do CB, Hinds DA, et al. Efficient replication of over 180 genetic associations with self-reported medical data . PLoS One. 2011; ; 6 : :e23473.. 7.
Hinds DA, McMahon G, Kiefer AK, et al. A genome-wide association meta-analysis of self-reported allergy identifies shared and allergy-specific susceptibility loci . Nat Genet. 2013; ; 45 : :907.–911. 8.
Hinds DA, Buil A, Ziemek D, et al. Genome-wide association analysis of self-reported events in 6,135 individuals and 252,827 controls identifies 8 loci associated with thrombosis . Hum Mol Genet. 2016 Feb 9. pii: ddw037. 9.
Hood L, Friend SH Predictive, personalized, preventive, participatory (P4) cancer medicine . Nat Rev Clin Oncol. 2011; ; 8 : :184.–187. 10.
Schizophrenia working group of the Psychiatric genomics consortium . Biological insights from 108 schizophrenia-associated genetic loci . Nature. 2014; ; 511 : :421.–427. 11.
Dorfman R. Falling prices and unfair competition in consumer genomics . Nat Biotechnol. 2013; ; 31 : :785.–786. |