| |
| Med Sci (Paris). 32(6): 640–645. doi: 10.1051/medsci/20163206029.Le cas CRISPR, mutations « ready-made » et évolution lamarckienne d’un système immunitaire adaptatif Didier Casane1,2 and Patrick Laurenti1,2* 1Laboratoire évolution, génomes, comportement, écologie, CNRS université Paris-Sud, UMR9191, IRD UMR247, avenue de la Terrasse, 91198Gif-sur-Yvette, France 2Université Paris-Diderot, Sorbonne Paris-Cité, Paris, France |
CRISPR-Cas permet de classer les biologistes en trois catégories qui ne sont pas mutuellement exclusives. Pour la plupart d’entre nous, c’est un acronyme synonyme de vagues de pourriels qui encombrent nos boîtes de courriers électroniques tant les publicités sont abondantes pour vanter les mérites des diverses compagnies qui commercialisent, sous ce nom, un système de mutagénèse dirigée. Pour beaucoup de biologistes, c’est en effet une nouvelle approche à la fois efficace, rapide et d’un coût raisonnable pour modifier des gènes, en particulier chez des espèces non-modèles [1] (→).
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n°12, décembre 2014, page 1066
Ce saut qualitatif de la maîtrise de la mutagénèse dirigée soulève beaucoup d’espoirs, en particulier dans le domaine de la thérapie génique [2, 3] (→), mais aussi beaucoup de craintes et d’interrogations éthiques [4] (→).
(→) Voir la Synthèse de J.P. Tremblay et la Chronique génomique de B. Jordan, m/s n°11, novembre 2015, pages 1014 et 1035
(→) Voir la Chronique génomique de B. Jordan, m/s n°6-7, juin-juillet 2015, page 691
Enfin, pour quelques biologistes, c’est un système d’immunité adaptative chez les procaryotes qui possède des caractéristiques extraordinaires quand on l’examine à la lumière des théories de l’évolution biologique [5, 6]. C’est dans cette dernière perspective que nous discuterons ici le système CRISPR-Cas. Deux théories furent développées au xix
e siècle pour rendre compte de la modification des espèces au cours du temps. Le transformisme de Lamarck s’opposa le premier au fixisme1 qui prévalait depuis l’antiquité en proposant que l’évolution des espèces reposât sur des modifications du phénotype induites par le milieu et le mode de vie des organismes (voir
Encadré
[7]). À l’inverse, l’évolutionnisme de Darwin proposa que les modifications héritables du phénotype se produisissent au hasard, c’est-à-dire indépendamment des effets bénéfiques ou délétères qui en découlent [8]. Dans ce cadre, les modifications des espèces au cours du temps ne sont pas dues à la modification des individus, mais à la modification des proportions des différentes formes qui coexistent dans les populations. L’avènement de la génétique au xx
e siècle porta un coup fatal au transformisme lamarckien en établissant l’antériorité de la mutation sur la sélection [9]. Dès lors, la biologie se développa dans le cadre conceptuel de la théorie de l’évolution de Darwin et le transformisme de Lamarck ne persista que dans le cadre relictuel du néo-lamarckisme français [10] ou dans celui, plus tragique, du lyssenkisme2 russe [11]. Beaucoup de concepts lamarckiens sont totalement caduques et, à ce jour, aucune étude de l’organisation et de l’évolution des génomes, ni de celles des relations entre le génotype et le phénotype, n’a permis d’identifier de mécanisme universel de « rétro-ingénierie » permettant aux organismes d’extraire de l’information liée à leur état ou au milieu, de la stocker dans leur génome, et de la transmettre à leur descendance. Une évolution lamarckienne peut toutefois être envisagée s’il y a une hérédité épigénétique de certains caractères, mais un tel processus reste à ce jour considéré comme très marginalement impliqué dans l’évolution à long terme. Il est certes indéniable que des états de caractères induisent des modifications épigénétiques différentes des cellules germinales qui sont transmises des parents aux descendants sans être codées dans l’ADN. De même, des traits culturels sont héritables sans être codés par l’ADN [12, 43] (→). Mais soulignons qu’il s’agit de modes d’évolution peu fréquents comparativement à ceux intégrés à la théorie de l’évolution depuis Darwin. En effet, la biologie évolutive a intégré de nombreux concepts nouveaux, comme notamment la prépondérance des effets du hasard ; et ce, non seulement au niveau de l’apparition des mutations, mais aussi au niveau de leur fixation ou de leur perte au cours du temps [13], de la complexification des systèmes biologiques [14, 15], voire de la formation de novo de nouveaux gènes [16] (→).
|
Lois de Lamarck Première Loi. Dans tout animal qui n’a point dépassé le terme de ses développements, l’emploi plus fréquent et soutenu d’un organe quelconque, fortifie peu à peu cet organe, le développe, l’agrandit, et lui donne une puissance proportionnée à la durée de cet emploi ; tandis que le défaut constant d’usage de tel organe, l’affaiblit insensiblement, le détériore, diminue progressivement ses facultés, et finit par le faire disparaître.
Deuxième Loi. Tout ce que la nature a fait acquérir ou perdre aux individus par l’influence des circonstances où leur race se trouve depuis longtemps exposée, et, par conséquent, par l’influence de l’emploi prédominant de tel organe, ou par celle d’un défaut constant d’usage de telle partie ; elle le conserve par la génération aux nouveaux individus qui en proviennent, pourvu que les changements acquis soient communs aux deux sexes, ou à ceux qui ont produit ces nouveaux individus.
Jean-Baptiste de Lamarck, Philosophie zoologique, 1809
|
(→) Voir la Synthèse de C. Junien et al., m/s n°1, janvier 2016, page 34
(→) Voir le Forum de D. Casane et P. Laurenti, m/s n°12, décembre 2014, page 1177
La résistance du lamarckisme contre l’hégémonie de la théorie actuelle de l’évolution (que nous qualifierons ici de darwinienne) est aujourd’hui un combat essentiellement hors de la sphère scientifique. Il s’agit en grande partie de résistances d’ordres religieux ou politique à une conception matérialiste de l’évolution qui ne propose aucune direction, aucune prévisibilité aux chemins empruntés par l’histoire évolutive de l’ensemble des organismes vivants, et donc aucun but vers lequel tendrait celle de l’homme [17]. Dans un cadre strictement scientifique, la discorde s’est focalisée essentiellement sur un point : existe-t-il des mutations qui se produisent préférentiellement quand elles apportent un avantage sélectif [18] ? Ce processus est souvent qualifié de lamarckien (bien que lié de façon très ténue aux idées de Lamarck), car il permet de réintroduire non seulement une relation plus étroite entre la mutation et l’adaptation à l’environnement, mais aussi une certaine prévisibilité, une direction dans la marche de l’évolution (Figure 1). Par le passé, des mécanismes lamarckiens furent plusieurs fois proposés, mais finalement toujours rejetés [19]. Ce n’est que très récemment que fut identifié pour la première fois un mécanisme permettant l’apparition de mutations préférentiellement utiles : le système immunitaire adaptatif CRISPR-Cas des bactéries et des archées [20].
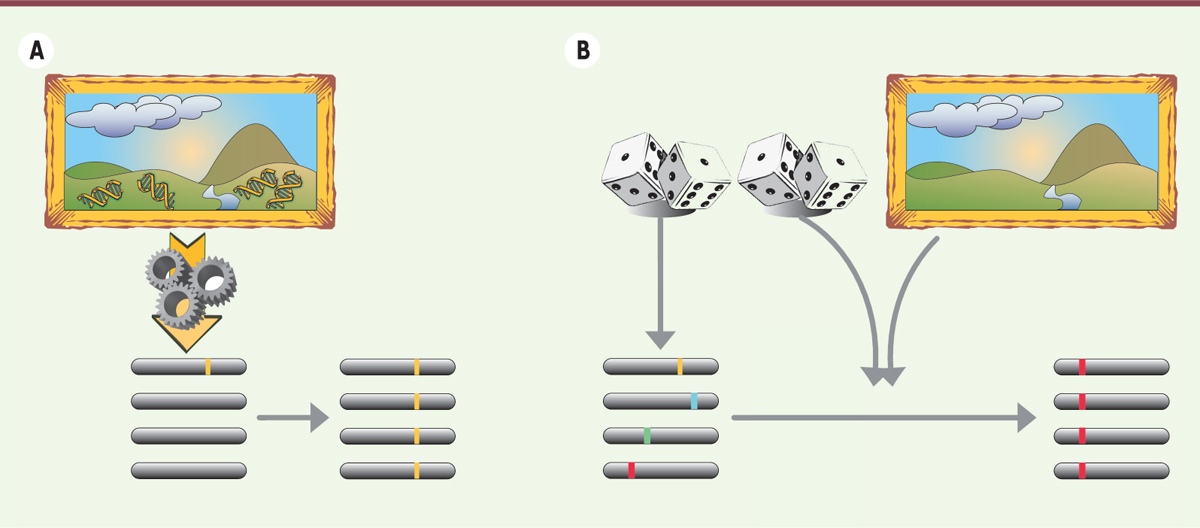
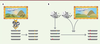 | Figure 1.
Deux modes d’évolution. A. Modèle lamarckien : par un processus de rétro-ingénierie utilisant l’information relative à l’environnement ou par extraction directe de l’ADN de cet environnement, une mutation utile à l’adaptation (favorable) est générée et se propage dans la population. B. Modèle darwinien : la diversité est générée indépendamment de l’effet favorable, neutre ou délétère des mutations. En fonction de l’intensité de la sélection, qui dépend des valeurs sélectives des différents génotypes et de la taille de la population, la mutation qui se fixe dans la population n’est pas nécessairement une mutation favorable. En effet, le hasard qui a un rôle très important dans les petites populations peut conduire à la disparition d’une mutation favorable ou à la fixation d’une mutation délétère. En revanche, dans une grande population, une mutation favorable aura une plus grande probabilité de se fixer (modifiée d’après Koonin et al. [ 5]). |
|
CRISPR-Cas : un système immunitaire adaptatif chez les procaryotes L’organisation et le fonctionnement du système CRISPR-Cas diffèrent de façon très importante selon les lignées procaryotes considérées [20–25]3, mais le principe général de fonctionnement est relativement stable et nous considèrerons ici, à titre d’exemple, le système tel qu’il a été décrit chez Escherichia coli. Un « protospacer » correspondant à un fragment d’ADN exogène, souvent de virus ou de plasmide, est intégré à l’extrémité du locus CRISPR entre deux séquences identiques. Ce locus CRISPR est transcrit en un long pré-ARNcr (pré-ARN CRISPR), puis ce transcrit est découpé en différents ARNcr correspondant à différents fragments d’ADN exogènes. Ces ARNcr, associés à des protéines Cas (CRISPR associated gene), vont constituer des unités de surveillance à l’intérieur du cytoplasme et, lors d’infections ultérieures, ils s’associeront aux séquences d’ADN exogènes complémentaires et les détruiront (Figure 2). Ainsi, l’intégration d’un fragment du génome d’un pathogène dans son propre génome permet à la cellule d’acquérir une résistance accrue à ce même pathogène si elle entre à nouveau en contact avec celui-ci. C’est donc bien un système immunitaire adaptatif dans la mesure où il est modifié au cours du temps pour être plus efficace en fonction des pathogènes qui ont infecté la cellule [26]. Dans la suite de cette revue, nous nous concentrerons sur ce rôle des systèmes CRISPR-Cas dans l’immunité. Signalons que, de plus, ces systèmes ont été impliqués récemment dans diverses autres fonctions comme la régulation de l’expression des gènes, la régulation des comportements de groupe, la régulation de la virulence virale, la régulation des éléments génétiques mobiles, la réparation de l’ADN, la dynamique des génomes, voire la dormance cellulaire et l’apoptose [27].
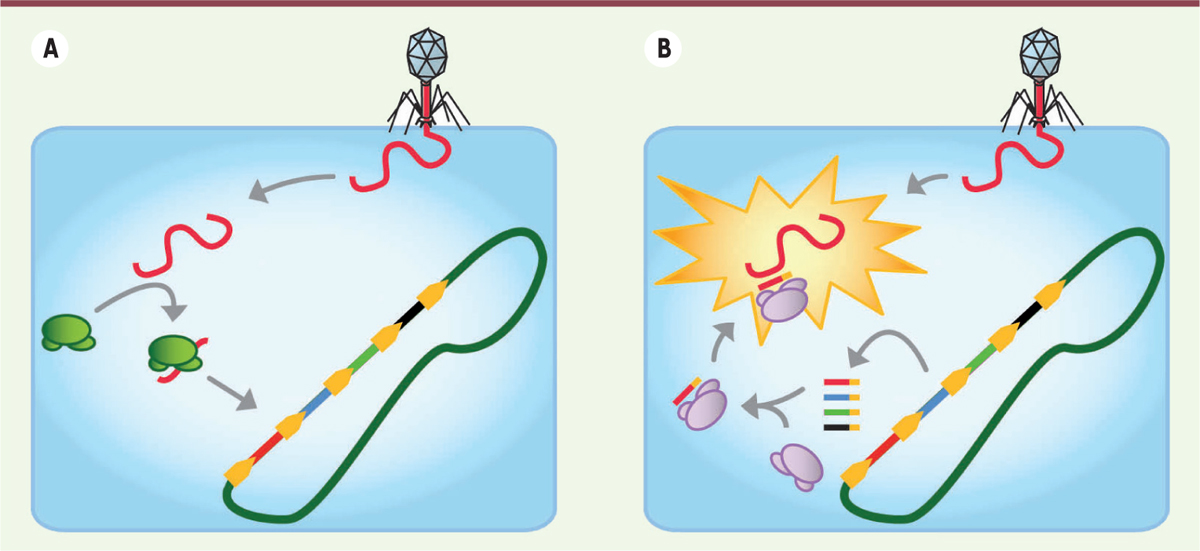
 | Figure 2.
Les étapes clés du fonctionnement du système immunitaire adaptatif CRISPR-Cas des procaryotes. A. Primo-infection : un fragment d’ADN du phage, le « protospacer », est découpé pour constituer un nouveau « spacer » qui est intégré entre deux séquences répétées au locus CRISPR. B. Contact ultérieur : l’association d’un ARN, transcrit du « spacer », avec des protéines Cas constitue un complexe moléculaire permettant l’identification et la destruction de l’ADN du phage. |
|
Origine du système CRISPR-Cas Hormis les séquences exogènes intégrées dans le génome, le système CRISPR-Cas implique d’une part des séquences répétées entre lesquelles les séquences exogènes s’intègrent et d’autre part un ensemble de protéines qui interviennent dans les différentes étapes nécessaires à la mise en place d’une protection efficace, soit : (1) l’acquisition et l’intégration dans le génome de fragments d’ADN exogènes, « les spacers » ; (2) le clivage du transcrit en ARNcr ; (3) l’association d’un ARNcr avec l’ADN exogène ; et (4) la destruction du génome exogène (Figure 2). Quand, dans quelle lignée et à partir de quels éléments, ces constituants du système CRISPR-Cas ont-ils été produits ? Ce sont là des questions auxquelles il est très difficile de répondre tant les systèmes qui ont été identifiés sont différents, en particulier pour ce qui est des protéines Cas. Par ailleurs, les gènes codant ces protéines évoluent si rapidement qu’il est très difficile d’identifier ceux qui sont homologues, c’est-à-dire hérités d’un ancêtre commun. Il a toutefois été proposé que la machinerie constituant le système CRISPR-Cas ancestral serait apparue chez des archées thermophiles, puis qu’elle se serait répandue dans d’autres lignées d’archées et de bactéries par transferts horizontaux [28]. Ce système a ensuite évolué dans des directions très variées, tant au niveau des étapes qui constituent le processus d’acquisition de l’immunité, que des gènes codant les protéines constituant la machinerie moléculaire. Les protéines Cas nécessaires pour manipuler l’ADN ont des activités de type nucléase, hélicase et polymérase. Il a été proposé que les gènes codant les protéines Cas1 et Cas9 auraient pour origine des gènes portés par des transposons qui sont des experts en découpage et intégration de fragments d’ADN [21, 22]. Il est frappant de constater que la recombinaison V(D)J, qui permet de créer une grande diversité de récepteurs des cellules T et d’immunoglobulines chez les gnathostomes (vertébrés à mâchoires), repose sur les protéines Rag (recombination activating gene) 1 et Rag2 qui descendent de gènes portés par un transposon [29]. Ainsi, et de façon indépendante, des systèmes immunitaires adaptatifs très différents auraient été bricolés à partir des capacités de manipulations de l’ADN codées par des transposons [23]. |
Évolution lamarckienne du locus CRISPR Le système CRISPR-Cas est un système immunitaire adaptatif, c’est-à-dire qu’il permet à une bactérie d’acquérir une protection dirigée contre un pathogène particulier avec lequel elle a été en contact. Comme cette protection repose sur l’insertion d’une séquence d’ADN du pathogène dans son propre génome, elle est transmissible à sa descendance. Cette modification du génome est avantageuse dans l’environnement dans lequel la bactérie vit à un moment donné. Ce processus d’adaptation suit assez précisément un schéma lamarckien si la modification du génome n’est pas aléatoire mais dirigée précisément afin d’augmenter la valeur sélective de l’individu (Figure 3). Des bactéries ont été identifiées, chez lesquelles l’intégration de spacers d’origine endogène (ADN de la bactérie) ou exogène (ADN de virus et plasmides) n’est pas biaisée. Dans ce cas, l’évolution de la résistance aux agents infectieux se fait de façon strictement darwinienne et au prix de la mort d’un très grand nombre de bactéries [6, 30]. Mais, chez d’autres bactéries, des mécanismes moléculaires limitent l’intégration de « spacers » inutiles voire nocifs, comme, par exemple, des fragments du génome de la cellule elle-même [31]. Cette évolution lamarckienne de la composante « spacer » du locus CRISPR est possible pour deux raisons. D’une part, parce qu’il n’y a pas de séparation entre cellules germinales et cellules somatiques chez les procaryotes et donc toute modification adaptative est transmise à la descendance. Chez un organisme multicellulaire, ce n’est pas aussi simple car une transformation de cellules somatiques doit conduire à, au moins, une mutation dans une cellule germinale pour avoir quelque chance d’être transmise à la descendance. D’autre part, les procaryotes ayant la capacité d’intégrer des fragments d’ADN présents dans le milieu [32], ils peuvent en extraire de l’information génétique à forte valeur adaptative sous une forme « ready-made »4. Dans le cas du système CRISPR-Cas, c’est un fragment d’ADN du pathogène qui est intégré. Il n’est donc pas nécessaire qu’il y ait un mécanisme de « rétro-ingénierie » permettant à la bactérie d’extraire une information du milieu et de la transformer en ADN pour la stocker dans son génome, avant de la transmettre à sa descendance.
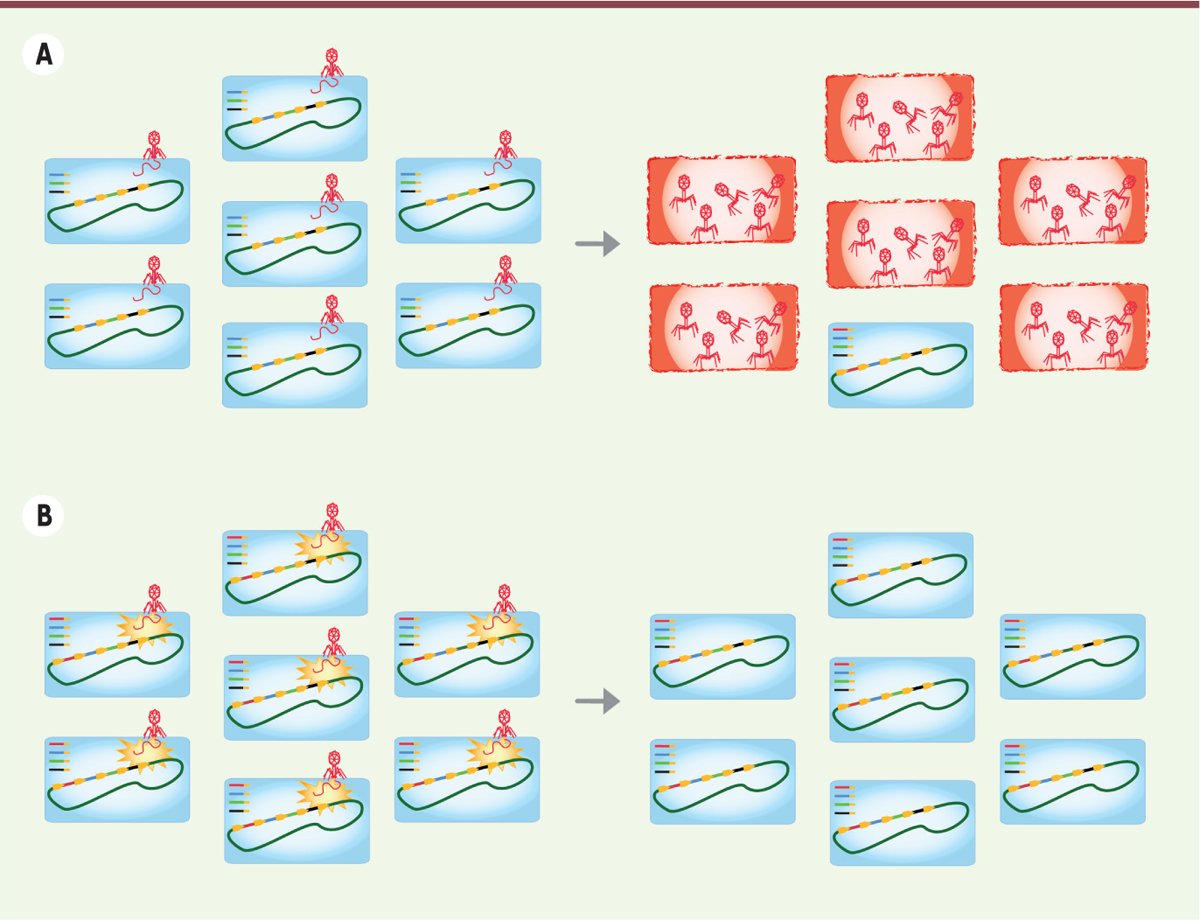
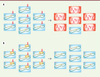 | Figure 3.
Effet d’un phage sur une population de bactéries. A. Bactéries naïves : si un phage prolifère dans une population de bactéries ne possédant pas de « spacer » correspondant à ce phage, la plupart des bactéries seront détruites, mais si au moins une bactérie intègre un fragment du phage dans son génome, elle sera immunisée et pourra constituer une population en présence de ce même phage. B. Bactéries immunisées : la population de bactéries n’est pas sensible au phage (modifié d’après Koonin et al. [ 5]). |
|
Pluralité des mécanismes évolutifs Dans l’état actuel des connaissances, un processus réellement lamarckien semble donc limité aux organismes unicellulaires ou aux lignées germinales des organismes multicellulaires, et doit impliquer le transfert d’information génétique « ready-made », donc d’ADN présent dans l’environnement. Seule l’évolution des « spacers » du locus CRISPR-Cas présente ces caractéristiques d’assez près pour être qualifiée de lamarckienne, même si, dans le détail, cette affirmation peut être discutée [33]. C’est toutefois un exemple isolé et il nous semble peu probable que beaucoup d’autres exemples soient découverts dans le futur. Les concepts actuels de la biologie évolutive ne nous paraissent donc pas en passe d’être profondément modifiés. Pourtant la renaissance d’un néo-lamarckisme de portée générale et la revitalisation de l’orthogenèse5 sont régulièrement annoncées dans le cadre d’études très médiatisées de l’hérédité épigénétique, mais la plupart des évolutionnistes considèrent que l’impact de l’hérédité épigénétique est négligeable sinon nul sur l’évolution à long terme de la grande majorité des caractères [34]6. Toutefois, il est également vrai que d’autres exemples de mutations induites par l’environnement chez les bactéries et les archées, mais aussi chez des eucaryotes, constituent des phénomènes qui peuvent être qualifiés de « quasi-lamarckiens ». Les transferts horizontaux de gènes appartiennent à cette catégorie. Les bactéries ont en effet la capacité d’échanger des gènes par l’intermédiaire de plasmides, de phages ou par simple transformation [32]. Cet ADN peut être ensuite intégré au génome. Il y a donc un lien direct entre la modification du génome et l’environnement, source de l’ADN intégré. Ce mécanisme permet, par exemple, la propagation rapide de résistances aux antibiotiques dans une multitude d’espèces non-apparentées sans réinvention récurrente de ces résistances. Toutefois, nous ne savons pas si les bactéries intègrent préférentiellement de l’ADN utile à l’adaptation à l’environnement ; par ailleurs, l’analyse massive d’un grand nombre de génomes montre que les transferts horizontaux de gènes sont une source « ready-made » d’information génétique qui ne concerne essentiellement que les organismes procaryotes [32]. Les transferts horizontaux de gènes ne participent donc que marginalement à la dynamique des génomes des eucaryotes actuels. Les mutations induites par le stress sont également un mécanisme d’évolution qualifiable de quasi-lamarckien. Dans des conditions de stress, c’est-à-dire dans un environnement sous optimal pour la survie de bactéries, celles-ci mettent en œuvre des mécanismes moléculaires qui augmentent le taux de mutation [19, 35]. Ce mécanisme n’est qu’apparemment lamarckien car, si les mutations sont bien induites par l’environnement, celles nécessaires à l’adaptation à cet environnement ne sont pas préférentiellement produites. Il y a seulement une élévation générale du taux de mutation qui augmente la probabilité d’apparition de mutations favorables, au prix de l’augmentation du taux de mutations délétères. Mais la sélection permet, dans un second temps, la fixation des mutations favorables et la disparition de celles qui sont délétères [36, 37]. Des cycles complexes d’apparition de bactéries produisant beaucoup de mutations dans des environnements perturbés suivis de la fixation de bactéries produisant moins de mutations dans des environnements stabilisés peuvent ainsi se mettre en place [38]. Chez les animaux, il a été identifié un système contrôlant la prolifération des transposons dans les lignées germinales qui dépend de petits ARN (piARN ou PIWI-interacting RNA) [39, 40]. Ces ARN sont souvent codés par des clusters de séquences dans le génome et ils correspondent aux transposons les plus actifs, c’est-à-dire aux transposons qui ont les effets potentiellement délétères les plus importants. Il y a donc, au cours de l’évolution, une accumulation de séquences de piARN qui dépend de l’« environnement » en transposons actifs. Dans le cas du système CRISPR-Cas, comme dans celui des piARN, l’évolution des séquences est très rapide et on ne trouve pas les mêmes « spacers », ou les mêmes piARN, chez des espèces pourtant proches. Ce résultat indique que l’environnement, c’est-à-dire les plasmides et les phages auxquels sont exposées les cellules bactériennes, ou les transposons actifs dans les cellules eucaryotes, se modifie rapidement, et que les génomes des hôtes doivent évoluer en conséquence [41]. Les génomes des virus et des plasmides évoluent en réponse, ce qui constitue un exemple de mécanisme de coévolution bien documenté [41, 42]. En conclusion, si la très grande majorité des processus évolutifs identifiés à ce jour est représentée par des mécanismes strictement darwiniens, il n’en existe pas moins une minorité qui relève de mécanismes plus ou moins fortement lamarckiens. Ce sont des processus rares, ce qui les rend d’autant plus précieux pour affiner notre connaissance des diverses modalités de l’évolution. Ces phénomènes qui reposent sur des mécanismes élucidés aux niveaux biochimique, génétique et évolutif sont aujourd’hui intégrés dans la théorie de l’évolution [33]. Il est donc assez déconcertant, et pour tout dire regrettable, de constater que ces questions scientifiques passionnantes et les observations qui démontrent la pluralité des mécanismes de l’évolution, soient l’occasion d’annonces tonitruantes de la fin de la théorie de l’évolution. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous tenons à exprimer ici toute notre gratitude à Cécile Combeau et Claude Gérin pour leur relecture attentive et critique de notre manuscrit, ainsi qu’à notre collègue Cushla Metcalfe, pour l’amélioration du titre et du résumé en anglais.
|
Footnotes |
1.
Gilgenkrantz H. La révolution des CRISPR est en marche . Med Sci (Paris). 2014; ; 30 : :1066.–1069. 2.
Tremblay JP. CRISPR, un système qui permet de corriger ou de modifier l’expression de gènes responsables de maladies héréditaires . Med Sci (Paris). 2015; ; 31 : :1014.–1022. 3.
Jordan B. CRISPR-Cas9, une nouvelle donne pour la thérapie génique . Med Sci (Paris). 2015; ; 31 : :1035.–1038. 4.
Jordan B. Thérapie génique germinale, le retour ? Med Sci (Paris). 2015; ; 31 : :691.–695. 5.
Koonin EV, Wolf YI. Is evolution Darwinian or/and Lamarckian? Biol Direct. 2009; ; 4 : :42.. 6.
Koonin EV, Wolf YI. Just how Lamarckian is CRISPR-Cas immunity: the continuum of evolvability mechanisms . Biol Direct. 2016 ; :11.. 7.
de Lamarck JB. Philosophie zoologique . Paris: : Dentu; , 1809. 8.
Darwin CR. On the origin of species by means of natural selection, or the preservation of favoured races in the struggle for life . London: : John Murray; , 1859. 9.
Luria SE, Delbruck M. Mutations of bacteria from virus sensitivity to virus resistance . Genetics. 1943; ; 28 : :491.–511. 10.
Loison L. French roots of French Neo-Lamarckisms, 1879–1985 . J Hist Biol. 2011; ; 44 : :713.–744. 11.
Medvedev J. Grandeur et chute de Lyssenko . Paris: : Gallimard; , 1971. 12.
Danchin E, Charmantier A, Champagne FA, et al. Beyond DNA: integrating inclusive inheritance into an extended theory of evolution . Nat Rev Genet. 2011; ; 12 : :475.–486. 13.
Kimura M. Evolutionary rate at molecular level . Nature. 1968; ; 217 : :624.–626. 14.
Lukes J, Archibald JM, Keeling PJ, et al. How a neutral evolutionary ratchet can build cellular complexity . Iubmb Life. 2011; ; 63 : :528.–537. 15.
Stoltzfus A. On the possibility of constructive neutral evolution . J Mol Evol. 1999; ; 49 : :169.–181. 16.
Casane D, Laurenti P. Syllogomanie moléculaire: l’ADN non codant enrichit le jeu des possibles . Med Sci (Paris). 2014; ; 30 : :1177.–1183. 17.
Lecointre G. Les sciences face aux créationnismes . Versailles: : Quæ; , 2011. 18.
Foster PL. Adaptive mutation: the uses of adversity . Annu Rev Microbiol. 1993; ; 47 : :467.–504. 19.
Foster PL. Stress-induced mutagenesis in bacteria . Crit Rev Biochem Mol Biol. 2007; ; 42 : :373.–397. 20.
Makarova KS, Grishin NV, Shabalina SA, et al. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action . Biol Direct. 2006; ; 1 : :7.. 21.
Chylinski K, Makarova KS, Charpentier E, Koonin EV. Classification and evolution of type II CRISPR-Cas systems . Nucleic Acids Res. 2014; ; 42 : :6091.–6105. 22.
Krupovic M, Makarova KS, Forterre P, et al. Casposons: a new superfamily of self-synthesizing DNA transposons at the origin of prokaryotic CRISPR-Cas immunity . BMC biol. 2014; ; 12 : :36.. 23.
Koonin EV, Krupovic M. Evolution of adaptive immunity from transposable elements combined with innate immune systems . Nat Rev Genet. 2015; ; 16 : :184.–192. 24.
Koonin EV, Makarova KS. CRISPR-Cas: evolution of an RNA-based adaptive immunity system in prokaryotes . RNA Biol. 2013; ; 10 : :679.–686. 25.
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems . Nat Rev Microbiol. 2015; ; 13 : :722.–736. 26.
Marraffini LA. CRISPR-Cas immunity in prokaryotes . Nature. 2015; ; 526 : :55.–61. 27.
Westra ER, Buckling A, Fineran PC. CRISPR-Cas systems: beyond adaptive immunity . Nat Rev Microbiol. 2014; ; 12 : :317.–326. 28.
Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems . Biol Direct. 2011; ; 6 : :38.. 29.
Kapitonov VV, Koonin EV. Evolution of the RAG1-RAG2 locus: both proteins came from the same transposon . Biol Direct. 2015; ; 10 : :20.. 30.
Wei Y, Terns RM, Terns MP. Cas9 function and host genome sampling in Type II-A CRISPR-Cas adaptation . Genes Dev. 2015; ; 29 : :356.–361. 31.
Levy A, Goren MG, Yosef I, et al. CRISPR adaptation biases explain preference for acquisition of foreign DNA . Nature. 2015; ; 520 : :505.–510. 32.
Koonin EV, Makarova KS, Aravind L. Horizontal gene transfer in prokaryotes: Quantification and classification . Annu Rev Microbiol. 2001; ; 55 : :709.–742. 33.
Weiss A. Lamarckian Illusions . Trends Ecol Evol. 2015; ; 30 : :566.–568. 34.
Heard E, Martienssen RA. Transgenerational epigenetic inheritance: myths and mechanisms . Cell. 2014; ; 157 : :95.–109. 35.
MacLean RC, Torres-Barcelo C, Moxon R. Evaluating evolutionary models of stress-induced mutagenesis in bacteria . Nat Rev Genet. 2013; ; 14 : :221.–227. 36.
Labat F, Pradillon O, Garry L, et al. Mutator phenotype confers advantage in Escherichia coli chronic urinary tract infection pathogenesis . FEMS Immunol Med Microbiol. 2005; ; 44 : :317.–321. 37.
Bjedov I, Tenaillon O, Gerard B, et al. Stress-induced mutagenesis in bacteria . Science. 2003; ; 300 : :1404.–1409. 38.
Taddei F, Radman M, MaynardSmith J, et al. Role of mutator alleles in adaptive evolution . Nature. 1997; ; 387 : :700.–702. 39.
Iwasaki YW, Siomi MC, Siomi H. PIWI-Interacting RNA: its biogenesis and functions . Annu Rev Biochem. 2015; ; 84 : :405.–433. 40.
Siomi MC, Sato K, Pezic D, Aravin AA. PIWI-interacting small RNAs: the vanguard of genome defence . Nat Rev Mol Cell Biol. 2011; ; 12 : :246.–258. 41.
Andersson AF, Banfield JF. Virus population dynamics and acquired virus resistance in natural microbial communities . Science. 2008; ; 320 : :1047.–1050. 42.
Bondy-Denomy J, Garcia B, Strum S, et al. Multiple mechanisms for CRISPR-Cas inhibition by anti-CRISPR proteins . Nature. 2015; ; 526 : :136.–139. 43.
Junien C, Panchenko P, Fneich S, et al. Épigénétique et réponses transgénérationnelles aux impacts de l’environnement : des faits aux lacunes . Med Sci (Paris). 2016; ; 32 : :35.–44. |