Les cellules souches sont des cellules le plus souvent indifférenciées ayant la capacité de produire d’autres types cellulaires tout en s’auto-renouvelant. Elles sont présentes chez tous les animaux et jouent un rôle central non seulement dans l’embryogenèse, mais également dans la production des gamètes, l’homéostasie des tissus, la régénération ou les nombreux modes de reproduction asexuée que l’on observe à travers l’arbre phylogénétique des métazoaires (comme le bourgeonnement, la fission, etc.) [1]. Par définition, les cellules souches n’existent pas chez les organismes unicellulaires, ce qui implique que leur acquisition est intimement liée à l’apparition de la multicellularité chez un ancêtre des métazoaires. Dès lors, comprendre les bases génétiques de l’acquisition des cellules souches peut nous aider à comprendre celles de l’émergence et de l’évolution des animaux.
Les éponges forment le groupe frère de tous les autres métazoaires (appelés eumétazoaires) [2, 3] et occupent à ce titre une position clé pour comprendre les premières étapes évolutives des animaux [15] (→).
(→) Voir la Perspective de D. Aubert, m/s n° 5, mai 2016, page 497
Depuis 2010, le séquençage du génome de trois espèces d’éponges et des transcriptomes d’une dizaine d’autres a révélé la présence de la plupart des familles et sous-familles de gènes de développement connus chez l’homme ou la drosophile [4]. Ces approches ont également révélé certaines homologies entre le plan d’organisation des éponges et celui des eumétazoaires. Dans notre étude [5], nous nous sommes intéressés à l’origine des cellules souches en cherchant à reconstruire le répertoire de gènes ancestralement exprimés par ces cellules chez les animaux. Pour cela nous avons identifié des gènes homologues exprimés par les cellules souches d’éponges et celles d’autres lignées, afin de déterminer les innovations génétiques ayant accompagné l’acquisition de ce type cellulaire il y a 700 millions d’années.
Ephydatia fluviatilis est une éponge d’eau douce appartenant au groupe des démosponges. Elle se reproduit principalement de façon asexuée, lorsque l’éponge-mère produit des milliers de gemmules (des sphères mesurant un demi-millimètre de diamètre, enveloppées d’un squelette siliceux) contenant des centaines de cellules en dormance [6]. Lorsqu’une gemmule se sépare de l’éponge-mère et trouve un substrat approprié, les cellules en dormance migrent en dehors de la sphère et prolifèrent activement. On les appelle alors archéocytes. Ce sont des cellules souches totipotentes qui s’auto-renouvellent tout en produisant les différents types cellulaires contribuant à former une éponge juvénile fonctionnelle. Les archéocytes peuvent également former la lignée germinale femelle (les ovocytes) et entrent donc dans la catégorie de ce que l’on appelle les unlimited primordial stem cells (uPriSC) [7]. Les spermatozoïdes, quant à eux, proviennent de la transdifférenciation d’un autre type cellulaire, les choanocytes. Ceux-ci sont munis d’un flagelle entouré d’une collerette de microvillosités ayant pour rôle de créer un courant d’eau et de capturer les particules alimentaires. Les éponges contiennent également divers types cellulaires incluant, par exemple, plusieurs types de cellules épithéliales, des cellules formant le squelette siliceux, des cellules contrôlant le flux d’entrée d’eau ou encore des cellules immunitaires [6].
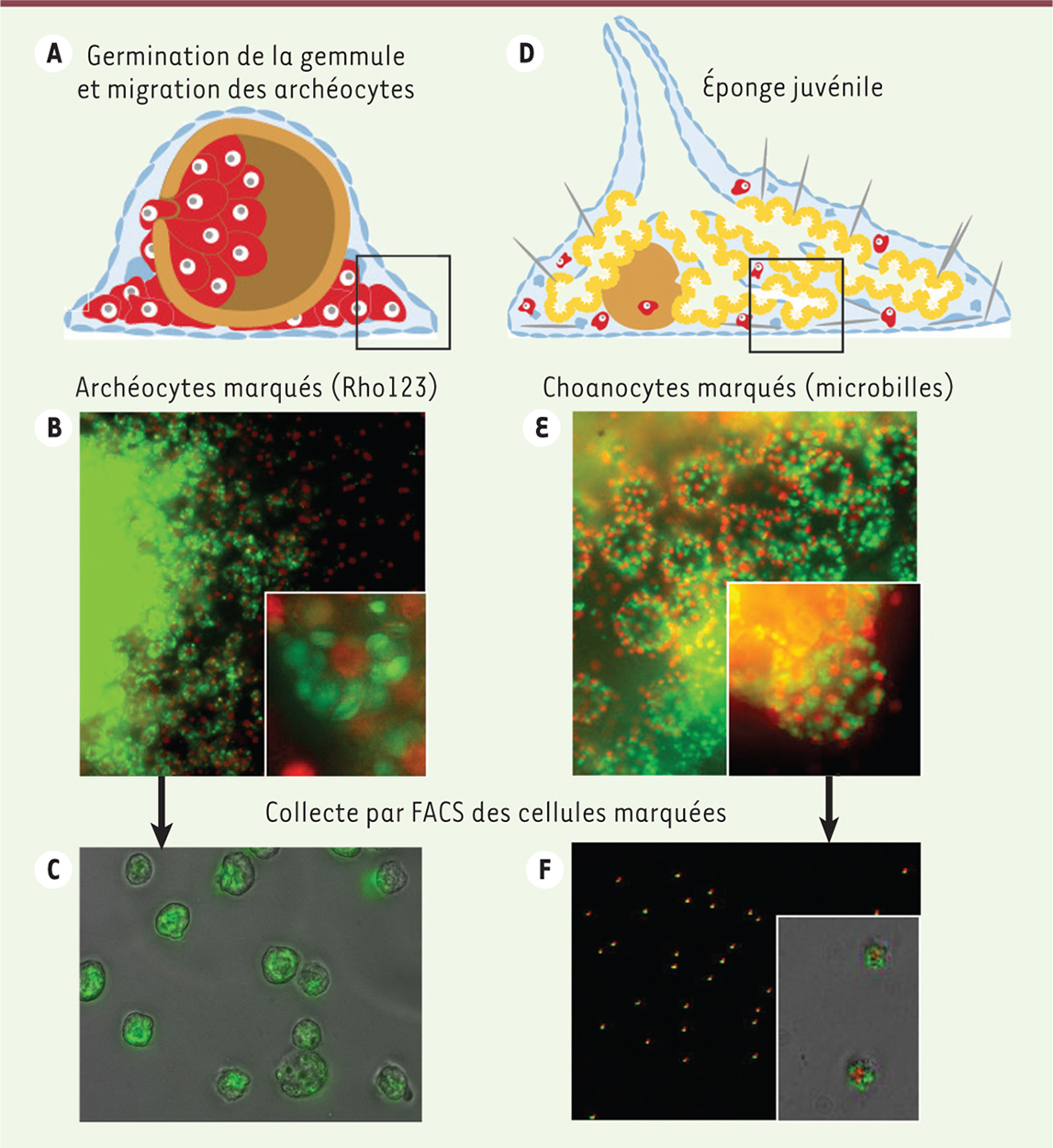
Durant la première étape de notre étude, nous avons obtenu une fraction pure d’archéocytes. Ceci a été rendu possible grâce à un marquage différentiel des différents types de cellules souches. Les archéocytes peuvent en effet être spécifiquement rendus fluorescents lorsque les éponges sont incubées en présence d’un marqueur mitochondrial, la rhodamine 123 (Rho123), qui marque les plaquettes vitellines des archéocytes. Les choanocytes peuvent, eux, être spécifiquement marqués en nourrissant les éponges avec des microbilles fluorescentes qui sont ingérées comme s’il s’agissait de particules alimentaires. Les autres types cellulaires ne sont pas marqués par ces deux traitements. En utilisant le tri cellulaire par FACS1, (fluorescence-activated cell sorting), nous avons donc pu isoler trois fractions cellulaires à partir d’éponges marquées : des archéocytes à partir d’éponges marquées à la Rho123, des choanocytes à partir d’éponges « nourries » avec des microbilles, et des cellules de types divers, correspondant aux cellules non fluorescentes, à partir d’éponges marquées par les deux traitements simultanés. Nous avons extrait instantanément les ARN messagers contenus dans ces fractions et les avons quantifiés par séquençage ARN. Nous avons ainsi identifié les gènes spécifiquement exprimés par les archéocytes. Nous avons ensuite cherché des gènes orthologues2, surexprimés dans les uPriSC d’autres lignées de métazoaires en utilisant les transcriptomes d’uPriSC publiés pour le cnidaire3 Hydra vulgaris [8] et la planaire4 Schmidtea mediterranea [9]. Nous avons ainsi reconstruit un répertoire de 180 gènes dont l’expression est conservée dans les uPriSC de métazoaires (Figure 1).
| Figure 1.
Purification des archéocytes et des choanocytes de l’éponge Ephydatia fluviatilis. A. Schema representant une gemmule en cours de germination et les archeocytes (en rouge) migrant hors de la gemmule. B. Region peripherique d’une eponge au stade represente en A, dont les archeocytes ont ete marques par la rhodamine 123 (les plaquettes vitellines fluorescent en vert) et par du Hoechst (les noyaux fluorescent en rouge). C. Archeocytes recoltes apres tri des cellules par FACS, toutes les cellules presentent de nombreuses plaquettes vitellines fluorescentes. D. Schema representant une eponge juvenile cinq jours apres germination, la plupart des types cellulaires sont en place, y compris les nombreux choanocytes organises en chambres choanocytaires (carre noir). E. Plusieurs chambres choanocytaires montrant les choanocytes marquees par des microbilles fluorescentes (vertes) et du Hoechst (noyau marque en rouge). F. Choanocytes recoltes apres tri des cellules par FACS. FACS : fluorescence-activated cell sorting. |
Comme nous l’attendions, ce répertoire contient une large proportion de gènes impliqués dans la réplication ou la réparation de l’ADN. De façon plus surprenante, il ne contient que trois facteurs de transcription, ce qui suggère que la grande majorité des facteurs de transcription associés aux cellules souches chez les animaux ne le sont pas depuis leur ancêtre commun. Par exemple, les facteurs de transcription ayant la capacité d’induire les cellules souches pluripotentes de mammifères – Sox2 (SRY [sex determining region Y]-box 2), Nanog et Oct4 (octamer-binding transcription factor 4) – n’ont pas d’orthologues en dehors des mammifères (pour Nanog et Sox2) ou des bilatériens5 (pour Oct4), ce qui implique que leur rôle dans le maintien des cellules souches est une innovation des mammifères, et non une caractéristique ancestrale. Néanmoins, les archéocytes d’Ephydatia sur-expriment les facteurs de transcription GATA, p53, Glis (GLI-similar family zinc finger) et Myc, dont les orthologues ont des fonctions connues dans les cellules souches de mammifères. Il est donc possible que les archéocytes d’éponges partagent avec les mammifères un réseau régulateur de l’expression des gènes, que l’on ne retrouve pas dans les uPriSC d’Hydra ou de Schmidtea. L’étude fonctionnelle de ces gènes chez les éponges est une voie très prometteuse pour comprendre les bases de la régulation génétique des cellules souches chez les animaux.
Contrairement aux facteurs de transcription, les régulateurs post-transcriptionnels sont très largement représentés dans le répertoire ancestral des uPriSC et comptent parmi les gènes les plus fortement exprimés. On y trouve par exemple des hélicases de la famille des DDX (DEAD-box proteins) qui régulent la traduction et divers aspects du métabolisme des ARN messager, ainsi que des gènes codant des protéines à domaine RRM (RNA recognition motif), impliquées dans l’épissage alternatif. Le rôle important des régulateurs post-transcriptionnels commence à peine à être compris dans les cellules souches de mammifères [10] mais nos résultats tendent à prouver que ce mode de régulation génétique est ancestralement utilisé par les cellules souches. Il est probable que la régulation post-transcriptionnelle de l’expression des gènes représente un avantage pour les cellules souches car le maintien ou la sortie de l’état pluripotent, et l’engagement dans telle ou telle voie de différenciation cellulaire, sont des phénomènes qui requièrent une régulation précise et coordonnée des gènes exprimés par la cellule. En régulant leurs gènes au niveau de la traduction, les cellules souches sont capables de modifier plus rapidement le répertoire de protéines qu’elles expriment. Chez les mammifères, des régulateurs post-transcriptionnels sont regroupés au sein de régulons6 qui coordonnent le métabolisme des ARNm durant le cycle cellulaire et la réponse inflammatoire [11, 12]. Dans le cas de la formation d’une éponge juvénile, une telle coordination est peut-être nécessaire pour que les archéocytes puissent construire en quelques jours une éponge fonctionnelle et organisée (Figure 2).
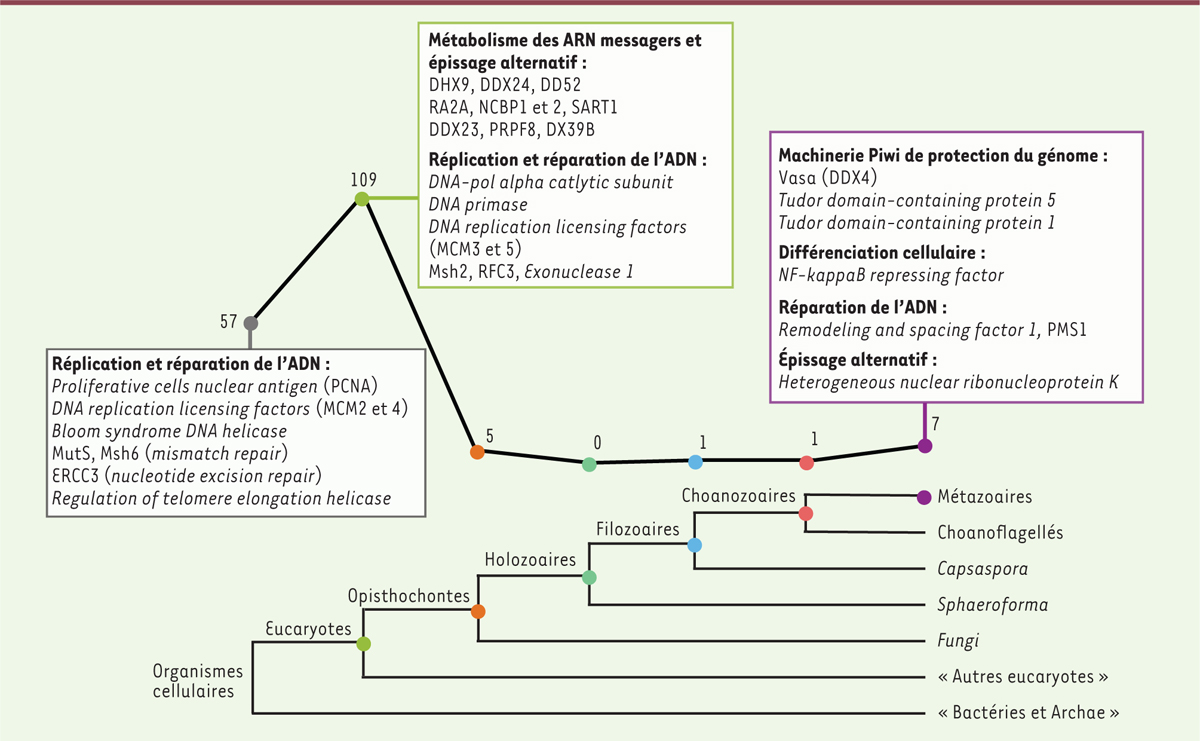
| Figure 2.
Profil phylogénétique des 180 gènes de la boîte à outils moléculaire ancestrale des cellules souches. En bas, un arbre phylogénétique du vivant montrant les noeuds auxquels les gènes ont été assignés. En haut, les chiffres représentent le nombre de gènes acquis à chaque noeud de l’arbre. Quelques gènes sont donnés en exemple. |
Pour comprendre comment la boîte à outils moléculaire ancestrale des cellules souches a été assemblée, nous avons retracé l’origine des 180 gènes la constituant, en cherchant les orthologues de ces gènes à travers l’arbre du vivant. Il apparaît que les gènes régulant le cycle cellulaire sont d’origine très ancienne : on en trouve des orthologues jusque chez les bactéries et les archae. Les régulateurs post-transcriptionnels, quant à eux, sont pour la plupart hérités d’un ancêtre eucaryote. La majorité des composants de la boîte à outils ancestrale des cellules souches sont donc des gènes plus anciens que les cellules souches elles-mêmes, ayant été recrutés chez un ancêtre commun des animaux. Seuls 7 des 180 gènes ont été acquis en même temps que les cellules souches chez un ancêtre des animaux. Parmi ces 7 gènes, on trouve les gènes Vasa, Tdrd1 (Tudor domain-containing 1) et Tdrd5 qui sont des composants d’une machinerie spéciale de protection du génome, la machinerie Piwi [13, 14] (→).
(→) Voir la Synthèse de S. Muller et al., m/s n° 5 mai 2013, page 487
D’autres gènes spécifiques des métazoaires ont un rôle central dans cette machinerie et sont sur-exprimés dans les archéocytes, incluant Piwi lui-même, Tdrd9 et Tdrkh (Tudor and KH domain-containing). La machinerie Piwi est notamment impliquée dans la destruction d’éléments génétiques mobiles (les transposons) qui perturbent l’intégrité du génome. Or, protéger le génome est fondamental pour les uPriSC puisqu’elles produisent des cellules différenciées tout au long de la vie d’un organisme, mais également la lignée germinale par laquelle les gènes sont transmis aux générations suivantes. Notre travail montre donc que l’acquisition des cellules souches chez un ancêtre des animaux a été rendue possible non seulement par le recrutement de nombreux régulateurs post-transcriptionnels d’origine ancienne, mais aussi par l’acquisition d’une machinerie nouvelle de protection du génome, et que ces caractéristiques moléculaires ont accompagné l’évolution des cellules souches à travers l’histoire évolutive des animaux.