| |
| Med Sci (Paris). 32(8-9): 677–680. doi: 10.1051/medsci/20163208006.Quand les gènes codant les anticorps s’adjoignent des fragments venus d’autres chromosomes Brice Laffleur,1 Zeinab Dalloul,2 Iman Dalloul,2 Sandrine Le Noir,2 and Michel Cogné2,3* 1Department of immunology, Columbia university, New York, États-Unis 2Université de Limoges, CNRS UMR 7276, Contrôle de la réponse immune et lymphoprolifération, faculté de médecine, 2, rue du Docteur Marcland, 87025Limoges, France 3Institut universitaire de France, Paris, France MeSH keywords: Animaux, Variation des antigènes, Chromosomes, Réarrangement des gènes des lymphocytes B, Réarrangement des gènes des lymphocytes T, Gènes d'immunoglobuline, Humains, Récepteur lymphocytaire T antigène, alpha-bêta, Recombinaison génétique, génétique |
Diversité du système immunitaire et recombinaisons chromosomiques L’immunité met en œuvre une multiplicité de cellules et molécules aptes à reconnaître un immense répertoire de structures moléculaires portées par les antigènes. Ceci permet à la fois la « tolérance du soi » en condition basale et une réponse à diverses agressions grâce à une immunité contre le « non-soi » (comme des microorganismes) ou le « soi modifié » (induit par des mutations, un cancer, etc.). Chez les invertébrés, l’immunité innée repose sur un répertoire limité de récepteurs invariants reconnaissant un spectre restreint d’antigènes. Elle a l’avantage de préexister à l’arrivée de tout antigène étranger mais elle présente peu de spécificité et d’affinité, et aucune adaptabilité. Chez les mammifères, s’ajoute une immunité adaptative qui est, elle, capable de lier une immense diversité d’antigènes avec spécificité et affinité via deux grands répertoires très diversifiés de cellules (les lymphocytes T et B) et de récepteurs ou molécules solubles : les récepteurs TCR (T cell receptor) et BCR (B cell receptor) ou les immunoglobulines (Ig) solubles. L’économie des gènes codant ces récepteurs permet de générer leur diversité de façon combinatoire, en réassociant des fragments de gènes à la manière d’un patchwork pour créer dans une cellule la combinaison codant une Ig ou un TCR donné. Des réarrangements chromosomiques génèrent ainsi une variété quasi-infinie de récepteurs à partir de seulement quelques dizaines de gènes ou briques élémentaires (Figure 1). Alors que ces mécanismes semblaient connus en détails, un type de réarrangement très inattendu vient d’être décrit comme permettant la génération d’anticorps de large réactivité contre le parasite responsable du paludisme, Plasmodium falciparum [1], remettant certains dogmes en questions…
 | Figure 1.
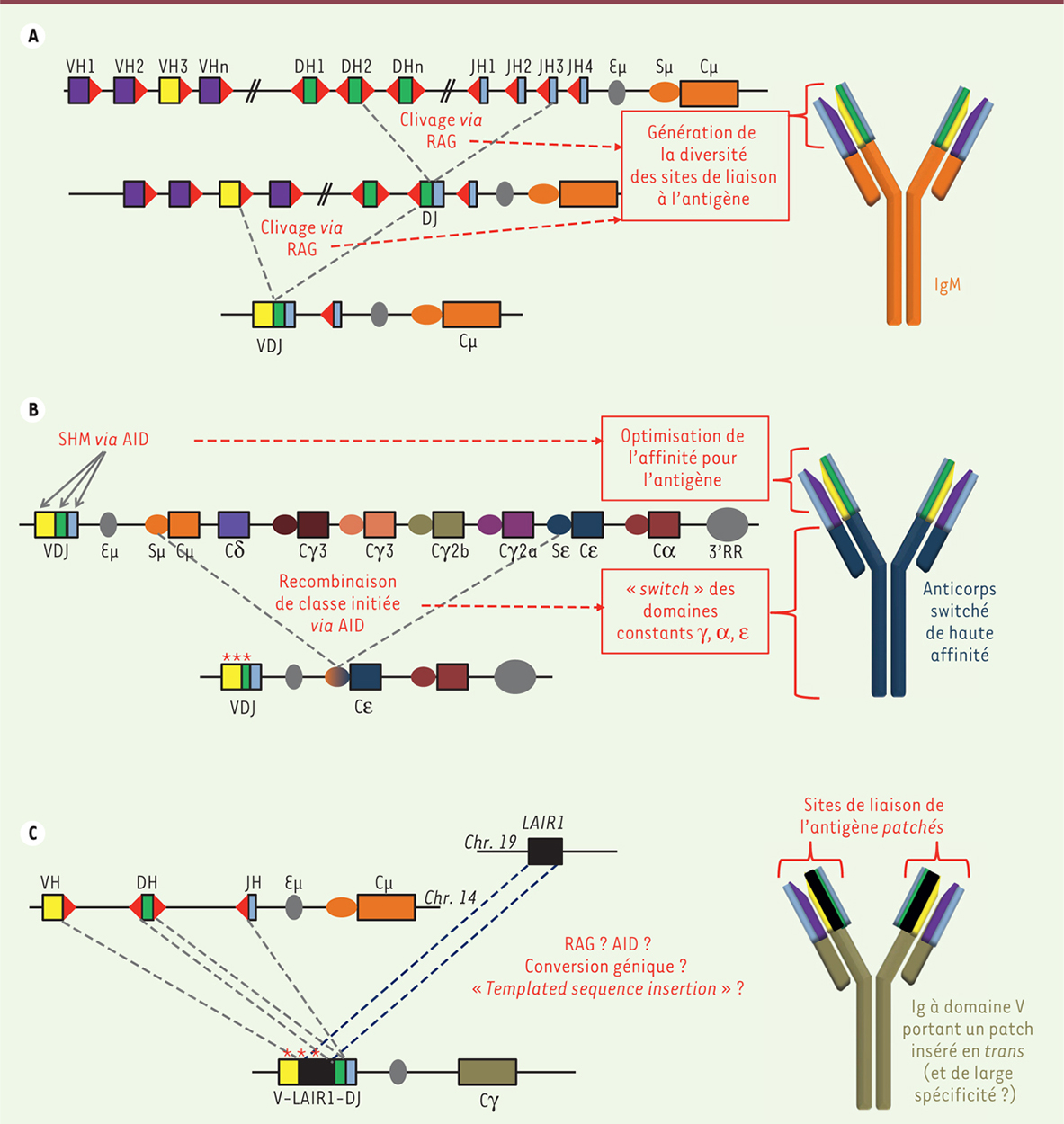
Mécanique des remaniements des gènes d’immunoglobulines. A. Génération initiale de la diversité des immunoglobulines (Ig) par RAG (recombination activating gene) et recombinaison VDJ au sein du locus des chaînes lourdes d’Ig (IgH). Les segments V, D, J et constant du locus IgH sont respectivement représentés en violet et jaune, vert, bleu et orange ; les RSS (recombination signal sequences) sont schématisés par des triangles rouges. La partie droite représente une IgM après recombinaison VDJ. B. Optimisation des Ig sous l’effet d’AID (activation induced cytidine deaminase) après rencontre de l’antigène, par hypermutation somatique et recombinaison de commutation de classe (switch ou CSR [class switch recombination]) du locus IgH. Les régions switch et les gènes constants sont respectivement représentés par des ronds et des carrés. Les étoiles correspondent aux mutations induites par AID sur le segment VDJ. La partie basse du schéma représente le locus IgH après CSR ; la partie droite représente une Ig après SHM (somatic hypermutation) et CSR, dans l’exemple du switch IgE. C. Insertion par recombinaison en « trans » d’une séquence non-Ig LAIR1 au sein d’un gène réarrangé VDJ du locus IgH et génération d’un domaine variable porteur d’un patch. Schéma représentatif d’une insertion du gène LAIR1 (leukocyte associated immunoglobulin like receptor 1) (représenté en noir) lors d’une recombinaison (VDJ-like ?) du locus IgH. Le schéma de droite représente une Ig avec un site de fixation à l’antigène comprenant l’addition d’un patch LAIR1. |
|
Génération de la diversité des récepteurs à l’antigène Les récepteurs à l’antigène, TCR et BCR, des lymphocytes T et B sont codés via la mécanique moléculaire commune de la « recombinaison V(D)J ». Celle-ci survient avant la rencontre avec l’antigène, dans le thymus pour les lymphocytes T, et dans la moelle osseuse pour les B. Elle assemble des segments de gènes nommés V (variable), D (diversité) et J (jonction) (Figure 1A). Le brassage combinatoire de ces quelques dizaines de gènes s’opère grâce aux enzymes spécialisées RAG (recombination activating gene) qui coupent l’ADN aux extrémités des segments V(D)J, au niveau de signaux dits RSS (recombination signal sequences). Les mécanismes classiques de réparation de l’ADN permettent ensuite de ressouder deux segments entre eux. À la diversité combinatoire, ces mécanismes de réparation ajoutent une certaine « imprécision » (par l’ajout ou le retrait de quelques bases au point de cassure qui a été réparé), augmentant ainsi la diversité des séquences générées (diversité dite « jonctionnelle ») [2, 3]. L’assemblage unique par chaque clone T ou B de tels segments « V(D)J », génère ainsi des populations lymphocytaires polyclonales qui expriment globalement un répertoire de récepteurs dont la diversité est quasi-infinie. Parmi ces millions de clones (chacun arborant un récepteur de spécificité définie), certains seront activés par la rencontre d’un antigène spécifique et participeront donc à la réponse immune. Les cellules T orchestrent ces réponses et peuvent détruire des cellules cibles ; les cellules B pourront sécréter leurs BCR sous la forme d’Ig solubles, les anticorps. Une mémoire immunitaire sera également générée permettant une réponse accélérée en cas de nouvelle rencontre avec le même antigène (vaccination). |
Maturation d’affinité et commutation de classe des immunoglobulines Afin d’adapter la réponse immune après la rencontre avec l’antigène, la lignée B est capable de prolonger le remaniement des gènes d’Ig grâce à l’enzyme AID (activation induced cytidine deaminase). Celle-ci initie deux processus : l’hypermutation somatique (SHM, pour somatic hypermutation) et la recombinaison de classe (CSR, pour class switch recombination) (Figure 1B) (pour revue voir [4]). Un segment VDJ préalablement assemblé pourra alors être modifié et son site de liaison à l’antigène amélioré par SHM. Un processus de sélection cellulaire favorise les cellules présentant le BCR ayant la plus haute affinité vis-à-vis de l’antigène. Plusieurs « classes » de BCR et d’Ig (IgM, puis IgG, IgA ou IgE, avec différents domaines constants) sont en outre successivement produites par un clone B (Figure 1B). Cette « commutation isotypique » ou « switch » résulte de la CSR entre des séquences d’ADN (régions « switch » des gènes d’Ig) dont le ciblage par AID crée des cassures double brin. Un dernier type AID-dépendant de recombinaison est la LSR (locus suicide recombination), une variante du switch qui supprime la totalité des gènes constants IgH (à l’origine de la chaîne lourde des Ig) et entraîne donc la perte du BCR et l’apoptose [5]. |
Interactions cis- et trans-chromosomiques Physiologiquement, dans l’immense majorité des cas, ces cassures programmées du génome réassocient in fine des fragments de gènes qui sont des partenaires légitimes (Ig-Ig dans les B, TCR-TCR dans les T). Les trans-recombinaisons avec d’autres chromosomes sont considérées comme rarissimes, relevant du champ de la pathologie. Les remaniements lymphoïde-spécifiques physiologiques du génome nécessitent donc un contrôle strict et leurs erreurs peuvent s’avérer dramatiques (comme la translocation d’un oncogène, à l’origine de leucémies ou de lymphomes) [10] (→).
(→) Voir la Synthèse de S. Gisselbrecht, m/s n° 2, février 2003, page 201
L’expression et la localisation nucléaire des enzymes RAG et AID ainsi que leur accès à la chromatine sont ainsi régulées dans le temps et l’espace, strictement dans les lignées lymphoïdes, et à des stades spécifiques de leur développement. Les chromosomes impliqués sont alors relocalisés dans des compartiments nucléaires particuliers qu’on peut dénommer « recombination factories »1. Différents activateurs transcriptionnels des gènes d’Ig ou de TCR orchestrent leurs remaniements en rapprochant les séquences cibles de recombinaison, créant d’éventuelles boucles chromosomiques lors de recombinaison « en cis » (sur un même chromosome). Les recombinaisons V(D)J sont réputées survenir quasi-exclusivement en cis lorsqu’elles surviennent entre des partenaires « légitimes » (c’est-à-dire entre segments V, D et J authentiques). Au contraire, les réarrangements de switch (qui surviennent de façon simultanée sur les deux allèles) impliquent fréquemment des recombinaisons inter-alléliques « en trans » (sortes de translocations légitimes entre chromosomes de la même paire) [6]. |
Un nouveau mécanisme de génération d’anticorps en transLe nouveau type de remaniement décrit par Tan et al. s’inscrit dans un contexte d’interactions trans-chromosomiques, mais avec la particularité d’impliquer, au moins partiellement, les recombinaisons V(D)J et d’apparier les locus d’Ig avec un gène autre situé sur un chromosome non homologue [1]. De façon étonnante, elles semblent cependant physiologiques puisqu’elles aboutissent à la production d’une molécule qui porte la plupart des caractéristiques d’un anticorps fonctionnel présentant une réactivité large et qui aurait vraisemblablement un rôle actif dans la réponse immune anti-plasmodiale. Ce nouveau type d’anticorps a été découvert chez deux individus différents, à l’occasion d’une étude portant sur les anticorps de réactivité large qui sont présents chez des sujets impaludés (le paludisme se traduit notamment par la présence de P. falciparum infectant les érythrocytes et induisant alors l’expression d’antigènes du parasite à la surface des hématies infectées). Alors que les malades peuvent présenter des accès sévères de paludisme en début d’infection ou de réinfection, les personnes vivant en zone impaludée développent une immunité et des anticorps dirigés contre l’isolat de parasite à l’origine de leur infection. Quelques sujets développent des anticorps dits « de large réactivité » qui sont capables d’agglutiner des hématies infectées par de multiples isolats indépendants de P. falciparum. L’étude de leur immunité présente donc un intérêt particulier, potentiellement révélateur de stratégies naturelles d’immunité anti-plasmodiale large. Ainsi, après avoir criblé des centaines d’échantillons, Tan et al. ont identifié quelques donneurs sains dont les anticorps sont capables de neutraliser des érythrocytes infectés par 6 isolats différents du parasite et d’entraîner leur élimination par phagocytose Ig-dépendante [1]. Les lymphocytes B de ces donneurs ont été immortalisés afin de déterminer la séquence de ces anticorps protecteurs de large réactivité. De façon totalement inattendue, le séquençage a révélé que le domaine de liaison à l’antigène de certains de ces anticorps était codé (chez deux patients indépendants) par un gène composite qui incluait une large portion étrangère aux gènes normaux d’Ig (qui sont situés sur le chromosome 14 humain) identifiable comme provenant du chromosome 19 mais recombinée et insérée entre les segments V et DJ. Ce segment de chromosome 19 correspond à une partie du gène LAIR1 (leukocyte associated immunoglobulin like receptor 1), un récepteur inhibiteur (à domaines ITIM [immunoreceptor tyrosine-based inhibition motif]) appartenant à la superfamille des Ig exprimé par les cellules B et liant, normalement, le collagène. Une séquence codant 98 acides aminés de LAIR1 est ainsi exprimée comme une sorte de « patch » inséré au sein d’un domaine variable d’Ig et permet, après hypermutation somatique, d’y lier non pas le collagène, mais des épitopes de la famille multigénique RIFIN (la plus large famille d’antigènes variants de Plasmodium, qui sont exprimés à la surface des érythrocytes infectés et impliqués en tant qu’adhésines dans certaines formes sévères du paludisme). De plus, une fois cette séquence insérée au sein du gène VDJ, elle subit comme un VDJ classique l’hypermutation permettant d’augmenter son affinité pour l’antigène plasmodial RIFIN (et de supprimer l’affinité de LAIR1 pour le collagène) (Figure 1C). |
Ce nouveau mécanisme démontre la capacité physiologique de nos cellules B à introduire au sein d’un gène d’Ig une séquence étrangère (non-Ig) afin de créer un néo-site de liaison à l’antigène. Le séquençage haut-débit devrait permettre d’évaluer la fréquence de telles insertions d’un patch LAIR1 dans des populations saines et éventuellement exposées à des infections chroniques. D’autres patchs de séquences non-Ig ainsi insérés pourraient également être découverts et il reste donc à savoir si le cas de LAIR1, pour les anticorps anti-Plasmodium, restera ou non un exemple unique d’anticorps fonctionnel naturel incluant un patch « non-Ig ». Le mécanisme de ce remaniement génique reste à préciser, mais il porte au moins la signature de RAG puisque l’insertion se fait au niveau du point de jonction entre les segments V et DJ. De plus, la séquence de LAIR1 se révèle flanquée d’introns incluant des séquences qui évoquent des RSS cryptiques. Une trans-recombinaison simple médiée par RAG semble exclue car les cellules B des clones concernés ont conservé 2 chromosomes 19 portant des copies intactes du gène LAIR1. Ceci évoquerait donc plutôt un phénomène de type conversion génique, ou encore l’insertion au point de cassure d’un gène d’Ig d’un pré-ARN messager LAIR1 rétrotranscrit (ce type de réparation mutagène des cassures double brin avec insertion d’une séquence rétrotransposée a récemment été décrit et dénommé « templated sequence insertion ») [7]. Pour les anticorps anti-Plasmodium, la complexité du phénomène décrit est encore accrue par l’implication claire d’AID dans les mutations secondaires du gène V-LAIR1-DJ réarrangé et du patch LAIR1. L’infection palustre entraîne une stimulation B chronique et une expression accrue d’AID, pouvant favoriser des translocations [8]. Il est possible qu’elle suscite également la trans-recombinaison de LAIR1 au sein d’un segment VDJ, puis son hypermutation. Dans ce contexte, on se rappellera aussi que même s’ils échappent au dogme et au schéma classique de la maturation B, des épisodes de co-expression de RAG et AID ont été démontrés [9]. Alors que ce processus est sans doute plus l’exception que la règle, il dévoile cependant des aptitudes insoupçonnées des lymphocytes B à poursuivre encore plus loin l’extrême diversification de leur répertoire afin d’optimiser et d’adapter les réponses immunes aux antigènes rencontrés, par l’usage d’un arsenal dont la description vient de s’enrichir d’une nouvelle arme ! Il est frappant de noter que cette description survient au cours de la caractérisation d’anticorps d’un grand intérêt potentiel en immunothérapie, actuellement traqués dans le cadre de la protection contre divers agents infectieux (VIH [virus de l’immunodéficience humaine], Norovirus, Influenza, P. falciparum, etc.), les anticorps neutralisants à vaste spectre de réactivité [11] (→).
(→) Voir la Nouvelle de T. Bruel et al. sur les anticorps bNAb, page 671 de ce numéro
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Tan J, Pieper K, Piccoli L, et al. A LAIR1 insertion generates broadly reactive antibodies against malaria variant antigens . Nature. 2016; ; 529 : :105.–109. 2.
Nishana M, Raghavan SC. Role of recombination activating genes in the generation of antigen receptor diversity and beyond . Immunology. 2012; ; 137 : :271.–281. 3.
Lieber MR, Yu K, Raghavan SC. Roles of nonhomologous DNA end joining, V(D)J recombination, and class switch recombination in chromosomal translocations . DNA Repair. 2006; ; 5 : :1234.–1245. 4.
Laffleur B, Denis-Lagache N, Péron S, et al. AID-induced remodeling of immunoglobulin genes and B cell fate . Oncotarget. 2014; ; 5 : :1118.–1131. 5.
Péron S, Laffleur B, Denis-Lagache N, et al. AID-driven deletion causes immunoglobulin heavy chain locus suicide recombination in B cells . Science. 2012; ; 336 : :931.–934. 6.
Laffleur B, Bardet SM, Garot A, et al. Immunoglobulin genes undergo legitimate repair in human B cells not only after cis- but also frequent trans-class switch recombination . Genes Immun. 2014; ; 15 : :341.–346. 7.
Onozawa M, Zhang Z, Kim YJ, et al. Repair of DNA double-strand breaks by templated nucleotide sequence insertions derived from distant regions of the genome . Proc Natl Acad Sci USA. 2014; ; 111 : :7729.–7734. 8.
Robbiani DF, Deroubaix S, Feldhahn N, et al. Plasmodium infection promotes genomic instability and AID-dependent B cell lymphoma . Cell. 2015; ; 162 : :727.–737. 9.
Wang JH, Gostissa M, Yan CT, et al. Mechanisms promoting translocations in editing and switching peripheral B cells . Nature. 2009; ; 460 : :231.–236. 10.
Gisselbrecht S. Oncogènes et leucémies : historique et perspectives . Med Sci (Paris). 2003; ; 19 : :201.–210. 11.
Bruel T, Mouquet H, Schwartz O. Des anticorps qui détruisent les cellules infectées par le VIH-1 . Med Sci (Paris). 2016; ; 32 : :671.–674. |