Depuis 1894 et la découverte par Alexandre Yersin de la bactérie responsable de la peste qui porte son nom, Yersinia pestis [1], l’étiologie des épidémies qui ont décimé d’abord le bassin méditerranéen, puis l’Europe, depuis deux mille ans, est maintenant connue [2]. Il y a 20 ans, nos travaux pionniers avaient montré que ces grandes épidémies qui déferlaient depuis le XIe siècle, étaient bien causées par Y. pestis et qu’il s’agissait sans aucun doute d’épidémies de peste [3, 4].
Même si certaines ont échoué [5], de très nombreuses équipes à travers le monde ont pu diagnostiquer la peste à partir de restes humains anciens. En utilisant différentes techniques de détection soit de séquences d’ADN soit d’antigènes protéiques spécifiques, elles ont montré que la plupart des épidémies qui avaient été décrites par les médecins contemporains des épidémies historiques, comme Guy De Chauliac, ou par les littérateurs ou les peintres, au cours des siècles précédents, étaient bien des épidémies de peste. Ces travaux de paléo-microbiologie ont ainsi permis d’obtenir des génomes (presque) complets de la bactérie Y. pestis isolée à partir de 18 individus provenant de quatre périodes différentes de l’histoire de la maladie (Tableau I). Ces résultats font entrer la paléo-épidémiologie de la peste dans une nouvelle dimension.
Tableau I.
Études publiées à ce jour sur les génomes anciens de Yersinia pestis. Le tableau recense les différentes études sur les génomes anciens de Y. pestis, les individus à partir desquels les génomes ont été reconstruits, la datation des échantillons et les épidémies ou événements concernés. La profondeur moyenne de séquençage par nucléotide (redondance) et le pourcentage de recouvrement des génomes de Y. pestis obtenus après séquençage sont également indiqués. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
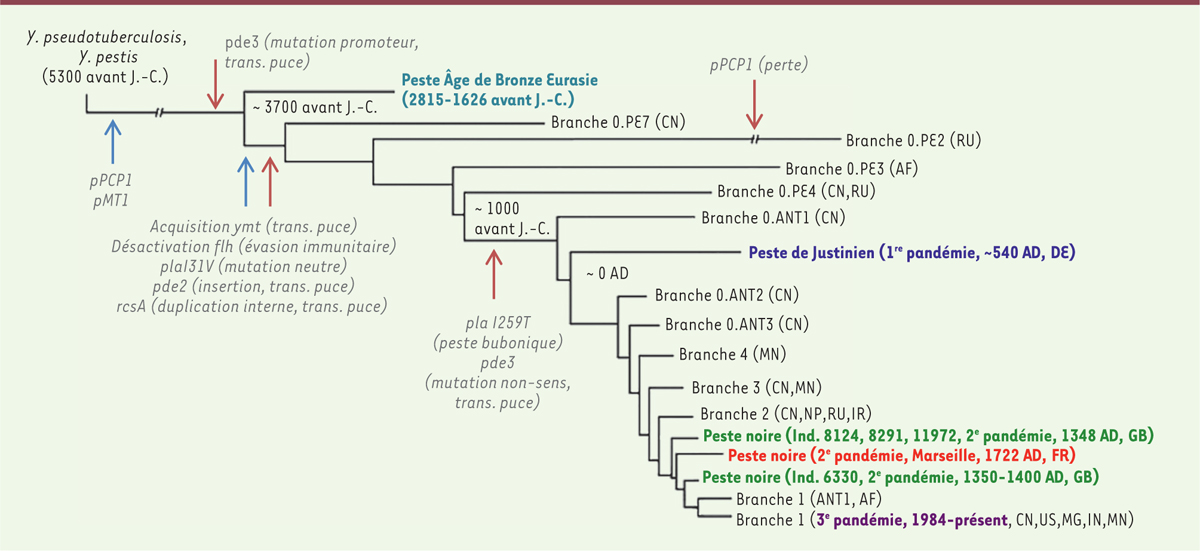
Le travail remarquable récemment publié par Rasmussen et collaborateurs [6] apporte une contribution importante en montrant que des populations, datées de 3 000 ans et donc vivant à l’âge de bronze, et issues d’une vaste région s’étendant depuis l’Europe jusqu’à l’Asie centrale, étaient également infectées par Y. pestis. La découverte et l’analyse des génomes de ces nouvelles souches, les plus anciennes décrites à ce jour, ont révélé que Y. pestis a en fait divergé il y a 5 300 ans, de Yersinia pseudotuberculosis, une autre bactérie pathogène pour l’homme (Figure 1). Ces résultats représentent ainsi une avancée dans la compréhension des mécanismes par lesquels Y. pestis s’est répandue, par vagues successives, dans les populations anciennes, provoquant ce qui est probablement l’une des plus grandes mortalités enregistrées par les données historiques. L’augmentation du nombre de génomes bactériens devenus disponibles et leur analyse fine devraient permettre de comprendre si un ou plusieurs clones de Y. pestis sont à l’origine des différentes vagues épidémiques successives qui ont été rapportées. Les données préliminaires que nous avions obtenues par une méthode pionnière à l’époque, le multi spacer typing (MST)1, et publiées avant la mise en évidence de génomes bactériens entiers, avaient montré que les souches anciennes européennes appartenaient au biotype Orientalis [7]. Les données génomiques récentes ne remettent pas en cause cette observation. Elles devraient cependant permettre de la préciser et de savoir si un ou plusieurs clones de biotype Orientalis-like ont été responsables des épidémies. L’analyse des clones de Y. pestis responsables des épidémies anciennes est en effet l’un des éléments qui permettrait de comprendre les mécanismes par lesquels les souches de cette bactérie ont pu induire une telle mortalité. Actuellement aucun élément en faveur d’une variation notable dans la virulence des souches anciennes par rapport aux souches modernes n’a pu être mis en évidence. C’est donc probablement le mode de transmission de ces souches anciennes dans les populations humaines qui permettrait de comprendre la grande mortalité qu’elles ont provoquée.
| Figure 1.
Représentation schématique de l’évolution de Yersinia pestis. Cette représentation combine les phylogénies présentées dans les travaux de Rasmussen et al. [6] et Bos et al. [13]. La topologie de l’arbre montre une relation évolutive approximative entre les différentes souches et n’est pas représentative d’une analyse de novo des données génomiques. Les événements évolutifs les plus significatifs sont indiqués par des flèches (rouge : pour une perte génétique ou fonctionnelle, bleu : pour un gain). Le registre historique des pandémies de peste est également indiqué avec une couleur différente en fonction de la période (les années sont indiquées entre parenthèses). trans. puce : transmission par puce ; CN : Chine ; RU : ancienne Union Soviétique ; AF : Afrique ; DE : Allemagne ; MN : Mongolie ; IR : Iran ; IN : Inde ; NP : Népal ; US : États-Unis ; MG : Madagascar ; FR : France ; GB : Grande-Bretagne ; AD : apr. J.-C. |
Le schéma classique de transmission de Y. pestis à l’homme à partir de populations de micro-rongeurs comme le rat par l’intermédiaire d’ectoparasites tels que la puce, est bien sûr réducteur. Il ne permet pas en effet de rendre compte des épidémies historiques de peste et plusieurs auteurs ont opposé de nombreux éléments de contradiction invalidant ce modèle. Nous avons proposé une alternative avec la possibilité d’une transmission interhumaine de Y. pestis, directement par des ectoparasites non pas de rongeurs mais d’homme, en particulier le pou de corps. Cette hypothèse est désormais validée par différentes observations : (1) la mise en évidence paléo-microbiologique de co-infection par Y. pestis et par Bartonnella quintana dont le vecteur principal est le pou de corps de l’homme [8], (2) l’identification récente de Y. pestis dans des poux de corps collectés chez des personnes vivant dans des régions de foyers de peste, au Congo [9] et (3) les données expérimentales qui montrent sans aucun doute la grande capacité des poux de corps à transmettre Y. pestis [10].
Ce modèle de transmission interhumaine ne permet cependant pas d’identifier les sources des infections de ces populations humaines et anciennes. S’il est tout à fait concevable que des populations de rongeurs accompagnant l’homme dans ses déplacements, en particulier ses pérégrinations maritimes dans le bassin méditerranéen, aient pu introduire Y. pestis dans les populations anciennes, la question des sources ultimes, et de leur persistance en Europe médiévale et moderne, reste cependant ouverte. La peste a totalement disparu de l’Europe depuis un siècle, les derniers cas ayant, par exemple, été diagnostiqués à Marseille en 1920. Dans les pays de la rive sud de la méditerranée, l’analyse génétique des souches, à l’occasion d’épisodes de peste récents, a montré qu’il s’agissait bien de souches autochtones et non de souches d’importation qui pourraient être liées au trafic maritime. Il existe en fait, et cela de façon certaine, des foyers de peste dans cette région, au Maghreb mais aussi au Proche et Moyen-Orient et jusqu’au Kurdistan. Les facteurs ayant déterminé la persistance dans ces régions de ces foyers de peste alors qu’il n’y en a plus au nord et en Europe, restent à déterminer et nous tentons actuellement de les identifier.