La culture microbienne a été dans l’histoire de la microbiologie la première technique permettant d’avoir une compréhension des microbes et des maladies qu’ils causaient [1]. Étant une technique longue, fastidieuse et coûteuse, les microbiologistes l’ont progressivement abandonnée à la suite de l’avènement des méthodes moléculaires, à la fin du XXe siècle [1]. La métagénomique appliquée pour la première fois en 2005 au microbiote digestif humain, qui promettait de découvrir l’incultivable, a été par la suite la seule et unique méthode utilisée par les chercheurs pour explorer les écosystèmes complexes, en particulier les microbiotes humains [2]. Cependant, cette technique est associée à de nombreux biais, comme celui dû à l’extraction ou celui résultant du choix des amorces (utilisées pour l’amplification des séquences nucléotidiques), expliquant que les résultats ne soient que rarement reproductibles entre différents laboratoires et que des données contradictoires soient parfois générées au sein d’un même laboratoire [2, 13] (→). Le biais de profondeur1, est probablement une des plus grandes limites actuelles de la métagénomique qui est incapable de détecter les espèces présentes à des concentrations inférieures à 105 ou 106 bactéries par gramme de selles [2, 3]. Plusieurs bactéries pathogènes pour l’homme, telles que Salmonella typhi, Yersinia enterocolitica ou Tropheryma whipplei sont pourtant responsables de pathologies humaines graves alors qu’elles sont présentes à de plus faibles concentrations dans les selles humaines [2]. Ainsi, malgré les progrès considérables que la métagénomique a permis pour étendre la définition du répertoire, une grande partie de ce dernier, dénommée usuellement la « dark matter » et qui correspond à un nombre incalculable de séquences nucléotidiques n’étant assignées à aucun microorganisme connu, résiste à cette avancée technologique et demeure totalement inconnue [4].
(→) Voir la Synthèse de J. Weissenbach et A. Sghir, page 937 de ce numéro
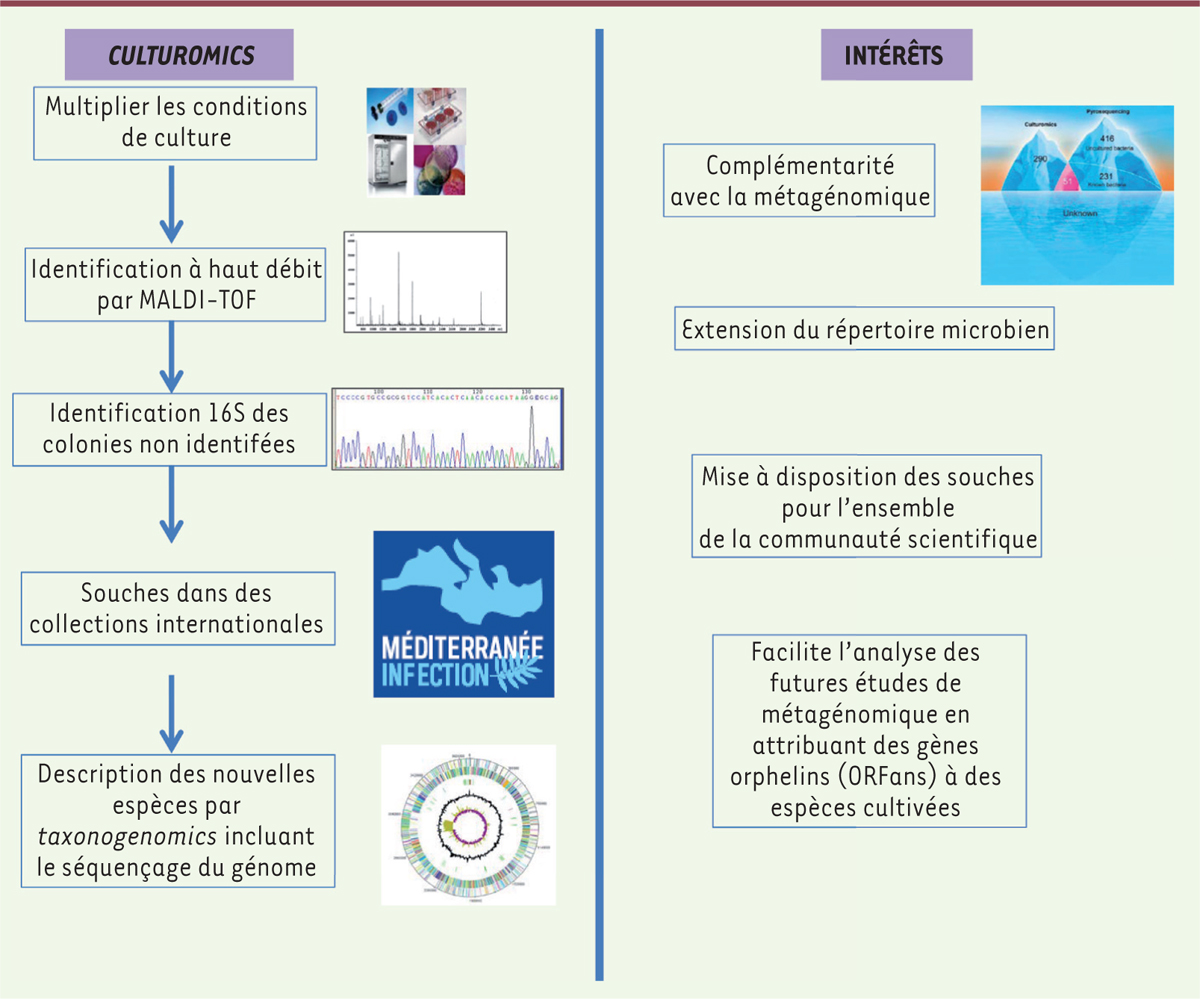
Jusqu’à présent, environ 13 000 espèces bactériennes ont été cultivées, incluant environ 2 200 espèces isolées à partir de prélèvements humains [5]. Depuis quelques années, la culture microbienne a en effet connu un renouveau important, en premier lieu grâce aux microbiologistes de l’environnement qui ont développé des techniques mimant la composition du milieu naturel des bactéries qu’ils cherchaient à isoler, facilitant ainsi leur croissance [2]. Ainsi, en utilisant des chambres de diffusion, Kaeberlein et al. ont montré que ce dispositif permettait d’isoler plus de 300 fois plus de colonies qu’en ensemençant directement les prélèvements sur gélose [6]. Ces 5 dernières années, la culture microbienne en microbiologie humaine a connu un essor considérable grâce à l’avènement et à la généralisation d’une méthode d’identification rapide et peu coûteuse par spectrométrie de masse de type MALDI-TOF (matrix assisted laser desorption ionisation/time of flight) qui permet, notamment, de détecter des espèces bactériennes pathogènes rares [7]. Cette technique qui est devenue la technique de référence d’identification bactérienne dans les laboratoires de microbiologie clinique, a permis également de développer un nouveau concept d’étude des microbiotes humains appelé « microbial culturomics » [2, 3]. Cette technique est fondée sur la multiplication des conditions de culture, en utilisant d’une part des conditions permettant de faciliter la croissance des espèces fastidieuses et, d’autre part, d’utiliser des inhibiteurs de croissance des espèces majoritaires, ce qui permet de sélectionner les espèces minoritaires afin de les identifier (Figure 1) [3]. Dans un travail préliminaire, cette approche a permis, grâce à l’utilisation de 212 conditions de culture différentes, et en analysant 31 500 colonies par MALDI-TOF, de cultiver 340 espèces bactériennes dont 31 nouvelles espèces ainsi que des espèces appartenant à des phylums rares (Synergistetes et Deinococcus-Thermus) [3]. En particulier, l’utilisation d’une pré-incubation des selles dans des flacons d’hémoculture ainsi que l’ajout de jus de rumen de mouton (un des compartiments de l’estomac des ruminants) stérilisé, afin de mimer le milieu naturel des bactéries du tube digestif, pour la plupart anaérobies, ont été deux des clés majeures permettant d’obtenir ces résultats [3]. La complémentarité de la culturomics avec la métagénomique a également été démontrée puisque seules 15 % des espèces analysées de façon concomitante par pyroséquençage ont été détectées avec les deux techniques [3]. Une analyse détaillée a permis de sélectionner successivement les 70 puis les 18 conditions de culture les plus appropriées pour explorer la plus grande diversité possible tout en réduisant la charge de travail nécessaire pour analyser chaque échantillon. D’autres travaux réalisés par notre équipe ont permis par la suite de cultiver plus de 50 % des espèces connues du tube digestif humain incluant 247 nouvelles espèces [2, 9].
Cette augmentation du nombre d’espèces bactériennes a conduit à devoir créer de nouvelles méthodes de description des espèces bactériennes découvertes. La taxonogenomics 2 implique ainsi que chaque nouvelle espèce voie son génome séquencé et comparé aux génomes des espèces les plus proches et que son spectre MALDI-TOF soit ajouté à sa description [8]. Le processus de description puis de publication étant relativement long, nous avons proposé récemment de créer des « new species announcements » de façon à ce que l’ensemble de la communauté scientifique puisse être informé en temps réel, dès l’isolement de la nouvelle espèce, afin d’en disposer le plus rapidement possible. Ces publications de description courtes incluent le numéro d’identification de l’amplification et du séquençage du gène 16S de ces nouvelles bactéries (numéro Genbank), le numéro de dépôt dans la collection de souches de l’unité des Rickettsies (CSUR)3, de façon à pouvoir être commandées rapidement, et quelques caractéristiques phénotypiques de base. La taxonogenomics a été reconnue. Quinze nouvelles espèces, décrites selon cette méthode, ont en effet été validées officiellement par le comité de taxonomie international de l’International Journal of Systematic and Evolutionary Microbiology [8]. Outre le fait de rendre obsolète l’hybridation ADN-ADN jusqu’alors gold standard pour décrire une nouvelle espèce (alors qu’il s’agit d’une méthode non reproductible entre laboratoires et non adaptée à de nombreuses espèces bactériennes [8]), le fait de séquencer chacun des génomes et de les déposer dans des banques de dépôt internationales (Genbank, EMBL [european molecular biology laboratory]), ce qui les rend disponibles à l’ensemble des chercheurs, permet ainsi d’attribuer à des espèces, des gènes qui étaient jusqu’alors orphelins (ORFans) et ainsi de faciliter l’interprétation des futures études de métagénomique. L’ensemble de ces nouvelles espèces sont disponibles dans des collections de souches internationales (CSUR et DSMZ [deutsche sammlung von mikroorganismen und zellkulturen]) accessibles à l’ensemble de la communauté scientifique, chacun pouvant les utiliser afin de rechercher un éventuel lien de causalité avec diverses pathologies.
Outre l’effort d’extension du répertoire (doublement du nombre d’espèces cultivées connues) qui permet de combler en partie la « dark matter » [9], la culture pure des bactéries demeure indispensable afin d’étudier leur virulence, leur sensibilité aux antibiotiques, ou pour développer des modèles expérimentaux [1]. Un lien entre des souches toxiniques4 de Clostridium butyricum et l’entérocolite ulcéro-nécrosante, touchant les enfants prématurés, a ainsi pu être démontré récemment par Cassir et al. [10]. C’est en effet grâce à la culturomics appliquée à leurs échantillons que ces auteurs ont mis en évidence une plus grande fréquence de cette bactérie chez les enfants atteints, par rapport aux témoins, et ont pu tester les toxines produites par les souches isolées [10]. De même, Zitvogel et al. ont récemment démontré le caractère immunomodulateur du microbiote digestif au cours de la réponse aux thérapies anti-cancéreuses [11, 12] (→).
(→) Voir la Synthèse de M. Vétizou et al., page 974 de ce numéro
Seul le fait de disposer de souches pures a permis à ces auteurs de tester une hypothèse qui n’aurait pu être vérifiée par la métagénomique seule.
Le concept de culturomics, appliqué aux microbiotes urinaire, vaginal ou cutané, permet des études similaires et l’obtention de résultats significatifs (résultats non publiés). La culturomics constitue donc un formidable effort pour la constitution du répertoire des bactéries isolées chez l’homme. Elle devrait donc permettre à des générations de chercheurs de disposer de souches pures leur permettant d’appréhender les relations existant entre bactéries composant le microbiote humain et santé humaine.