| |
| Med Sci (Paris). 32(11): 999–1002. doi: 10.1051/medsci/20163211016.Microbiote intestinal, la clé pour une meilleure alimentation ? Patrick Veiga,1 Julien Tap,1 and Muriel Derrien1* 1Danone Nutricia Research, Avenue de la Vauve, 91120Palaiseau, France |

L’adoption du mode de vie occidental s’accompagne de changements du microbiote intestinal, associés à l’émergence de maladies chroniques [34, 35] (→).
(→) Voir les Synthèses de O. Rahmouni et al., et de R. Burcelin et al., pages 968 et 952 de ce numéro
Au cours de la dernière décennie, le développement de thérapies ciblant le microbiote d’individus malades a connu un fort engouement. Cependant, les solutions préventives aidant à maintenir la symbiose hôte - microbe pour faire face aux facteurs de la vie moderne font encore défaut. Dans cette revue, nous discutons du rôle clé de l’alimentation dans l’évolution de la symbiose microbe-hôte, via notamment son influence sur la production de molécules bioactives. Nous présentons également des résultats récents qui soutiennent le concept selon lequel la personnalisation de l’alimentation en fonction du génotype et du microbiote de chaque individu est une opportunité et un défi scientifique prometteur. |
Les changements alimentaires au cours de l’évolution humaine L’évolution de l’homme s’est accompagnée de changements périodiques dans son mode de vie qui se reflètent dans ses habitudes alimentaires. Durant la période paléolithique, les humains étaient des chasseurs-cueilleurs nomades, accédant à une nourriture dépendant des saisons. À partir de 10 000 ans avant JC (période néolithique), les humains ont commencé à se sédentariser, pratiquant un nouveau style de vie fondé sur l’agriculture et l’élevage. De nouveaux aliments, comme les céréales et les produits laitiers, sont apparus, et la densité de population a fortement augmenté [1]. Un changement drastique dans les habitudes alimentaires humaines est apparu avec la révolution industrielle, il y a moins de 200 ans, marquant le début de la consommation de produits raffinés, s’éloignant des aliments saisonniers produits localement. L’alimentation moderne des pays occidentalisés est ainsi caractérisée par une forte consommation de produits d’origine animale et riches en sucres et gras, l’utilisation de conservateurs, et une plus faible consommation d’aliments à base de plantes comme les fruits, les légumes et les céréales complètes [1]. De nombreuses études récentes ont exploré l’impact de l’occidentalisation sur le microbiote intestinal dans des populations humaines rurales, qui n’ont pas encore adopté un mode de vie occidental, en Afrique, en Amérique du Sud, et en Asie. Ces études nous ont appris que le microbiote de ces populations est « enrichi » en termes de diversité microbienne par rapport à celui des populations occidentales. Ces observations ont été modélisées expérimentalement chez la souris et les singes en captivité et ont été reliées à la quantité et à la diversité de fibres alimentaires constitutives des régimes alimentaires [2, 3]. En Afrique rurale, les populations peuvent consommer jusqu’à 120 g de fibres par jour alors que, dans les pays occidentaux, la moyenne de consommation s’élève en moyenne à 15 g par jour [4]. Cet appauvrissement en diversité microbienne dans les populations occidentales s’accompagne de facto de la perte de fonctions microbiennes qui sont redondantes. Cela entraîne la mise en péril de la capacité de l’écosystème à se restaurer après une exposition à des éléments perturbateurs externes (également appelée « résilience »), comme les antibiotiques et le stress. L’autre conséquence de la réduction de consommation de fibres sur le microbiote est une diminution de la production d’acides gras à chaîne courte (acétate, propionate, butyrate), qui contribuent à inhiber la croissance d’entérobactéries, une famille bactérienne comprenant de nombreuses bactéries pathogènes opportunistes [5]. |
Le microbiote, bioactivateur de molécules issues de l’alimentation Il est aujourd’hui admis qu’une myriade de molécules produites par le microbiote intestinal interagit directement sur la santé métabolique ou le système immunitaire ou nerveux de l’hôte [34, 36] (→).
(→) Voir les Synthèses de R. Burcelin et al., et de V. Gaboriau-Routhiau et N. Cerf-Bensussan, pages 952 et 961 de ce numéro
Beaucoup de ces précurseurs sont apportés par l’alimentation. C’est le cas du tryptophane, des polyphénols, des fibres, du glutamate, de la carnitine, de la choline [6]. L’identification des voies métaboliques responsables de la production de ces molécules permettra d’identifier les aliments qui sont sources d’ingrédients prébiotiques, et ceux, fermentés, qui sont enrichis en souches à potentiel probiotique, permettant de remédier aux lacunes fonctionnelles du microbiote intestinal. Ainsi, H. Sokol et ses collaborateurs ont montré que l’administration de souches de Lactobacillus, capables de produire des dérivés d’indole à partir du tryptophane, permettaient de réduire l’inflammation intestinale causée par un déficit de leur production par le microbiote endogène [7, 37] (→).
(→) Voir la Nouvelle de B. Lamas et al., page 933 de ce numéro
|
L’écologie microbienne au service de la santé S’il est difficile aujourd’hui de définir ce qu’est un « bon » microbiote, il est en revanche possible de l’analyser sous le prisme des indices propres à l’écologie microbienne : richesse, diversité, stabilité. Des études cliniques ont montré que la richesse (ou alpha-diversité) en espèces et gènes des microbiotes intestinaux constituait une variable essentielle permettant leur comparaison. Cette richesse est déterminée soit par le séquençage ciblé du gène codant la sous-unité 16S de l’ARN ribosomal, soit par la construction non supervisée d’un catalogue de gènes issus d’une approche métagénomique par séquençage direct [8, 38] (→).
(→) Voir la Synthèse de J. Weissenbach et A. Sghir, page 937 de ce numéro
Plusieurs de ces études ont montré l’intérêt de quantifier la richesse du microbiote en regard des paramètres métaboliques d’un individu et sa réponse vis-à-vis d’une intervention nutritionnelle [9–11]. Il est possible de comparer la biodiversité du microbiote (ou diversité bêta) entre individus ou de données cliniques ou alimentaires (par exemple, l’indice de masse corporelle, le taux de protéine contenu dans le bol alimentaire). La stratégie qui consiste à intégrer la mesure de diversité bêta pour stratifier les individus est très répandue dans l’analyse du microbiote [12, 13]. L’étude de cette diversité bêta a, en particulier, permis de faire émerger le concept d’entérotypes [13] qui repose sur la stratification des individus selon des types constituant leur microbiote. Ce concept met l’accent sur le fait que toutes les variations structurelles du microbiote sont régies par des interactions entre bactéries. Plusieurs études montrent, par exemple, que l’entérotype enrichi avec le genre Prevotella est plus prévalent dans les populations rurales en Afrique que dans les populations occidentales [5, 14]. Wu et al. ont montré que les entérotypes peuvent être associés aux habitudes alimentaires [15]. |
Le microbiote en haute définition Lors d’une approche métagénomique par séquençage direct d’un échantillon d’ADN, il est surtout possible d’accéder aux informations génétiques des espèces microbiennes dont se compose le microbiote intestinal. Il existe plusieurs outils pour assembler, détecter et annoter les gènes à partir des lectures issues d’un séquençage à haut débit [16–19]. Le goulot d’étranglement de l’analyse se situe au niveau des bases de données publiques disponibles qui souffrent d’un manque de représentativité des microorganismes anaérobies (majoritaires dans le microbiote intestinal) et des gènes non annotés fonctionnellement [8, 20, 38] (→).
(→) Voir la Synthèse de J. Weissenbach et A. Sghir, page 937 de ce numéro
L’utilisation de bases de données d’annotation potentielle de protéines telles que les bases CAZy (Carbohydrate-active enzymes) ou KEGG (Kyoto encyclopedia of genes and genomes) [16, 21, 22] permettent d’inférer les fonctions des enzymes impliquées respectivement dans la dégradation des sucres complexes ou des voies métaboliques. Cette approche a récemment permis de montrer que les fonctions de dégradation des fibres du microbiote intestinal étaient moins abondantes chez les patients atteints de cancer colorectal [23]. Couplés à des analyses de méta-transcriptomique, ces outils bio-informatiques permettent de capturer les voies métaboliques les plus activées en réponse à une intervention nutritionnelle. Cette approche a ainsi montré que la consommation d’un produit laitier fermenté entraîne la surexpression des gènes codant des enzymes capables de dégrader les fibres alimentaires [24]. L’évolution récente des technologies et des catalogues génétiques d’espèces microbiennes conduit à la possibilité d’identifier différentes souches d’une même espèce à partir d’un jeu de données métagénomiques. Ainsi, il a été récemment montré l’existence de trois souches majoritaires d’Eubacterium rectale : deux souches spécifiques des populations occidentales et une spécifique de la population chinoise [25]. Ces approches de détection des souches sont possibles grâce à la détection de la variation polymorphique de chaque nucléotide (SNP, pour single-nucleotide polymorphism) ou l’établissement du pangénome de chaque espèce microbienne [25]. L’utilisation de l’approche SNP a montré, sur une période d’un an, que chaque individu conserve les souches qui lui sont propres [26]. L’analyse pangénomique réalisée à partir de données métagénomiques de l’espèce Bifidobacterium longum a permis plus récemment de prédire la capacité de persistance dans le microbiote intestinal d’une souche de Bifidobacterium longum (AH1206) administrée oralement [27]. |
Inégalitarisme du microbiote Il est maintenant reconnu qu’un régime alimentaire donné peut avoir différents impacts chez différents individus [28, 29]. Une étude a ainsi montré, chez une population d’obèses ou en surpoids, qu’un régime alimentaire hypocalorique, riche en fibres, augmente la richesse du microbiote chez les individus dont le microbiote était « pauvre » mais n’augmente pas la richesse du microbiote chez les individus ayant un microbiote qui était déjà « riche » [11], confirmant qu’un écosystème « riche », ou divers, est plus stable [30]. Dans cette même étude, les auteurs ont montré que l’amélioration des paramètres d’inflammation apportée par l’intervention nutritionnelle était accrue dans le cas d’un microbiote « riche ». Dans une autre étude, Kovatcheva et al. ont rapporté que les individus en bonne santé présentant une plus grande abondance de Prevotella copri ont mieux répondu à un régime de trois jours enrichi en orge, que ceux ayant des niveaux inférieurs de cette bactérie, cela résultant d’une amélioration du métabolisme du glucose apportée par la bactérie [31]. De même, les effets d’un produit laitier fermenté, contenant une souche de Bifidobacterium animalis subsp lactis, sur le microbiote intestinal, dépendent de la composition du microbiote de l’individu qui l’ingère [32]. La composition du microbiote résident des sujets recrutés pour les études cliniques ou l’élaboration de nouveaux produits alimentaires fonctionnels devra donc être prise en compte dans le futur. |
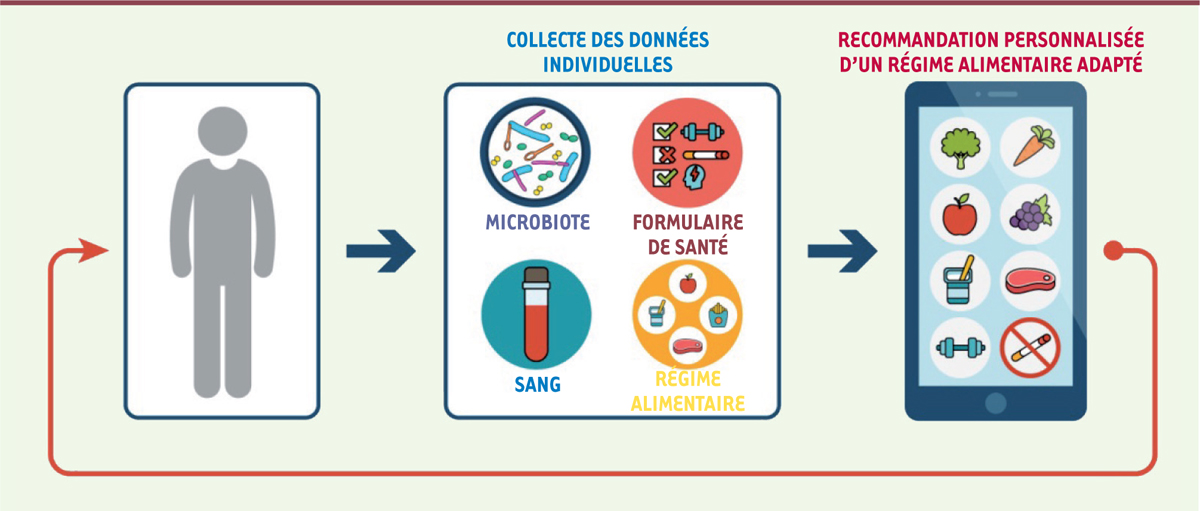
Vers une nutrition sur mesure ? L’un des défis futurs dans le domaine de la nutrition, sera la personnalisation de cette nutrition, tenant compte du microbiote intestinal de l’hôte et des facteurs environnementaux (Figure 1).
 | Figure 1.
Le concept de stratification d’individus fondé sur des variables telles que le microbiote intestinal, l’alimentation, le système immunitaire, permettra dans un proche avenir d’améliorer les recommandations nutritionnelles. |
Les données issues des études du microbiote intestinal sont cependant à mettre en regard des données cliniques et alimentaires des individus. Il est en effet important d’établir des critères qualité pour le recueil des analyses des données qui sont supposées être confrontées à celles concernant le microbiote intestinal. Par ailleurs, les méthodes statistiques classiques ne sont pas adaptées à l’analyse de ce type de données [33]. Il est alors nécessaire d’utiliser des méthodes calculatoires modernes telles que l’apprentissage machine (en anglais, machine learning). Dans une étude récente, Zeevi et al. ont utilisé ces techniques d’apprentissage machine pour générer des recommandations nutritionnelles personnalisées, calculées à partir des données cliniques, alimentaires et microbiennes. Ces recommandations sur mesure ont ainsi permis de réduire la réponse glycémique postprandiale [28]. |
Nous assistons aujourd’hui à une explosion des efforts de recherche publique et privée sur des approches thérapeutiques visant à « traiter » le microbiote de personnes déjà atteintes de maladies. Toutefois, il est important de ne pas délaisser l’étude des causes de la dysbiose à l’origine de ces pathologies, et d’augmenter l’effort scientifique pour trouver des solutions préventives qui préserveront l’eubiose dans le contexte dans lequel nous vivons actuellement. Des régimes plus personnalisés, avec notamment le développement d’une nouvelle génération de souches bactériennes à potentiel probiotique, capables de pallier les déficits fonctionnels du microbiote des individus, seront sans aucun doute à la base des solutions préventives qui seront déployées dans le futur. |
Les auteurs déclarent avoir un contrat de travail avec l’entreprise Danone.
|
1.
Cordain L, Eaton SB, Sebastian A, et al. Origins and evolution of the Western diet: health implications for the 21st century . Am J Clin Nutr. 2005; ; 81 : :341.–354. 2.
Sonnenburg ED, Smits SA, Tikhonov M, et al. Diet-induced extinctions in the gut microbiota compound over generations . Nature. 2016; ; 529 : :212.–215. 3.
Clayton JB, Vangay P, Huang H, et al. Captivity humanizes the primate microbiome . Proc Natl Acad Sci USA. 2016; ; 113 : :10376.–10381. 4.
Eaton S, Eaton S, III, Konner M. Paleolithic nutrition revisited: A twelve-year retrospective on its nature and implications . Eur J Clin Nutr. 1997; ; 51 : :207.–216. 5.
De Filippo C, Cavalieri D, Di Paola M, et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa . Proc Natl Acad Sci USA. 2010; ; 107 : :14691.–14696. 6.
Spanogiannopoulos P, Bess EN, Carmody RN, Turnbaugh PJ. The microbial pharmacists within us: a metagenomic view of xenobiotic metabolism . Nat Rev Microbiol. 2016; ; 14 : :273.–287. 7.
Lamas B, Richard ML, Leducq V, et al. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands . Nat Med. 2016; ; 22 : :598.–605. 8.
Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing . Nature. 2010; ; 464 : :59.–65. 9.
Forslund K, Hildebrand F, Nielsen T, et al. Disentangling type 2 diabetes and metformin treatment signatures in the human gut microbiota . Nature. 2015; ; 528 : :262.–266. 10.
Pedersen HK, Gudmundsdottir V, Nielsen HB, et al. Human gut microbes impact host serum metabolome and insulin sensitivity . Nature. 2016; ; 535 : :376.–381. 11.
Cotillard A, Kennedy SP, Kong LC, et al. Dietary intervention impact on gut microbial gene richness . Nature. 2013; ; 500 : :585.–588. 12.
Caporaso JG, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data . Nat Methods. 2010; ; 7 : :335.–336. 13.
Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome . Nature. 2011; ; 473 : :174.–180. 14.
Yatsunenko T, Rey FE, Manary MJ, et al. Human gut microbiome viewed across age and geography . Nature. 2012; ; 486 : :222.–227. 15.
Wu GD, Chen J, Hoffmann C, et al. Linking long-term dietary patterns with gut microbial enterotypes . Science. 2011; ; 334 : :105.–108. 16.
Kultima JR, Coelho LP, Forslund K, et al. MOCAT2: a metagenomic assembly, annotation and profiling framework . Bioinformatics. 2016; ; 32 : :2520.–2523. 17.
Treangen TJ, Koren S, Sommer DD, et al. MetAMOS: a modular and open source metagenomic assembly and analysis pipeline . Genome Biol. 2013; ; 14 : :R2.. 18.
Segata N, Waldron L, Ballarini A, et al. Metagenomic microbial community profiling using unique clade-specific marker genes . Nat Methods. 2012; ; 9 : :811.–814. 19.
Huson DH, Auch AF, Qi J, Schuster SC. MEGAN analysis of metagenomic data . Genome Res. 2007; ; 17 : :377.–386. 20.
Heintz-Buschart A, May P, Laczny CC, et al. Integrated multi-omics of the human gut microbiome in a case study of familial type 1 diabetes . Nat Microbiol. 2016; ; 2 : :16180.. 21.
Cantarel BL, Coutinho PM, Rancurel C, et al. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics . Nucleic Acids Res. 2009; ; 37 : :D233.–D2D8. 22.
Kanehisa M, Goto S, Sato Y, et al. Data, information, knowledge and principle: back to metabolism in KEGG . Nucleic Acids Res. 2014; ; 42 : :D199.–D205. 23.
Zeller G, Tap J, Voigt AY, et al. Potential of fecal microbiota for early-stage detection of colorectal cancer . Mol Syst Biol. 2014; ; 10 : :766.. 24.
McNulty NP, Wu M, Erickson AR, et al. Effects of diet on resource utilization by a model human gut microbiota containing Bacteroides cellulosilyticus WH2, a symbiont with an extensive glycobiome . PLoS Biol. 2013; ; 11 : :e1001637.. 25.
Scholz M, Ward DV, Pasolli E, et al. Strain-level microbial epidemiology and population genomics from shotgun metagenomics . Nat Meth. 2016; ; 13 : :435.–438. 26.
Schloissnig S, Arumugam M, Sunagawa S, et al. Genomic variation landscape of the human gut microbiome . Nature. 2013; ; 493 : :45.–50. 27.
Maldonado-Gómez María X, Martínez I, Bottacini F, et al. Stable engraftment of Bifidobacterium longum AH1206 in the human gut depends on individualized features of the resident microbiome . Cell Host Microbe. 2016; ; 20 : :515.–526. 28.
Zeevi D, Korem T, Zmora N, et al. Personalized nutrition by prediction of glycemic responses . Cell. 2015; ; 163 : :1079.–1094. 29.
Salonen A, Lahti L, Salojarvi J, et al. Impact of diet and individual variation on intestinal microbiota composition and fermentation products in obese men . ISME J. 2014; ; 8 : :2218.–2230. 30.
Jeffery IB, Lynch DB, O’Toole PW. Composition and temporal stability of the gut microbiota in older persons . ISME J. 2016; ; 10 : :170.–182. 31.
Kovatcheva-Datchary P, Nilsson A, Akrami R, et al. Dietary fiber-induced improvement in glucose metabolism is associated with increased abundance of Prevotella . Cell Metab. 2015; ; 22 : :971.–982. 32.
Zhang C, Derrien M, Levenez F, et al. Ecological robustness of the gut microbiota in response to ingestion of transient food-borne microbes . ISME J. 2016; ; 10 : :2235.–2245. 33.
Knights D, Costello EK, Knight R. Supervised classification of human microbiota . FEMS Microbiol Rev. 2011; ; 35 : :343.–359. 34.
Burcelin R, Nicolas S, Blasco-Baque V. Microbiotes et maladies métaboliques : de nouveaux concepts pour de nouvelles stratégies thérapeutiques . Med Sci (Paris). 2016; ; 32 : :952.–960. 35.
Rahmouni O, Dubuquoy L, Desreumaux P, Neut C. Microbiote intestinal et développement des maladies inflammatoires chroniques de l’intestin . Med Sci (Paris). 2016; ; 32 : :968.–973. 36.
Gaboriau-Routhiau V, Cerf-Bensussan N. Microbiote intestinal et développement du système immunitaire . Med Sci (Paris). 2016; ; 32 : :961.–967. 37.
Lamas B, Richard ML, Sokol H. CARD9 et colite : un pont entre dysbiose et immunité . Med Sci (Paris). 2016; ; 32 : :933.–936. 38.
Weissenbach J, Sghir A. Microbiotes et Métagénomique . Med Sci (Paris). 2016; ; 32 : :937.–943. |