| |
| Med Sci (Paris). 32(11): 1017–1019. doi: 10.1051/medsci/20163211019.Les éléments génétiques mobiles d’insectes sautent fréquemment dans les génomes de virus Clément Gilbert1* and Jean Peccoud1 1Laboratoire Écologie et Biologie des Interactions, équipe Écologie Évolution Symbiose, université de Poitiers, UMR CNRS 7267, 6, rue Michel Brunet, F-86073Poitiers, France MeSH keywords: Animaux, Éléments transposables d'ADN, ADN viral, Évolution moléculaire, Transfert horizontal de gène, Génome viral, Interactions hôte-pathogène, Humains, Mutagenèse par insertion, génétique, virologie |
Les virus ne peuvent se répliquer que lorsqu’ils infectent des cellules de leurs hôtes. La réplication virale implique donc une forte proximité physique entre génomes viraux et génomes cellulaires. Cette proximité offre de nombreuses opportunités de recombinaison, ou d’échanges de gènes, entre les virus et leurs hôtes. On sait aujourd’hui que de nombreux gènes cellulaires ont été capturés par des lignées de virus au cours d’évènements de recombinaison accidentels. Le premier gène connu de ce type est src, découvert dans le génome du virus du sarcome du poulet (rous sarcoma virus, RSV) et codant une tyrosine kinase impliquée dans le contrôle de la prolifération cellulaire. Depuis la fin des années 1970, on sait que RSV a hérité src de son hôte [1]. Le mécanisme de ce transfert est bien connu. RSV étant un rétrovirus, c’est la machinerie transcriptionnelle cellulaire qui réplique son génome (à ARN simple brin) intégré dans celui de son hôte. Une copie de RSV s’est ainsi intégrée dans le génome du poulet en amont du gène src, ce qui a engendré la production de longs transcrits accolant le génome RSV à l’ARNm de src. Cette fusion transcriptionnelle, ou transduction, a généré un nouveau génome viral qui a pu être encapsidé et répliqué. La capture de src s’est avérée bénéfique à RSV puisque la surexpression de ce gène suite à l’infection par le virus stimule la prolifération cellulaire, augmentant la production de particules virales. Dans le même temps, cette prolifération cellulaire induit la formation de tumeurs chez le poulet. Src est donc aussi le premier oncogène viral d’origine cellulaire à avoir été découvert [2]. Depuis, de nombreux gènes cellulaires (oncogènes ou autres) ont été trouvés dans divers types de virus, notamment des virus à grands génomes d’ADN double brin [3, 4]. L’herpèsvirus humain de type 8 est par exemple ainsi connu pour avoir hérité de près du tiers de ses 82 gènes d’un hôte vertébré ancestral [5]. |
Les transferts horizontaux d’ADN d’hôtes à virus sont peu étudiés Une forte proportion des gènes cellulaires capturés par les virus à ADN contribuent à l’atténuation de la réponse antivirale des organismes infectés [6]. Le poxvirus MCV (molluscum contagiosum virus) possède de tels gènes, qui codent des protéines fixant l’interleukine 18, diminuant ainsi la production d’interféron gamma et la réponse antivirale médiée par les lymphocytes T1 auxiliaires [7]. Il semble logique que la plupart des gènes d’origine cellulaire caractérisés dans les virus leur soient bénéfiques. En effet, un gène cellulaire ne peut être détecté qu’après avoir atteint une fréquence importante dans l’espèce virale étudiée, ce qui exclut un effet délétère sur le taux de croissance virale. De fait, on ne sait presque rien sur le nombre et la nature des transferts de gènes cellulaires non bénéfiques aux virus et présents à faibles fréquences dans les populations virales. Pourtant, on peut raisonnablement penser que ces transferts sont les plus fréquents, comme le sont les mutations génétiques non bénéfiques en général. |
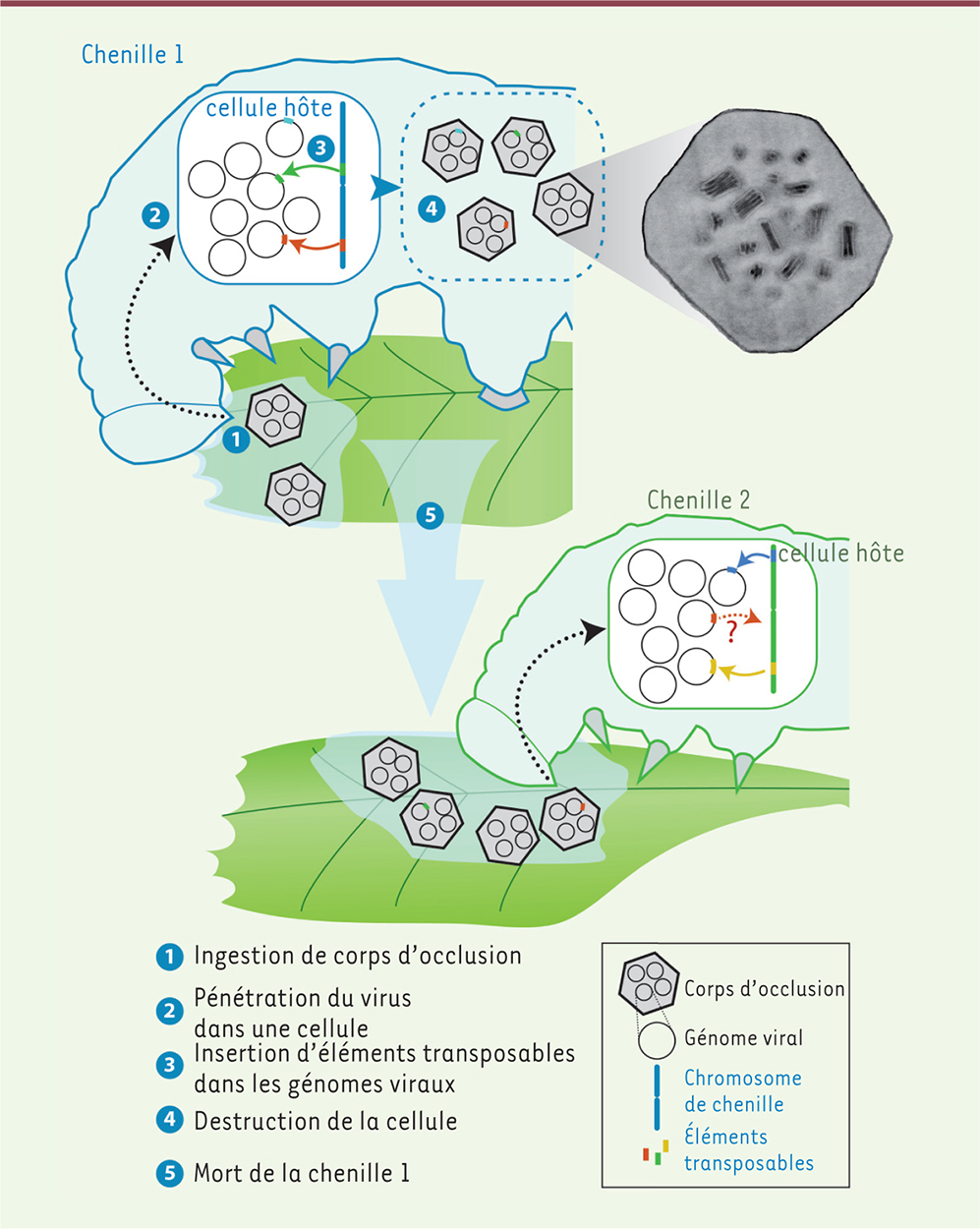
De nombreuses séquences d’ADN hôte sautent dans les génomes viraux lors d’une infection Afin de mieux comprendre la dynamique micro-évolutive des transferts de gènes des hôtes vers leur virus, nous avons récemment analysé des génomes de populations virales au cours d’infections [8]. Nous avons séquencé un grand nombre de génomes de plusieurs lignées expérimentales du virus Autographa californica multicapsid nucleopolyhedrovirus (AcMNPV, de la famille des Baculoviridae). AcMNPV possède un grand génome à ADN double brin circulaire de 134 kb et infecte de nombreuses espèces de lépidoptères (papillons). Nous avons généré dix lignées d’infections au laboratoire sur des chenilles d’un papillon appelé légionnaire de la betterave, et dix autres lignées sur des chenilles de la fausse arpenteuse du chou. Pour chacune des vingt lignées, le virus a été propagé pendant dix cycles d’infection successifs. Nous avons extrait les particules virales issues des dixièmes cycles d’infection pour les vingt lignées, ainsi que la population de virus ayant servi à initier ces lignées. L’ADN viral purifié nous a permis de séquencer l’équivalent d’environ 500 000 génomes d’AcMNPV. Les millions de séquences obtenues représentent de courts fragments d’ADN d’environ 100 nucléotides et sont appelées « lectures ». Afin de détecter et caractériser des gènes non viraux présents dans les particules virales, nous avons extrait de nos séquences les lectures ne présentant aucune ressemblance avec le génome d’AcMNPV (sa séquence étant connue), et nous les avons assemblées pour reconstituer des séquences plus longues appelées « contigs ». La majorité de ces contigs s’est avérée appartenir aux génomes des papillons. Afin d’évaluer le nombre de transferts de gènes des papillons au virus au cours de ces infections expérimentales, et d’exclure une simple contamination de notre échantillon par de l’ADN de papillon, nous avons recherché les lectures composées pour partie d’ADN viral, et pour partie d’ADN de papillon (homologue à un contig). Cette approche nous a permis de découvrir pas moins de 27 504 fragments d’ADN de papillon intégrés dans l’ensemble des génomes viraux séquencés. Ces intégrations sont réparties dans la totalité des 20 lignées d’infection. Ceci suggère qu’à chaque fois qu’AcMNPV infecte un papillon, un grand nombre de fragments d’ADN sont transférés des chromosomes du papillon vers le virus (Figure 1).
 | 3Figure 1.
Mécanisme de transferts d’ADN entre insecte hôte et baculovirus. En haut, une chenille ingère des corps d’occlusion d’un baculovirus contenant des virions (un corps d’occlusion en coupe est photographié par microscopie électronique à transmission). Ceux-ci répliquent leur génome à ADN double brin circulaire dans les noyaux des cellules de la chenille. Une partie de ces génomes (environ 5 %) acquiert des éléments transposables (segments vert, rouge et bleu) provenant des chromosomes de l’insecte. Certains de ces génomes sont ensuite encapsidés dans des corps d’occlusion qui persistent après la mort de la chenille, liquéfiée par le virus. Une autre chenille (en bas) pourra ingérer ces corps d’occlusion, de sorte que ses éléments transposables continuent d’alimenter le flux d’ADN d’insecte à virus. Par ailleurs, certains éléments transposables (en rouge) hérités de la chenille précédemment infectée, et véhiculés par les génomes viraux ingérés, pourraient éventuellement sauter dans les chromosomes de la seconde chenille (flèche en pointillés rouge), entraînant un transfert horizontal d’ADN entre lignées d’insectes (©Julien Gaillard, Département des Microscopies, Université François Rabelais de Tours). |
|
Un flux continu d’ADN hôte dans les génomes viraux L’ensemble des 27 504 fragments d’ADN de papillons intégrés a pu être attribué à 86 contigs différents. La plupart de ces contigs se sont donc intégrés de multiples fois, à différents sites des génomes viraux. De manière frappante, la majorité des 86 contigs (plus précisément 69 contigs, représentant 19 899 fragments d’ADN) se sont avérés être des éléments transposables (ET). Les ET sont des séquences capables de se mouvoir d’un locus génomique à un autre tout en se dupliquant dans les génomes [9]. Les 17 contigs restants ne ressemblent à aucun ET ou gène connu et annoté de papillon. L’abondance d’ET et l’absence d’autres gènes parmi les séquences de papillons intégrées dans les populations virales contrastent avec nos connaissances de la composition des génomes viraux, dans lesquels les gènes cellulaires sont bien plus fréquents que les ET. Il est néanmoins intuitif que les transferts d’ET, capables de sauter dans les génomes, soient beaucoup plus fréquents que les transferts de gènes « non-sauteurs ». Par ailleurs, l’insertion d’un ET, qui n’a généralement pas d’autre fonction que de se déplacer, a peu de chance de profiter au virus. Cette supposition est confirmée par le fait qu’aucune des insertions d’ADN de papillon identifiées dans la population de virus initiale n’a été retrouvée après 10 cycles d’infection dans les 20 lignées virales. Les virus porteurs d’un fragment d’ADN de papillon sont donc rapidement éliminés des populations virales, en moins de 10 cycles d’infection, sans doute parce qu’ils se répliquent moins bien que ceux ne portant pas d’insertion. Dans le même temps, à chaque infection, de nouvelles séquences de papillons s’intègrent dans les génomes nouvellement répliqués. Il y aurait donc un flux et un renouvellement constant de séquences de papillons s’intégrant dans les génomes viraux à mesure que le virus se réplique et se propage d’un individu hôte à l’autre. Le transfert et la fixation de gènes cellulaires (non ET) dans les populations virales seraient des évènements bien plus rares, n’ayant pas été observés dans nos lignées expérimentales. Ce flux soutenu d’ET dans les populations virales soulève la question de son impact sur le virus mais aussi sur l’hôte. Nous avons calculé qu’en moyenne 5 génomes viraux sur 100 portent au moins une séquence de papillon. Les dizaines de milliers de virus qu’une chenille ingère en s’infectant portent ainsi une centaine d’ET, dont certains pourraient prendre le trajet retour et sauter du génome du virus à celui du papillon nouvellement infecté. Ainsi, les résultats de cette étude renforcent l’hypothèse selon laquelle les virus pourraient faciliter les transferts horizontaux de matériel génétique entre animaux [10–12] (→).
(→) Voir la Nouvelle de C. Gilbert et al., m/s décembre 2010, page 1025
En accord avec cette hypothèse, nous avons montré que 21 des 69 ET de papillons intégrés dans les génomes d’AcMNPV avaient été transférés horizontalement entre plusieurs espèces d’insectes, dont certains papillons hôtes de baculovirus. Enfin, de par leur probable effet délétère sur les virus et leur fréquence relativement élevée, les insertions d’ET dans les génomes viraux pourraient être bénéfiques aux hôtes infectés. Il serait intéressant de tester l’hypothèse provoquante selon laquelle l’activité des ET d’un organisme pourrait contribuer à le protéger contre les virus en inactivant certains de leurs gènes et/ou en induisant de nombreuses cassures d’ADN dans leurs génomes. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
4.
Stehelin D, Varmus HE, Bishop JM, Vogt PK. DNA related to the transforming gene(s) of avian sarcoma viruses is present in normal avian DNA . Nature. 1976; ; 260 : :170.–173. 5.
Vogt PK. Retroviral oncogenes: a historical primer . Nat Rev Cancer. 2012; ; 12 : :639.–648. 6.
Theze J, Takatsuka J, Nakai M, et al. Gene acquisition convergence between entomopoxviruses and baculoviruses . Viruses. 2015; ; 7 : :1960.–1974. 7.
Hughes AL, Irausquin S, Friedman R. The evolutionary biology of poxviruses . Infect Genet Evol. 2010; ; 10 : :50.–59. 8.
Holzerlandt R, Orengo C, Kellam P, Alba MM. Identification of new herpesvirus gene homologs in the human genome . Genome Res. 2002; ; 12 : :1739.–1748. 9.
McFadden G, Murphy PM. Host-related immunomodulators encoded by poxviruses and herpesviruses . Curr Opin Microbiol. 2000; ; 3 : :371.–378. 10.
Xiang Y, Moss B. IL-18 binding and inhibition of interferon gamma induction by human poxvirus-encoded proteins . Proc Natl Acad Sci USA. 1999; ; 96 : :11537.–11542. 11.
Gilbert C, Peccoud J, Chateigner A, et al. Continuous influx of genetic material from host to virus populations . PLoS Genet. 2016; ; 12 : :e1005838.. 12.
Craig NLCR, Craigie R, Gellert M, Lambowitz AM. Mobile DNA II . Washington (DC): : American Society for Microbiology Press; , 2002 : :1254. p. 13.
Schaack S, Gilbert C, Feschotte C. Promiscuous DNA: horizontal transfer of transposable elements and why it matters for eukaryotic evolution . Trends Ecol Evol. 2010; ; 25 : :537.–546. 14.
Gilbert C, Chateigner A, Ernenwein L, et al. Population genomics supports baculoviruses as vectors of horizontal transfer of insect transposons . Nat Commun. 2014; ; 5 : :3348.. 15.
Gilbert C, Schaack S, Feschotte C. Quand les éléments génétiques mobiles bondissent entre espèces animales . Med Sci (Paris). 2010; ; 26 : :1025.–1027. |