| |
| Med Sci (Paris). 33(11): 971–978. doi: 10.1051/medsci/20173311014.Les nouvelles technologies d’analyses ADN au service des enquêtes judiciaires François-Xavier Laurent,1a Geoffrey Vibrac,2 Aurélien Rubio,3* Marie-Thérèse Thévenot,1 and Laurent Pène1 1Institut national de police scientifique, laboratoire de police scientifique de Lyon, 31, avenue Franklin Roosevelt, 69134Écully Cedex, France 2Université de Lorraine, Institut François Gény - Institut de Sciences criminelles et de Droit médical (EA 7301), 13, place Carnot, 54000Nancy, France 3Cour d’appel de Nancy, 3, rue Suzanne Regnault-Gousset, 54000Nancy, France |
Un déplacement toutes les minutes : c’est le rythme d’intervention moyen de la police technique et scientifique en France. Devenue incontournable dans le système judiciaire français et mondial, elle suscite beaucoup d’intrigues et de fascinations. Si quelques clichés découlent des séries télévisées, avec des scénarios rocambolesques et des experts multitâches à la pointe de la technologie, elles témoignent tout de même d’une discipline scientifique en perpétuelle évolution, la criminalistique. Cette discipline regroupe l’ensemble des méthodes d’analyses scientifiques mises en œuvre par la justice et la police pour concourir à la preuve de la commission d’une infraction et d’en identifier son auteur ou discriminer un individu. Fondée sur le postulat d’Edmond Locard, professeur de médecine légale et fondateur à Lyon, en 1910, du premier laboratoire de police scientifique au monde, elle repose sur le fait qu’un malfaiteur laisse sur les lieux de l’infraction des traces de son passage et emporte avec lui des éléments qui détermineront sa présence et son action sur la scène d’infraction. Depuis près de trente années, la biologie a révolutionné les preuves scientifiques dans le monde judiciaire. Cette synthèse a donc pour but de faire un point de situation sur l’apport des nouvelles technologies d’analyse ADN en génétique criminalistique. |
ADN, profil génétique et criminalistique En matière pénale, la méthode d’identification de référence est l’identification par empreinte génétique. Initiée par Sir Alec Jeffreys, en 1983, elle consistait en l’analyse par Southern blot, de polymorphismes de longueur de fragments de restriction (ou RFLP pour restriction fragment length polymorphism) [1]. L’inconvénient majeur de cette technique étant les grandes quantités d’ADN nécessaires, elle fût rapidement remplacée par la PCR (polymerase chain reaction), méthode d’amplification de l’ADN in vitro [2]. L’utilisation de la PCR en criminalistique décupla les capacités d’analyse, permettant d’obtenir des empreintes génétiques à partir de traces contenant peu d’ADN, ou de l’ADN dégradé. Depuis près de 30 ans, les variations génétiques recherchées concernent les microsatellites, désignés par l’acronyme STR (short tandem repeats), des séquences courtes de 2 à 6 paires nucléotidiques répétées jusqu’à plusieurs dizaines de fois [3,4]. Les STR sont amplifiés par PCR multiplexe à l’aide d’amorces spécifiques dont l’une est couplée à un fluorochrome. Les amplicons ainsi obtenus sont ensuite séparés et détectés par électrophorèse capillaire (
Figure 1
). Les résultats sont représentés par des pics de fluorescence dont la position sur l’axe des abscisses correspond à la taille du fragment amplifié, constitué du STR et des régions flanquantes. La validation des profils génétiques permet d’éliminer les artefacts d’analyse pour ne garder que les pics correspondant aux allèles présents dans la trace biologique [5]. Si les marqueurs STR sont polymorphes, le nombre d’allèles, pour chacun, est relativement faible (quelques dizaines au maximum) et sont partagés par 5 à 20 % de la population, en moyenne. Le pouvoir de discrimination de cette méthode repose sur l’analyse combinée d’une vingtaine de marqueurs STR autosomaux, ce qui permet d’obtenir une probabilité d’apparition d’un profil génétique dans une population de référence, de l’ordre d’une sur plusieurs milliards de milliards. En France, la liste des marqueurs génétiques autosomaux dont l’analyse est explicitement autorisée dans le cadre d’une enquête judiciaire figure dans l’article A38 du Code de procédure pénale. Ce sont uniquement ces marqueurs qui peuvent être utilisés pour alimenter les profils référencés dans le Fichier national automatisé des empreintes génétiques (FNAEG), une base de données nationale créée en 1998 [6].
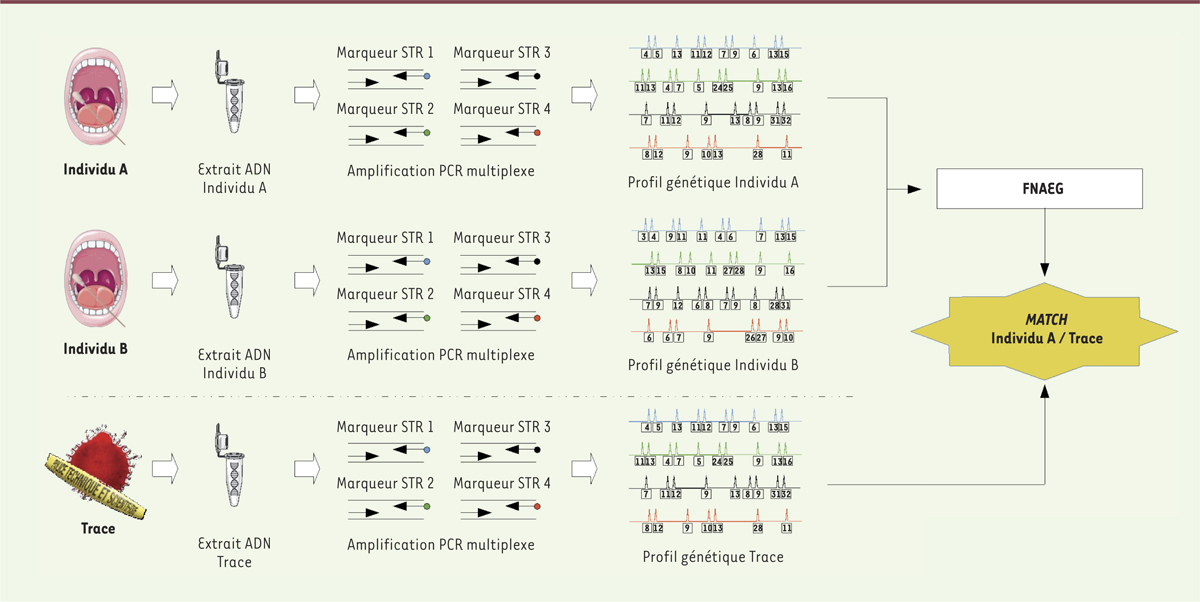
 | Figure 1.
Principe de l’identification génétique par établissement d’un profil génétique STR. Les prélèvements buccaux des individus A et B sont utilisés pour en extraire l’ADN. L’amplification par PCR d’une vingtaine de marqueurs STR et la séparation des amplicons par leur taille permet l’établissement d’un profil génétique unique. Ces profils génétiques sont envoyés au Fichier national automatisé des empreintes génétiques (FNAEG). L’analyse d’une trace de sang, découverte sur une scène de crime, permet l’établissement d’un profil génétique identique à celui de l’individu A. STR : short tandem repeats ; MATCH : concordance. |
Les infractions passibles1, d’un prélèvement génétique et permettant d’alimenter cette base de données, correspondent à celles qui impliquent l’ensemble des personnes mises en cause ou condamnées pour des actes délictuels ou criminels, à l’exception des infractions au Code de la route, des outrages simples et des délits financiers. Le FNAEG permet donc de centraliser les profils génétiques obtenus à partir des traces biologiques prélevées sur les scènes d’infraction, et ceux des individus mis en cause ou condamnés. Les laboratoires de police scientifique réalisent l’extraction de l’ADN à partir de tout type de matrices biologiques, qu’elles soient des traces « riches » (sang, sperme, salive) ou « pauvres » en ADN (traces de contact contenant des cellules épithéliales, éléments pileux, os, pellicules capillaires, sueur, excréments, etc.). Au 31 mai 2017, le nombre de profils génétiques d’individus inscrits au FNAEG était de l’ordre de 2,5 millions, le nombre de traces, d’environ 400 0002,. Le FNAEG comporte également un moteur de rapprochement qui permet de proposer des correspondances entre un individu et une trace (qui peuvent indiquer, grâce au principe de l’échange de Locard [voir plus haut], la présence de l’individu sur la scène d’infraction), des correspondances entre des traces (indiquant l’implication d’un individu dans des infractions en série) et même, des correspondances entre des individus (les délinquants utilisant parfois des alias pour éviter l’aggravation de leurs peines en cas de récidive). D’autres types de profils génétiques peuvent être enregistrés au FNAEG, pour des cas particuliers (découverte de cadavre non identifié, ou profil établi à partir d’objets personnels ayant appartenu à une personne disparue). Lors d’un rapprochement proposé par le fichier, l’expert3 en charge du dossier effectue une confirmation de la correspondance. Cette confirmation nécessite une analyse statistique qui repose sur le calcul de la probabilité que deux individus pris au hasard dans une même population aient le même génotype, c’est le « random match probability » [7]. À l’échelle de l’individu, l’ADN est une molécule extrêmement stable, ce qui garantit que l’intégrité de l’information qu’elle contient soit conservée au cours des divisions cellulaires. C’est d’ailleurs cette notion de faible intra-variabilité génétique qui est relevée par Raphaël Coquoz et al. dans leur ouvrage « Preuve par l’ADN, la génétique au service de la Justice » [8]. Les auteurs expliquent que « [l’] on peut admettre que n’importe quel prélèvement de n’importe quelle partie du corps prélevée à n’importe quel moment de l’existence de l’individu produit toujours le même profil ADN ». C’est d’ailleurs grâce à ce principe que des rapprochements de profils génétiques peuvent être réalisés dans des affaires classées (cold cases) avec parfois plusieurs dizaines d’années entre une trace récupérée sur une scène d’infraction, et un prélèvement salivaire de l’individu mis en cause. Si la méthode d’identification par génotypage STR paraît relativement simple, la validation des profils génétiques peut être sérieusement compliquée lors de l’analyse d’échantillons biologiques complexes. Le droit encadrant très précisément ce type d’analyses génétiques, de même que les conditions d’alimentation du FNAEG, il est relativement courant qu’un certain nombre de traces ne puissent pas produire de profils génétiques exploitables, empêchant de facto une aide précieuse à l’enquête judiciaire. L’objectif de cette revue est de présenter les récentes évolutions techniques en génétique criminalistique et des exemples pour lesquels ces nouvelles technologies permettent d’optimiser les chances d’identification de suspects. |
Dégradation de l’ADN et interférences dans l’analyse criminalistique En génétique médicale, les échantillons biologiques analysés sont généralement riches en ADN et conservés dans des conditions de température et d’humidité optimales afin de garantir leur intégrité. Ce n’est évidemment pas le cas pour ceux issus des traces biologiques prélevées sur des scènes d’infraction, compte-tenu de leur diversité et de leur intégrité très souvent altérée (à cause de variations importantes de température et/ou d’humidité, ou de l’exposition prolongée aux rayons ultra-violets solaires4). Le dépôt de traces sur des supports non contrôlés (tissus et objets de la vie courante) peut également être contaminé par des produits (le calcium, le colorant indigo, le collagène, etc.) qui seront co-purifiés avec l’ADN et qui pourront interférer de façon partielle ou totale avec les analyses génétiques réalisées par amplifications par PCR [9]. Il n’est donc pas surprenant qu’un grand nombre de profils génétiques analysés en génétique criminalistique soient des profils partiels pour lesquels un ou plusieurs marqueurs examinés ne peuvent pas être pris en compte car ne répondant pas à l’ensemble des critères de validation des profils génétiques généralement mis en place par chaque laboratoire (intensité de fluorescence trop faible, balance d’hétérozygotie sous le seuil fixé, etc.). D’après les chiffres observés au sein du laboratoire de police scientifique de Lyon, le taux d’échantillons présentant un profil partiel dû à une dégradation est d’environ 15 %. Ce chiffre élevé a deux explications : la première correspond à la proportion d’échantillons qui proviennent de traces de contact (correspondant à des cellules épidermiques déposées sur un support solide, lors de la manipulation d’un objet par exemple) dans lesquelles l’ADN a été fragmenté au cours du processus de desquamation. L’autre raison découle directement du type de marqueurs génétiques utilisé par les laboratoires de génétique criminalistique : les STR. En effet, leur analyse nécessite une amplification de séquences pouvant aller jusqu’à 450 paires de bases (pb). Or les grands fragments d’ADN sont sensibles à la dégradation. Ils sont donc les premiers marqueurs à ne pouvoir être amplifiés lors de l’étape de PCR. Outre le fait que l’amplification soit impossible pour certains de ces marqueurs, la dégradation peut affecter sélectivement l’allèle le plus grand chez un individu hétérozygote, c’est ce que l’on appelle un « drop-out » (dans ce cas, seul l’allèle intact, le plus court, sera détecté). Le risque majeur de ce biais est la transmission au fichier d’un profil génétique issu d’une trace qui soit erroné, contenant un marqueur qui sera noté comme homozygote alors qu’en réalité, celui-ci était hétérozygote. Une correspondance peut être établie par le FNAEG5, en présence d’un seul marqueur erroné. Cependant, aucun rapprochement ne sera accepté si le nombre de marqueurs mal interprétés est supérieur, puisque la probabilité de concordance aléatoire augmentera alors fortement. Dans ce cas, qui s’avère relativement courant, l’expert peut décider de rejeter l’un ou plusieurs des marqueurs, et transmettre néanmoins le profil partiel au fichier. Si ce correctif permet d’éviter un rejet du profil, le fait de disposer de profils génétiques incomplets a une incidence sur le calcul de la probabilité de coïncidence fortuite pour deux profils génétiques comparés, réduisant ainsi la valeur de la force probante. Ceci provoque une augmentation du nombre de profils qui ont été rapprochés et, de manière directe, rend complexe les enquêtes de police puisque le fichier propose alors autant de pistes à élucider. Le Code de procédure pénale ne donne pas d’indication quant au nombre minimal de marqueurs génétiques nécessaires pour valider un profil et être transmis au fichier. Cependant, afin de limiter un nombre de rapprochements trop important, dans le cas de transmission de profils très partiels, un nombre minimal de 6 marqueurs génétiques renseignés dans un profil (dont l’amélogénine, marqueur discriminant le sexe de l’individu6) a été défini. Certains kits commerciaux comprennent des amorces qui s’apparient au plus près des STR, ce qui permet de réduire autant que possible la taille des amplicons [10]. Mais, avec les nouvelles méthodes de séquençage (next generation sequencing ou NGS), une révolution technologique est apparue en criminalistique. La grande nouveauté de ces techniques est leur indépendance par rapport aux électrophorèses capillaires, qui présentent des capacités d’analyse de fluorescence limitées. Les analyses classiques nécessitent en effet que la taille de tous les amplicons (correspondants aux différentes formes alléliques d’un marqueur génétique) soit unique pour un fluorochrome donné. Ceci implique donc que les intervalles de taille de chaque marqueur ne se chevauchent pas, ce qui rendrait impossible l’identification de l’allèle par sa taille, d’où la présence de tailles alléliques élevées (plus de 450 pb). Le séquençage s’affranchit de cette limitation. Cela permet de réduire drastiquement la taille de l’ensemble des amplicons nécessaires à l’analyse. Ainsi, la moyenne des tailles maximales des amplicons pour l’ensemble des marqueurs STR autosomaux (en prenant le nombre maximal de répétitions pour chaque STR) du kit GlobalFiler (Applied Biosystems) est de 232 pb contre 188 pb pour le kit ForenSeq DNA Signature Prep (Illumina), analysable sur le séquenceur MiSeq FGx (Illumina) [11,12]. Cependant, si les amorces restent positionnables au plus près du STR, dans des régions flanquantes sans mutations ponctuelles connues (pour garantir une amplification chez l’ensemble des individus), une taille minimale, correspondant à la répétition du motif lui-même, reste une limite. La technologie NGS apporte néanmoins une innovation supplémentaire pour des traces biologiques très dégradées pour lesquelles même des profils génétiques extrêmement partiels peuvent être obtenus. Plusieurs solutions permettent désormais l’analyse d’autres polymorphismes dont les SNP (single nucleotide polymorphism, ou polymorphisme nucléotidique), des mutations isolées qui sont parfaitement adaptées à la criminalistique. Par leur caractère ponctuel, la taille des amplicons pour l’analyse des SNP peut en effet se limiter à une cinquantaine de nucléotides. Le caractère bi-allélique des SNP ne leur confère cependant pas le même pouvoir discriminant que des STR autosomaux qui, eux, bénéficient d’un nombre important d’allèles dus au polymorphisme de taille. Ce caractère limite donc fortement leur intérêt en cas de mélange de plusieurs ADN. Statistiquement, l’analyse d’une centaine de SNP autosomaux pourrait ainsi égaler le pouvoir de discrimination obtenu avec une vingtaine de STR autosomaux. Ce nombre pourrait être réduit, par l’utilisation de plusieurs polymorphismes touchant le même amplicon, ce qui pourrait générer de nombreux haplotypes et augmenter le pouvoir de discrimination [13]. Cependant, la restriction de l’article A38 du Code de procédure pénale, limitant la validation des profils pour leur inscription au FNAEG, ou aux autres bases de données internationales (selon le traité de Prüm7), à une liste définie de marqueurs STR, empêche de facto la transmission de profils SNP. L’analyse de ce type de polymorphisme reste néanmoins possible dans le cadre d’affaires spécifiques dans lesquelles un profil génétique issu d’une trace SNP, provenant d’une trace très dégradée, peut faire l’objet d’une comparaison avec un profil génétique SNP d’un individu suspecté. Si les nouvelles technologies peuvent aider à analyser de nouvelles informations génétiques dans un but d’identification, elles ne permettent pas en revanche, d’obtenir des données à partir de séquences génétiques définitivement détruites par des conditions environnementales extrêmes ou de conservation inadaptées. |
La difficulté des mélanges d’ADN Si la détérioration de l’ADN est, en criminalistique, à l’origine de nombreuses difficultés, ce sont les mélanges d’ADN qui représentent le plus grand défi. Tout d’abord parce que les règles de validation et d’interprétation d’une trace biologique, composée de deux ou plusieurs ADN, et les règles utilisées pour la validation de profils génétiques provenant de contributeurs uniques sont incompatibles entre elles. Ensuite parce que le fichier de référence n’autorise pas l’enregistrement de profils mélangés : il sera nécessaire que l’expert interprète ce mélange afin de tenter de déduire l’implication d’un ou plusieurs contributeurs. Loin d’être un cas marginal, les mélanges d’ADN représentent plus de 30 % de l’ensemble des profils génétiques analysés au laboratoire de police scientifique de Lyon. Dès lors qu’un objet est manipulé par plusieurs individus, des traces biologiques peuvent en effet être prélevées en même temps conduisant ainsi à un mélange d’ADN au sein de l’échantillon. Dès la mise en évidence, sur au moins deux marqueurs, de plus de deux allèles, l’hypothèse de la présence d’ADN issu d’au moins deux individus, au sein de la trace biologique, doit être testée. Cette hypothèse est renforcée par la présence de balances d’hétérozygotie déséquilibrées pour certains marqueurs, preuve d’un effet cumulatif d’allèles de taille identique provenant d’individus différents qui se traduit par des intensités de fluorescence variables [14–17]. Les mélanges d’ADN peuvent être séparés en plusieurs catégories (
Figure 2
). Les mélanges dont l’un des contributeurs est qualifié de « majoritaire » ; ils se caractérisent par des allèles qui se distinguent par leur intensité de fluorescence lors de l’analyse, plus élevée que les autres allèles du mélange. L’intensité des différents allèles étant corrélée à la proportion respective de chaque contributeur dans le mélange, les allèles les plus intenses seront associés au contributeur majoritaire, les allèles les moins intenses, aux contributeurs minoritaires. Selon la qualité du profil, il pourra être difficile de distinguer un allèle faible d’un artéfact analytique (stutter, pull-up, épaulement, ou bruit de fond important), très couramment rencontré dans les mélanges. Les profils génétiques obtenus à partir de mélanges peuvent être également qualifiés de « valides ». Ce sont des mélanges d’ADN comportant deux contributeurs dans lesquels la présence du contributeur minoritaire est supérieure à 25 %. Il est alors impossible d’extraire l’ADN provenant d’un contributeur du mélange, les règles de contributions appliquées dans le cas de profils majoritaires n’étant pas utilisables dans ce cas, sauf si le profil génétique du deuxième contributeur est connu. L’un des exemples souvent rencontré de ce type de mélange concerne celui constitué de l’ADN d’une victime et de celui d’un agresseur présumé. Le profil de la victime étant recueilli lors de l’enquête (pour discriminer les traces relevées sur la scène d’infraction), il est néanmoins possible de supprimer les allèles caractéristiques de la victime et donc de déduire le profil de l’agresseur. Les profils qualifiés de « mélanges à comparer » sont, quant à eux, des mélanges à forte intensité, composés de deux ou trois contributeurs, et pour lesquels aucun profil majoritaire n’est exploitable. Dans ces cas particuliers, la probabilité que l’ADN d’un suspect, pour lequel il existe des indices graves et concordants, puisse être présent dans le mélange sera évaluée. Si les allèles du suspect ne sont pas présents sur l’un ou plusieurs marqueurs du profil génétique, il sera possible d’exclure l’hypothèse de sa présence. Dans le cas contraire, il sera indispensable d’évaluer statistiquement le poids de la preuve correspondant à un rapport de vraisemblance (likelihood ratio en anglais) [18–20]. Deux hypothèses, mutuellement exclusives, correspondant, l’une, à la présence de l’ADN de l’individu dans le mélange (hypothèse de l’accusation) et, l’autre, à la présence d’un autre individu pris au hasard dans la population (hypothèse de la défense) seront alors confrontées. La valeur du rapport de vraisemblance (c’est-à-dire le rapport des probabilités d’observer un résultat analytique en considérant les deux hypothèses) aiguillera l’expert pour déterminer la présence probable, ou non, de l’ADN du suspect dans le mélange. Dans ce cas, les calculs statistiques semblent donc incontournables pour juger de la pertinence d’une hypothèse. Cependant, le développement de nouvelles méthodes innovantes, inspirées de celles employées en biologie cellulaire et en génomique, apparaissent très prometteuses quant à la déconvolution des mélanges complexes. Elles concernent, tout d’abord, la phase pré-analytique, c’est à dire le prélèvement de la trace biologique et l’extraction du matériel biologique qu’elle contient. Dans les cas d’agressions sexuelles, par exemple, les prélèvements vaginaux ou anaux peuvent produire des mélanges d’ADN dans lesquels le profil de la victime sera très majoritaire, principalement à cause du détachement de cellules au niveau des muqueuses. Ces prélèvements donnent généralement des profils masculins faibles, ou uniquement féminins. L’analyse de marqueurs STR du chromosome Y (donc présents uniquement dans le profil masculin), pourront alors permettre d’identifier un haplotype et donc de remonter une lignée paternelle8. Une autre option est de procéder à une lyse différentielle [21,22]. Cette méthode d’isolement permet la séparation des spermatozoïdes des autres cellules (essentiellement celles de la victime) en deux fractions qui pourront être analysées séparément, les profils masculins uniques recueillis seront alors identifiés et pourront être référencés au FNAEG. Cette méthode de lyse différentielle ne semble cependant pas pouvoir être appliquée pour d’autres types cellulaires, notamment les cellules salivaires, sanguines ou de contact qui seront en fait recueillies dans une même fraction et donc indiscernables. Une dernière alternative consiste en la séparation des cellules par microdissection laser [23]. Cette technique a été employée à l’origine en cancérologie, pour séparer les cellules saines des cellules pathologiques. Les différents types cellulaires sont marqués spécifiquement par immunohistochimie et un découpage sélectif permet leur analyse séparée [24]. Des techniques de cytométrie de flux adaptées à la criminalistique, notamment la technologie DEPArray (Menarini Silicon Biosystems) permettent également l’établissement d’un profil génétique à partir d’une cellule unique [25,26]. Cette approche nécessite cependant un prélèvement supplémentaire, à partir de l’échantillon initialement récolté et qui n’a pas encore été traité, la séparation des cellules devant être réalisée avant la lyse cellulaire. Or ce nouveau prélèvement peut ne pas contenir le mélange observé avec le premier prélèvement, ou comprendre des ADN dans des proportions de contributeurs qui ne sont pas identiques à celles du premier prélèvement. Il est également envisageable que ce nouveau prélèvement soit impossible à réaliser lorsque, par exemple, la totalité de la trace biologique a déjà été extraite.
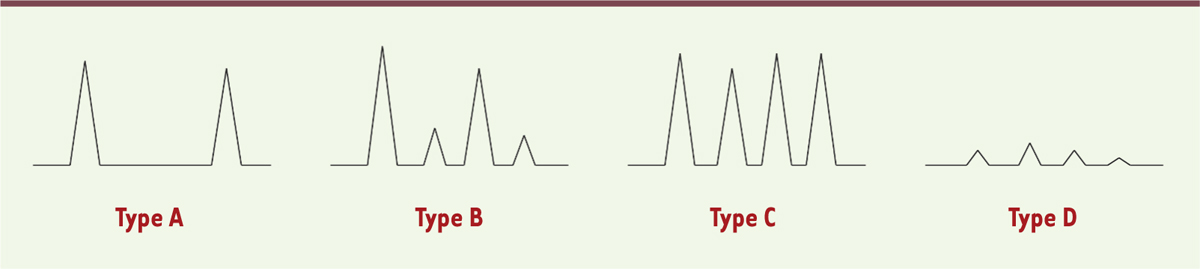
 | Figure 2.
Exemples de profils génétiques observables dans différentes conditions de mélange. Le type A représente un profil unique (hétérozygote) ou un mélange de deux individus possédant les mêmes allèles. Le type B correspond à un profil en mélange où il est possible de séparer le contributeur minoritaire (pics de faible intensité) du contributeur majoritaire (pics de haute intensité). Le type C correspond à un profil en mélange de deux contributeurs de type « mélange à comparer » où il n’est pas possible de séparer les contributeurs. Le type D correspond à un profil ADN en mélange obtenu à partir d’une quantité très faible d’ADN pouvant générer des disparités d’amplification caractérisées par la présence d’effets stochastiques. Ce type de profil n’est pas interprétable. |
Le séquençage haut-débit des mélanges d’ADN semble alors être une alternative plus réaliste, au moins à court terme. En effet, si la séparation par la taille des amplicons d’un marqueur génétique permet d’identifier l’allèle correspondant, aucune information concernant la séquence du microsatellite n’est apportée par l’électrophorèse capillaire. La plupart des polymorphismes des STR examinés ne concernent pas le nombre de répétitions du motif, mais leur séquence nucléotidique [27–30]. La présence de nombreux microvariants, référencés dans des bases de données internationales, ouvre ainsi une voie importante à la déconvolution des mélanges grâce à la séparation possible des allèles de même taille s’ils présentent des différences nucléotidiques (
Figure 3
). Si ces mutations ponctuelles semblent affecter différentiellement les marqueurs génétiques avec une préférence pour les microsatellites, dans les kits commerciaux, le séquençage des amplicons, voire des régions adjacentes de ces marqueurs, devrait permettre d’accroître le nombre de profils, extraits de mélanges, qui soient possiblement admissibles par le fichier FNAEG. À noter cependant que le fichier national ne prend pas en compte les différentes mutations ponctuelles… Il est en effet actuellement illusoire de pouvoir définir une nomenclature internationale qui inclurait la totalité des mutations pour l’ensemble des séquences nucléotidiques. Bien que cela soit peu probable dans un futur proche, une adaptation circonstancielle du fichier serait pourtant nécessaire afin de prendre en compte ces informations supplémentaires.
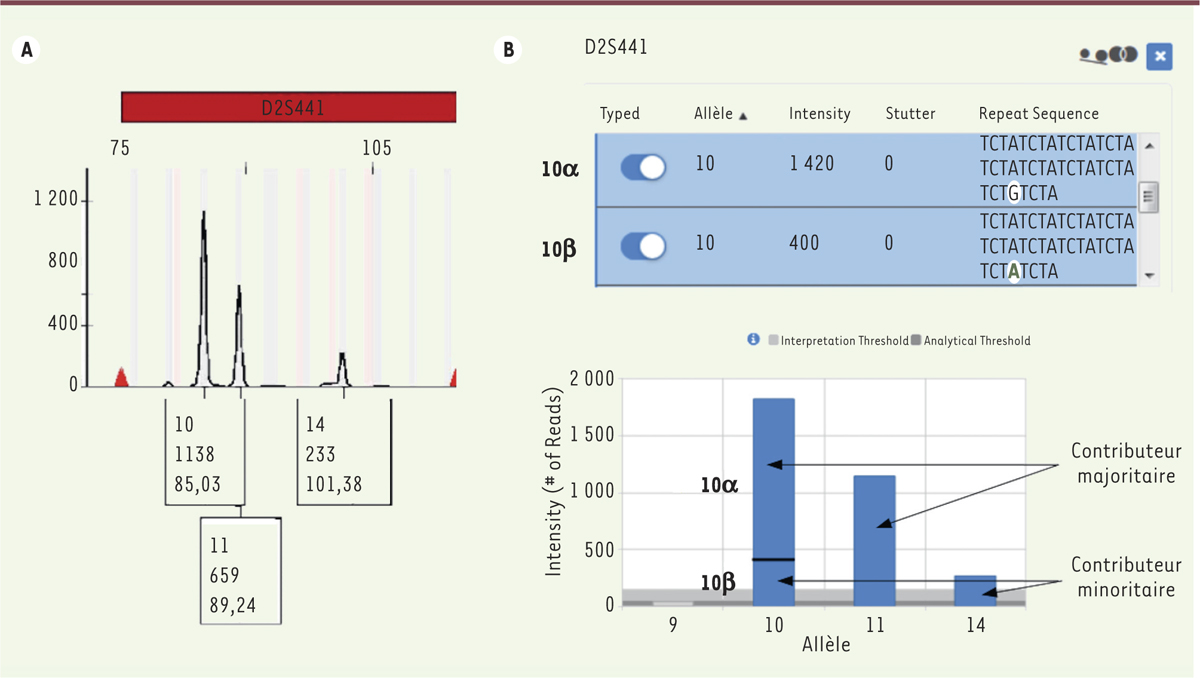
 | Figure 3.
Résultat de l’analyse du marqueur D2S441 dans un mélange ADN par séparation des amplicons par électrophorèse capillaire (A) et par séquençage NGS (B). A. Trois pics de fluorescence sont identifiés (10, 11 et 14 - dans chaque rectangle, le premier chiffre correspond à l’allèle, le deuxième au niveau de fluorescence et le troisième à la taille du fragment en nombre de bases). Cependant, il est très difficile de les associer aux contributeurs du mélange. Il pourrait s’agir d’un contributeur majoritaire homozygote pour l’allèle 10 et d’un contributeur minoritaire 11,14 avec une balance d’hétérozygotie mauvaise ou encore d’un majoritaire 10,11 et un minoritaire avec un allèle 14 sans certitude concernant l’autre allèle. En tout état de cause, ce marqueur serait considéré comme non valide. B.L’analyse des séquences du STR D2S441 montre la présence d’une isomutation (SNP G ou A) dans l’allèle 10 qui permet de détecter deux allèles 10 (α et β). La proportion respective de chaque contributeur dans le mélange est donc facilement analysable avec un contributeur majoritaire 10(α),11 et un contributeur minoritaire 10(β),14. |
|
L’application des avancées réalisées en recherche académique (génétique, génomique, biologie cellulaire, analyse statistique, etc.) au domaine de la criminalistique a pour ambition de faciliter le travail de l’expert afin qu’il puisse rendre des conclusions à la justice, qui soient les plus éclairées possibles. La diminution continuelle des coûts d’analyse par séquençage à haut-débit rend possible ce type d’analyse par les laboratoires de criminalistique. L’analyse de plus de 230 marqueurs génétiques (STR autosomaux, STR sexuels sur les chromosomes X et Y, SNP d’identité, SNP d’origine biogéographiques et phénotypiques) en une seule réaction, est déjà possible [12]. L’exhaustivité des données produites par l’analyse de l’ensemble de ces marqueurs génétiques ne sera pas utile pour chaque affaire. Elle permettra néanmoins, dans les cas complexes, d’optimiser l’analyse des traces et des indices utiles à la manifestation de la vérité. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jeffreys AJ, Wilson V, Thein SL. Individual-specific fingerprints of human DNA . Nature. 1985; ; 316 : :76.–79. 2.
Saiki RK, Gelfand DH, Stoffel S, et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase . Science. 1988; ; 239 : :487.–491. 3.
Butler JM. Short tandem repeat typing technologies used in human identity testing . BioTechniques. 2007;; 43 : :ii.–v. 4.
Fan H, Chu JY. A Brief review of short tandem repeat mutation . Genomics Proteomics Bioinformatics. 2007; ; 5 : :7.–14. 5.
Butler JM. STR Alleles and amplification artifacts. Chapter 3. Advanced topics in forensic DNA typing: interpretation . San Diego: : Academic Press; , 2015 : :47.–86. 6.
Jalby C La police technique et scientifique . Paris: : PUF; , 2014 : 128 p 7.
Butler JM. DNA Profile frequency estimates and match probabilities. Chapter 11. Advanced topics in forensic DNA typing: interpretation . San Diego: : Academic Press; , 2015 : :281.–308. 8.
Coquoz R, Taroni F. Preuve par l’ADN : la génétique au service de la justice . Paris: : PPUR Presses Polytechniques; , 2013 : :370p.. 9.
Alaeddini R. Forensic implications of PCR inhibition: a review . Forensic Sci Int Genet. 2012; ; 6 : :297.–305. 10.
Mulero JJ, Chang CW, Lagacé RE, et al. Development and validation of the AmpFSTR MiniFilerTM PCR amplification kit: a MiniSTR multiplex for the analysis of degraded and/or PCR inhibited DNA . Minifiler kit validation. J Forensic Sci. 2008; ; 53 : :838.–852. 11.
Flores S, Sun J, King J, et al. Internal validation of the GlobalFilerTM Express PCR amplification kit for the direct amplification of reference DNA samples on a high-throughput automated workflow . Forensic Sci Int Genet. 2014; ; 10 : :33.–39. 12.
Jäger AC, Alvarez ML, Davis CP, et al. Developmental validation of the MiSeq FGx forensic genomics system for targeted next generation sequencing in forensic DNA casework and database laboratories . Forensic Sci Int Genet. 2017; ; 28 : :52.–70. 13.
Hiroaki N, Koji F, Tetsushi K, et al. Approaches for identifying multiple-SNP haplotype blocks for use in human identification . Leg Med Tokyo Jpn. 2015; ; 17 : :415.–420. 14.
Schneider PM, Fimmers R, Keil W, et al. The german stain commission: recommendations for the interpretation of mixed stains . Int J Legal Med. 2009; ; 123 : :1.–5. 15.
Bieber FR, Buckleton JS, Budowle B, et al. Evaluation of forensic DNA mixture evidence: protocol for evaluation, interpretation, and statistical calculations using the combined probability of inclusion . BMC Genet. 2016; ; 17 : :125.. 16.
Nurit B, Anat G, Michal S, et al. Evaluating the prevalence of DNA mixtures found in fingernail samples from victims and suspects in homicide cases . Forensic Sci Int Genet. 2011; ; 5 : :532.–537. 17.
Bill M, Gill P, Curran J, et al. PENDULUM: a guideline-based approach to the interpretation of STR mixtures . Forensic Sci Int. 2005; ; 148 : :181.–189. 18.
Haned H, Benschop CCG, Gill PD, et al. Complex DNA mixture analysis in a forensic context: evaluating the probative value using a likelihood ratio model . Forensic Sci Int Genet. 2015; ; 16 : :17.–25. 19.
Slooten KJ. Accurate assessment of the weight of evidence for DNA mixtures by integrating the likelihood ratio . Forensic Sci Int Genet. 2017; ; 27 : :1.–16. 20.
Slooten KJ, Egeland T. Exclusion probabilities and likelihood ratios with applications to mixtures . Int J Legal Med. 2016; ; 130 : :39.–57. 21.
Vuichard S, Borer U, Bottinelli M, et al. Differential DNA extraction of challenging simulated sexual-assault samples: a Swiss collaborative study . Investig Genet. 2011; ; 2 : :11.. 22.
Klein SB, Buoncristiani MR. Evaluating the efficacy of DNA differential extraction methods for sexual assault evidence . Forensic Sci Int Genet. 2017; ; 29 : :109.–117. 23.
Vandewoestyne M, Deforce D. Laser capture microdissection in forensic research: a review . Int J Legal Med. 2010; ; 124 : :513.–521. 24.
Bruijns B, van Asten A, Tiggelaar R, et al. Microfluidic devices for forensic DNA analysis: a review . Biosensors. 2016; ; 6 : 25.
Fontana F, Rapone C, Bregola G, et al. Isolation and genetic analysis of pure cells from forensic biological mixtures: the precision of a digital approach . Forensic Sci Int Genet. 2017; ; 29 : :225.–241. 26.
Hansson O, Gill P. Characterisation of artefacts and drop-in events using STR-validator and single-cell analysis . Forensic Sci Int Genet. 2017; ; 30 : :57.–65. 27.
Rockenbauer E, Hansen S, Mikkelsen M, et al. Characterization of mutations and sequence variants in the D21S11 locus by next generation sequencing . Forensic Sci Int Genet. 2014; ; 8 : :68.–72. 28.
Gettings KB, Aponte RA, Vallone PM, et al. STR allele sequence variation: Current knowledge and future issues . Forensic Sci Int Genet. 2015; ; 18 : :118.–130. 29.
Gettings KB, Kiesler KM, Faith SA, et al. Sequence variation of 22 autosomal STR loci detected by next generation sequencing . Forensic Sci Int Genet. 2016; ; 21 : :15.–21. 30.
Børsting C, Morling N. Next generation sequencing and its applications in forensic genetics . Forensic Sci Int Genet. 2015; ; 18 : :78.–89. |