| |
| Med Sci (Paris). 33(12): 1111–1117. doi: 10.1051/medsci/20173212019.Prix Nobel de Chimie 2017 : Jacques Dubochet, Joachim Frank et Richard Henderson La révolution de la résolution en cryo-microscopie électronique Emmanuelle Neumann,1 Leandro Farias Estrozi,1 Grégory Effantin,1 Cécile Breyton,1 and Guy Schoehn1a 1Univ. Grenoble Alpes, CNRS, CEA, Institut de biologie structurale, 71, rue Jules Horowitz, CS10090, 38044Grenoble Cedex 09, France |

Vignette (Photo © Inserm/Musée Jules Verne - Ville de Nantes / Dehausse, Éric). Afin d’explorer le monde qui nous entoure, l’homme a depuis longtemps tenté de rendre visible « l’invisible ». Ainsi peut se définir la microscopie : l’examen de l’infiniment petit. Il existe différents types de microscopes et, après le prix Nobel de Physique décerné à Ernst Ruska en 1986 pour l’invention du microscope électronique, celui de Chimie de 2017 a été attribué à Jacques Dubochet, de l’université de Lausanne, en Suisse, à Joachim Frank, de l’université de Columbia, aux États-Unis, et à Richard Henderson, du laboratoire de biologie moléculaire de Cambridge, au Royaume-Uni, pour leurs contributions essentielles au développement de la « cryo-microscopie électronique ». La cryo-microscopie électronique est principalement dédiée aux sciences du vivant. Elle contribue à comprendre de nombreux processus moléculaires. Les microscopes électroniques de dernière génération permettent désormais d’obtenir des structures tridimensionnelles (3D) d’objets biologiques à des résolutions qui sont quasi-atomiques (2,26 Å [1], 1,8 Å [2], 2,2 Å [3], ou 3,2 Å [4]), alors que les résolutions moyennes qui étaient atteintes jusqu’en 2014, stagnaient aux environs de 15 Å. Ces résolutions permettaient d’ailleurs aux détracteurs de la cryo-microscopie électronique de la qualifier de « blob-ologie », ou science des « blobs »1
(Figure 1).
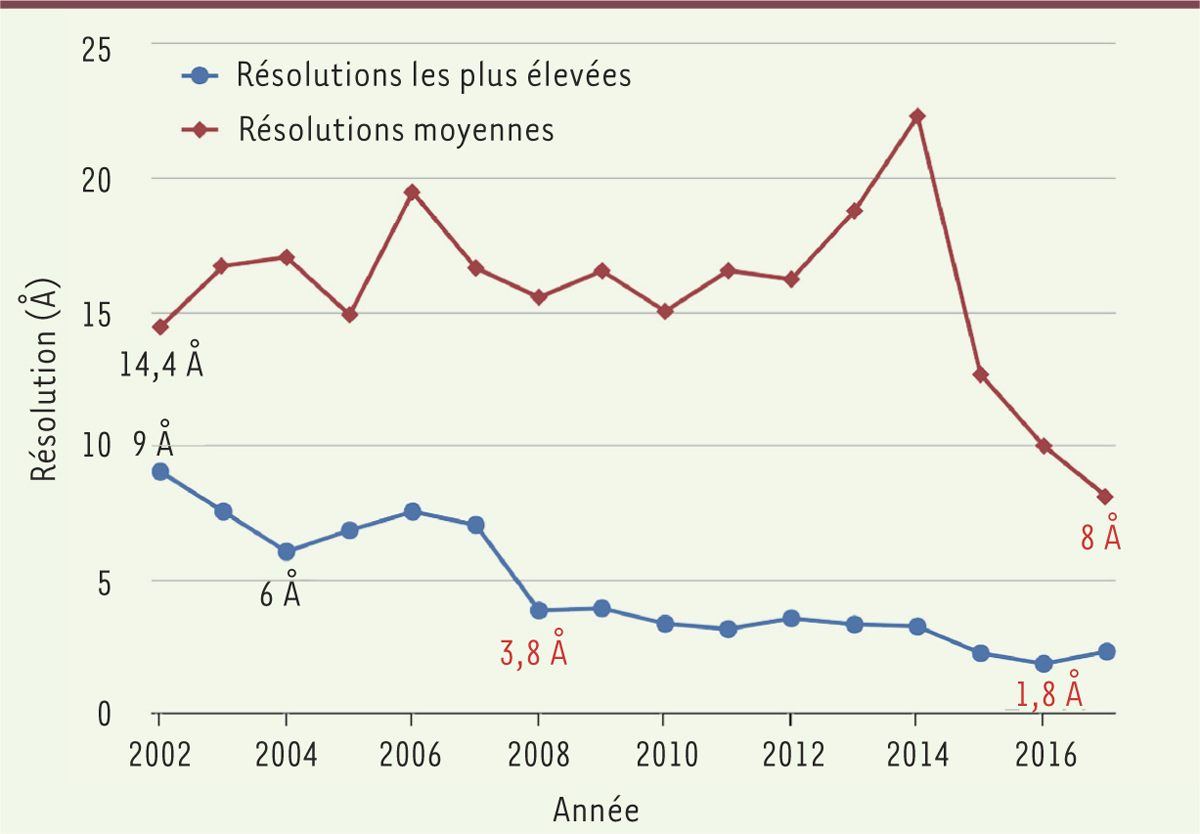
 | Figure 1.
Résolutions moyennes et les plus élevées des cartes tridimensionnelles de microscopie électronique déposées dans la base de données EMDB en fonction des années (https://www.ebi.ac.uk/pdbe/emdb/statistics_main.html/). |
Cette méthode pouvait paraître comme complètement impensable il y a quelques décennies. En effet, comment un échantillon biologique peut-il être observé, sous vide et sous un faisceau d’électrons, sans être instantanément détruit ? Pour trouver une explication et des solutions, revenons à l’origine de la microscopie. Les premiers microscopes furent des microscopes optiques, inventés en Hollande au xviie siècle. Ils évoluèrent sans cesse au cours des siècles, permettant d’apporter de nombreuses connaissances dans divers domaines, allant de la physique à la biologie. Le pouvoir de résolution d’un microscope, ou sa capacité à discerner deux points adjacents, est lié à la longueur d’onde de la source de lumière (la résolution étant d’environ λ/2, c’est-à-dire longueur d’onde divisée par 2). Pour un microscope optique « classique », sa résolution est limitée à 0,2 µm. Afin d’étudier des objets de taille inférieure, il est donc nécessaire d’utiliser des ondes qui peuvent atteindre des longueurs d’onde suffisamment faibles pour pouvoir observer des objets de la taille des atomes, comme par exemple les électrons (λ ~ 10-3 nm). Ce n’est qu’au début des années 1930, qu’Ernst Ruska et Max Knoll [5] ont pu mettre au point un microscope « électronique ». Ils ont ainsi été à l’origine des premières images ayant une résolution de quelques dizaines de nanomètres. Différents types d’instruments ont ensuite été développés permettant d’observer, par exemple, les virus jusqu’alors invisibles : des microscopes électroniques à balayage en réflexion, qui permettent l’analyse de la surface d’échantillons épais, et des microscopes à transmission capables (en particulier) d’analyses à très haute résolution d’échantillons suffisamment minces pour que les électrons transmis puissent les traverser. L’observation par un microscope électronique repose sur une double contrainte : l’échantillon doit supporter le vide présent au sein de la colonne de transmission, et qui est nécessaire pour éviter toute dispersion des électrons, et il ne doit pas être détruit par le faisceau d’électrons : la quantité d’électrons que l’échantillon reçoit doit donc être limitée et contrôlée. |
Le microscope électronique Un microscope électronique à transmission est constitué d’un système de pompage qui permet d’obtenir un vide poussé (jusqu’à 10-9 Pascal – la pression atmosphérique est d’environ 105 Pascal), d’une colonne d’optique électronique (une source d’électrons et des lentilles électromagnétiques), d’une chambre d’introduction de l’échantillon, et d’un, ou plusieurs, systèmes de détection : écran fluorescent, caméras CCD (charge-coupled device)/CMOS (complementary metal oxide semiconductor) et/ou à détection directe d’électrons. Désormais, les avancées technologiques ont conduit au développement d’instruments qui sont très performants. Ils génèrent des données qui, après analyse d’images, permettent d’accéder, pour des échantillons biologiques, à des informations d’une résolution quasi-atomique. Ces microscopes de dernière génération possèdent tous des sources d’électrons à effet de champ [31]. Ils ont pour caractéristique principale de générer un faisceau d’électrons très brillant, très cohérent et parallèle. Dès leur apparition, ces instruments ont permis d’accéder à des informations sub-nanométriques permettant des reconstructions tridimensionnelles d’objets biologiques [6–8]. Une nouvelle génération de détecteurs, les caméras à détection directe d’électrons, a, ces dernières années, littéralement révolutionné la discipline. Pour des objets biologiques au-delà de 150 kDa, la cryo-microscopie est ainsi devenue incontournable pour toute approche structurale, comme le prouve le nombre croissant de structures tridimensionnelles qui ont été réalisées récemment (Figure 2).
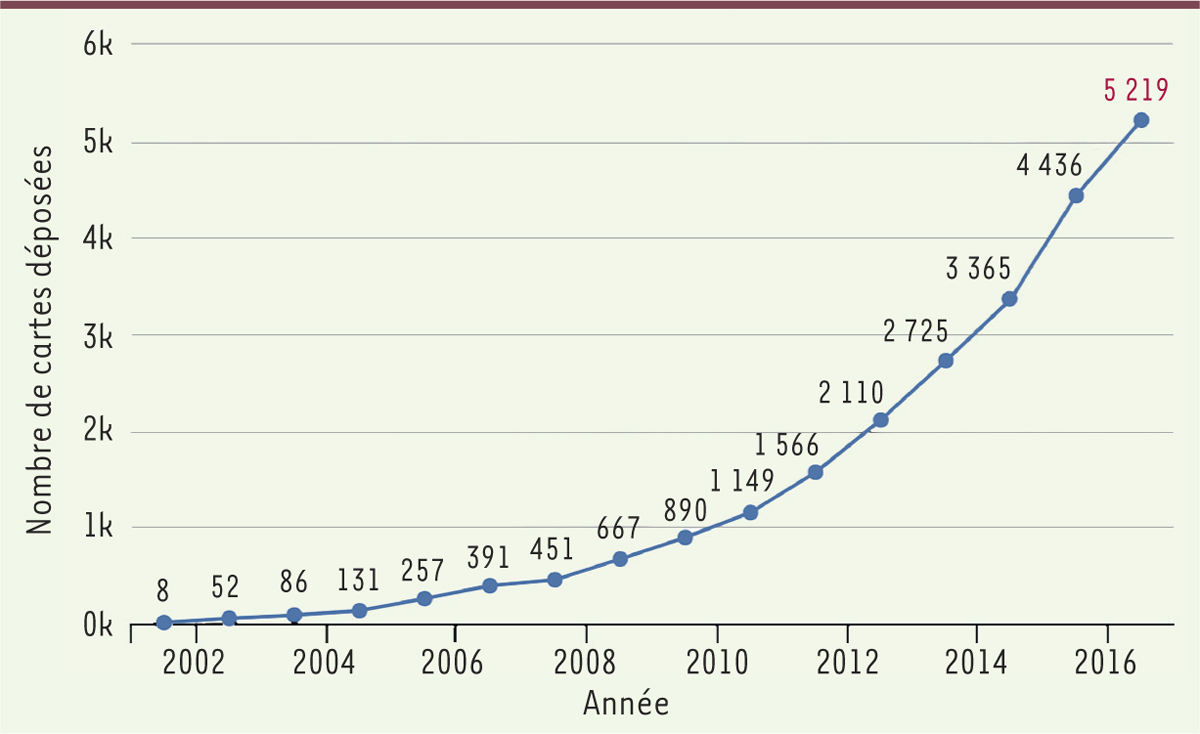
 | Figure 2.
Nombre de structures tridimensionnelles déposées dans la base de données de microscopie électronique EMDB en fonction des années (https://www.ebi.ac.uk/pdbe/emdb/statistics_main.html/). |
Richard Henderson, récompensé pour ses travaux par le prix Nobel de Chimie de cette année 2017, a contribué (étant à la tête d’un consortium européen) au développement et à l’émergence de ces caméras de nouvelle génération. D’autres avancées technologiques ont contribué à ces développements importants : les correcteurs de l’aberration de sphéricité de la lentille objectif (un exemple de résultat à une résolution inférieure à 3 Å est donné dans l’article [9]) ; l’utilisation de « phase plate » (la structure du nucléosome, une particule de 200 kDa a ainsi été déterminée à 3,9 Å de résolution [10]) ; et, plus récemment, la structure de l’hémoglobine de 64 kDa qui a été obtenue à 3,2 Å de résolution [11]. Il faut également souligner, qu’au-delà de ces développements, l’amélioration de la puissance des ordinateurs et le développement continu des techniques d’analyse d’images, dont Joachim Frank fut l’un des pionniers, a largement participé à l’augmentation du nombre de reconstructions 3D à résolution quasi-atomique d’objets biologiques. Tout ceci n’a été possible que grâce au développement, au début des années 1980, sous l’impulsion de Jacques Dubochet, d’une technique révolutionnaire de préparation des échantillons biologiques (electron microscopy of frozen biological suspensions [12]). Pour faire face aux dégâts engendrés par l’introduction d’un échantillon biologique dans l’environnement sous vide d’une colonne de microscope électronique, et afin de le maintenir dans un état morphologique aussi proche que possible de son état naturel, différentes techniques ont été développées et optimisées en fonction de l’échantillon : des méthodes de fixation (chimique ou par congélation sous haute pression) ; d’inclusion dans des résines ; de coupes et de marquage/coloration pour l’étude de tissus cellulaires ; des méthodes d’ombrage, de coloration négative et d’échantillons congelés hydratés pour l’étude de particules isolées [13]. Cette dernière technique, la cryo-microscopie électronique, permet d’obtenir un film mince (de l’ordre de 100 nm d’épaisseur) de glace vitreuse qui inclut l’échantillon à observer dans un état natif (voir plus loin). Ces méthodes permettent un accès à la structure interne de l’objet, l’image résultante étant une projection de l’objet à travers toute son épaisseur. Les étapes principales des progrès effectués en microscopie électronique dans le domaine particulier de la biologie structurale sont résumées dans la Figure 3.
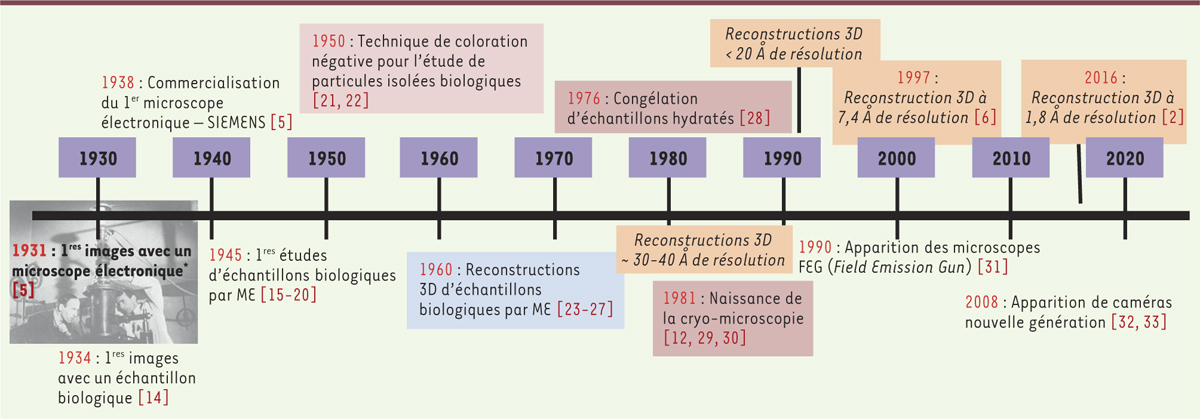
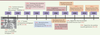 | Figure 3.
Frise chronologique montrant les étapes principales majeures franchies par la microscopie électronique (ME) structurale en biologie depuis 1930 (*photo : Max Knoll et Ernst Ruska, 1944). |
Mais qui sont ces 3 scientifiques, à qui nous devons toute cette effervescence autour de la cryo-microscopie électronique, et qui sont aujourd’hui récompensés par ce prix Nobel de Chimie ? |

Depuis son plus jeune âge, Jacques Dubochet a cherché à comprendre les choses pour ne plus en avoir peur. Cela l’a conduit à faire une thèse : « Je n’ai pas essayé de gagner le prix Nobel mais de faire de la science ». Après sa thèse en Biophysique effectuée à Genève avec Eduard Kellenberger et soutenue en 1973, il dépose en 1978 un projet pour devenir responsable d’un groupe de recherche, au Laboratoire européen de biologie moléculaire (EMBL), à Heidelberg. Ce projet visait à utiliser le froid afin de pouvoir appliquer la microscopie électronique structurale au vivant et ainsi contourner le problème de l’observation d’objets hydratés dans le vide poussé de l’enceinte du microscope. Dans un tel environnement, l’eau contenue dans l’échantillon s’évapore en effet, de manière presque instantanée. Les conséquences sur les macromolécules biologiques sont ainsi catastrophiques : elles s’effondrent sur elles-mêmes. Leur structure tridimensionnelle fine est donc perdue. Ce problème peut néanmoins être contourné : en générant une empreinte de l’échantillon insensible au vide (technique dite de coloration négative), ou en congelant l’échantillon à suffisamment basse température afin que l’eau ne puisse plus s’évaporer. Dans la première technique, ce n’est pas le complexe macromoléculaire qui est visualisé mais son empreinte et les détails de la structure dépendent de la taille des grains composant la substance qui est utilisée pour la coloration négative (des sels d’atomes lourds) (Figure 4A). L’information ainsi obtenue n’est en fait qu’une information de surface (le colorant entoure l’objet mais ne pénètre que peu à l’intérieur). L’objet peut également s’effondrer sur lui-même et être déformé, avant que le moule ne se soit solidifié. Cette technique ne peut donc être utilisée pour la biologie structurale à haute résolution. La deuxième technique (congélation) a ses limites : lorsque l’eau est congelée, elle cristallise et augmente de volume. Les cristaux ainsi formés détruisent la structure des objets en solution. C’est là qu’intervient Jacques Dubochet : « J’ai inventé l’eau froide », a-t-il déclaré à la suite de l’annonce de son prix Nobel. Effectivement, il a mis au point une technique de congélation ultra rapide d’une couche d’eau d’épaisseur sub-micrométrique… La vitesse de congélation dans de l’éthane liquide (à –170 °C), de l’ordre de 10 000 °C par seconde, empêche en effet la formation de cristaux de glace. L’eau est alors figée dans un état très proche de son état liquide. Cela s’appelle « glace vitreuse » ou « amorphe » (d’où l’appellation « eau froide » employée par Jacques Dubochet). Cette glace de même densité que l’eau liquide, et qui occupe le même volume, est translucide aux électrons (si toutefois elle est suffisamment fine). La congélation ultra rapide de l’eau va également conserver tout ce qu’elle contient, et donc les macromolécules biologiques dont on désire étudier la structure (Figure 4B). Entre 1981 et 1988, Dubochet et ses collègues publient cette nouvelle méthode dans plusieurs revues [12, 29, 30] dont celle intitulée : « Cryo-electron microscopy of vitrified specimens » [34], qui est, aujourd’hui, citée près de 2 000 fois, par d’autres publications. Après son passage à l’EMBL d’Heidelberg, Jacques Dubochet a été recruté par l’université de Lausanne. Il a alors poursuivi ses travaux sur la microscopie électronique en s’intéressant à la congélation et à la coupe ultra fine d’objets biologiques plus épais après vitrification. Cette nouvelle technique a été baptisée CEMOVIS : cryo-electron microscopy of vitrious section [35].
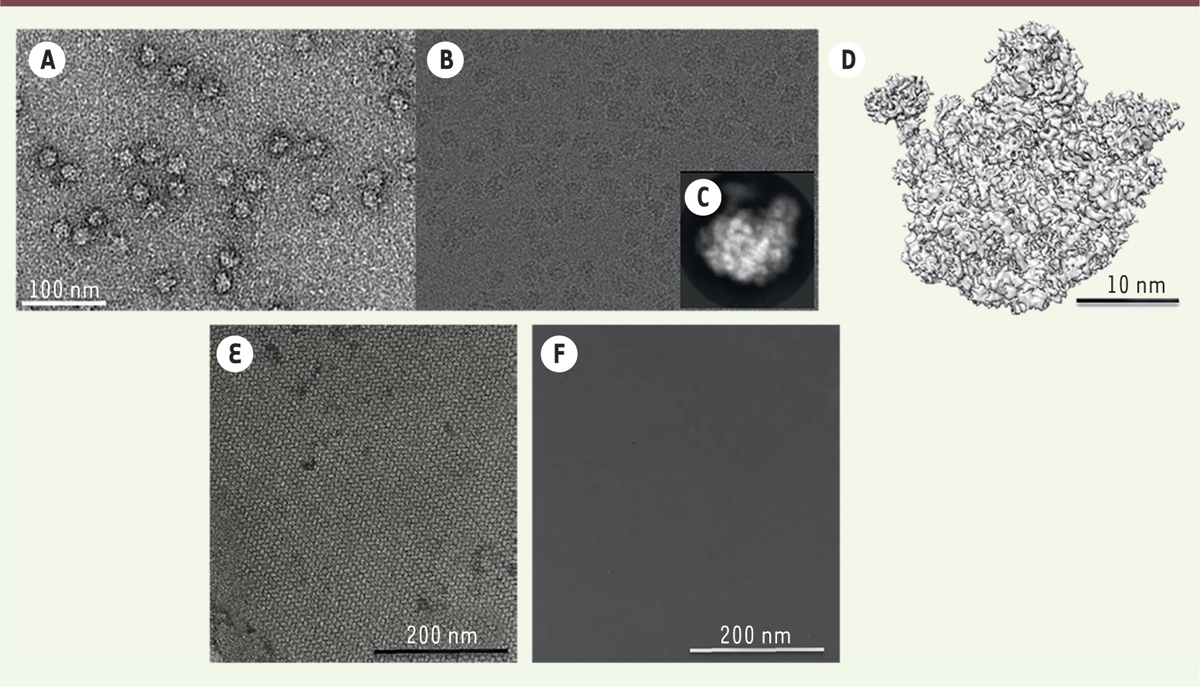
 | Figure 4.
Image de coloration négative (A) et de cryo-microscopie (B) de la sous-unité 50S du ribosome bactérien. C. Classe 2D (moyenne) obtenue à partir de particules sélectionnées dans (B). D. Reconstruction 3D de l’échantillon à 7 Å de résolution. Cristaux 2D visualisés en coloration négative (E) et en cryo (F) du complexe b6 f de la chaîne photosynthétique de Chlamydomonas reinhardtii. |
Jacques Dubochet a ainsi rendu possible l’observation de la structure native d’échantillons biologiques, de macromolécules à des cellules entières. Malgré ses contributions restées majeures, il conclut toujours ces entretiens de manière très modeste, en disant que tout cela n’aurait pas été possible sans le soutien des différentes institutions dans lesquelles il a travaillé, et qu’« un scientifique, c’est un homme dont le seul maître est la nature »… |

Un microscope électronique permet d’acquérir des images d’un objet. Les images obtenues par cryo-microscopie électronique contiennent de l’information à haute résolution de cet échantillon. Pour limiter les dommages d’irradiation que cet échantillon biologique peut subir afin de le visualiser, la dose d’électrons à laquelle il est soumis doit être limitée, ce qui génère, de facto, des images qui seront bruitées et quasiment dénuées de toute utilité. Seule la moyenne d’un grand nombre d’images présente un intérêt pour « récupérer » l’information à haute résolution. Joachim Frank fut l’un des premiers à le comprendre. Il développa, dès les années 1970, des méthodes mathématiques afin de combiner un grand nombre d’images bruitées d’un même objet et ainsi, d’en obtenir la structure 3D. Joachim Frank est donc l’un des pionniers de l’analyse d’images en cryo-microscopie électronique. Il est à l’origine du concept de « fonction enveloppe », qui décrit la façon dont la cohérence partielle des ondes électroniques affecte la formation des images en microscopie électronique en transmission [36–38]. Il est aussi à l’origine de l’utilisation de méthodes de classification des particules, par statistiques multivariées, qui permettent de regrouper en classes des particules statistiquement similaires [39, 40]. Ces méthodes de classification et de calcul de moyennes de classes, développées par Jean-Pierre Bretaudière [41] et utilisées dans le laboratoire de Joachim Frank, ont grandement contribué au développement des logiciels d’analyse d’images qui permettent d’obtenir des reconstructions en trois dimensions de molécules biologiques (Figure 4C). Les améliorations, à la fois technologiques et des logiciels, permettent désormais de déterminer des structures de complexes biologiques à résolution quasi atomique. Toutes les avancées méthodologiques et d’analyse d’images réalisées par Joachim Frank, ont été mises à la disposition de la communauté scientifique par le biais d’un logiciel modulaire d’analyse d’images appelé SPIDER (system for processing of image data in electron microscopy and related field) [42]. Par ailleurs, Joachim Frank a un objet de prédilection. Il est expert dans la biologie des ribosomes. Ces complexes ribonucléoprotéiques existent chez tous les organismes vivants et sont essentiels pour la synthèse des protéines à partir des ARN messagers. L’étude de la structure et de la fonction du ribosome a d’ailleurs été récompensée par le prix Nobel de chimie en 2009 (attribué à Venkatraman Ramakrishnan, Thomas A. Steitz et Ada E. Yonath). Grâce à une analyse d’images issues de ses travaux, Joachim Frank publie, en 1981, les premières projections moyennées de ribosome par microscopie électronique [40], suivi des premières reconstructions 3D, par coloration négative, en 1987 [43], et par cryo-microscopie électronique, en 1991 [44] (Figure 4D). L’une de ses découvertes majeures fut la mise en évidence du pivotement de la petite sous-unité du ribosome (30S) par rapport à la plus grande (50S). Cette rotation est associée au mouvement des ARN de transfert à l’intérieur du ribosome [45]. Joachim Frank a également grandement contribué à la compréhension détaillée de ce qui advient durant le déplacement du ribosome le long de l’ARN messager (phénomène de translocation), en explorant notamment le rôle de différents facteurs d’élongation et de terminaison par cryo-microscopie électronique [46]. La contribution de Joachim Frank a donc été double : pionnier de l’analyse d’images et des études majeures sur la biologie du ribosome par cryo-microscopie électronique. |

Richard Henderson a été récompensé pour avoir déterminé la structure d’une protéine à résolution atomique par cristallographie électronique. Il montrait ainsi toutes les potentialités de cette technique pour observer les molécules du vivant. Henderson s’est initialement intéressé à la microscopie électronique sur un échantillon biologique bien particulier : la membrane pourpre. La membrane pourpre est une membrane naturelle, purifiée à partir de la bactérie Halobacterium salinarum qui a la capacité de se développer dans des milieux saturés en sel. Cette membrane est composée de lipides et d’une seule protéine, la bacteriorhodopsine, organisée en réseau (un cristal bidimensionnel de protéine). Un cofacteur, qui absorbe la lumière, est associé à la protéine et lui donne sa couleur pourpre caractéristique. La bacteriorhodopsine permet à la bactérie de transformer l’énergie lumineuse en un gradient de protons, une énergie chimique directement utilisable par la cellule. Dans les années 1970, alors que la cristallographie aux rayons X avait montré toute son efficacité pour déterminer la structure de protéines à partir de cristaux 3D, il était alors admis que les protéines membranaires resteraient réfractaires à cette technique. Il était en effet nécessaire d’utiliser des détergents incompatibles avec la cristalographie, afin de préserver leur solubilité en dehors de la membrane. Des techniques alternatives étaient donc nécessaires pour que leur structure puisse être déterminée. Afin de s’affranchir de la présence d’eau, pourtant nécessaire à la stabilité de la protéine, mais qui à l’époque était incompatible avec le vide de la colonne du microscope, Henderson et Unwin ont eu l’idée de protéger la membrane pourpre par du glucose. Ce sucre permet en effet de satisfaire les liaisons hydrogènes de la surface de la membrane pourpre et donc d’en préserver la structure native. Afin de pallier le problème de la destruction de l’échantillon par le faisceau d’électrons, ils « illuminent » les cristaux de membrane pourpre avec une très faible dose d’électrons : contrairement à celles obtenues après coloration négative (Figure 4E), les images obtenues sans colorant ne présentent alors aucun détail visible à l’œil (Figure 4F). La nature cristalline de l’échantillon va cependant permettre de moyenner un grand nombre de molécules (le nombre moyen de protéines dans un cristal de membrane pourpre d’un micromètre de diamètre est de 120 000). Ces protéines forment en effet un réseau bidimensionnel régulier (cristallin) et font diffracter les électrons (de manière similaire aux cristaux tridimensionnels de protéines qui font diffracter les rayons X dans les synchrotrons). Henderson et Unwin ont utilisé ces clichés de diffraction électronique pour calculer, en passant par l’espace de Fourier, la structure tridimensionnelle de la protéine formant ces réseaux. En 1975, Henderson et Unwin publient ainsi la première structure d’une protéine membranaire, celle de la bactériorhodopsine, à 7 Å de résolution [47]. À cette résolution, la protéine apparaît formée de 7 densités allongées, perpendiculaires à la membrane ou inclinées au maximum de 20°, distantes de 10 à 12 Å, et longues de 35 à 40 Å. Ils interprètent ces densités comme des hélices transmembranaires. Ainsi, seulement 3 ans après que le modèle de la membrane en mosaïque fluide a été proposé par Singer et Nicholson [48], Henderson et Unwin apportaient la preuve expérimentale que les protéines membranaires ont une structure définie et qu’elles traversent, en effet, la bicouche lipidique formant un faisceau d’hélices alpha. Il aura fallu quinze ans de plus pour que Henderson et ses collègues parviennent à déterminer la structure de la protéine à une résolution permettant le positionnement de chaque atome (3,5 Å) [49]. Ce résultat aura pu être atteint par l’optimisation de la préparation de l’échantillon, toujours préservé dans le glucose. Il aura également nécessité le développement de méthodes de traitement de l’image des cristaux. En particulier, les réseaux bidimensionnel de protéines n’étant pas totalement réguliers, les distorsions les affectant ont pu être corrigées pour permettre un meilleur « moyennage » des molécules et donc atteindre une meilleure résolution finale. Outre le fait que l’obtention de la structure atomique de la bacteriorhodopsine a permis d’expliquer nombre de résultats qui avaient été obtenus précédemment, elle a surtout permis de comprendre le mécanisme de transformation par cette protéine, de l’énergie lumineuse en énergie chimique ! La microscopie électronique a donc amorcé une « révolution de la résolution », et l’analyse de particules isolées atteint actuellement des résolutions qui n’avaient été jusqu’à présent jamais espérées, sur des protéines qui ont une taille de plus en plus petite. Cette évolution repose également, en partie, sur le développement de détecteurs à électrons directs, plus sensibles que les précédents détecteurs, et qui permettent d’acquérir des images avec des doses d’électrons encore plus faibles. Des films remplacent les images, et permettent de corriger le mouvement de l’échantillon qui est induit par l’illumination. Cette correction autorise un bien meilleur alignement et « moyennage » des molécules étudiées, et donc, in fine, une bien meilleure résolution. Bien que Richard Henderson n’ait pas été reconnu par le comité Nobel pour cet aspect, il mérite doublement cette récompense puisqu’il a aussi participé au développement de ces nouveaux détecteurs. Grâce aux contributions essentielles des trois lauréats du prix Nobel de Chimie de cette année 2017, et aux développements successifs qui ont permis d’optimiser la technique, la cryo-microscopie est actuellement l’une des techniques phares en biologie structurale. Par un accès à des informations à l’échelle atomique ou quasi atomique, elle permet de comprendre dans le détail un grand nombre de processus biologiques qui sont d’un intérêt majeur pour la santé publique. En France, cette révolution s’amorce (lentement) avec la présence sur le territoire national (à l’IGBMC de Strasbourg et à l’ESRF de Grenoble) de deux cryo-microscopes de toute dernière génération (Krios, FEI) équipés des meilleurs détecteurs actuels sur le marché (détecteurs directs d’électrons). |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Dong Y, Liu Y, Jiang W, et al. Antibody-induced uncoating of human rhinovirus B14 . Proc Natl Acad Sci USA. 2017; ; 114 : :8017.–8022. 2.
Merk A, Bartesaghi A, Banerjee S, et al. Breaking Cryo-EM resolution barriers to facilitate drug discovery . Cell. 2016; ; 165 : :1698.–1707. 3.
Amunts A, Brown A, Toots J, et al. Ribosome. The structure of the human mitochondrial ribosome . Science. 2015; ; 348 : :95.–98. 4.
Bartesaghi A, Matthies D, Banerjee S, et al. Structure of β-galactosidase at 3.2-Å resolution obtained by cryo-electron microscopy . Proc Natl Acad Sci USA. 2014;; 111 : :11709.–14. 5.
Ruska E. Nobel lecture. The development of the electron microscope and of electron microscopy . Biosci Rep. 1987; ; 7 : :607.–629. 6.
Böttcher B, Kiselev NA, Stel’Mashchuk VY, et al. Three-dimensional structure of infectious bursal disease virus determined by electron cryomicroscopy . J Virol. 1997; ; 71 : :325.–330. 7.
Conway JF, Cheng N, Zlotnick A, et al. Visualization of a 4-helix bundle in the hepatitis B virus capsid by cryo-electron microscopy . Nature. 1997; ; 386 : :91.–94. 8.
Fujiyoshi Y. The structural study of membrane proteins by electron crystallography . Adv Biophys. 1998; ; 35 : :25.–80. 9.
Fischer N, Neumann P, Konevega AL, et al. Structure of the E. coli ribosome-EF-Tu complex at <3 Å resolution by Cs-corrected cryo-EM . Nature. 2015; ; 520 : :567.–570. 10.
Chua EY, Vogirala VK, Inian O, et al. 3,9 Å structure of the nucleosome core particle determined by phase-plate cryo-EM . Nucleic Acids Res. 2016; ; 44 : :8013.–8019. 11.
Khoshouei M, Radjainia M, Baumeister W, Danev R. Cryo-EM structure of haemoglobin at 3,2 Å determined with the Volta phase plate . Nat Commun. 2017; ; 8 : :16099.. 12.
Lepault J, Booy FP, Dubochet J. Electron microscopy of frozen biological suspensions . J Microsc. 1983; ; 129 : :89.–102. 13.
Fender P, Moriscot C, Ruigrok RWH, Schoehn G. Electron microscopy of viruses: Techniques to prepare viruses and viral proteins for observation by electron microscopy . Reference Module in Biomedical Sciences. 2014. 14.
Marton L. The electron microscope: A new tool for bacteriological research . J Bacteriol. 1941; ; 41 : :397.–413. 15.
Marton L, Gupta NN, Marton C. Modifications of specimens in electron microscopy . Science. 1946; ; 104 : :35.–36. 16.
Williams RC, Wyckoff RW. Electron shadow micrography of the tobacco mosaic virus protein . Science. 1945; ; 101 : :594.–596. 17.
Price WC, Williams RC, Wyckoff RW. The electron micrography of crystalline plant Viruses . Science. 1945; ; 102 : :277.–278. 18.
Loring HS, Marton L, Schwerdt CE. Electron microscopy of purified Lansing virus . Proc Soc Exp Biol Med. 1946; ; 62 : :291.. 19.
Sharp DG, Taylor AR, et al. Rabbit papilloma and vaccinia viruses and T2 bacteriophage of E. coli in shadow electron micrographs . Proc Soc Exp Biol Med. 1946; ; 61 : :259.–265. 20.
Wyckoff RW. Electron microscopic study of viruses . J Am Med Assoc. 1948; ; 136 : :1081.–1083. 21.
Hall CE. Electron densitometry of stained virus particles . Biophys Biochem Cytol. 1955; ; 1 : :1.–12. 22.
Brenner S, Horne RW. A negative staining method for high resolution electron microscopy of viruses . Biochim Biophys Acta. 1959; ; 34 : :103.–110. 23.
Klug A, Caspar DL. The structure of small viruses . Adv Virus Res. 1960; ; 7 : :225.–325. 24.
Horne RW, Wildy P. Virus structure revealed by negative staining . Adv Virus Res. 1963; ; 10 : :101.–170. 25.
Klug A, Berger JE. An optical method for the analysis of periodicities in electron micrographs, and some observations on the mechanism of negative staining . J Mol Biol. 1964; ; 10 : :565.–569. 26.
Crowther RA, Amos LA, Finch JT, et al. Three dimensional reconstructions of spherical viruses by fourier synthesis from electron micrographs . Nature. 1970; ; 226 : :421.–425. 27.
DeRosier DJ, Moore PB. Reconstruction of three-dimensional images from electron micrographs of structures with helical symmetry . J Mol Biol. 1970; ; 52 : :355.–369. 28.
Taylor KA, Glaeser RM. Electron microscopy of frozen hydrated biological specimens . J Ultrastruct Res. 1976; ; 55 : :448.–456. 29.
Dbochet J, Booy FP, Freeman R, et al. Low temperature electron microscopy . Annu Rev Biophys Bioeng. 1981; ; 10 : :133.–149. 30.
Adrian M, Dubochet J, Lepault J, McDowall AW. Cryo-electron microscopy of viruses . Nature. 1984; ; 308 : :32.–36. 31.
Zhou ZH, Chiu W. Prospects for using an IVEM with a FEG for imaging macromolecules towards atomic resolution . Ultramicroscopy. 1993; ; 49 : :407.–416. 32.
Xuong NH, Jin L, Kleinfelder S, et al. Future directions for camera systems in electron microscopy . Methods Cell Biol. 2007; ; 79 : :721.–739. 33.
Jin L, Milazzo AC, Kleinfelder S, et al. Applications of direct detection device in transmission electron microscopy . J Struct Biol. 2008; ; 161 : :352.–358. 34.
Dubochet J, Adrian M, Chang JJ, et al. Cryo-electron microscopy of vitrified specimens . Q Rev Biophys. 1988; ; 21 : :129.–228. 35.
Al-Amoudi A, Norlen LP, Dubochet J. Cryo-electron microscopy of vitreous sections of native biological cells and tissues . J Struct Biol. 2004; ; 148 : :131.–135. 36.
Frank J. The envelope of electron microscopic transfer functions for partially coherent illumination . Optik. 1973; ; 38 : :519.–536. 37.
Frank J, Al-Ali L. Signal-to-noise ratio of electron micrographs obtained by cross correlation . Nature. 1975; ; 256 : :376.–379. 38.
Frank J. Averaging of low exposure electron micrographs of non-periodic objects . Ultramicroscopy. 1975; ; 1 : :159.–162. 39.
Van Heel M, Frank J. Use of multivariate statistics in analysing the images of biological macromolecules . Ultramicroscopy. 1981; ; 6 : :187.–194. 40.
Frank J, Verschoor A, Boublik M. Multivariate statistical analysis of ribosome electron micrographs. L and R lateral views of the 40 S subunit from HeLa cells . J Mol Biol. 1982; ; 161 : :107.–133. 41.
Van Heel M, Portugal R, Schatz M. Multivariate stastitical analysis in single particle (cryo)electron microscopy . In: Orlova E, Verkleij A, eds. Handbook on DVD: 3D-EM in life sciences . London: : 3D-EM Network of Excellence; , 2009. 42.
Frank J, Shimkin B, Dowse H. SPIDER: a modular software system for electron image processing . Ultramicroscopy. 1981; ; 6 : :343.–358. 43.
Radermacher M, Wagenknecht T, Verschoor A, Frank J. Three-dimensional reconstruction from a single-exposure, random conical tilt series applied to the 50S ribosomal subunit of Escherichia coli . J Microsc. 1987; ; 146 : :113.–136. 44.
Frank J, Penczek P, Grassucci R, Srivastava S. Three-dimensional reconstruction of the 70S Escherichia coli ribosome in ice: the distribution of ribosomal RNA . J Cell Biol. 1991; ; 115 : :597.–605. 45.
Frank J, Agrawal RK. A ratchet-like inter-subunit reorganization of the ribosome during translocation . Nature. 2000; ; 406 : :318.–322. 46.
Frank J, Gao H, Sengupta J, et al. The process of mRNA-tRNA translocation . Proc Natl Acad Sci USA. 2007; ; 104 : :19671.–19678. 47.
Henderson R, Unwin PN. Three-dimensional model of purple membrane obtained by electron microscopy . Nature. 1975; ; 257 : :28.–32. 48.
Singer SJ, Nicolson GL. The fluid mosaic model of the structure of cell membranes . Science. 1972; ; 175 : :720.–731. 49.
Henderson R, Baldwin JM, Ceska TA. Model for the structure of bacteriorhodopsin based on high-resolution electron cryo-microscopy . J Mol Biol. 1990; ; 21 : :899.–929. |