| |
| Med Sci (Paris). 34(1): 63–71. doi: 10.1051/medsci/20183401015.Mettre la cellule au coeur de la recherche contre le cancer Bruno Goud1* and Daniel Louvard1** 1Institut Curie, université de recherche Paris sciences et lettres (PSL), CNRS, UMR 144, 26, rue d'Ulm, 75248 Paris Cedex 05, France |
Il n’est pas surprenant de constater que les principales questions liées à la compréhension des mécanismes moléculaires du développement, de la survie et de la mort des organismes, et des processus de dérégulation qui se produisent dans des maladies telles que le cancer, les maladies neurodégénératives ou les maladies rares, convergent vers la cellule. En d’autres termes, la cellule, unité de base du vivant, reste plus que jamais un niveau essentiel pour la compréhension de la vie et des pathologies. Nous souhaitons illustrer dans cette revue comment une approche cellulaire intégrée a apporté et portera les progrès futurs de la recherche contre le cancer. |
Historique et état des lieux Si le médecin allemand Rudolf Virchow (1821-1902) fut le premier à proposer, en 1855, que les pathologies ont comme origine un dysfonctionnement des cellules [1], c’est le biologiste Theodore Boveri (1862-1915) qui émit, en 1902, l’hypothèse d’une base cellulaire du cancer [2]. En se fondant sur les très beaux dessins de mitoses anormales dans les cellules cancéreuses réalisés par Léo Hansemann et publiés en 1890 [3] et sur ses propres travaux sur le développement des embryons d’oursin, Boveri proposa en effet qu’une tumeur dérive d’une seule cellule qui se divise de manière incontrôlée et qui prolifère à cause d’un nombre élevé de centrosomes (supérieur à 2), ce qui conduit à une aneuploïdie et à un fonctionnement anormal de ses chromosomes. Même si sa théorie cellulaire fut rejetée à l’époque par ses pairs, beaucoup de prédictions de Boveri se révélèrent exactes comme l’illustre, en particulier, la découverte, presque 100 ans après, des oncogènes et des gènes suppresseurs de tumeurs majoritairement impliqués dans la prolifération ou le cycle cellulaire. La recherche fondamentale sur le cancer a mis l’accent, à juste titre, sur l’étude de la fonction et de la régulation de ces oncogènes (en particulier KRAS et MYC) et suppresseurs de tumeur (TP53 [tumor protein 53], RB [rétinoblastome], BRCA1 [breast cancer 1] et APC [adenomatous polyposis coli]) qui entraînent l’apparition de cancers quand ils sont activés par mutations, ou quand leur expression est perdue par délétion [4-7]. On sait aujourd’hui que la réalité est beaucoup plus complexe, l’activation d’un oncogène ou la perte d’un gène suppresseur de tumeur ne suffisant en effet pas à induire les étapes de la progression tumorale. La grande majorité des cancers résulte d’altérations séquentielles de plusieurs oncogènes et gènes suppresseurs de tumeur, comme l’a proposé Bert Vogelstein dans son article princeps sur le cancer colorectal [8]. Mais cette hypothèse a aussi ses limites et n’explique pas, par exemple, l’inadéquation entre le développement rapide de certains cancers et le taux de mutations plus faible qu’attendu, ou l’observation que les cancers impliquent souvent des gènes très différents, selon l’organe touché. Une vision qui s’arrêterait seulement à l’identification des gènes impliqués ne rend pas compte de la diversité phénotypique observée dans les cellules au sein d’une même tumeur, des nombreuses aberrations chromosomiques, ou de l’apparition de la résistance aux thérapies. L’étendue des fonctions que certains oncogènes pourraient jouer en tant que master regulators1 de la structure de la chromatine et de la transcription, peut cependant en partie expliquer cette diversité. Cela pourrait être le cas, par exemple, des protéines Myc, produits de l’oncogène MYC, qui sont surexprimées dans 60 à 70 % des cancers [9]. Depuis une dizaine d’années, l’accent est mis sur l’importance des modifications épigénétiques dans la progression tumorale avec, en premier lieu, celles touchant la méthylation de l’ADN, comme le suggère les travaux montrant que les oncogènes sont hypométhylés et les gènes suppresseurs de tumeur, hyperméthylés [10]. Les modifications post-traductionnelles des histones, en particulier par acétylation, jouent aussi un rôle majeur dans la régulation épigénétique de l’expression des gènes [11]. Les gènes impliqués dans la progression tumorale ne sont donc pas nécessairement mutés, mais ils peuvent être inactivés, ou leur expression modifiée, par des mécanismes épigénétiques. L’épigénétique du cancer suscite aujourd’hui beaucoup d’intérêt et d’espoir même si des questions fondamentales demeurent : les modifications épigénétiques ne sont-elles que le résultat de mutations dans la séquence des gènes ou, à l’inverse, conduisent-elles au cancer en altérant l’expression des gènes [12] ? La génomique et l’épigénomique du cancer ont bénéficié des progrès extraordinaires des techniques de séquençage à haut-débit (aussi appelées NGS pour next generation sequencing). Ces techniques qui ont considérablement réduit le temps et le coût du séquençage complet du génome des cellules cancéreuses, ont permis la classification moléculaire des tumeurs, l’identification de nombreux biomarqueurs, et ont ouvert la voie à « la médecine de précision » et aux thérapies ciblées. Elles ont aussi permis de mettre récemment en évidence, dans différents types de tumeurs et de leucémies, des mutations dans les gènes codant les méthylases et les déméthylases (DNMT[DNA methyltransferase] 1, 3A, 3B, ou TET[TET methylcytosine dioxygenase]1, 2, 3) ainsi que dans ceux codant les histones acétylases (HAT) et les désacétylases (HDAC), suggérant que les facteurs épigénétiques sont des moteurs potentiels de la tumorigenèse [12]. |
Questions et objectifs pour le futur Les altérations génétiques, et très probablement épigénétiques, conduisent à une dérégulation générale des grandes fonctions cellulaires, en premier lieu, celles gouvernant l’homéostasie cellulaire, les voies de signalisation, le cycle cellulaire, l’adhérence entre cellules et matrice extracellulaire et entre cellules qui contribuent à leur migration hors de leur tissu d’origine et aux métastases. Mais par quels mécanismes ces altérations impactent les grandes fonctions cellulaires reste encore très mal compris. La génomique des cellules cancéreuses a ainsi permis d’identifier des listes de gènes, dont il faut maintenant étudier précisément le rôle au niveau du fonctionnement global de la cellule et des tissus au sein desquels la cellule maligne prolifère. La question centrale qui demeure est la compréhension des liens existant entre génotype et phénotype. Sur des informations génomiques, peut-on prédire l’agressivité de certaines cellules tumorales et leur résistance aux traitements ? Pourquoi différentes signatures moléculaires pour différents cancers ? Pourquoi des gènes d’expression ubiquitaire, comme APC ou BRCA1, n’ont-ils des incidences que dans certains cancers, en l’occurrence, le cancer du côlon et le cancer du sein, respectivement ? Quelle est l’origine et quelles sont les conséquences de l’hétérogénéité intra-tumorale ? Pour répondre à ces questions, il semble maintenant essentiel d’étudier la fonction des gènes impliqués dans les cancers, et des processus épigénétiques, à la lumière de l’économie de la cellule normale et cancéreuse et de ses interactions avec son environnement. |
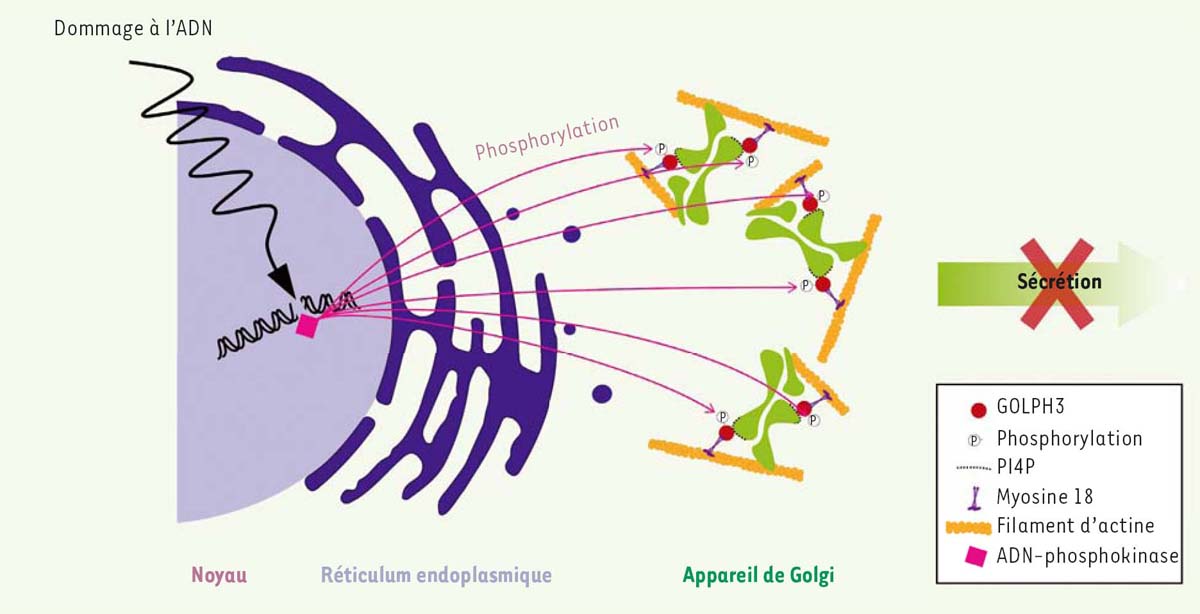
Au niveau intracellulaire Une approche cellulaire intégrée doit se situer en premier lieu au niveau des organites (compartiments) sur lesquels repose le fonctionnement des cellules eucaryotes. Des travaux entrepris dans les années récentes, montrent que les modifications de l’homéostasie des organites intracellulaires jouent un rôle clé dans les étapes précoces de la transformation cellulaire. Un des exemples les mieux connus illustrant ces recherches est celui du centrosome qui joue un rôle essentiel dans l’organisation du réseau des microtubules et du fuseau mitotique des cellules eucaryotes, et dont Boveri avait prédit que l’amplification conduisait au processus d’aneuploïdie et au cancer [2]. Même si on ne doit pas exclure l’hypothèse que l’amplification du nombre de centrosomes est une conséquence de l’activité des oncogènes, des travaux réalisés dans plusieurs modèles montrent que l’augmentation du nombre de centrosomes joue un rôle essentiel dans l’initiation du cancer et la progression tumorale. L’équipe de David Pellman a ainsi démontré que l’augmentation du nombre de centrosomes (obtenue en surexprimant la sérine/thréonine protéine kinase PLK4 [polo-like kinase 4]) induit un phénotype invasif similaire à celui obtenu en surexprimant l’oncogène ERBB2 (ou HER2 [human epidermal growth factor receptor 2]) [13]. Les équipes de Cédric Blanpain et Renata Basto ont montré que l’aneuploïdie résultant de l’amplification centrosomale accélère l’apparition de cancers de l’épiderme de souris déficientes en p53 [14], et ceux de l’équipe d’Andrew Holland montrent que, chez la souris, l’amplification des centrosomes est suffisante pour promouvoir une tumorigenèse spontanée [15]. Un autre exemple de fonction cellulaire directement impliquée dans l’oncogenèse qui fait actuellement l’objet de très nombreux travaux est l’autophagie ( ). ). ( ) Voir le numéro thématique Autophagie, m/s n° 3, mars 2017 ) Voir le numéro thématique Autophagie, m/s n° 3, mars 2017 L’autophagie est une fonction essentielle permettant de contrôler en permanence l’homéostasie cellulaire en conditions physiologiques et dont les bases moléculaires des mécanismes ont été établies par les biologistes cellulaires [16]. L’autophagie a d’abord été considérée comme un mécanisme suppresseur de tumeur : une délétion d’un gène essentiel de l’autophagie, ATG6/BECN1 codant la Bécline-1, étant observée dans de nombreux cancers [17]. Mais on sait aujourd’hui qu’elle est aussi un promoteur de tumeurs, en particulier dans les cancers où KRAS est activé [18]. Les travaux futurs devront établir si le rôle de l’autophagie dans la progression tumorale est plus important dans certains cancers que dans d’autres, et si des drogues ciblant les protéines impliquées dans l’autophagie sont efficaces [19]. Une approche cellulaire intégrée est également essentielle pour comprendre la régulation spatio-temporelle des voies de signalisation. Il est maintenant établi que les récepteurs de surface activés par la fixation de leur ligand ne signalent pas seulement depuis la surface cellulaire, et que leur endocytose ne résulte pas uniquement en l’atténuation de la propagation du signal; la signalisation se poursuit au cours du processus d’endocytose. Elle concerne tous les compartiments de cette voie (endosomes précoces, tardifs et lysosomes) comme cela a été démontré pour les récepteurs des facteurs de croissance comme l’EGFR (epidermal growth factor receptor) qui, de surcroît, peut activer des voies de signalisation au niveau du noyau [20]. L’impact de l’oncogenèse sur la dynamique des compartiments d’endocytose et sur les voies de signalisation qui y sont associées reste encore très mal connu et mériterait de nouvelles recherches. D’autres organites sont des acteurs majeurs de l’oncogenèse. Il est maintenant bien établi que la réponse au stress du réticulum endoplasmique, en particulier l’UPR (unfolded protein response) joue un rôle majeur dans le développement du cancer et les mécanismes de résistance aux traitements anti-cancéreux [21]. De même, l’activité des mitochondries, à commencer par la production d’énergie, contribue à de multiples niveaux à la tumorigenèse [22]. De manière peut-être plus inattendue, il a été montré récemment que l’appareil de Golgi, organite central de la voie de biosynthèse/sécrétion des cellules, remplit des fonctions qui vont bien au-delà de son rôle dans le transport intracellulaire. Par exemple, les voies de signalisation activées par Ras dépendent de la localisation de la protéine, et le pool golgien de Ras joue un rôle important dans l’apoptose où il peut exercer un effet antagoniste sur les signaux de différenciation [23, 24]. Un lien direct entre l’appareil de Golgi et le cancer a aussi été établi par les travaux montrant que la protéine GOLPH3 (Golgi phosphoprotein 3) est un oncogène [25]. La surexpression de GOLPH3 est observée dans de nombreux cancers et confère une résistance à la mort cellulaire par des drogues qui endommagent l’ADN (24). GOLPH3 se fixe sur les membranes golgiennes via le PI4P (phosphoinositide-4-phosphate), et il est important de rappeler qu’une surexpression de la PI4-kinase est observée dans de nombreux cancers [26]. GOLPH3 interagit aussi avec la Myosine 18, dont le gène est muté ou hyperméthylé dans plusieurs cancers [27] (Figure 1).  | Figure 1. Effets des dommages à l’ADN sur l’appareil de Golgi. En réponse à un dommage à l’ADN, DNA-PK (DNA-phosphokinase) phosphoryle GOLPH3 (Golgi phosphoprotein 3), une protéine golgienne se liant au PI4P (phosphatidylinositol-4-phosphate, le phosphoinositide le plus abondant de l’appareil de Golgi). GOLPH3 phosphorylé se lie à la Myosine 18 et au cytosquelette d’actine et induit une fragmentation de l’appareil de Golgi ainsi qu’une inhibition du processus de sécrétion, ce qui favorise la survie cellulaire. Les facteurs sécrétés impliqués n’ont pas été encore identifiés. |
Soulignons par ailleurs qu’il est essentiel de comprendre les multiples voies de transport qui connectent les organites entre eux pour optimiser le ciblage intracellulaire des drogues anti-cancéreuses. Les travaux des équipes de L. Johannes et d’E. Tartour sur l’utilisation du fragment B de la toxine de Shiga2 à des fins de vaccination anti-tumorale illustrent parfaitement ce constat [28]. |
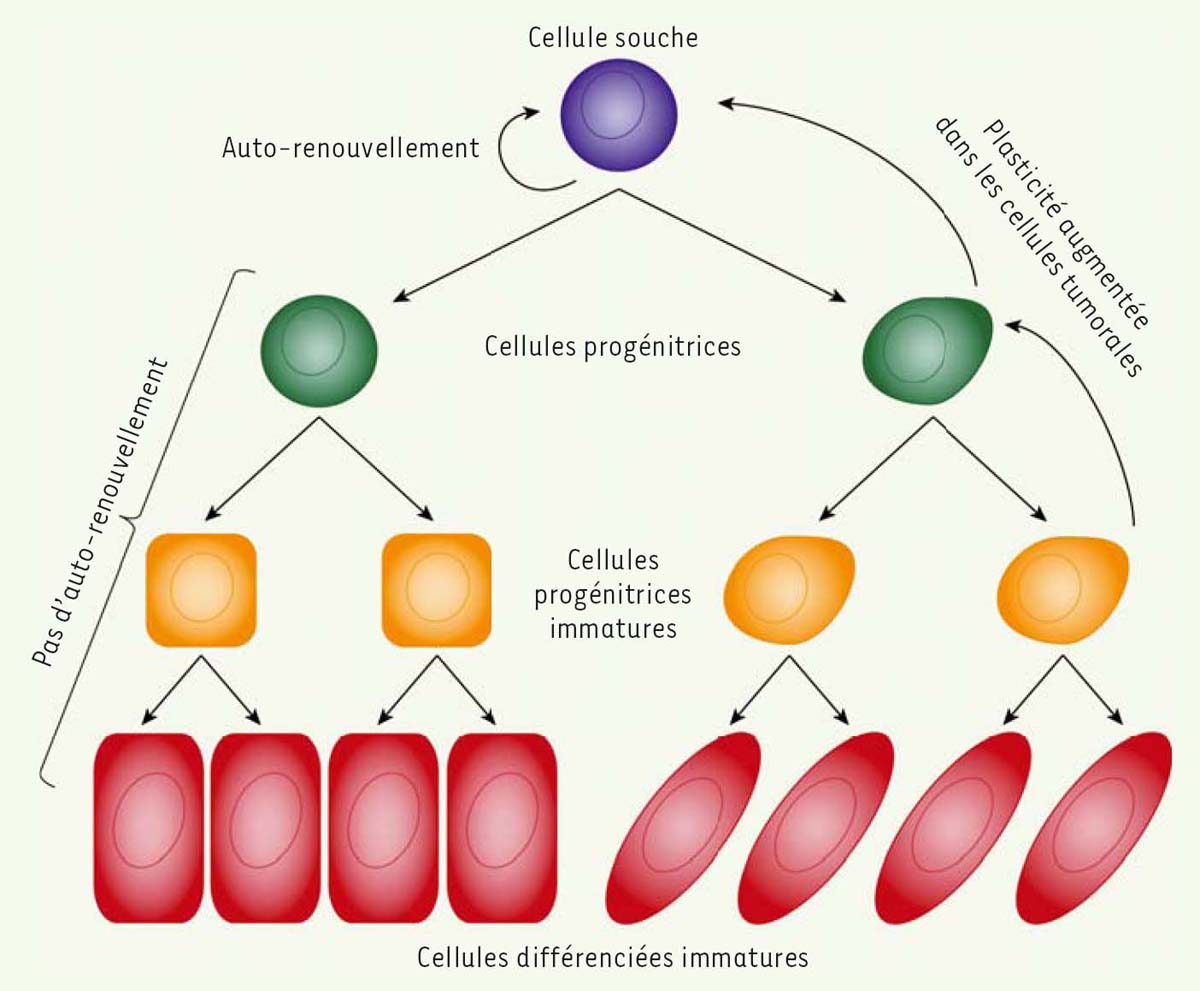
Depuis 20-25 ans, les chercheurs ont redécouvert une propriété intrinsèque des cellules normales, plus particulièrement des cellules épithéliales : leur plasticité phénotypique. Les modifications morphologiques et fonctionnelles de ces cellules sont liées à un changement majeur de l’expression du programme de différenciation que sous-tend une modulation du profil d’expression génique des cellules. Les biologistes du développement ont décrit depuis longtemps les propriétés des cellules épithéliales embryonnaires. Ce phénomène appelé transition épithélio-mésenchymateuse (TEM ou EMT, en anglais) est fréquent et essentiel au cours de l’embryogénèse. La TEM peut être réactivée dans les cellules d’organismes adultes dans des situations physiologiques aiguës (cicatrisation, renouvellement des tissus après une phase aiguë de mortalité cellulaire) ou physiopathologiques (fibroses, cancers, etc.). La TEM est réversible (par transition mésenchymo-épithéliale, TME ou MET). La plasticité des cellules épithéliales leur permet une « transformation » en cellules mésenchymateuses et vice versa [29]. Depuis plusieurs décennies, les cancérologues se sont interrogés sur l’origine cellulaire des cellules malignes. S’agit-il d’un processus stochastique dans lequel toutes les cellules d’un tissu ont la même probabilité d’acquérir et de propager les altérations génomiques, ou d’un processus hiérarchisé dans lequel seules certaines cellules ont la capacité de former une tumeur ? L’hypothèse des cellules souches tumorales (tumor stem cell) ou des cellules initiatrices des tumeurs (tumor initiating cell) a fait l’objet de nombreuses controverses qui ne sont pas complétement résolues. Les hématologistes ont cependant accumulé un grand nombre de preuves expérimentales en faveur de cette hypothèse pour les cancers des cellules sanguines [30]. Pour les tumeurs solides, la situation est plus nuancée. Elle pourrait dépendre du tissu d’origine. La plasticité des cellules tumorales des tumeurs solides (sein, côlon, peau, pancréas, poumon) pourrait justifier la complexité des interprétations. Une cellule maligne différenciée peut acquérir des propriétés de cellule souche maligne analogues à celles d’une cellule souche normale ayant accumulée des mutations oncogéniques. La réversibilité de la TEM/TME, probablement induite par des changements épigénétiques, contribuerait ainsi à l’hétérogénéité cellulaire et génotypique observées dans les tumeurs solides (Figure 2). 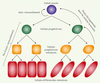 | Figure 2.Potentiel et plasticité des cellules souches. Les cellules souches permettent le renouvellement des tissus adultes. Les divisions des cellules souches normales en cellules progénitrices s’effectuent essentielle-ment de façon unidirectionelle. Au contraire, les cellules souches et progénitrices malignes porteuses de mutations oncogéniques ont une plasticité accrue leur permettant d’accroitre la population de cellules souches malignes (adapté de [ 31]). |
Il est important de souligner que la recherche de solutions thérapeutiques pour éradiquer les tumeurs ne peut ignorer l’existence de l’hétérogénéité cellulaire au sein d’une tumeur. Toutes les cellules d’une tumeur n’ont pas la même susceptibilité ou résistance aux agents anti-cancéreux. L’hypothèse communément admise de résistance acquise par mutation de la cible moléculaire lors d’un traitement ne peut ignorer le fait que les cellules souches normales et tumorales peuvent être intrinsèquement résistantes aux agents anti-cancéreux. À terme, il sera essentiel de développer des drogues contre les cellules malignes les plus nombreuses dans la tumeur et contre les cellules à l’origine de la tumeur, moins nombreuses mais plus résistantes. La stratégie qui s’impose repose, comme pour d’autres approches thérapeutiques des cancers (thérapies ciblées), sur l’utilisation rationnelle et adaptée d’une combinatoire d’agents thérapeutiques ciblant les voies de signalisation modifiées par mutations génétiques et épigénétiques. Les travaux d’Eric Lander et Robert Weinberg dans ce domaine ont ouvert la voie à l’identification de molécules susceptibles de tuer les cellules souches tumorales mais présentant une faible toxicité pour les cellules souches normales [31]. Raphael Rodriguez et son équipe ont poursuivi ces recherches, en étudiant le mécanisme d’action de la salinomycine, un antibiotique connu3 qui a également des propriétés anti-tumorales. Ils ont pu montrer que cette molécule bloque en fait la translocation du fer au niveau des lysosomes, provoquant la production d’espéces réactives de l’oxygène (ROS) et une mort cellulaire qui s’apparente à la ferroptose [32, 53] ( ). ). ( ) Voir la Synthèse de A. Hamaï et M. Mehrpour, m/s n° 3, mars 2017, page 260 ) Voir la Synthèse de A. Hamaï et M. Mehrpour, m/s n° 3, mars 2017, page 260 |
Au niveau de la cellule en interaction avec son microenvironnement La compréhension des mécanismes complexes qui contrôlent la formation de métastases (migration, dissémination, extravasion, adaptation aux sites secondaires) est un défi pour les biologistes cellulaires. Pour mettre en œuvre de nouvelles thérapies efficaces, on ne pourra faire l’économie de nouvelles recherches au regard des échecs des thérapies pour éradiquer les métastases. Les quelques exemples qui suivent illustrent combien il est important de tenir compte du microenvironnement de la tumeur et d’expérimenter dans un contexte in vivo ou dans des conditions de culture tridimensionnelle (3D) qui miment la situation in vivo. |
Signalisation et migration Les voies de signalisation ne se limitent pas à la surface cellulaire. L’équipe de Johanna Ivaska a montré que la kinase FAK (focal adhesion kinase), qui joue un rôle essentiel dans la dynamique des adhérences focales, contribue à cette voie via les endosomes précoces [33]. Cette voie de signalisation à partir des endosomes inhibe la mort cellulaire et promeut la croissance cellulaire en l’absence d’adhérence sur un substrat. Inversement, si elle est bloquée, les cellules cancéreuses redeviennent sensibles à la mort cellulaire induite par détachement du substrat (anoikis4) et leur potentiel métastatique en est alors altéré. Les cellules métastatiques utilisent les intégrines pour leur migration. Les travaux récents de Guillaume Montagnac [34] ont révélé, in vivo, un mécanisme original de migration des cellules tumorales le long des fibres de collagène. Les intégrines fermement liées à ces fibres ne peuvent être internalisées par la cellule. Elles génèrent cependant une force adhésive qui permet à la cellule tumorale de migrer dans un environnement en trois dimensions (3D). Les recherches de Danijela Vignjevic apportent un éclairage nouveau sur le rôle des fibroblastes « activés » en contact ou à proximité des cellules tumorales (les CAF, ou cancer-associated fibroblast). Ces recherches montrent une coopération entre fibroblastes et cellules tumorales impliquant les forces mécaniques générées par les fibroblastes, les facteurs secrétés, les intégrines et la fibronectine [35]. Ces recherches exploitent des techniques d’imagerie in vivo et des cultures cellulaires en 3D sur des substrats obtenus par microfabrication. Outre leur intérêt cognitif, ces travaux apportent des réponses pour expliquer, par exemple, les échecs des inhibiteurs de métalloprotéases et ouvrent des possibilités pour mieux les utiliser à l’avenir. Cette conception d’une approche globale des cellules et de leur environnement est essentielle pour définir les meilleures combinaisons d’agents anti-métastatiques [36]. |
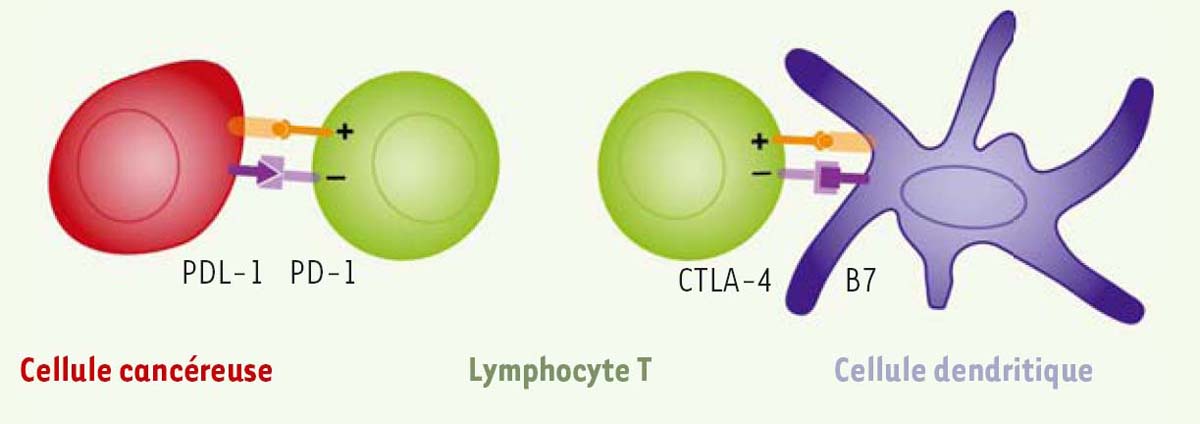
Système immunitaire et microbiote Les recherches en immunologie cellulaire récentes ont clairement montré que l’inflammation et le système immunitaire jouent des rôles importants dans la carcinogenèse et le développement des tumeurs. Si l’inflammation chronique (et les infections chroniques) augmente la fréquence de cancers, les réponses immunitaires adaptatives (spécifiques) peuvent provoquer le rejet de tumeurs, même à des stades tardifs. L’immunothérapie utilisant les anticorps bloquant PD-1 (programmed cell death 1), un récepteur inhibiteur des lymphocytes T, a des effets thérapeutiques spectaculaires dans certains cancers métastatiques avancés (comme les mélanomes). Ces succès thérapeutiques issus de recherches fondamentales en biologie cellulaire montrent, pour la première fois, que la manipulation des cellules de l’environnement tumoral (en l’occurrence les lymphocytes) peut modifier l’évolution clinique du cancer [37, 38] ( ). ). ( ) Voir la Synthèse de W.H. Fridman et C. Sautès-Fridman, m/s n° 4, avril 2014, page 359 ) Voir la Synthèse de W.H. Fridman et C. Sautès-Fridman, m/s n° 4, avril 2014, page 359 Ce changement de paradigme (on cible l’environnement tumoral et non la tumeur elle-même) a provoqué un intérêt accru pour les cellules de l’environnement tumoral : les tumeurs sont ainsi devenues un véritable tissu, dont l’équilibre biologique doit être compris et peut-être manipulé à des fins thérapeutiques. De nombreux travaux tentent d’identifier les cellules de l’environnement tumoral et de comprendre leur fonctionnement ( ). ). ( ) Voir le numéro thématique Microenvironnements tumoraux, m/s n° 4, avril 2014 ) Voir le numéro thématique Microenvironnements tumoraux, m/s n° 4, avril 2014 On connaît de mieux en mieux les mécanismes par lesquels les tumeurs influencent leur environnement immunitaire en empêchant l’activation des lymphocytes. L’étude de ces mécanismes de suppression a permis de mieux comprendre les mécanismes généraux de maintien de la tolérance immunitaire dans les tissus sains. Des centaines d’essais cliniques qui manipulent les différentes voies de suppression immunitaire sont désormais en cours (Figure 3). Cette révolution ne doit cependant pas sous-estimer les effets secondaires sévères potentiels ou révélés (auto-immunité, réaction inflammatoire, etc.) que doivent gérer les cliniciens en charge des patients.  | Figure 3. L’immunothérapie des cancers exploite les interactions entre cellules via des récepteurs de la surface cellulaire. Certaines associations de récepteurs activent le système immunitaire (+), d’autres l’inhibent (-). Par exemple, CTLA-4(cytotoxic T-lymphocyte-associated protein 4)/B7(CD80/CD86) entre cellules dendritiques et lymphocytes T et PDL1 (programmed death-ligand 1) et PD1 (programmed cell death 1) entre cellules tumorales et lymphocytes T inhibent la réponse immunitaire. Grâce à des anticorps anti-PDL1 ou anti-PD1 ou encore anti-CTLA-4, il est possible de rompre ces interactions et d’activer une réponse immunitaire contre les cellules tumorales. PD-L1 est aussi exprimé sur des cellules myéloïdes infiltrant la tumeur, qui pourraient jouer un rôle important d’immunosuppression et rendraient compte de l’efficacité des anti-PD-L1 même lorsque les cellules tumorales n’expriment pas ou très peu cette molécule. |
Une autre série de travaux récents relie le cancer à son environnement. Ils concernent le microbiote, c’est-à-dire les bactéries commensales qui vivent dans nos organismes (principalement en contact des muqueuses, dont celles de notre intestin) et dont le nombre est dix fois supérieur à celui des cellules d’un individu ( ). ). ( ) Voir le numéro thématique Microbiote, m/s n° 11, novembre 2016 ) Voir le numéro thématique Microbiote, m/s n° 11, novembre 2016 Des espèces particulières de bactéries commensales contribuent à différents aspects des réponses immunitaires, notamment aux réponses immunitaires contre les tumeurs. La manipulation de la composition du microbiote afin d’optimiser l’immunothérapie du cancer est l’une des voies prometteuses actuellement en cours d’étude préclinique et clinique [39]. |
Nombre de voies de transduction du signal ont été découvertes comme pouvant être activées en réponse à des sollicitations mécaniques subies par les cellules en culture. Or, les tumeurs modifient les propriétés mécaniques du tissu qu’elles constituent, par une augmentation de sa rigidité, liée à la fibrose, aux stades avancés de la tumorigenèse. Ainsi, chez la souris, les cellules épithéliales de la glande mammaire en culture ont des contractions adaptatives de type acto-myosine, ce qui provoque l’activation mécanique de la kinase Akt (protéine kinase B), et l’induction de la progression tumorale [40]. Une « pression mitotique » de croissance tumorale, présente dès les stades les plus précoces de la tumorigenèse, s’applique alors de plus aux cellules saines environnantes. In vivo, la voie tumorigène β-caténine se trouve ainsi activée mécaniquement, ce qui induit l’hyper-prolifération et la tumorigenèse des cellules épithéliales de l’intestin [41]. |
Interactions métaboliques Les facteurs de l’environnement tumoral (hypoxie, acidose, etc.) agissent sur les voies de signalisation qui sont régulées par les oncogènes et les gènes suppresseurs de tumeur, et sur le métabolisme des cellules cancéreuses (et réciproquement). Comprendre l’implication des voies métaboliques dans le dialogue entre cellules cancéreuses et stroma nécessite une approche intégrée de biologie cellulaire. L’étude des cellules étoilées du pancréas illustre l’importance de cette thématique. Ces cellules produisent en effet un acide aminé non-essentiel, l’alanine, qui stimule la croissance des cellules de la tumeur [42]. Un autre exemple est CLIC3 (chloride intracellular channel protein 3), qui présente une activité glutathion-dependent oxydoreductase, qui est surexprimée dans les cancers de l’ovaire et du pancréas et régule les voies de recyclage dépendant de la GTPase RAB25 [43]. Cette enzyme est secrétée par les CAF, ce qui a pour conséquence de stimuler l’angiogenèse et l’invasion tumorale [44]. |
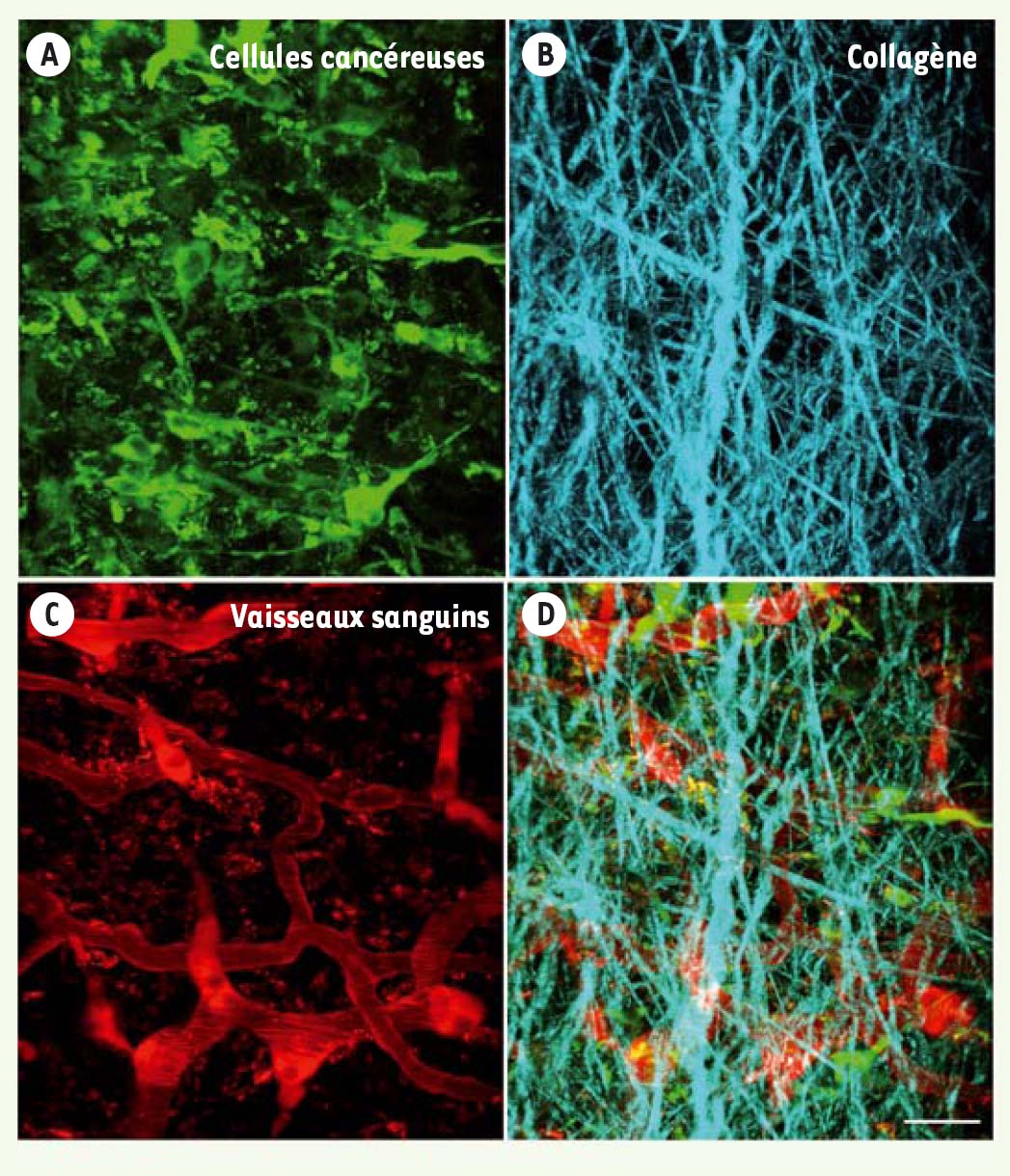
Les nouveaux outils de la biologie cellulaire Les biologistes cellulaires disposent aujourd’hui de nouveaux outils, tant méthodologiques que conceptuels, pour faire face aux défis de demain. Sur le plan méthodologique, au-delà de la révolution de l’édition (ou correction) du génome par la méthode fondée en particulier sur CRISPR/Cas9, qui permet d’étudier la fonction des protéines dans un contexte physiologique, on assiste à de remarquables avancées en imagerie. Les microscopies à molécule unique (PALM/STORM et STED)5 [54] ( ) permettent de franchir la limite de résolution de la lumière (soit 200 nm), et la microscopie à « feuilles de lumière » [55] ( ) permettent de franchir la limite de résolution de la lumière (soit 200 nm), et la microscopie à « feuilles de lumière » [55] ( ) de visualiser des molécules au sein des cellules et organismes avec une très haute résolution spatio-temporelle [45]. La microscopie intravitale à deux ou trois photons permet de suivre in vivo le processus métastatique (Figure 4). ) de visualiser des molécules au sein des cellules et organismes avec une très haute résolution spatio-temporelle [45]. La microscopie intravitale à deux ou trois photons permet de suivre in vivo le processus métastatique (Figure 4). ( ) Voir la Nouvelle de R. Chéreau et al., page 17 de ce numéro ) Voir la Nouvelle de R. Chéreau et al., page 17 de ce numéro ( ) Voir la Synthèse de P.P. Girard et B.C. Forget, m/s n° 8-9, aoûtseptembre 2011, page 753 ) Voir la Synthèse de P.P. Girard et B.C. Forget, m/s n° 8-9, aoûtseptembre 2011, page 753 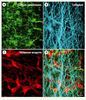 | Figure 4. Cellules cancéreuses observées in vivo par microscopie intra-vitale à 2 photons. Les cellules cancéreuses CT26 ont été injectées par voie sous-cutanée dans le dos d’une souris Nude et analysées en microscopie six semaines plus tard. A. Cellules cancéreuses exprimant la GFP (green fluorescent protein) (en vert). B. Les fibres de collagène du stroma sont visualisées en bleu grâce à leur auto-fluorescence intrinsèque (seconde harmonique). C. Les vaisseaux sanguins sont marqués par du dextrane injecté par voie intraveineuse (en rouge). D. Image composite superposant les images A, B et C. Reconstitution 3D de coupes optiques de la tumeur (46 images sériées de 3 μm d’épaisseur); échelle : 30 μm (© clichés de Danijela Vignjevic et Luc Fetler, Institut Curie, Paris, France). |
L’optogénétique permet, quant à elle, de contrôler in vivo par la lumière les interactions entre protéines et de manipuler localement des fonctions cellulaires [46]. L’imagerie électronique a aussi fait des progrès remarquables, en particulier avec les développements récents de la cryo-electron microscopy [56]( ) qui permet d’étudier la structure de complexes macromoléculaires à quelques angströms de résolution [47]. D’autres outils, fondés sur les techniques de microfluidique ou de microfabrication, permettent maintenant d’analyser en 3 dimensions et en temps réel les interactions entre cellules tout en contrôlant leur microenvironnement [48]. ) qui permet d’étudier la structure de complexes macromoléculaires à quelques angströms de résolution [47]. D’autres outils, fondés sur les techniques de microfluidique ou de microfabrication, permettent maintenant d’analyser en 3 dimensions et en temps réel les interactions entre cellules tout en contrôlant leur microenvironnement [48]. ( ) Voir le Dossier technique de J.A. Boutin et al., m/s n° 8-9, août-septembre 2016, page 758 ) Voir le Dossier technique de J.A. Boutin et al., m/s n° 8-9, août-septembre 2016, page 758 Les organoïdes, obtenus à partir de cellules souches normales ou malignes, reproduisent in vitro l’architecture et la complexité des tissus normaux ou des tumeurs. Cette technologie introduite par Hans Clevers avec des cellules de l’intestin (minigut) [49] et généralisable à de nombreux organes, est promise à de nombreux développements et découvertes. Les organoïdes sont des modèles de choix pour l’étude en 3 dimensions des propriétés d’ensembles cellulaires dans des conditions expérimentales bien définies. La recherche de combinatoires de drogues pour détruire des cellules tumorales de profil génomique connu est déjà bien illustrée par les travaux récents de l’équipe de H. Clevers [50]. De façon spectaculaire, les résultats obtenus par cette équipe sur des organoïdes d’intestin provenant de patients souffrant de mucoviscidose (présentant un gène CFTR [cystic fibrosis transmembrane conductance regulator] muté) ont permis de cribler des chimiothèques et d’identifier des molécules capables de restaurer la fonction du gène déficient in vitro, et in vivo sur des malades [51]. Sur le plan conceptuel, la cellule doit être considérée comme une entité dynamique fonctionnelle dont l’organisation spatio-temporelle est étroitement couplée à son environnement physico-chimique. Les apports de la physique, des mathématiques et de la bio-informatique (systems biology) sont déterminants. La biologie cellulaire est devenue une science quantitative. Les fonctions cellulaires commencent à faire l’objet d’une modélisation mathématique, ce qui permettra de prédire pourquoi et comment une cellule devient cancéreuse. La bio-informatique a par exemple permis à notre équipe de créer un modèle de souris développant des métastases de tumeurs intestinales, projet qui avait échoué dans de nombreuses équipes dans le monde [52]. De même on peut anticiper qu’il sera possible de prédire pour une tumeur dont les déficits génétiques sont identifiés quelles résistances et quelles sensibilités aux drogues sont attendues, et par conséquent de proposer aux malades des thérapies ciblées adaptées à leur tumeur (médecine de précision) [57] ( ). ). ( ) Voir la Synthèse de Z. Saidak et al., m/s n° 12, décembre 2017, page 1055 ) Voir la Synthèse de Z. Saidak et al., m/s n° 12, décembre 2017, page 1055 |
Les thérapies anti-cancéreuses développées récemment ont surtout pris en compte jusqu’à présent les données « omiques », essentiellement génomiques et transcriptomiques. Il nous paraît essentiel dans l’avenir d’intégrer ces données dans le contexte de la cellule normale et cancéreuse. La biologie cellulaire, se nourrissant des apports d’autres disciplines comme la physique, la chimie ou encore les mathématiques, contribuera dans le futur à comprendre les principes de l’organisation des cellules et des tissus, et favorisera l’essor d’une « system cancer cell biology ». Cette approche pluridisciplinaire apportera des connaissances essentielles pour la conception de nouvelles thérapies ciblées. |
Nous remercions Sebastian Amigorena, Emmanuel Farge, Edith Heard, et Danijela Vignjevic pour leur contribution à l’écriture de ce texte. Nous remercions Michel Bornens, Anne Houdusse, et Renata Basto pour leur lecture critique du manuscrit. Nous remercions Bruno Latgé pour le dessin des Figures 1, 2 et 3. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1. Virchow R. Die Cellularpathologie in ihrer Begründung auf physiologische und pathologische Gewebelehre. Berlin : Verlag: , 1858. 2. Boveri T. Concerning the origin of malignant tumors by Theodor Boveri. Translated and annotated by Henry Harris . J Cell Sci. 2008;; 121 : :1.-84. 3. Hansemann L. Ueber asymmetrische zelltheilung in epithelkrebsen und deren biologische bedeutung . Arch Pathol Anat Physiol Klin Med. 1890;; 119 : :299.-326. 4. Bishop JM. Cancer: the rise of the genetic paradigm . Genes Dev. 1995;; 9 : :1309.-15. 5. Varmus H. The transformation of oncology . Science. 2016;; 352 : :123.. 6. Knudson AG. Mutation and cancer: statistical study of retinoblastoma . Proc Natl Acad Sci USA. 1971;; 68 : :820.-3. 7. Weinberg RA. The retinoblastoma protein and cell cycle control . Cell. 1995;; 81 : :323.-30. 8. Fearon ER, Vogelstein B. A genetic model for colorectal tumorigenesis . Cell. 1990;; 61 : :759.-67. 9. Poole CJ, van Riggelen J. MYC-Master Regulator of the cancer epigenome and transcriptome . Genes. 2017;; 8 : :5.. 10. Hollyday R. A new theory of carcinogenesis . Br J Cancer. 1979;; 40 : :513.. 11. Shen H, Laird PW. Interplay between the cancer genome and epigenome . Cell. 2013;; 153 : :38.-55. 12. Jones PA, Issa JP, Baylin S. Targeting the cancer epigenome for therapy . Nat Rev Genet. 2016;; 17 : :630.-41. 13. Godinho SA, Picone R, Burute M, et al. . Oncogene-like induction of cellular invasion from centrosome amplification . Nature. 2014;; 510 : :167.-71. 14. Sercin O, Larsimont JC, Karambelas AE, et al. . Transient PLK4 overexpression accelerates tumorigenesis in p53-deficient epidermis . Nat Cell Biol. 2016;; 18 : :100.-10. 15. Levine MS, Bakker B, Boeckx B et al. . Centrosome amplification is sufficient to promote spontaneous tumorigenesis in mammals . Dev Cell. 2017;; 40 : :313.-22. 16. Harnett MM, Pineda MA, Latré de Laté P, et al. . From Christian de Duve to Yoshinori Ohsumi: more to autophagy than just dining at home . Biomed J. 2017;; 40 : :9.-22. 17. Qu X, Yu J, Bhagat G, et al. . Promotion of tumorigenesis by heterozygous disruption of the beclin 1 autophagy gene . J Clin Invest. 2003;; 112 : :1809.-20. 18. Guo JY, Xia B, White E. Autophagy mediated tumor promotion . Cell. 2013;; 155 : :1216.-9. 19. Galluzzi L, Pietrocola F, Bravo-San Pedro JM, et al. . Autophagy in malignant transformation and ca ncer progression . EMBO J. 2015;; 34 : :856.-80. 20. Bergeron JJ, Di Guglielmo GM, Dahan S, et al. . Spatial and temporal regulation of receptor tyrosine kinase activation and intracellular signal transduction . Annu Rev Biochem. 2016;; 85 : :573.-9. 21. Avril T, Vauléon E, Chevet E. Endoplasmic reticulum stress signaling and chemotherapy resistance in solid cancers. Oncogenesis. 2017;; 6 : :e373.. 22. Vyas S, Zaganjor E, Haigis MC. Mitochondria and cancer . Cell. 2016;; 166 : :555.-66. 23. Herrero A, Casar B, Colón-Bolea P, et al. . Defined spatiotemporal features of RAS-ERK signals dictate cell fate in MCF-7 mammary epithelial cells . Mol Biol Cell. 2016;; 12 : :1958.-68. 24. Hernandez-Valladares M, Prior IA. Comparative proteomic analysis of compartmentalized Ras signaling . Sci Rep. 2015;; 5 : :17307.. 25. Farber-Katz SE, Dippold HC, Buschman MD, et al. . DNA damage triggers Golgi dispersal via DNA-PK and GOLPH3 . Cell. 2014;; 156 : :413.-27. 26. Waugh MG. Phosphatidylinositol 4-kinases, phosphatidylinositol 4-phosphate and cancer . Cancer Lett. 2012;; 325 : :125.-31. 27. Ouderkirk, JL, Krendel M. Non-muscle myosins in tumor progression, cancer cell invasion, and metastasis . Cytoskeleton. 2014;; 71 : :447.-63. 28. Tran T, Diniz MO, Dransart E, et al. . A therapeutic Her2/neu vaccine targeting dendritic cells preferentially inhibits the groxth of low Her/neu-expressing tumor in HLA-A2 transgenic mice . Clin Cancer Res. 2016;; 22 : :4133.-44. 29. Thiery JP, Acloque H, Huang RY, Nieto MA. Epithelial-mesenchymal transitions in development and disease . Cell. 2009;; 139 : :871.-90. 30. Lapidot T, Sirard C, Vormoor J, et al. . A cell initiating human acute myeloid leukaemia after transplantation into SCID mice . Nature. 1994;; 367 : :645.-8. 31. Gupta PB, Onder TT, Jiang G, et al. . Identification of selective inhibitors of cancer stem cells by high-throughput screening . Cell. 2009;; 138 : :645.-59. 32. Mai TT, Hamaï A, Hienzsch A et al. . Salinomycin kills cancer stem cells by sequestering iron in lysosomes . Nat Chem Biol. 2017;; 9 : :1025.-33. 33. Alanko J, Mai A, Jacquemet G et al. . Integrin endosomal signalling suppresses anoikis . Nat Cell Biol. 2015;; 17 : :1412.-21. 34. Elkhatib N, Bresteau E, Baschieri F, et al. . Tubular clathrin/AP-2 lattices pinch collagen fibers to support 3D cell migration . Science. 2017;; 356 : :6343.. 35. Glentis A, Oertle P, Mariani P, et al. . Carcinoma-associated fibroblasts induce metalloprotease-independent cancer cell invasion of the basement membrane . Nat Commun. 2017;; 8 : :924.. 36. Correia AL, Bissell MJ. The tumor microenvironment is a dominant force in multidrug resistance . Drug Resist Update. 2012;; 15 : :39.-49. 37. Chen DS, Mellman I. Elements of cancer immunity and the cancer-immune set point . Nature. 2017;; 541 : :321.-30. 38. Fridman WH, Sautès-Fridman C. Le microenvironnement tumoral : matrice nourricière, champ de bataille et cible thérapeutique des cancers . Med Sci (Paris). 2014;; 30 : :359.-65. 39. Zitvogel L, Daillère R, Roberti MP, et al. . Anticancer effects of the microbiome and its products . Nat Rev Microbiol. 2017;; 8 : :465.-78. 40. Rubashkin MG, Cassereau L, Bainer R et al. . Force engages vinculin and promotes tumor progression by enhancing PI3K activation of phosphatidylinositol (3,4,5)-triphosphate . Cancer Res. 2014;; 74 : :4597.-611. 41. Fernandez-Sanchez ME, Barbier S, Whitehead J, et al. . Mechanical induction of the tumorigenic beta-catenin pathway by tumour growth pressure . Nature. 2015;; 523 : :92.-5. 42. Sousa CM, Biancur DE, Wang X, et al. . Pancreatic stellate cells support tumour metabolism through autophagic alanine secretion . Nature. 2016;; 536 : :479.-83. 43. Dozynkiewicz MA, Jamieson NB, Macpherson I, et al. . Rab25 and CLIC3 collaborate to promote integrin recycling from late endosomes/lysosomes and drive cancer progression . Dev Cell. 2012;; 22 : :131.-45. 44. Hernandez-Fernaud JR, Ruengeler E, Casazza A, et al. . Secreted CLIC3 drives cancer progression through its glutathione-dependent oxidoreductase activity . Nat Commun. 2017;; 8 : :14206.. 45. Chen BC, Legant WR, Wang K, et al. . Lattice light-sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution . Science. 2014;; 346 : :1257998.. 46. Kennedy MJ, Hughes RM, Peteya LA, et al. . Rapid blue-light-mediated induction of protein interactions in living cells . Nat Methods. 2010;; 7 : :973.-5. 47. Orlov I, Myasnikov AG, Andronov L, et al. . The integrative role of cryo electron microscopy in molecular and cellular structural biology . Biol Cell. 2017;; 109 : :81.-93. 48. Sung KE, Beebe DJ. Microfluidic 3D models of cancer. Adv Drug Deliv Rev. 2014;; 79-80 : :68.-78. 49. Fatehullah A, Tan SH, Barker N. Organoids as an in vitro model of human development and disease . Nat Cell Biol. 2016;; 18 : :246.-54. 50. van de Wetering M, Francies HE et al. . Prospective derivation of a living organoid biobank of colorectal cancer patients . Cell. 2015;; 16 : :933.-45. 51. Saini A. Cystic fibrosis patients benefit from mini guts. Cell Stem Cell. 2016;; 19: :425.-7. 52. Chanrion M, Kuperstein I, Barrière C, et al. . Concomitant Notch activation and p53 deletion trigger epithelial-to-mesenchymal transition and metastasis in mouse gut . Nat Commun. 2014;; 5 : :5005.. 53. Hamaï A, Mehrpour M. Homéostasie du fer et autophagie . Med Sci (Paris). 2017;; 33 : :260.-7. 54. Chéreau R, Angibaud J, Nägerl UV. L'imagerie super-résolution STED révèle un nouveau type de plasticité axonale. Med Sci (Paris). 2018;; 34 : :17.-20. 55. Girard PP, Forget BC. Microscopie de fluorescence à feuille de lumière : la face cachée de l'échantillon enfin dévoilée . Med Sci (Paris). 2011;; 27 : :753.-62. 56. Boutin JA, Li Z, Vuillard L, Vénien-Bryan C. La cryo-microscopie, une alternative à la cristallographie aux rayons X ? Med Sci (Paris). 2016;; 32 : :758.-67. 57. Saidak Z, Giaccobi AS, Chenda Ly M, et al. . La modélisation mathématique, un outil essentiel pour l'étude du ciblage thérapeutique des tumeurs solides . Med Sci (Paris). 2017;; 33 : :1055.-62. |



 ).
).