| |
| Med Sci (Paris). 34(2): 155–160. doi: 10.1051/medsci/20183402013.Le mélanome uvéal Une maladie modèle des anomalies de l’épissage et de l’oncogenèse Samar Alsafadi,1,2 Lenha Mobuchon,2 Manuel Rodrigues,2 and Marc-Henri Stern2* 1Département de recherche translationnelle, Institut Curie, université Paris Sciences et Lettres - PSL Research University, 26, rue d’Ulm, 75248Cedex Paris, France 2Inserm U830, Institut Curie, université Paris Sciences et Lettres - PSL Research University, 26, rue d’Ulm, 75248Cedex Paris, France |
Vignette (Photo © CIML/Inserm/Cnrs/Delfini, Marcello/Fallet, Mathieu). Le mélanome uvéal est un cancer rare de l’adulte, dont les événements oncogéniques très stéréotypés ont été décryptés ces 10 dernières années. Ses particularités épidémiolo-giques, génétiques et transcriptionnelles en font un modèle remarquable de l’oncogenèse. La transformation maligne implique de façon presque mutuellement exclusive de grands processus biologiques, comme la régulation chro-matinienne par inactivation de BAP1, l’épissage par mutations de SF3B1, et la traduction par mutations d’EIF1AX. L’étude du mélanome uvéal a permis de découvrir les mécanismes de l’épissage anormal lié aux mutations de SF3B1. La compréhension du lien entre ces anomalies et la transformation maligne sera la prochaine étape, dans l’espoir d’en déduire de nouvelles pistes thérapeutiques. Bien que rare, le mélanome de l’uvée (MU) est la tumeur maligne intraoculaire primitive la plus fréquente de l’adulte. Il représente 5 % de tous les mélanomes avec une incidence de 500 à 600 nouveaux cas par an en France. Il se développe à partir du tractus uvéal, incluant l’iris, les corps ciliaires et la choroïde, la localisation la plus fréquente étant au pôle postérieur de la choroïde. La tumeur primitive est le plus souvent bien contrôlée par les traitements actuels qui visent à préserver le mieux possible l’œil et la vision. Le traitement de la tumeur primaire est adapté à sa taille et à sa localisation, avec irradiation pour la plupart des tumeurs (curiethérapie par disque d’iode 125 ou protonthérapie en fonction de la taille et de la localisation) et énucléation pour les très grosses tumeurs. La gravité du MU est liée à sa progression métastatique dans 30 à 50 % des cas, avec un tropisme hépatique remarquable, puisqu’environ 90 % des lésions secondaires s’y développent sans autre atteinte viscérale et sans que cela ne soit expliqué par un lien anatomique. Le pronostic est alors très péjoratif avec une survie médiane de moins d’un an. Aucune chimiothérapie, thérapie ciblée, immunothérapie, en phase adjuvante ou au stade métastatique, n’a démontré d’efficacité dans la prolongation de la survie globale [1]. Contrastant avec le peu d’avancées thérapeutiques, la compréhension des mécanismes moléculaires de l’oncogenèse du MU a fait des progrès considérables ces dernières années. Le MU apparaît comme un cancer de l’adulte avec des singularités qui font l’objet de cette revue. |
Épidémiologie du mélanome uvéal Comme le mélanome cutané, le MU est associé à la peau et aux yeux clairs. Les données épidémiologiques américaines de la base de données SEER (surveillance, epidemiology, and end results) montrent un risque relatif environ 10 fois plus faible pour les populations d’origine afro-américaine ou asiatique par rapport à la population d’origine européenne [2, 3], suggérant que le rayonnement ultraviolet (UV) représente le facteur étiologique le plus probable. Cependant, l’incidence du MU est similaire dans les populations d’origine européenne quelque soit le continent de résidence. Contrairement au mélanome cutané, le MU n’a pas connu la progression de l’incidence observée en Europe ou en Australie, associée à l’augmentation de l’exposition aux UV [4]. L’analyse par séquençage complet du génome de 12 MU nous a permis d’éliminer le rôle mutagène des UV dans cette maladie en montrant l’un des plus faibles taux de mutations jamais rapportés pour une tumeur solide de l’adulte, et l’absence de signature mutationnelle associée aux UV, éléments qui ont été largement confirmés depuis [5-7]. L’hypothèse la plus probable pour expliquer le risque plus élevé de MU dans la population européenne, était la prévalence de variants génétiques de risque dans cette population. Nous avons donc réalisé une étude d’association pan-génomique (genome-wide association study ou GWAS), comparant des individus européens atteints de MU et sains. Nous avons identifié une région à risque autour des locus TERT (telomerase reverse transcriptase) et CLPTM1L (cleft lip and palate trans-membrane 1-like) [8]. Cependant, ces allèles à risque ont une prévalence similaire ou plus élevée dans les populations africaines et n’expliquent donc pas l’épidémiologie. Une autre étude d’association ciblée sur les locus associés au mélanome cutané a retrouvé certains gènes de la pigmentation [9], confirmés par notre GWAS. Ces données confirment donc les nombreuses observations cliniques sur une relation pigmentation/MU, sans que le mécanisme oncogénique sous-jacent ne soit encore compris. |
Les aberrations génomiques associées au mélanome uvéal Le profil génomique établi par cytogénétique ou puces à ADN est relativement simple, le plus souvent diploïde avec des gains et des pertes récurrents de chromosomes (monosomie 3) ou de bras chromosomiques (1p-, 6p+, 6q-, 8p-, 8q+). Ce profil est très utile en clinique car il permet de classer les tumeurs à haut risque (monosomie 3 associée à 8q+), risque intermédiaire (une seule de ces altérations) et faible risque (aucune de ces altérations) d’évolution métastatique [10]. |
Comme mentionné précédemment, le MU est la ou l’une des tumeurs de l’adulte avec le plus faible taux de mutations somatiques. Parmi ces rares mutations, les mutations prédominantes correspondent à des transitions C>T (cytosine> thymine) dans un contexte d’îlots CpG (cyto-sine-phosphate-guanine), attribuées à la désamination de méthylcytosines. Le taux de ces mutations n’est pas lié à l’âge au diagnostic, contrairement à la plupart des autres tumeurs [11]. Cette extrême stabilité du génome des MU reste mystérieuse. Différentes hypothèses peuvent être évoquées comme un très lent renouvellement des cellules souches [12], ou des mécanismes de réparation sur-actifs [13]. L’inefficacité relative de l’immunothérapie (inhibiteurs des points de contrôle [checkpoints] immunitaires) dans ce cancer est probablement expliquée par le très faible taux de mutations [14]. |
Les événements oncogéniques Au niveau moléculaire, le MU est associé à des altérations oncogéniques très stéréotypées que l’on classe en 2 catégories : (1) les mutations activatrices de la voie des récepteurs membranaires couplés aux protéines Gαq, et (2) les mutations des gènes dits « BES » (BAP1, EIF1AX et SF3B1) (Figure 1).  | Figure 1. Mécanismes de transformation maligne du mélanome uvéal. A. Altérations de la voie de Gαq dans le mélanome uvéal. Les étoiles indiquent les mutations activatrices. Plus de 85 % des MU sont porteurs de mutations activatrices de GNAQ ou GNA11, codant des unités Ga des protéines G hétéro-trimériques. Des mutations activatrices de CYSTRL2, codant un récepteur des leucotriènes couplé aux protéines G (GPCR) ou de PLCB4, codant l’isoforme β4 de la phospholi-pase C (PLCβ), sont retrouvées plus rarement. Ces mutations mutuellement exclusives aboutissent à l’activation des mêmes voies de transduction du signal, comme l’activation de la voie YAP (Yes-associated protein). B. Événements génomiques et génétiques dans le mélanome uvéal. L’événement initiateur est une mutation aboutissant à l’activation de la voie des Gαq. Le deuxième événement oncogénique inclut des mutations presque mutuellement exclusives de BAP1 (BRCA1 [breast cancer 1]-associated protein 1), EIF1AX (eukaryotic translation initiation factor 1A, X-linked) et SF3B1 (splicing factor 3B subunit 1). Les mutations de BAP1 sont associées à la monosomie 3 et définissent les tumeurs à haut risque métastatique. Les mutations SF3B1 et EIF1AX sont associées à un faible risque métastatique. AP1 : activator protein 1; BES : BAP1, EIF1AX, SF3B1; CYSLTR2 : cysteinyl leukotriene receptor 2; ERK : extracellular signal-regulated kinase; GNAQ : G protein subunit alpha Q; GNA11 : G protein subunit alpha 11; GPCR : récepteurs couplés aux protéines G; PKC : protéine kinase C ; PLCβ; phospholipase C beta; PLCB4; phospholipase C beta 4; TEAD; TEA domain transcription factor 1; YAP; Yes-associated protein. |
La voie des Gαq Les Gαq sont une sous-famille (incluant Gαq, Gα11, Gα14 et Gα15, codés respectivement par GNAQ, GNA11, GNA14 et GNA15) des unités alpha des protéines G hétéro-trimériques 1,. Les protéines G permettent la transduction du signal à partir des récepteurs mem-branaires couplés aux protéines G (G protein-coupled receptor ou GPCR). Même si le lien de temporalité n’est pas rigoureusement établi, on considère que l’événement initiateur du MU est une mutation aboutissant à l’activation de la voie de Gαq (Figure 1A). En effet, les mutations touchant les gènes GNAQ et GNA11 sont retrouvées dans des tumeurs mélanocytaires bénignes telles que les naevi bleus et naevi d’Ota 2, ces derniers étant un facteur de risque de MU. Plus de 85 % des MU sont porteurs de mutations activatrices hétérozygotes sur les codons Q209 ou R183 des gènes GNAQ et GNA11 [15, 16]. La mutation L129Q de CYSTRL2, qui code un récepteur des leucotriènes couplé aux protéines G, ou la mutation D630Y de PLCB4, qui code l’isoforme β4 de la phospholipase C, sont retrouvées beaucoup plus rarement [17, 18]. Ces mutations mutuellement exclusives aboutissent à l’activation des mêmes voies de transduction du signal, comme l’activation de la voie YAP (Yes-associated protein), jouant probablement un rôle majeur dans la transformation maligne. Plusieurs essais thérapeutiques en cours évaluent l’efficacité des inhibiteurs de ces voies, notamment avec des inhibiteurs des voies MAPK (mitogen-activated protein kinases) ou PKC (proté ine kinase C). L’événement BES (BAP1, EIF1AX et SF3B1) Le deuxième évènement oncogénique inclut des mutations presque mutuellement exclusives de 3 gènes impliqués dans des processus très divers : BAP1, EIF1AX et SF3B1 (BES), codant des protéines impliquées respectivement dans la régulation transcriptionnelle, la traduction protéique, et l’épissage (Figure 1B) [5, 19-21]. Les mutations de BAP1 (BRCA1 [breast cancer 1]-associated protein 1) ont été les premières identifiées [19]. Ces mutations sont le plus souvent somatiques, associées à la monosomie du chromosome 3 où est localisé le gène BAP1, et définissent les tumeurs à haut risque métastatique. De rares mutations germinales de BAP1 sont responsables d’un syndrome de prédisposition à certaines tumeurs incluant le MU (et les rares formes familiales de MU), les mésothéliomes, ainsi que certains mélanomes cutanés et carcinomes du rein à cellules claires [22-24]. BAP1 code une désubiquitinase qui se lie, entre autres, aux isoformes ASXL1 (additional sex combs like-1), L2 et L3 avec qui elle forme le complexe qui désubiquitine en particulier l’histone H2A, et probablement d’autres substrats. La mutation de Calypso, son orthologue chez la drosophile, entraîne un phénotype de type Polycomb, c’est-à-dire une dérégulation antéro-postérieure des gènes du développement [25]. Toutefois, il n’est pas certain que cette propriété soit conservée chez les mammifères chez lesquels la fonction de BAP1 sur la régulation de la chromatine est certaine, mais reste encore mal comprise. Son étude est compliquée par les profondes modifications du cycle, du métabolisme et des caractéristiques cellulaires résultant des modulations expérimentales du niveau d’expression de BAP1 [26, 27]. Les mutations EIF1AX (eukaryotic translation initiation factor 1A, X-linked) ont été associées aux MU de faible risque métastatique [20]. EIF1AX code un facteur d’initiation de la traduction dont le rôle précis est encore inconnu. Les mutations, toujours localisées dans la partie 5’ du gène, aboutiraient à la production d’une protéine dont la troncation altérerait sa fonction, et donc les capacités de traduction de la cellule tumorale [28], sans qu’il soit encore clair s’il s’agit d’une diminution ou d’un changement de fonction des protéines résultantes de ces mutations qui affectent le gène EIF1AX. Des mutations récurrentes sur le gène SF3B1 (splicing factor 3B subunit 1) ont aussi été décrites par plusieurs équipes dont la nôtre en 2013, dans environ 20 % des MU [5, 20, 21]. Les mutations de ce gène avaient été rapportées auparavant dans des syndromes myélodysplasiques et des leucémies lymphoïdes chroniques B. Elles touchent les codons R625, K666 et K700 (mutations dites hotspots)3. Alors que ces mutations semblent conduire aux mêmes conséquences sur l’épissage, le codon R625 est préférentiellement muté dans les MU, et le codon K700, dans les hémopathies malignes. Avec SF3B1, plusieurs autres gènes codant des composants essentiels de la machinerie d’épissage (SRSF2 [serine/arginine-rich splicing factor 2], U2AF1 [U2 small nuclear RNA auxiliary factor 1] et ZRSR2 [U2 small nuclear ribonucleoprotein auxiliary factor 35 kDa subunit-related protein 2], pour les principaux), sont mutés de façon récurrente, le plus souvent sur les sites hotspots, dans d’autres pathologies malignes et hémopathies malignes. Ces gènes forment donc une nouvelle classe d’oncogènes, et si les conséquences de leurs mutations sur l’épissage ont été clarifiées ces deux dernières années, leurs mécanismes oncogéniques sont largement incompris à l’heure actuelle. |
Les anomalies de l’épissage liées aux mutations SF3B1L’épissage de l’ARN est un processus fondamental et complexe qui a fait l’objet d’une récente revue dans médecine/sciences [29] ➔. SF3B1 est un composant essentiel de snRNP U2 (U2 small nuclear ribo-nucleoprotein), un complexe en charge de la reconnaissance du point de branchement (PB). SF3B1 présente une région hydrophile N-terminale contenant un motif de liaison à U2AF, et une région C-terminale formée de 22 répétitions non-identiques HEAT (Huntingtin, elongation factor 3, protein phosphatase 2A, targets of rapamycin 1). Les mutations faux sens hotspots (R625, K666 et K700) de SF3B1 ciblent le 4e, 5e et 6e domaine HEAT. Ces altérations affectent ces résidus spatialement très proches les uns des autres, ce qui pourrait modifier la conformation tripartite du complexe SF3B1/p14/snRNA U2 [30]. De façon remarquable, les travaux parallèles de 3 équipes dont la nôtre, cherchant à comprendre les conséquences de ces mutations du gène SF3B1, ont abouti à des conclusions identiques en utilisant des approches différentes et des modèles tumoraux distincts [31-33]. (➔)Voir la Synthèse de G. Dujardin et al., m/s n° 12, décembre 2016, page 1103 Le transcriptome réalisé par RNA-Seq (séquençage de l’ARN) à grande profondeur, de 74 MU primaires, dont 16 porteurs d’une mutation du gène SF3B1 nous à permis de répertorier toutes les jonctions d’épissage dans ces échantillons, puis d’identifier un nombre restreint de jonctions différentiellement exprimées entre les cas mutés et sauvages pour SF3B1 (1 469 sur les 142 458 jonctions détectées). Ces jonctions différentielles étaient principalement dues à l’utilisation d’un site accepteur anormal situé en amont du site canonique. Leur analyse a permis de retrouver deux particularités remarquables : (1) la présence de l’accepteur alternatif AG dans un contexte SF3B1mut à environ 20 bases en amont de l’accepteur canonique, dans une région normalement dépourvue d’AG; (2) la présence d’une adénosine à environ 12 bases en amont de l’accepteur alternatif, qui s’est avérée être un point de branchement (PB) alternatif. Par la modélisation de ces sites d’épissage dans des vecteurs plasmidiques, et la mutagenèse systématique des résidus clés, nous avons montré que la compétition des PB en fonction de leur affinité pour snRNP U2 explique le choix entre ces accepteurs alternatifs. Les mutations SF3B1 sont donc des mutations « changement de fonction », le complexe snRNP U2* contenant SF3B1 muté ne pouvant reconnaître que les PB de forte affinité, même éloignés du site de fixation U2AF, normalement déterminant pour la fixation de snRNP U2 (Figure 2) [31]. L’utilisation de l’accepteur alternatif aboutit donc à l’insertion d’une vingtaine de bases introniques dans l’ARN messager final. Les conséquences sur le produit protéique sont variables en fonction du nombre de bases insérées : insertion d’environ 6 acides aminés, si le cadre de lecture est en phase, ou séquence peptidique totalement changée en aval de la jonction, en cas de changement de phase. Le changement de phase aboutit souvent à un codon stop prématuré, qui conduit à la dégradation des transcrits par le processus de NMD (nonsense-mediated mRNA decay) [33]. In fine, les épissages anormaux liés aux mutations de SF3B1 aboutissent à l’altération de l’expression et de la séquence de milliers de protéines, sans que la (ou les) cible(s) oncogénique(s) de ce processus soient encore connues. 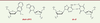 | Figure 2. Représentation schématique du mécanisme des mutants SF3B1 reconnaissant des points de branchement (PB) alternatifs (PB’) et résultant en ARNm aberrants. mut : mutated; p14 : splicing factor 3B, 14 kDa subunit; SF3B1 : splicing factor 3B subunit 1; U2AF : U2 small nuclear RNA auxiliary factor 2; U2 snARN : U2 small nuclear RNA; U2AF 35 kDa (35) and 65 kDa (65) subunits. |
|
Vers un mécanisme unique de l’oncogenèse du MU Deux observations paradoxales peuvent donc être réalisées :
- Les différentes mutations des gènes de protéines d’épissage surviennent de façon mutuellement exclusive : SF3B1, SRSF2, U2AF1 et ZRSR2 dans les myélodysplasies [34]; SF3B1 et SRSF2 dans le MU, comme récemment rapporté [7]. Cette exclusivité suggère une relation épistatique entre ces évènements dans une dysfonction oncogénique commune. Or, les conséquences maintenant connues de ces différentes mutations sont très différentes : accepteurs alternatifs pour SF3B1mut, saut d’exon ou rétention d’intron pour U2AF1mut [35, 36], rétention d’intron du processus mineur d’épissage dit U12 pour ZRSR2mut [37]. Pour SRSF2mut, les altérations de l’épissage - inclusion ou exclusion d’exons cassettes - sont différentes en fonction du type de mutations [38, 39]. De plus, les mêmes sites d’épissage ne sont pas altérés par les différentes mutations.
- Les conséquences des différentes altérations oncogéniques BES (BAP1, SF3B1 et EIF1AX) dans le MU touchent des processus qui ne sont a priori pas liés : régulation chromatinienne pour BAP1, épissage pour SF3B1 et traduction pour EIF1AX. Or, ces altérations sont à l’origine du même type tumoral pour le clinicien et le pathologiste, mais de pronostics différents.
Ces observations nous conduisent à proposer comme hypothèse de travail que les différentes mutations des gènes de protéines d’épissage aboutissent à un mécanisme commun d’oncogenèse, et que les anomalies de BAP1, SF3B1 et EIF1AX convergent vers ce mécanisme commun (Figure 3). Notre objectif à terme est évidemment de décrypter ces mécanismes d’oncogenèse, afin de développer de nouvelles approches thérapeutiques pour cette tumeur de mauvais pronostic.  | Figure 3. Représentation schématique de l’hypothèse d’un mécanisme unique de l’oncogenèse du MU. BAP1 : BRCA1 [breast cancer 1]-associated protein 1; EIF1AX : eukaryotic translation initiation factor 1A, X-linked; mut : muté; SF3B1 : splicing factor 3B subunit 1; SRSF2 : serine/arginine-rich splicing factor 2; U2AF1 : U2 small nuclear RNA auxiliary factor 1; ZRSR2; zinc finger CCCH- type, RNA binding motif and serine/arginine-rich 2. |
|
Les auteurs remercient l’Inserm, l’Institut Curie, la Cancéropôle Île-de-France, l’INCa et la Commission européenne (UMCURE) pour leur soutien à la recherche. S.A. est financée par le programme SIRIC, L.M. par le programme H2020 UMCURE, et M.R. par la bourse doctorale INCa/ITMO/ AVIESAN « Formation à la recherche translationnelle ». |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1. Carvajal RD, Schwartz GK, Tezel T, et al. Metastatic disease from uveal melanoma: treatment options and future prospects . Br J Ophthalmol. 2017;; 101 : :38.-44. 2. Singh AD, Turell ME, Topham AK. Uveal melanoma: trends in incidence, treatment, and survival. Ophthalmology. 2011;; 118 : :1881.-5. 3. Park SJ, Oh CM, Kim BW, et al. Nationwide incidence of ocular melanoma in South Korea by using the National Cancer Registry Database. (1999-2011).. Invest Ophthalmol Vis Sci. 2015;; 56 : :4719.-24. 4. Andreoli MT, Mieler WF, Leiderman YI. Epidemiological trends in uveal melanoma. Br J Ophthalmol. 2015;; 99 : :1550.-3. 5. Furney SJ, Pedersen M, Gentien D, et al. SF3B1 mutations are associated with alternative splicing in uveal melanoma. Cancer Discov. 2013;; 3 : :1122.-9. 6. Royer-Bertrand B, Torsello M, Rimoldi D, et al. Comprehensive genetic landscape of uveal melanoma by whole-genome sequencing . Am J Hum Genet. 2016;; 99 : :1190.-8. 7. Robertson AG, Shih J, Yau C, et al. Integrative analysis identifies four molecular and clinical subsets in uveal melanoma. Cancer Cell. 2017;; 32 : :204.-20 e15.. 8. Mobuchon L, Battistella A, Bardel C, et al. A GWAS in uveal melanoma identifies risk polymorphisms in the CLPTM1L locus. NPJ Genom Med. 2017; :2.. 9. Ferguson R, Vogelsang M, Ucisik-Akkaya E, et al. Genetic markers of pigmentation are novel risk loci for uveal melanoma. Sci Rep. 2016;; 6 : :31191.. 10. Cassoux N, Rodrigues MJ, Plancher C, et al. Genome-wide profiling is a clinically relevant and affordable prognostic test in posterior uveal melanoma. Br J Ophthalmol. 2014;; 98 : :769.-74. 11. Alexandrov LB, Jones PH, Wedge DC, et al. Clock-like mutational processes in human somatic cells. Nat Genet. 2015;; 47 : :1402.-7. 12. Tomasetti C, Vogelstein B. Cancer etiology. Variation in cancer risk among tissues can be explained by the number of stem cell divisions. Science. 2015;; 347 : :78.-81. 13. Helleday T, Eshtad S, Nik-Zainal S. Mechanisms underlying mutational signatures in human cancers. Nat Rev Genet. 2014;; 15 : :585.-98. 14. Kottschade LA, McWilliams RR, Markovic SN, et al. The use of pembrolizumab for the treatment of metastatic uveal melanoma. Melanoma Res. 2016;; 26 : :300.-3. 15. Van Raamsdonk CD, Bezrookove V, Green G, et al. Frequent somatic mutations of GNAQ in uveal melanoma and blue naevi. Nature. 2009;; 457 : :599.-602. 16. Van Raamsdonk CD, Griewank KG, Crosby MB, et al. Mutations in GNA11 in uveal melanoma. N Engl J Med. 2010;; 363 : :2191.-9. 17. Moore AR, Ceraudo E, Sher JJ, et al. Recurrent activating mutations of G-protein-coupled receptor CYSLTR2 in uveal melanoma. Nat Genet. 2016;; 48 : :675.-80. 18. Johansson P, Aoude LG, Wadt K, et al. Deep sequencing of uveal melanoma identifies a recurrent mutation in PLCB4. Oncotarget. 2016;; 7 : :4624.-31. 19. Harbour JW, Onken MD, Roberson ED, et al. Frequent mutation of BAP1 in metastasizing uveal melanomas. Science. 2010;; 330 : :1410.-3. 20. Martin M, Masshofer L, Temming P, et al. Exome sequencing identifies recurrent somatic mutations in EIF1AX and SF3B1 in uveal melanoma with disomy 3. Nat Genet. 2013;; 45 : :933.-6. 21. Harbour JW, Roberson ED, Anbunathan H, et al. Recurrent mutations at codon 625 of the splicing factor SF3B1 in uveal melanoma. Nat Genet. 2013;; 45 : :133.-5. 22. Wiesner T, Obenauf AC, Murali R, et al. Germline mutations in BAP1 predispose to melanocytic tumors. Nat Genet. 2011;; 43 : :1018.-21. 23. Testa JR, Cheung M, Pei J, et al. Germline BAP1 mutations predispose to malignant mesothelioma. Nat Genet. 2011;; 43 : :1022.-5. 24. Popova T, Hebert L, Jacquemin V, et al. Germline BAP1 mutations predispose to renal cell carcinomas. Am J Hum Genet. 2013;; 92 : :974.-80. 25. Scheuermann JC, de Ayala Alonso AG, Oktaba K, et al. Histone H2A deubiquitinase activity of the Polycomb repressive complex PR-DUB. Nature. 2010;; 465 : :243.-7. 26. Hebert L, Bellanger D, Guillas C, et al. Modulating BAP1 expression affects ROS homeostasis, cell motility and mitochondrial function. Oncotarget. 2017;; 8 : :72513.-27. 27. Bononi A, Yang H, Giorgi C, et al. Germline BAP1 mutations induce a Warburg effect. Cell Death Differ. 2017;; 24 : :1694.-704. 28. Johnson CP, Kim IK, Esmaeli B, et al. Systematic genomic and translational efficiency studies of uveal melanoma. PLoS One. 2017;; 12 : e0178189.. 29. Dujardin G, Daguenet E, Bernard DG, et al. L’épissage des ARN pré-messagers : quand le splicéosome perd pied. Med Sci (Paris). 2016;; 32 : :1103.-10. 30. Cretu C, Schmitzova J, Ponce-Salvatierra A, et al. Molecular architecture of SF3b and structural consequences of its cancer-related mutations. Mol Cell. 2016;; 64 : :307.-19. 31. Alsafadi S, Houy A, Battistella A, et al. Cancer-associated SF3B1 mutations affect alternative splicing by promoting alternative branchpoint usage. Nat Commun. 2016;; 7 : :10615.. 32. DeBoever C, Ghia EM, Shepard PJ, et al. Transcriptome sequencing reveals potential mechanism of cryptic 3’ splice site selection in SF3B1-mutated cancers. PLoS Comput Biol. 2015;; 11 : e1004105.. 33. Darman RB, Seiler M, Agrawal AA, et al. Cancer-associated SF3B1 hotspot mutations induce cryptic 3’ splice site selection through use of a different branch point. Cell Rep. 2015;; 13 : :1033.-45. 34. Joshi P, Halene S, Abdel-Wahab O. How do messenger RNA splicing alterations drive myelodysplasia? Blood. 2017;; 129 : :2465.-70. 35. Shirai CL, Ley JN, White BS, et al. Mutant U2AF1 expression alters hematopoiesis and pre-mRNA splicing in vivo. Cancer Cell. 2015;; 27 : :631.-43. 36. Ilagan JO, Ramakrishnan A, Hayes B, et al. U2AF1 mutations alter splice site recognition in hematological malignancies. Genome Res. 2015;; 25 : :14.-26. 37. Madan V, Kanojia D, Li J, et al. Aberrant splicing of U12-type introns is the hallmark of ZRSR2 mutant myelodysplastic syndrome. Nat Commun. 2015;; 6 : :6042.. 38. Zhang J, Lieu YK, Ali AM, et al. Disease-associated mutation in SRSF2 misregulates splicing by altering RNA-binding affinities. Proc Natl Acad Sci USA. 2015;; 112 : :E4726.-34. 39. Kim E, Ilagan JO, Liang Y, et al. SRSF2 mutations contribute to myelodysplasia by mutant-specific effects on exon recognition. Cancer Cell. 2015;; 27 : :617.-30. |