Pour survivre, toute cellule eucaryote doit s’adapter à des changements d’environnement (nutriments, température, etc.), répondre à des stimulations extérieures (hormones, neurotransmetteurs, etc.), et effectuer des processus dynamiques complexes (cycle cellulaire, réparation de l’ADN, etc.). Elle doit notamment moduler la production de protéines en fonction de ses besoins. Elle peut contrôler dans le noyau la production des ARN messagers (ARNm) codant ces protéines - régulations dites transcriptionnelles - ou contrôler dans le cytoplasme la traduction des ARNm en protéines, en dégradant ces ARNm ou en bloquant leur traduction - régulations dites post-transcriptionnelles.
De façon remarquable, un grand nombre des facteurs impliqués dans la dégradation des ARNm, le contrôle de la traduction et l’interférence par les microARN, s’accumulent dans de minuscules granules cytoplasmiques appelés P-bodies (pour processing-bodies).
Les P-bodies sont observés dans tous les organismes eucaryotes, animaux et végétaux [ 1]. Ils appartiennent à un ensemble de granules, dont les caractéristiques morphologiques et compositionnelles dépendent du type cellulaire (granules germinaux, granules neuronaux) ou des conditions d’apparition (granules de stress) [ 2].➔
➔ Voir la nouvelle de J. Nikolic et D. Blondel, m/s n° 11, novembre 2017, page 921
Contrairement aux autres organelles du cytoplasme (mitochondries, Golgi, etc.), les P-bodies sont dépourvus de membrane [ 3]. Ils se comportent comme des gouttelettes épaisses concentrant des ARN et des protéines, au sein d’un cytoplasme lui-même liquide [ 4].
La fonction des P-bodies est énigmatique. Il a d’abord été proposé qu’ils soient des sites de dégradation des ARN. En effet, ils contiennent presque tous les facteurs de dégradation de l’extrémité 5’ des ARN, et leur nombre et taille augmentent quand la dégradation des ARN est bloquée, comme s’ils accumulaient des molécules en attente de dégradation [1]. Cependant, d’autres études ont proposé qu’ils stockent des ARN non traduits en vue d’une utilisation ultérieure. De fait, les facteurs essentiels pour la formation des P-bodies sont des répresseurs de la traduction [ 5]. De plus, quelques études ont montré que certains ARNm s’accumulent dans les P-bodies lorsque leur traduction est bloquée par un microARN, et qu’ils sont libérés dans le cytosol pour être traduits lorsque ce microARN est inactivé [ 6]. Un long débat s’est alors installé, associant aux P-bodies deux fonctions paradoxales, dégradation des ARN et stockage d’ARN non traduits, sans savoir comment se faisait le choix [1].
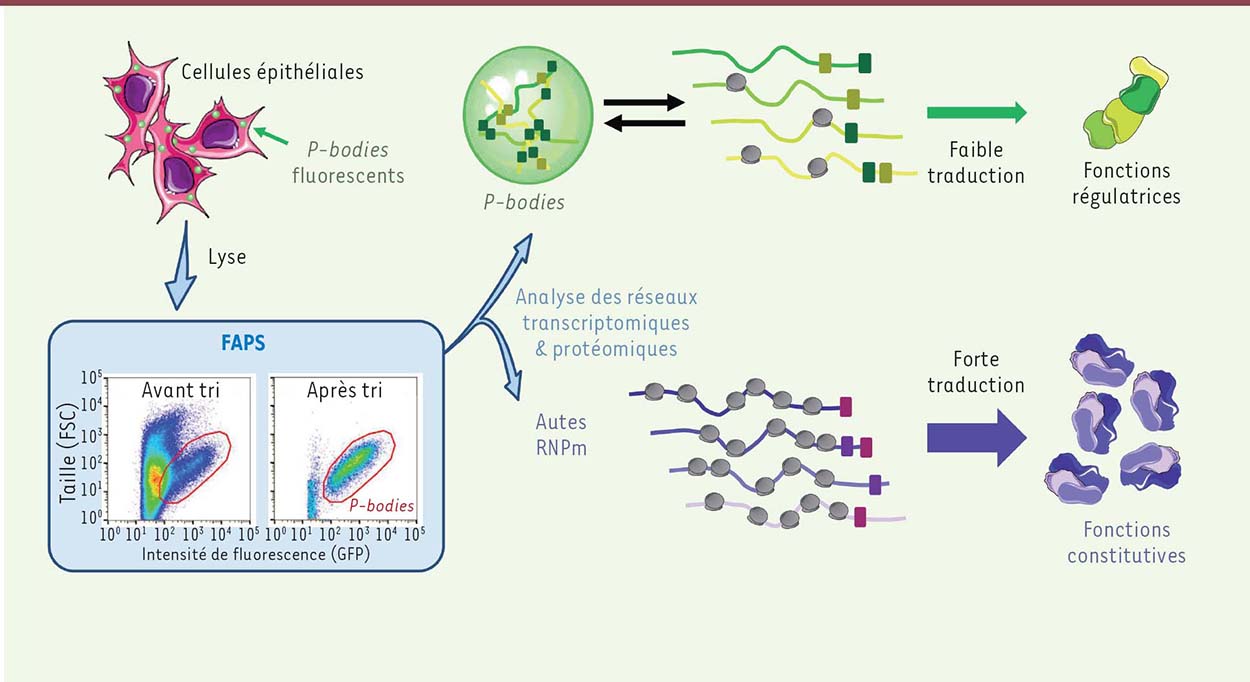
Le contenu en ARN des P-bodies est une boîte noire : s’agit-il d’ARNm ou d’ARN non-codants ? De n’importe quel transcrit cellulaire ou de quelques ARN particuliers ? Seule certitude, ils ne contiennent pas d’ARN ribosomique. Pour répondre à ces questions, le moyen le plus direct était de purifier les P-bodies. Les méthodes classiques de centrifugation différentielle n’ayant pas réussi à séparer les P-bodies des autres organelles cytoplasmiques, nous avons opté pour une stratégie innovante, le FAPS (fluorescence-activated particle sorting) [ 7]. Nous avons généré des cellules humaines exprimant un marqueur fluorescent des P-bodies, le GFP-LSM14A1 (fusion green fluorescent protein-LSM14A), puis trié les P-bodies rendus fluorescents à partir d’un lysat cytoplasmique à l’aide d’un FACS (fluorescence-activated cell sorter), appareil habituellement utilisé pour trier des cellules fluorescentes (Figure 1). Étant donnés la taille des P-bodies (500 nm de diamètre), à la limite de détection de l’appareil, et leur faible nombre (environ 5 par cellule), le défi était de taille ! Une semaine de tri fut nécessaire pour obtenir suffisamment de matériel pour identifier les protéines par spectrométrie de masse et les ARN par séquençage.
| Figure 1.
Trier des P-bodies humains pour comprendre leur fonction. Des cellules épithéliales humaines ont été utilisées pour purifier des P-bodies par la méthode de FAPS (fluorescence-activated particle sorting). L’analyse de leur contenu en ARN et protéines a permis de dessiner le tableau des complexes ARNm-protéines (RNPm) ciblés dans les P-bodies, ou qui en sont exclus. Les ARNm (messagers) des P-bodies sont peu traduits et codent essentiellement des protéines régulatrices, alors que les ARNm exclus des P-bodies sont fortement traduits et codent des protéines dites de ménage. |
Plus de cent protéines ont été détectées dans les P-bodies, la plupart liant des ARN, comme on pouvait l’anticiper et, parmi elles, les composants des P-bodies déjà connus. Le contenu des P-bodies en ARN est très varié, avec plusieurs milliers de transcrits. Ce sont essentiellement des ARNm, alors que la plupart des ARN non-codants (ARN nucléolaires, longs ARN non-codants, ARN antisens, etc.) en sont exclus. Lorsque les P-bodies sont supprimés expérimentalement des cellules, les ARN qu’ils contiennent normalement semblent intacts et leur abondance reste inchangée. Ainsi, rien n’étaye l’hypothèse qu’ils étaient en cours de dégradation. Au contraire, ces ARNm ont en commun d’être abondants mais traduits de façon inefficace, et leur traduction est plus active en l’absence de P-bodies. Les P-bodies sont donc des lieux de stockage d’ARNm non traduits. Ils s’apparentent ainsi fonctionnellement aux granules germinaux dans les ovocytes en attente de fécondation, ou aux granules neuronaux présents sous les synapses [6].
Au-delà de ces révélations sur la fonction des P-bodies dans le métabolisme des ARN, nos données apportent aussi des informations sur leur formation. En les combinant avec les données de la littérature portant sur les interactions entre protéines et ARN [ 8], par des approches relevant de la biologie des systèmes, nous avons vu émerger, à l’échelle de la cellule entière, l’image d’un réseau complexe de protéines et d’ARN, qui détermine quels ARN se condensent dans les P-bodies et quels ARN en sont exclus. Ainsi, ce sont les combinaisons de protéines liant l’ARN qui contrôlent leur condensation dans les P-bodies (Figure 1).
Plus inattendu, les milliers d’ARN présents dans les P-bodies codent des protéines impliquées dans divers aspects de la physiologie cellulaire, mais avec pour point commun d’être essentiellement des protéines régulatrices, tandis que les ARNm exclus codent des protéines dites « de ménage » (Figure 1). Trois exemples pour illustrer ces résultats : (1) le protéasome est un gros complexe cytoplasmique qui dégrade les protéines lorsqu’elles ont été couplées à un peptide ubiquitine par des enzymes appelées ubiquitine ligases. Les ARNm codant les sous-unités du protéasome, effecteur de la protéolyse, sont massivement exclus des P-bodies, alors que la plupart des ARNm codant les ubiquitines ligases, régulant la protéolyse, y sont stockés; (2) les ARNm codant les protéines du ribosome, effecteur de la traduction, sont massivement exclus des P-bodies, quand la plupart des ARNm codant des régulateurs de la traduction s’y accumulent. Parmi ces régulateurs se trouvent notamment les protéines des P-bodies eux-mêmes; (3) les ARNm des histones, qui forment les nucléosomes sur la chromatine, sont exclus, alors que ceux codant des modificateurs d’histone, régulateurs de la transcription, sont stockés dans les P-bodies. On peut ainsi évoquer la notion de régulons post-transcriptionnels, c’est-à-dire d’ensembles de transcrits de fonction similaire, qui sont soit exclus, soit stockés dans les P-bodies.
Une analyse plus détaillée montre que les ARNm des protéines présentes dans un même complexe multi-protéique tendent à être collectivement exclus, ou collectivement stockés dans les P-bodies. Nous avons cité précédemment les ARNm codant les sous-unités du protéasome, des ribosomes et des nucléosomes, qui sont exclus des P-bodies, mais d’autres complexes sont également concernés : la RNAse H (ribonucléase H), l’ATP synthase, etc. Au contraire, les ARNm codant les sous-unités de la phosphoinositide 3-kinase, du facteur de transcription TFIIH (transcription factor IIH), du centrosome et des complexes condensine/cohésine impliqués dans le cycle cellulaire sont stockés dans les P-bodies. Cette observation suggère que les P-bodies contribuent à adapter la production des protéines individuelles à leur stœchiométrie au sein de complexes multi-protéines.
Ce paysage global de régulons est né de l’analyse des P-bodies dans un contexte précis : des cellules épithéliales humaines, cultivées dans des conditions contrôlées in vitro. On peut anticiper que le contenu des P-bodies diffèrera selon l’environnement et le type cellulaires, sinon ces organelles ne pourraient servir de levier pour une régulation. Notre étude, au-delà de fournir des outils méthodologiques pour explorer le potentiel de stockage des P-bodies dans d’autres modèles, révèle plus largement l’existence de régulons post-transcriptionnels, dont les recherches à venir détermineront l’importance pour la physiologie cellulaire.