Vignette (Photo © Inserm - Patrick Delapierre). Depuis l’avènement des séquenceurs de seconde génération, le volume d’expertises génomiques s’est accru, avec des technologies qui ont largement investi les centres hospitaliers. Cette méthodologie désormais employée tant à des fins d’expertises diagnostiques qu’en recherche en santé humaine peut être qualifiée de « couteau suisse » génomique permettant d’accéder à des informations susceptibles d’orienter les thérapies qui seront appliquées au patient. Ainsi, l’une des premières applications agréées par la FDA (food and drug administration) en 2013 a concerné la caractérisation des 139 variants du gène CFTR (cystic fibrosis transmembrane conductance regulator) associés à la fibrose kystique (ou mucoviscidose). Parallèlement, les technologies n’ont eu de cesse d’évoluer, affichant des débits de séquençage à la hausse, pendant que les coûts inhérents à la génération de ces séquences diminuaient. Pour répondre aux exigences de la médecine de précision, nécessitant de considérer les singularités du patient, ces informations génomiques peuvent désormais être intégrées à un arbre de décision thérapeutique. Cette seconde génération de séquençage est d’ores et déjà implantée dans les laboratoires hospitaliers, et de nouvelles applications cliniques, fondées sur ces nouvelles technologies qui remplacent les anciennes pour réduire les temps et les coûts, se développent. Alors que cette seconde génération est encore en plein essor, une troisième génération de séquençage par nanopores émerge [
21] ➔.
➔
Voir la Synthèse de F. Montel, m/s n° 2, février 2018, page 161
Plusieurs acteurs de biotech se sont lancés dans le développement de technologies fondées sur ces nanopores. Genia, dont Roche a fait l’acquisition, et la Quantapore, fondée sur l’interaction quantique-optique entre nanopore et ADN le traversant, sont encore en phase de développement. Oxford Nanopore Technologies (ONT) occupe le devant de la scène, avec la commercialisation du MinION, un séquenceur portatif au format d’une clé USB. Tous ont l’ambition de lancer sur le marché une technologie portative permettant un séquençage à bas coûts et d’étendre son accessibilité. Certaines caractéristiques propres à cette nouvelle génération de séquenceurs, comme la grande taille des lectures (ou read en anglais) produites, la distingue de celle qui l’a précédée, la 2e génération (Tableau I). Comment cette innovation pourrait trouver prochainement sa place aux côtés des autres technologies utilisées en clinique ? Un récent article de Bertrand Jordan [
1] ➔ revenait sur l’historique et les promesses accompagnant la venue sur le marché de la technologie d’ONT. La possibilité pour certaines applications cliniques d’obtenir des résultats extrêmement rapidement et l’emploi du séquenceur sur le terrain, comme cela a été le cas pour le virus Ebola en Afrique, ont largement contribué à la promotion de cette technologie. Tableau I.
| Technologie |
2e génération |
3e génération |
|
Matrice de départ
|
ADN |
ADN ou ARN |
|
Détection des bases méthylées
|
Non |
Oui |
|
Modalité d’applications
|
Un laboratoire orienté biologie moléculaire |
Décentralisation complète envisageable |
|
Maturité technologique
|
Méthode en voie d’appropriation |
Méthode émergente |
|
Coût investissement
|
Coût d’investissement relativement élevé |
Coûts d’investissement marginal |
|
Format des données
|
Données peu entachées d’erreurs (taux d’erreurs < 1 %) |
Données erronées (taux brut d’erreurs légèrement supérieur à 15 %) |
|
Taille des lectures
|
Lectures courtes 400 bases pour Ion Torrent à 2 x 300 bases pour Illumina |
Lectures pouvant dépasser 100 kilobases |
|
Exploitation des données
|
Variable selon les technologies (4h - 24h) |
Temps réel |
|
Principales limites
|
- Coût d’investissement- Débit d’échantillons minimal pour optimiser un run
|
- Stabilisation des protocoles- Coût à l’échantillon, selon les applications, encore relativement élevé |
Comparaison des principales caractéristiques associées aux deux générations actuelles de séquençage haut-débit. |
➔
Voir la Chronique génomique de B. Jordan, m/s n° 8-9, août-septembre 2017, page 801
En abordant des applications diagnostiques en infectiologie, pour le diagnostic prénatal non invasif, la mise en évidence de modifications de génome initiatrices de cancers ou pour son utilisation en recherche, cette revue propose de comprendre dans quelle mesure cette technologie de séquençage de 3e génération portative pourrait répondre à un besoin de précision thérapeutique. |
Une application en infectiologie Pour le traitement des maladies infectieuses sévères, l’identification rapide des agents pathogènes est primordiale. Dans les cas de bactériémies, il est ainsi hautement recommandé de commencer un traitement antibiotique dans l’heure qui suit le diagnostic. Cependant, l’identification des pathogènes incriminés est une étape qui reste souvent longue. Les méthodes d’identification phénotypiques conventionnelles, fondées sur des cultures sur des milieux sélectifs et des galeries d’identification, nécessitent souvent plusieurs jours, voire plusieurs semaines pour obtenir un résultat. Le développement de protocoles visant à identifier le plus rapidement possible les souches incriminées mais aussi à prédire leurs susceptibilités et leurs résistances aux traitements antibiotiques, est donc l’une des priorités de la recherche clinique en microbiologie depuis quelques années. L’accès au séquençage [
2] ➔ et plus particulièrement au séquençage de 3e génération ouvre de nouvelles perspectives qui devraient faciliter le diagnostic et permettre une mise en place rapide du traitement approprié. Trois grandes pistes sont activement explorées : (1) la métagénomique ciblée permettant de caractériser un microbiote; (2) l’identification ciblée et directe des îlots de résistance et des gènes de susceptibilité aux antibiotiques pour les bactéries; et (3) le diagnostic par identification, sans culture et sans amplification spécifique, directement à partir de l’extraction de l’ADN total d’un échantillon clinique.
➔
Voir la Synthèse de C. Audebert et al., m/s n° 12, décembre 2014, page 1144
La métagénomique ciblée La métagénomique ciblée consiste en l’amplification d’un locus génomique considéré comme ubiquitaire chez l’ensemble des micro-organismes constitutifs du microbiote étudié et comportant des régions conservées, qui seront le plus souvent amplifiées par un même couple d’amorces, mais aussi des régions variables d’un organisme à l’autre afin de les discriminer. Ainsi, pour les bactéries, l’ADNr (ADN ribosomique) 16S est le locus le plus souvent utilisé. Les applications en métagénomique se sont très fortement développées depuis une dizaine d’années. Elles se sont démocratisées avec l’arrivée du séquençage de 2 e génération. Cette approche est en effet rapidement devenue une méthode de référence de profilage d’un microbiote avec des applications en environnement [
3], en santé [
4,
5] mais aussi dans l’industrie plus spécifiquement agro-alimentaire [
6]. Bien qu’elle présente de nombreux avantages, elle montre également certaines limites. Les technologies de séquençage de 2 e génération s’adressent à des régions d’environ 500 nucléotides, représentant environ 30 % de l’ADNr 16S. Cette limite de taille implique donc un biais sur le choix de la région variable séquencée. Certaines régions variables sont susceptibles de mieux discriminer certains groupes de bactéries (région V2-V3 par exemple pour les bactéries lactiques). Se limiter à une seule région variable implique d’atteindre une résolution au niveau de la famille et, au mieux, du genre. Il est ainsi illusoire d’espérer atteindre une identification au niveau de l’espèce par cette approche. D’autres méthodes faisant appel à des amplifications par PCR (polymerase chain reaction) multiplexes existent pour tenter de capter une plus grande quantité de zones variables. Le kit 16S metagenomics (Thermo Fisher Scientific) contient ainsi deux ensembles d’amorces permettant le séquençage de 7 régions hyper-variables. Utiliser plusieurs régions permet de diminuer le biais inhérent au choix de la région séquencée et d’augmenter la probabilité d’identifier un maximum d’organismes présents dans l’échantillon. Cependant, dans ce type d’expérience, l’information n’est pas phasée : les régions séquencées sont indépendantes les unes des autres, ce qui ne permet pas l’utilisation corrélée de l’ensemble des cibles séquencées; le gain en résolution taxonomique est donc limité. Le bénéfice en précision taxonomique est également affecté par les préparations des banques qui restent laborieuses et coûteuses. Le séquençage de 3 e génération ouvre de nouvelles perspectives en métagénomique ciblée : en séquençant des régions plus longues, la résolution taxonomique devrait être améliorée. Il reste cependant à déterminer si le taux d’erreurs, plus élevé, pénalisera l’association d’une lecture à un bon taxon. En 2016, une étude portant sur l’utilisation du MinION (ONT) pour la métagénomique ciblée [
7] a montré qu’il était possible, en utilisant des amorces « universelles » permettant d’amplifier plus de 90 % de l’ADNr 16S, d’identifier les bactéries au niveau de l’espèce en dépit d’un taux d’erreurs élevé. Des biais d’abondances, dus aux réactions de PCR, ont cependant été retrouvés. Des conclusions similaires ont été obtenues dans une étude menée sur le microbiote intestinal de souris [
8]. Ces deux premières études ont été réalisées avec une version de chimie (l’ensemble des réactifs utilisés pour le séquençage) qui est devenue obsolète (ONT R7, qui corrigeait les erreurs de lecture en séquençant les deux brins d’ADN sens et antisens, correspondant au mode 2D) et qui donnait un taux d’erreurs de séquençage d’environ 15 %. Par la suite, la chimie ONT R9 en mode 2D a permis de diminuer le nombre d’erreurs de séquençage, passant ainsi à environ 5 %. Le mode 2D a depuis disparu; il a été remplacé par un nouveau protocole, réduisant le temps de préparation à seulement 10 minutes, et dans lequel seul le brin sens est lu (protocole 1D only). Ce protocole est donc plus rapide mais de plus faible qualité (avec 20 à 30 % d’erreurs). Le débit de séquençage s’est trouvé ainsi largement augmenté. L’ADNr 16S quasi complet d’une communauté artificielle contenant 20 espèces en proportion équimolaire a été séquencé avec ce protocole rapide [
9]. Les résultats ont montré une meilleure sensibilité au niveau de l’espèce et une grande rapidité (seulement 5 minutes de séquençage suffisent pour obtenir des résultats comparables à ceux qui auparavant nécessitaient 4 heures). La longueur des lectures doit permettre de séquencer l’ADNr 23S, plus long que l’ADNr 16S, ou même l’opéron d’ADNr complet, et ainsi, peut-être, d’atteindre une meilleure résolution pour l’identification des bactéries. Dans une étude récente [
10], 4,5 kilobases de l’opéron ribosomique ont pu être séquencées avec ce nouveau protocole, et un plus grand nombre de lectures ont été classées correctement au niveau de l’espèce, la longueur de l’amplicon compensant le taux d’erreurs plus élevé. Bien que les amorces nécessitent d’être optimisées pour diminuer les biais de couvertures, et que les banques de séquences pour l’opéron ribosomique doivent être complétées, cette étude montre qu’il est concevable d’identifier des bactéries au niveau de la souche. Malgré certains biais, la métagénomique ciblée reste facile à mettre en place. Elle permet de définir un profil microbien de plus en plus précis, ouvrant de nouvelles perspectives en clinique où le temps est un facteur déterminant. Bien évidemment, des protocoles standardisés, éprouvés et adaptés aux hôpitaux, doivent encore être développés et l’absence de standardisation représente le verrou actuel majeur pour l’emploi des approches métagénomiques en clinique. Détection de la susceptibilité et de la résistance aux antibiotiques La résistance aux antibiotiques constitue aujourd’hui l’un des plus importants problèmes de santé mondiale. L’augmentation du nombre de bactéries résistantes nécessite la mise en place de méthodes de détection rapides afin de déterminer leurs profils de susceptibilité et de résistance. Le séquençage haut-débit apporte des solutions efficaces à ces problématiques. Les îlots de résistance et de pathogénicité sont souvent constitués, ou flanqués, de régions répétées, difficiles à identifier en utilisant des lectures génomiques courtes. En combinant des lectures courtes, obtenues par la technique Illumina, contenant très peu d’erreurs, et des lectures longues, obtenue par MinION, Ashton et al. [
11] sont parvenus, en 2014, à déterminer la position et la structure d’un îlot de résistance aux antibiotiques dans le génome de Salmonella typhi Haplotype 58, qui n’avait jusqu’alors pas été identifié, démontrant ainsi l’intérêt de cette nouvelle génération de séquençage qui aboutit à des génomes bactériens complètement assemblés. Des isolats bactériens cliniques résistants à la plupart des traitements antibiotiques ont été rapportés pour de nombreuses espèces, rendant les tests de sensibilité aux antibiotiques cruciaux pour définir le traitement de ces infections. Aujourd’hui, ils reposent sur des identifications phénotypiques après culture bactérienne. Bien que ces tests apportent une réponse en un ou deux jours pour les bactéries à croissance rapide, plusieurs semaines peuvent être nécessaires pour les bactéries à croissance lente comme Mycobacterium turbeculosis, agent responsable de la tuberculose. Le séquençage des génomes bactériens permet d’augmenter significativement la vitesse d’identification exhaustive des résistances et susceptibilités aux antibiotiques. Une preuve de concept de l’utilisation du séquençage en infectiologie clinique dans ce contexte a été apportée [
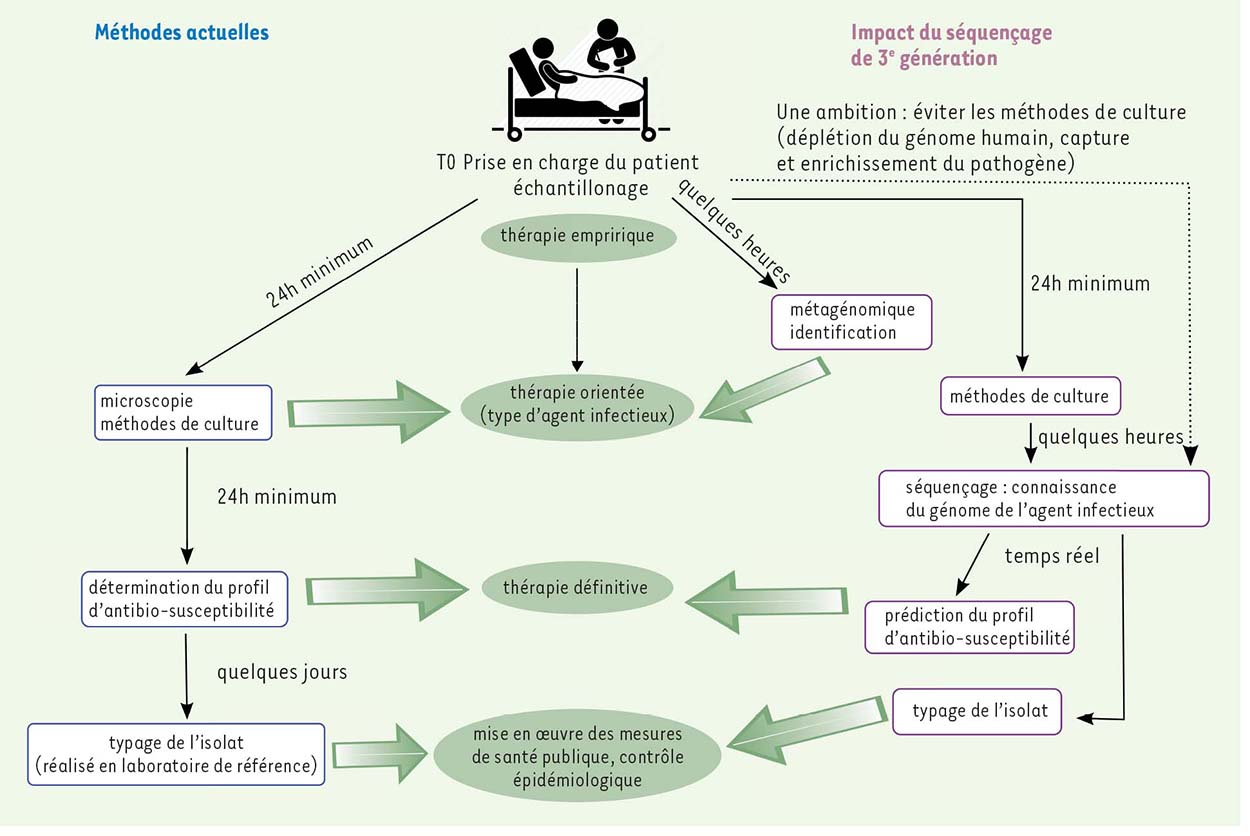
12]. Un protocole est ainsi proposé pour l’évaluation de la susceptibilité aux antibiotiques et des résistances induites par des gènes ou des mutations ponctuelles. Une implémentation concrète de ce protocole a été réalisée pour deux espèces multi-résistantes, Staphylococcus aureus et M. turbeculosis, avec des lectures courtes Illumina. Pour S. aureus, la sensibilité et la spécificité qui ont été obtenues sont comparables à celles recueillies avec les méthodes phénotypiques standards, démontrant la faisabilité du protocole. Pour M. tuberculosis, la sensibilité observée s’est avérée en revanche plus faible avec le protocole qu’avec les méthodes standards, probablement à cause de mécanismes de résistance méconnus qui ne figurent pas dans la base de données utilisée. Ceci révèle l’une des limites de cette méthode qui nécessite un catalogue des résistances ou un jeu de données d’apprentissage très important. Si dans un cadre strictement clinique ce type de données posent problème (en raison de l’absence de validation), elles sont la base de recherches visant à relier phénotype et causalité génomique. Pour la détection des résistances, un gain de 12 h est observé pour S. aureus contre un gain de temps de 6 à 8 semaines pour M. tuberculosis, ce délai serait réduit à 2 semaines en l’absence de culture. Cette preuve de concept a été étendue avec une implémentation du protocole pour des données MinION : les longues lectures suffisent dans ce cas pour retrouver des résultats de qualité identique aux méthodes classiques, en réduisant encore le temps du séquençage. Malgré quelques limites restant à franchir, comme par exemple l’amélioration des connaissances des mécanismes de résistance au niveau génétique, cette preuve de concept démontre l’intérêt du séquençage haut-débit pour la détection des résistances aux antibiotiques. Cette méthodologie peut être étendue à d’autres types de mécanismes bactériens reposant sur des gènes comme ceux codant des facteurs de virulence. L’amélioration des technologies de séquençage de 3e génération (permettant moins d’erreurs et plus de débit) ferait prendre un nouveau virage en microbiologie clinique, éliminant totalement l’étape la plus chronophage : la culture (Figure 1).
 | Figure 1.
Enchaînement des tâches visant à prendre en compte les caractéristiques d’un pathogène pour adapter un traitement, et mise en perspective des apports présents et envisageables du séquençage haut-débit de troisième génération.
|
Diagnostic et génome des pathogènes Plusieurs études ont montré l’intérêt des lectures longues pour l’obtention de séquences complètes de génomes microbiens, et le diagnostic génomique présente trois caractéristiques qui en font une approche de choix pour des applications cliniques :
-
il s’agit d’un diagnostic sans a priori (pas de biais PCR [polymerase chain reaction]);
-
ce diagnostic peut être envisagé sans culture préalable;
-
il peut s’avérer conclusif pour l’élucidation de co-infections.
Le diagnostic génomique réalisé à l’aide d’un séquenceur de 3e génération permet un formidable gain de temps, en évitant les phases de culture, avec la possibilité d’être employé au plus près du patient et non, comme c’est souvent le cas actuellement, au sein de centres spécialisés. Néanmoins, compte tenu du débit actuel des séquenceurs, il est nécessaire de limiter la contamination en génome humain en amont du séquençage, par centrifugations sélectives afin de séparer les cellules humaines des micro-organismes, suivies d’une étape de lyse différentielle pour éliminer le génome humain [
13]. Le diagnostic génomique permet de révéler les coinfections plus aisément que les autres méthodes. Les approches de 3e génération ajoutent la précision d’identification grâce à la taille de leurs lectures de plusieurs kilobases. Même erronées, les données générées apparaissent suffisantes pour une identification fine au niveau de la souche [11]. Portabilité, analyse en temps réel et lectures longues (Tableau I) sont autant de caractéristiques qui font des technologies de séquençage par nanopores des outils d’investigation génomique permettant de gagner du temps, de la précision et de pallier les difficultés liées aux méthodes de cultures. Exempte de toute amplification, cette approche nécessite cependant quelques centaines de nanogrammes d’ADN purifié, ce qui pourrait constituer une limite à son utilisation pour des échantillons cliniques. Ce verrou est néanmoins en passe d’être levé notamment par l’utilisation de kit d’amplification pangénomique (de type REPLI-g WGA, Qiagen) permettant une analyse sur moins de 1 ng de matériel génétique isolé. Analyses génomiques du patient Si caractériser le génome des agents infectieux représente déjà une avancée dans le sens de la médecine de précision, s’intéresser au génome de l’hôte afin de mieux le soigner est un sujet en plein développement. Proposer des traitements ciblés nécessite d’identifier les singularités d’un patient avec, souvent, le recours à des technologies d’analyse génomique. L’accès aux technologies de séquençage devient ainsi un enjeu fondamental, en particulier pour les diagnostics prénataux non invasifs et en cancérologie, ouvrant de nouvelles perspectives avec la possibilité d’utiliser des biopsies liquides. Diagnostic prénatal non invasif Le risque de fausses couches accompagnant l’amniocentèse réalisée à des fins de diagnostic d’anomalies génétiques a motivé la recherche de solutions alternatives aux tests génétiques prénataux existants. Des solutions non invasives tirant profit de la faible proportion de cellules fœtales circulantes (une cellule fœtale pour 10 5 à 10 8 cellules maternelles), ou de l’ADN fœtal présent dans le sang maternel, dont le taux augmente au cours de la grossesse, ont été développées, notamment sur la base du séquençage haut-débit de 2 e génération avec, pour la recherche d’aneuploïdie fœtale, par exemple, des sensibilités et spécificités proches de 99 %. Principalement en raison du coût et de l’environnement des séquenceurs de 2 e génération, des implémentations sur des séquenceurs portatifs de 3 e génération ont été proposées. Malgré le taux d’erreurs encore relativement important, la faisabilité de cette technologie a été démontrée [
14]. Diagnostic en cancérologie La prise en compte du statut des mutations du gène TP53 (codant le suppresseur de tumeur p53) est devenue quasi-routinière dans les pratiques cliniques concernant les patients atteints de leucémie lymphoïde chronique. Du séquençage Sanger au séquençage de 2 e génération, plusieurs technologies ont servi de base à un déploiement méthodologique rationalisant, en termes de temps, de coût et de sensibilité, le séquençage de ce gène, et des recommandations, fixées par l’European research initiative afin de limiter les variabilités interlaboratoires, ont permis l’application de ces technologies pour dresser le profil de ses mutations. La généralisation des approches visant à prendre en compte la singularité moléculaire des tumeurs des patients repose directement sur le développement de l’accès aux technologies employées pour cette caractérisation. Ainsi, en 2014, 117 000 tests génétiques, intéressant 70 000 patients, ont été réalisés en France, pour un coût de 24 millions d’euros. En 2015, d’après les données de l’Institut national du cancer, les thérapies ciblées représentaient 24 % de l’arsenal thérapeutique autorisé dans le cancer. L’implémentation des technologies de séquençage haut-débit de 2e génération a constitué un virage majeur avec la possibilité de multiplexer les échantillons et d’étudier simultanément un nombre élevé de gènes (plus de 30 aujourd’hui) conduisant à une baisse drastique des coûts. Cette mise en œuvre implique des réorganisations au sein des laboratoires avec l’acquisition de nouvelles compétences devenues primordiales pour l’analyse des résultats. Récemment, Minervini et al. [
15] ont montré que l’utilisation du séquenceur MinION permettait un gain de sensibilité par rapport à un séquençage Sanger et qu’il pourrait être utilisé comme alternative au séquençage de 2e génération dans un contexte de décentralisation, même si son débit de séquençage demeure limité pour la détection précoce des tumeurs solides. Le séquençage de 2e génération répond à l’exigence que nécessitent des analyses diagnostiques comme le profilage du gène TP53 et les séquenceurs nanopore ne paraissent pas apporter actuellement une plus-value significative. En revanche, la grande taille des lectures de séquençage qu’ils permettent devrait s’intégrer parfaitement pour les études de variations structurales. De nombreux cancers (cancer des poumons, ovarien, œsophagien) résultent de variations structurales somatiques constituées de délétions, de duplications, de variations du nombre de copies, d’insertions, d’inversions ou de translocations de relativement grande taille, allant de 1 kilobase (kb) à 3 mégabases (Mb). Ainsi, dans le cas des leucémies aiguës, plusieurs réarrangements chromosomiques ont été décrits résultant souvent en la fusion de deux gènes et l’expression de protéines hybrides. L’évaluation de ces modifications structurales fournit des informations cruciales pour les traitements et l’évolution de la maladie. La cytogénétique conventionnelle est la méthode de référence. Cependant, cette méthode, qui nécessite expertise et temps, n’est pas adaptée à la détection de réarrangements cryptiques. Elle est ainsi souvent associée à des méthodes d’hybridation in situ par fluorescence (FISH) et de RT-PCR (reverse transcriptase-polymerase chain reaction), dont le coût peut rapidement devenir prohibitif. Fondée sur le séquençage ciblé multiplexé, une méthode par ligation-dependant RT-PCR (LD-RTPCR) a été développée [
16]. Les ARN messagers isolés des échantillons de patients sont convertis en ADN complémentaires (ADNc) et incubés avec des sondes spécifiques des régions de réarrangements connus. Si une fusion est observée les sondes sont ligaturées, amplifiées puis séquencées par des approches de 2e génération. Cette méthode permet de détecter la plupart des variants structuraux identifiés par les méthodes conventionnelles mais aussi des variants additionnels, en évitant des analyses FISH. Elle peut être adaptée (nouvelles sondes) mais elle est très dépendante des connaissances a priori des jonctions dans l’ARNm de fusion dont une seule variation dans le site peut empêcher la détection. Les caractéristiques mêmes des variants structuraux en font des objets génomiques constitués de séquences répétées qui restent difficiles à identifier par les technologies de séquençage de 2e génération ne proposant que des lectures courtes. Les séquenceurs de 3e génération permettent, par contre, avec des lectures pouvant atteindre plusieurs dizaines de kb, l’identification de ces variants. Ainsi, Norris et al. [
17] ont montré l’application possible de la technologie ONT pour détecter, après une PCR préalable, des variations structurales caractérisées incluant de larges délétions, des inversions et des translocations qui inactivent les gènes suppresseurs de tumeurs CDKN2A(cyclin-dependent kinase inhibitor 2A)/p16 et SMAD4(mothers against DPP homolog 4)/DPC4(deleted in pancreatic cancer-4) dans le cancer pancréatique. Une récente étude montre que les longues lectures apportent un bénéfice significatif dans la détection de remaniements chromosomiques massifs de novo de type chromothripsis1 [
18] démontrant ainsi l’apport du séquençage par nanopores pour l’étude des variants structuraux. L’une des promesses du séquençage par nanopores, qui pourrait devenir un argument de poids, est la possibilité d’accéder aux profils du méthylome par ces technologies. Une preuve de concept a en effet permis de montrer l’efficience de l’approche de 3e génération pour une application difficile à appréhender par une méthode employant une génération précédente [
19]. Cette étude, fondée sur le séquençage de deux lignées cellulaires mammaires humaines, la lignée MCF10A (cellules épithéliales non tumorigènes) et la lignée MDA-MB-231 (cellules de métastase agressives), a montré la possibilité d’accéder à l’information des 5-méthylcytosines par la technologie ONT. |
1.
Jordan
B.
Séquençage d’ADN : l’offensive des nanopores.
Med Sci (Paris).
2017;; 33 : :801.-4. 2.
Audebert
C
,
Hot
D
,
Lemoine
Y
,
Caboche
S.
Le séquençage haut-débit : vers un diagnostic basé sur la séquence complète du génome de l’agent infectieux.
Med Sci (Paris).
2014;; 30 : :1144.-51. 3.
Bouhajja
E
,
Agathos
SN
,
George
IF.
Metagenomics: probing pollutant fate in natural and engineered ecosystems . Biotechnol Adv.
2016;; 34 : :1413.-26. 4.
Hilton
SK
,
Castro-Nallar
E
,
Pérez-Losada
M
, et al.
Metataxonomic and metagenomic approaches vs. culture-based techniques for clinical pathology. , Front Microbiol.
2016;; 7 : :484.. 5.
Deurenberg
RH
,
Bathoorn
E
,
Chlebowicz
MA
, et al.
Application of next generation sequencing in clinical microbiology and infection prevention . J Biotechnol.
2017;; 243 : :16.-24. 6.
Ercolini
D.
High-throughput sequencing and metagenomics: moving forward in the culture-independent analysis of food microbial ecology . Appl Environ Microbiol.
2013;; 79 : :3148.-55. 7.
Benítez-Paez
A
,
Portune
KJ
,
Sanz
Y.
Species-level resolution of 16S rRNA gene amplicons sequenced through the MinION™ portable nanopore sequencer . Gigascience.
2016;; 28 : :4.. 8.
Shin
J
,
Lee
S
,
Go
MJ
, et al.
Analysis of the mouse gut microbiome using full-length 16S rRNA amplicon sequencing . Sci Rep.
2016;; 6 : :29681.. 9.
Mitsuhashi
S
,
Kryukov
K
,
Nakagawa
S
, et al.
A portable system for rapid bacterial composition analysis using a nanopore-based sequencer and laptop computer . Sci Rep.
2017;; 7 : :5657.. 10.
Benítez-Paez
A
,
Sanz
Y.
Multi-locus and long amplicon sequencing approach to study microbial diversity at species level using the MinION™ portable nanopore sequencer . Gigascience.
2017;; 6 : :1.-12. 11.
Ashton
PM
,
Nair
S
,
Dallman
T
, et al.
MinION nanopore sequencing identifies the position and structure of a bacterial antibiotic resistance island . Nat Biotechnol.
2015;; 33 : :296.-300. 12.
Bradley
P
,
Gordon
NC
,
Walker
TM
, et al.
Rapid antibiotic-resistance predictions from genome sequence data for Staphylococcus aureus and Mycobacterium tuberculosis . Nat Commun.
2015;; 6 : :10063.. 13.
Schmidt
K
,
Mwaigwisya
S
,
Crossman
LC
, et al.
Identification of bacterial pathogens and antimicrobial resistance directly from clinical urines by nanopore-based metagenomic sequencing . J Antimicrob Chemother.
2017;; 72 : :104.-14. 14.
Cheng
SH
,
Jiang
P
,
Sun
K
, et al.
Noninvasive prenatal testing by nanopore sequencing of maternal plasma DNA: feasibility assessment . Clin Chem.
2015;; 61 : :1305.-6. 15.
Minervini
CF
,
Cumbo
C
,
Orsini
P
, et al.
TP53 gene mutation analysis in chronic lymphocytic leukemia by nanopore MinION sequencing . Diagn Pathol.
2016;; 11 : :96.. 16.
Ruminy
P
,
Marchand
V
,
Buchbinder
N
, et al.
Multiplexed targeted sequencing of recurrent fusion genes in acute leukaemia . Leukemia.
2016;; 30 : :757.-60. 17.
Norris
AL
,
Workman
RE
,
Fan
Y
, et al.
Nanopore sequencing detects structural variants in cancer . Cancer Biol Ther.
2016;; 17 : :246.-53. 18.
Cretu Stancu
M
,
van Roosmalen
MJ
,
Renkens
I
, et al.
Mapping and phasing of structural variation in patient genomes using nanopore sequencing . Nat Commun.
2017;; 8 : :1326.. 19.
Simpson
JT
,
Workman
RE
,
Zuzarte
PC
, et al.
Detecting DNA cytosine methylation using nanopore sequencing . Nat Methods.
2017;; 14 : :407.-10. 20.
Jordan
B.
La génération suivante, déjà . Med Sci (Paris).
2009;; 25 : :649.-50. 21.
Montel
F.
Séquençage de l’ADN par nanopores : résultats et perspectives . Med Sci (Paris).
2018;; 34 : :161.-5. |