| |
| Med Sci (Paris). 34(4): 363–365. doi: 10.1051/medsci/20183404018.Chroniques génomiques - Dépister les cancers asymptomatiques ? Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/ CNRS; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 09, France MeSH keywords: Marqueurs biologiques tumoraux, ADN tumoral, Dépistage précoce du cancer, Humains, Dépistage de masse, Protéines tumorales, Tumeurs, Oncogènes, Valeur prédictive des tests, Sensibilité et spécificité, analyse, méthodes, normes, diagnostic |

Une étude parue au début de l’année dans la revue Science [
1] a fait grand bruit : tendant à révéler la présence d’un cancer par l’analyse de l’ADN tumoral circulant dans le sang, elle affiche des performances intéressantes en termes de sensibilité et de spécificité, et a été saluée comme une étape vers la mise au point d’un test de dépistage en l’absence de signes cliniques [
2]. Dans l’idéal, un tel test pourrait être pratiqué à grande échelle et permettrait la détection de cancers encore peu développés et susceptibles d’un traitement efficace. C’est, on le sait, le but de plusieurs laboratoires et entreprises, notamment la firme Grail déjà évoquée dans ces chroniques [
3] 
 Voir la Chronique génomique de B. Jordan, m/s n° 4, avril 2017, page 441
Voir la Chronique génomique de B. Jordan, m/s n° 4, avril 2017, page 441
Je voudrais ici indiquer brièvement ce qu’apporte de nouveau ce travail, évaluer ses limites, rappeler en quoi la notion même de dépistage peut être problématique et m’interroger sur les raisons de l’engouement médiatique et financier1 qui entoure ces tentatives. |
Une analyse sophistiquée impliquant ADN et protéines aboutit à un test performant L’article de Science [1] émane d’un ensemble d’excellents laboratoires aux États-Unis et en Australie, et décrit un test appelé CancerSEEK qui constitue une version sophistiquée de la technique dite de « biopsie liquide » [
4] 
 Voir la Chronique génomique de B. Jordan, m/s n° 8-9, août-septembre 2015, page 805
Voir la Chronique génomique de B. Jordan, m/s n° 8-9, août-septembre 2015, page 805
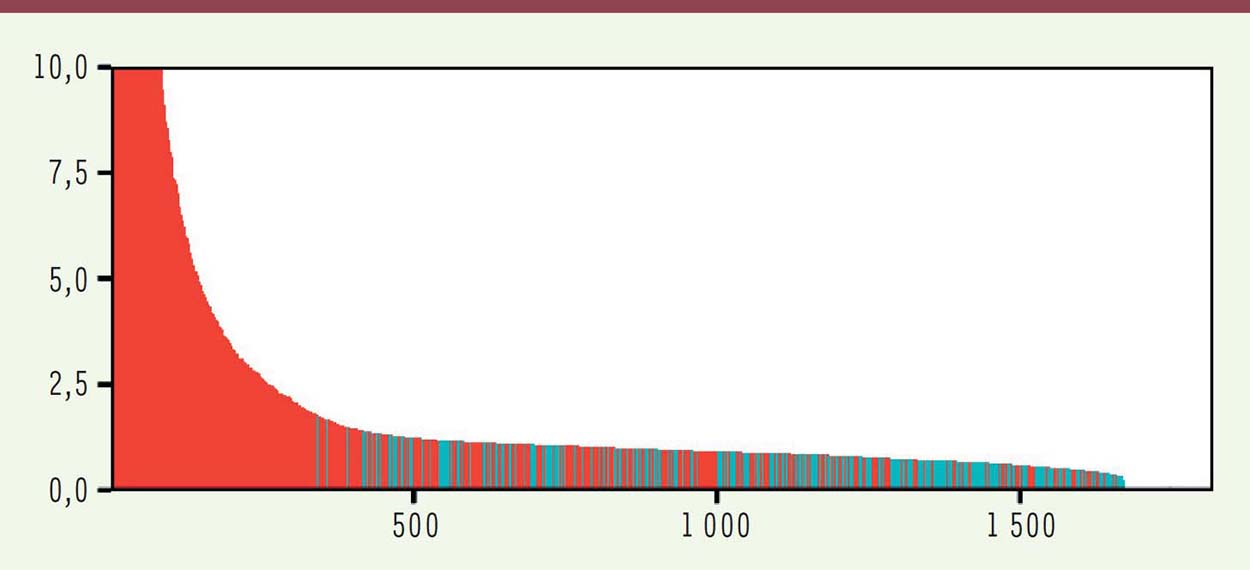
Celle-ci vise en général à analyser, dans un échantillon de sang, les (rares) cellules tumorales circulantes ou les fragments d’ADN libérés par la tumeur. Dans le cas présent, les auteurs procèdent en fait à une double analyse, d’une part la recherche, dans l’ADN circulant, de mutations au sein d’une soixantaine d’amplicons2, couvrant les régions critiques des gènes les plus souvent impliqués dans des cancers, et d’autre part la mesure (par immuno-essai) du niveau plasmatique de huit protéines elles aussi liées à différents cancers. Ces différents marqueurs ont été sélectionnés au sein d’un ensemble plus vaste (200 amplicons et 39 protéines) après des essais montrant qu’ils sont les plus informatifs. Ils sont alors utilisés pour étudier une série de 1 005 patients présentant un cancer non métastatique de stade I à III3 appartenant à l’un des huit types les plus fréquents (ovaire, foie, estomac, pancréas, œsophage, colo-rectum, poumon et sein), ainsi qu’une cohorte « témoin » comprenant 812 personnes indemnes de cancer. Un algorithme prenant en compte la présence de mutations aux 2 001 positions dans l’ADN couvertes par les amplicons, et les niveaux plasmatiques des huit protéines mesurées, est alors construit et utilisé pour évaluer les patients et les témoins; la
Figure 1
montre la distribution des valeurs de l’index obtenu pour l’ensemble de ces personnes après application de l’algorithme.  | Figure 1.
Performance du test CancerSEEK sur l’ensemble des sujets (patients et témoins). En ordonnée, la valeur de l’index obtenu par application de l’algorithme; en abscisse, le nombre de sujets. Chaque personne est représentée par une ligne verticale. Les témoins (en bleu) présentent des valeurs plus faibles de l’index que les patients (en rouge), mais un certain recouvrement est visible (extrait partiel remanié de la figure S2 des données supplémentaires de [ 1]). |
Comme toujours pour un test, la sensibilité et la spécificité ne sont pas indépendantes : si l’on choisissait comme seuil de positivité la valeur 2,5 de l’index (Figure 1), la spécificité serait de 100 % (aucun faux positif) mais la sensibilité n’atteindrait qu’environ 25 % (approximativement 250 positifs sur 1 005 patients). En fonction de leurs études statistiques, les auteurs ont adopté un seuil qui aboutit à une sensibilité médiane (sur cet échantillon) de 70 % et une spécificité légèrement supérieure à 99 % (7 « faux positifs » sur 812 témoins). Si l’on rentre dans le détail, on note que la sensibilité varie avec le type de cancer : presque 100 % pour l’ovaire, moins de 40 % pour le sein. Comme l’on pouvait s’y attendre, elle est aussi nettement plus faible pour les cancers de stade I (43 %) que pour ceux de stade II (73 %) ou III (78 %). Deux remarques pour terminer cette brève description : il est concevable que certains des sept faux positifs correspondent en fait à un cancer non encore visible, la spécificité serait alors meilleure qu’annoncé; en revanche, on peut reprocher à cette étude le fait que l’algorithme mis au point n’ait pas été utilisé sur un autre ensemble indépendant de patients de manière à vérifier rigoureusement sa pertinence. Notons encore que les auteurs montrent qu’un autre algorithme appliqué à leurs données permet, dans plus de la moitié des cas, d’indiquer la localisation du cancer, une donnée très importante du point de vue clinique. Au total, il s’agit bien là d’un travail significatif dont les résultats sont globalement supérieurs à tout ce qui a été publié jusqu’ici dans ce domaine, ce qui explique son retentissement [2]. Faut-il pour autant en conclure qu’un dépistage systématique du cancer (en l’absence de tout symptôme) est à portée de main ? |
Des inconnues de taille, une justification discutable Notons d’abord la faible sensibilité de CancerSEEK pour les tumeurs au stade I (43 %) : elle sera logiquement encore plus faible pour des tumeurs plus précoces, celles que l’on cherchera à détecter. La technique d’analyse employée est déjà très sophistiquée (même si les auteurs affirment qu’elle pourra ne coûter que 500 dollars, un chiffre qui ne semble pas très réaliste compte tenu des marges pratiquées par les industriels), peut-elle être encore perfectionnée et rendue plus sensible sans augmenter le taux de faux positifs [
5] ? Et la validation d’un test commercial, incontournable pour sa mise sur le marché, risque d’être longue et coûteuse : il faudrait idéalement tester plusieurs milliers d’individus puis les suivre sur cinq ou dix ans pour voir lesquels développeront un cancer et déterminer s’ils avaient été repérés par le test… Plus globalement, on se pose de plus en plus de questions sur le bien-fondé des campagnes de dépistage, dont le rapport bénéfice/risque est loin d’être aussi évident que ne l’affirment leurs promoteurs [
6,
7]. C’est le cas du dépistage du cancer de la prostate par le dosage du niveau du PSA (prostate specific antigen) pour lequel la preuve a été faite qu’un dépistage large et systématique faisait plus de mal que de bien; la question se pose sérieusement pour le dépistage du cancer du sein [
8] et même pour celui du poumon [
9]. Le bénéfice a été clairement établi, en revanche, pour le cancer colo-rectal et pour celui du col de l’utérus. Pour revenir à CancerSEEK, les problèmes se situent à deux niveaux : les faux positifs, nombreux en cas de dépistage large même si la spécificité est de 99 %, soumettent des personnes indemnes à des explorations complémentaires (biopsies, etc.) comportant certains risques. L’autre écueil important est la détection de cancers à évolution très lente qui n’auraient vraisemblablement pas posé de problème, suivie de leur traitement et des morbidités qui y sont associées. Et n’oublions pas, si la sensibilité est de 43 %, les 57 % de « faux négatifs » (cancer présent mais non détecté) qui vont être rassurés à tort ! Des campagnes de dépistage trop larges menées avec des tests encore imparfaits transforment ainsi nombre d’individus en « patients » embarqués dans une série d’analyses et parfois d’interventions souvent inutiles mais toujours anxiogènes et non dénuées de risque - tout en rassurant à tort une bonne partie d’entre eux. Le test CancerSEEK va être utilisé pour 50 000 femmes âgées de 65 à 75 ans qui seront suivies sur cinq ans [2], une étude qui sera certainement instructive mais qui montre l’importance et la durée des essais nécessaires pour valider de telles analyses… |
Les raisons d’un engouement Dans ces conditions, on peut se demander comment il se fait que ces travaux rencontrent un écho aussi favorable et suscitent de tels investissements. Il faut noter qu’ils se situent dans un contexte où se développe une wellness industry
4 [
10,
11] censée assurer notre santé et notre bien-être grâce à l’analyse approfondie de multiples paramètres biologiques mesurés en continu par divers objets connectés : avec les techniques d’intelligence artificielle et de deep learning
5,, on pourrait ainsi détecter des anomalies subtiles et les corriger avant qu’elles n’aient des conséquences physiologiques. Un dépistage des cancers « avant qu’ils n’apparaissent » est évidemment cohérent avec ce type de démarche, d’autant plus que des décennies de promotion des campagnes de dépistage ont installé dans l’opinion la conviction que « le dépistage sauve des vies » - quelles que soient les performances du test mis en œuvre. Le marché potentiel est donc bien présent, et ces projets qui mettent en œuvre un ensemble de techniques de pointe s’avèrent très séduisants pour les investisseurs. L’institution médicale y trouve aussi son compte, surtout lorsqu’elle est largement privatisée comme aux États-Unis : un volume important de tests sophistiqués (et donc onéreux), suivis pour certains d’investigations complémentaires, et débouchant sur un traitement quasi-systématique… Les auteurs de l’article de Science évoquent bien le problème du surdiagnostic, mais c’est pour affirmer aussitôt que le véritable problème est celui d’un éventuel surtraitement (traitement de tumeurs qui n’auraient pas posé de problème au patient) et qu’il sera évité grâce à l’expertise des oncologues [2]. On ne peut qu’être sceptique sur ce point, la tendance naturelle des patients et des cliniciens étant d’intervenir dès la détection d’une anomalie. On voit qu’au total il existe un contexte fort qui pousse à la mise au point et à l’emploi de ce type d’analyse même si son utilité réelle et son rapport bénéfice/risque ne sont pas clairement établis. |
Un avenir qui reste problématique Il est certain que ces travaux vont se poursuivre, tant dans le cadre universitaire qu’au sein d’entreprises comme Grail ou Guardant Health [3]  . Les résultats présenteront un intérêt scientifique indéniable et apporteront des informations précieuses sur l’histoire naturelle des cancers, et, au prix d’une plus grande complexité technique, les performances des tests proposés seront sans doute améliorées. Comme déjà mentionné [3], ils déboucheront sur des applications solides pour le suivi des cancers en cours de traitement, domaine pour lequel l’analyse est à la fois plus facile (abondance d’ADN tumoral, mutations connues) et indubitablement pertinente. Auront-ils un réel impact en termes de dépistage ? On peut rester sceptique sur ce point, sauf si l’on envisage des sous-populations particulières dans lesquelles le risque de cancer est élevé, ce qui minimise l’impact des faux positifs. En tout état de cause, l’idée que dans quelques années l’on pourra par une simple analyse de sang détecter un cancer au « stade zéro » me semble à peu près aussi chimérique que l’affirmation que « l’homme qui vivra mille ans est déjà né »… [
12]. . Les résultats présenteront un intérêt scientifique indéniable et apporteront des informations précieuses sur l’histoire naturelle des cancers, et, au prix d’une plus grande complexité technique, les performances des tests proposés seront sans doute améliorées. Comme déjà mentionné [3], ils déboucheront sur des applications solides pour le suivi des cancers en cours de traitement, domaine pour lequel l’analyse est à la fois plus facile (abondance d’ADN tumoral, mutations connues) et indubitablement pertinente. Auront-ils un réel impact en termes de dépistage ? On peut rester sceptique sur ce point, sauf si l’on envisage des sous-populations particulières dans lesquelles le risque de cancer est élevé, ce qui minimise l’impact des faux positifs. En tout état de cause, l’idée que dans quelques années l’on pourra par une simple analyse de sang détecter un cancer au « stade zéro » me semble à peu près aussi chimérique que l’affirmation que « l’homme qui vivra mille ans est déjà né »… [
12].
 Voir la Chronique génomique de B. Jordan, m/s n°4, avril 2017, page 441
Voir la Chronique génomique de B. Jordan, m/s n°4, avril 2017, page 441
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Cohen
JD
,
Li
L
,
Wang
Y
, et al.
Detection and localization of surgically resectable cancers with a multi-analyte blood test . Science.
2018;; 359 : :926.-30. 2.
Kaiser
J.
Liquid biopsy for cancer promises early detection.
Science.
2018;; 359 : :259.. 3.
Jordan
B
. Les chimères du dépistage . Med Sci (Paris).
2017;; 33 : :441.-4. 4.
Jordan
B.
Biopsies liquides, une révolution en cancérologie ?
Med Sci (Paris).
2015;; 31 : :805.-7. 5.
Haque
IS
,
Elemento
O
. Challenges in using ctDNA to achieve early detection of cancer . bioRxiv. ( preprint first posted online dec. 21, 2017). doi: https://doi.org/10.1101/237578.. 6.
Prasad
V
,
Lenzer
J
,
Newman
DH
. Why cancer screening has never been shown to save lives and what we can do about it . Br Med J.
2016;; 352 : :h6080.. 7.
Esserman
LJ
,
Thompson
IM
,
Reid
B
, et al.
Addressing overdiagnosis and overtreatment in cancer: a prescription for change.
Lancet Oncol.
2014;; 15 : :e234.-42. 8.
Welch
HG
,
Prorok
PC
,
O’Malley
AJ
,
Kramer
BS
. Breast-cancer tumor size, overdiagnosis, and mammography screening effectiveness.
N Engl J Med.
2016;; 375 : :1438.-47. 9.
Kinsinger
LS
,
Anderson
C
,
Kim
J
, et al.
Implementation of lung cancer screening in the veterans health administration . JAMA Intern Med.
2017;; 177 : :399.-406. 10.
Gibson
G
. Wellness and health omics linked to the environment: the WHOLE approach to personalized medicine . Adv Exp Med Biol.
2014;; 799 : :1.-14. 11.
Saqi
M
,
Pellet
J
,
Roznovat
I
, et al.
Systems medicine: the future of medical genomics, healthcare, and wellness . Methods Mol Biol.
2016;; 1386 : :43.-60. 12.
Alexandre
L
. L’homme qui vivra mille ans est déjà né (interview) . Le Point,
30 juillet.
2014. |


 Voir la Chronique génomique de B. Jordan, m/s n° 4, avril 2017, page 441
Voir la Chronique génomique de B. Jordan, m/s n° 4, avril 2017, page 441