| |
| Med Sci (Paris). 34(8-9): 749–751. doi: 10.1051/medsci/20183408025.De HGP-write aux « super-cellules » Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS ; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Escherichia coli, Génome humain, Projet génome humain, Humains, Annotation de séquence moléculaire, Édition, Écriture, génétique, physiologie, économie, organisation et administration, méthodes |

On se souvient sans doute du projet HGP-write, annoncé en juin 2016 par Jeff Boeke et George Church [1], et qui visait à réaliser la synthèse intégrale du génome humain - rien que cela ! Comme la plupart des commentateurs, j’étais assez sceptique sur la possibilité de réaliser un tel programme [2] (→). (→) Voir la Chronique génomique de B. Jordan, m/s n° 10, octobre 2016, page 898 De fait, près de deux ans plus tard, ce projet n’a pas réussi à décrocher des financements significatifs (on parle ici de centaines de millions de dollars) et ne semble pas avoir réellement débuté, même s’il suscite un intérêt soutenu et implique plusieurs dizaines d’institutions. Au contraire, la synthèse d’un génome complet de levure, Saccharomyces cerevisiae [3], a bien avancé et devrait aboutir à la fin de cette année [4] : mais il s’agit là d’un génome de 12 mégabases environ, alors que l’ADN humain (haploïde) s’étend sur 3 000 mégabases. Cependant, voici qu’apparaît un nouvel avatar de ce projet, la construction d’une lignée de « super-cellules » humaines, résistante à toute infection par les virus. Présenté par Nature comme un downsizing du plan initial [5], ce projet a été discuté lors d’une réunion scientifique du Genome Project-write et a fait l’objet d’un communiqué de presse détaillé [6]. Il me semble qu’il mérite d’être examiné avec attention, et c’est l’objectif de cette chronique. |
En fait, l’équipe de Church explore depuis plusieurs années, sur la bactérie Escherichia coli, la construction d’organismes résistants aux virus, par une approche assez radicale : l’élimination dans le génome de tous les exemplaires d’un codon (et son remplacement par un codon synonyme), suivie de la suppression de l’ARN de transfert spécifique du codon éliminé. Dans ces conditions la bactérie est viable mais l’ARN provenant d’un virus ne peut être traduit puisqu’il contient fatalement le codon « interdit ». Le procédé a d’abord été utilisé [7] pour éliminer les 321 exemplaires d’un codon stop UAG et le remplacer par UAA, avec succès : la souche résultante avait une croissance un peu ralentie mais un phénotype normal, avec une résistance accrue aux bactériophages. Une tentative beaucoup plus ambitieuse, visant à éliminer sept codons du génome de la bactérie (toujours remplacés par des codons synonymes) est bien avancée [8] mais n’a pas encore totalement abouti – il faut dire que dans ce cas, l’on doit modifier en tout 62 214 codons dans le génome bactérien (17 par gène, en moyenne). Son achèvement est néanmoins annoncé pour fin 2018. Pour l’essentiel, l’approche consiste à définir in silico la nouvelle séquence du génome bactérien, puis à la réaliser par synthèse d’oligonucléotides et assemblage de segments mesurant environ 50 kilobases, et enfin, à tester la viabilité de ces segments en les substituant un par un à leurs équivalents « sauvages » dans le génome bactérien. Il faut noter que ces travaux sont aussi connectés avec les efforts dont nous avons récemment parlé pour faire produire aux bactéries des protéines incluant de nouveaux acides aminés [9] (→). (→) Voir la Chronique génomique de B. Jordan, m/s n° 2, février 2018, page 179 Le dernier article faisant le point sur ce projet [8] donne une estimation du coût total d’une telle modification du génome d’E. coli, évalué à environ un million de dollars1 : chiffre à garder en mémoire lorsqu’on discutera la possibilité de passer d’E. coli à Homo sapiens… |
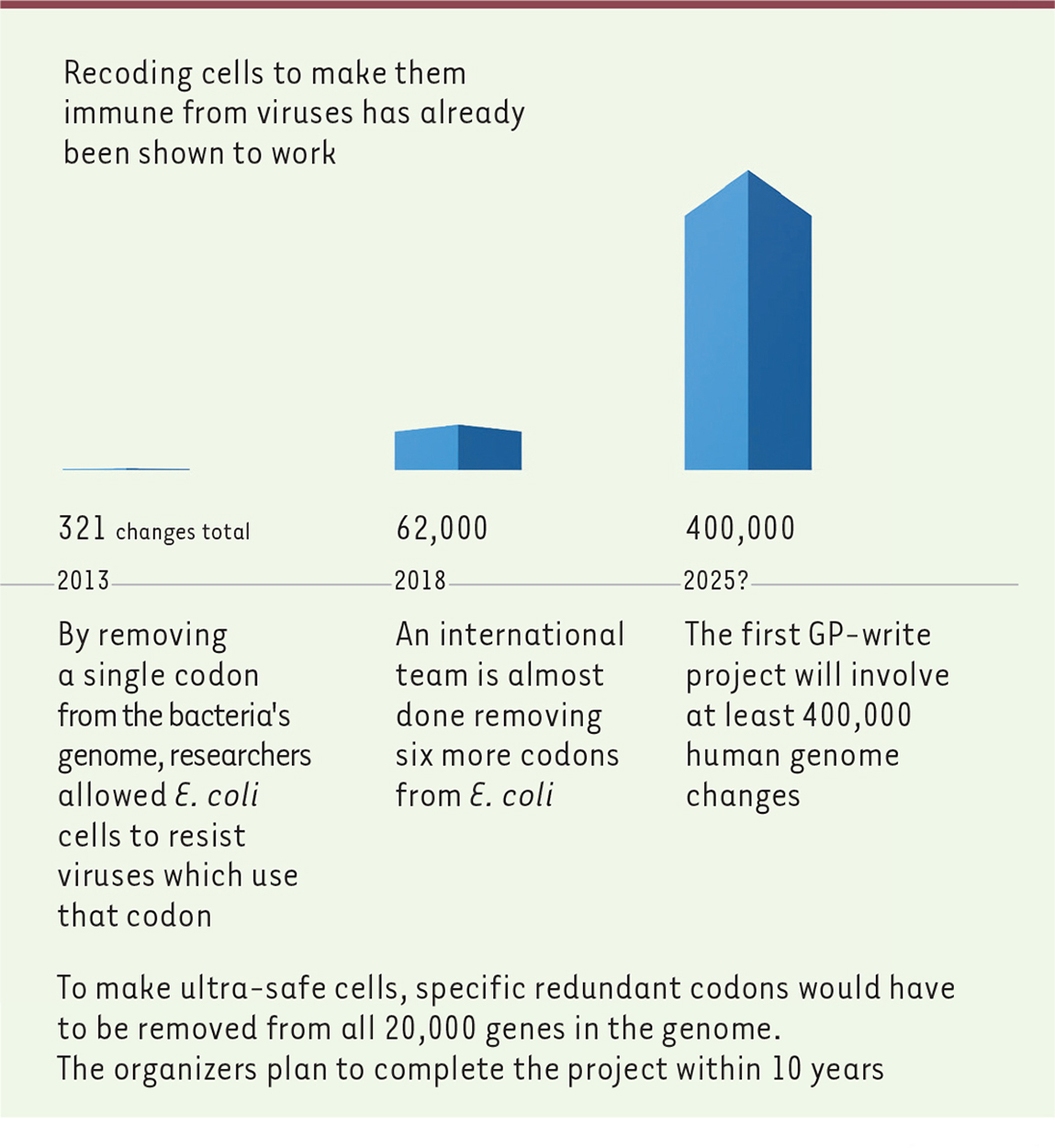
Le projet, présenté lors de la deuxième réunion du programme HGP-write [6] tenue le 1er mai à Boston, vise donc à appliquer ce procédé à une lignée de cellules humaines, dans le but d’obtenir une lignée résistante à tous les virus connus (et inconnus, tout virus devant obligatoirement faire synthétiser des protéines à sa cellule-hôte). Les proposants sont bien conscients du changement d’échelle que cela représente (Figure 1).
 | Figure 1.
Comparaison du recodage d’Escherichia coli pour un codon (à gauche), sept codons (au centre), avec le recodage du génome humain (extrait du site de HGP-write : http://engineeringbiologycenter.org/ultrasafecells/). |
Compte tenu du nombre et de la taille des gènes humains, il faut en effet prévoir environ 400 000 changements (remplacement d’un codon par un codon synonyme, grâce à une substitution de base). Il faut impérativement que tous ces changements soient effectués puisque l’on va ensuite supprimer l’ARN de transfert correspondant. Les auteurs se proposent d’ailleurs d’en profiter pour effectuer d’autres modifications, comme, par exemple, l’élimination du gène du prion, l’inactivation des éléments mobiles, la stimulation des systèmes de réparation de l’ADN… toute une série de changements visant à faire de cette lignée « recodée » un outil universel pour la biotechnologie. C’est ce qui est réalisé actuellement dans le programme « Levure » [11], et il sera intéressant de voir si cette « super-levure » est accueillie avec enthousiasme par l’industrie. Les modifications à réaliser sur l’ADN humain pourraient en théorie être effectuées par la technique CRISPR/Cas9, mais, compte tenu de leur nombre, la voie de la synthèse d’ADN apparaît plus réaliste. Ici encore, le travail bientôt abouti sur la levure [4] joue le rôle de projet-pilote et donne à penser que l’objectif n’est pas totalement chimérique : c’est un saut quantitatif de deux bons ordres de grandeur (250 fois), et les techniques génomiques ont montré à plusieurs reprises des sauts bien plus importants, que l’on pense au coût du séquençage d’ADN ou à la complexité des microarrays. Il est néanmoins clair que cela ne se fera pas en un jour, et que des financements très significatifs seront indispensables. |
Obstacles financiers et manque d’objectifs intermédiaires La question du financement est essentielle ; or, en deux ans d’existence, le projet HGP-write n’a pas réussi à déclencher des soutiens à la hauteur de la tâche. Même si son site annonce 232 millions de dollars, et si Church parle de 500 millions [5], il s’agit en fait de subventions attribuées à des laboratoires participants mais qui ne sont pas spécifiques au projet, et surtout de fonds levés par des entreprises, dont la connexion à HGP-write est parfois ténue. Or il est clair qu’il faudrait de l’ordre du milliard de dollars pour alimenter un tel programme durant les dix années prévues. Le parallèle souvent fait avec le programme Génome humain, très critiqué et parfois considéré comme irréaliste à ses débuts, fait en réalité ressortir une différence importante. Ce qui a en grande partie permis le développement du programme Génome, c’est qu’il est rapidement apparu que la construction des cartes génétiques et physiques (activité principale durant les premières années) avait des retombées immédiates et très importantes en génétique médicale et accélérait considérablement l’élucidation des maladies génétiques. Le projet trouvait là une justification évidente, même s’il avait dû – comme certains le craignaient – échouer à obtenir une séquence de bonne qualité dans un délai raisonnable. On voit mal, dans le cas des super-cellules, l’utilité d’une telle entité en cours de construction, dans laquelle une partie des substitutions seulement aurait été effectuée : seul le produit final présente un intérêt pour la science et la biotechnologie. Certes, les avancées techniques que devrait impulser l’entreprise, par exemple pour la synthèse d’ADN ou l’assemblage de mini-chromosomes, auront une utilité certaine ; mais le cœur de projet, la lignée de super-cellules, n’est intéressante et utile qu’une fois finalisée. C’est une faiblesse importante pour un projet d’une telle ampleur. |
Ces considérations conduisent à un certain scepticisme sur les chances de réalisation effective de cette nouvelle mouture du projet HGP-write. Il faut néanmoins moduler cette réserve en observant la multiplication des travaux de biologie synthétique sur l’ADN, ce qui ouvre la possibilité de synergies inattendues. Le projet-pilote sur la levure va aboutir très bientôt, et il porte sur une version de ce génome qui a été largement optimisée pour les applications industrielles [11] : s’il s’avère que c’est un succès, si cette super-levure est largement adoptée par l’industrie, cela aura un effet d’entraînement sur tout le secteur. Les travaux de recodage sur E. coli, notamment ceux qui permettent la production de protéines « exotiques » [9] (→) peuvent eux aussi avoir des retombées importantes. Et, même s’il s’agit encore de démonstrations de faisabilité, les essais de stockage d’information dans l’ADN [10] (→) (→) Voir la Chronique génomique de B. Jordan, m/s n° 2, février 2018, page 179 (→) Voir la Chronique génomique de B. Jordan, m/s n° 6-7, juin-juillet 2018, page 622 poussent, eux aussi, au progrès des techniques de synthèse et d’assemblage. Ne soyons donc pas trop pessimistes : les super-cellules deviendront peut-être un jour une réalité. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1. Boeke JD, Church G, Hessel A, et al. The genome project-write . Science. 2016; ; 353 : :126.–127. 2. Jordan B. HGP-write : après lalecture, l’écriture ? Med/Sci (Paris). 2016; ; 32 : :898.–901. 3. Richardson SM, Mitchell LA, Stracquadanio G, et al. Design of a synthetic yeast genome . Science. 2017; ; 355 : :1040.–1044. 4. Pretorius IS, Boeke JD. Yeast 2.0-connecting the dots in the construction of the world’s first functional synthetic eukaryotic genome . FEMS Yeast Res. 2018; ; 18 : :foy032.. 5. Dolgin E. Scientists downsize bold plan to make human genome from scratch . Nature. 2018; ; 557 : :16.–17 7. Lajoie MJ, Rovner AJ, Goodman DB, et al. Genomically recoded organisms expand biological functions . Science. 2013; ; 342 : :357.–360. 8. Ostrov N, Landon M, Guell M, et al. Design, synthesis, and testing toward a 57-codon genome . Science. 2016; ; 353 : :819.–822. 9. Jordan B. Bases alternatives et organismes synthétiques. Med/Sci (Paris). 2018; ; 34 : :179.–184. 10. Jordan B. L’ADN comme mémoire informatique ? Med/Sci (Paris). 2018; ; 34 : :622.–625. 11. Kannan K, Gibson DG. Yeast genome, by design . Science. 2017; ; 355 : :1024.–1025. |