| |
| Med Sci (Paris). 34(12): 1116–1122. doi: 10.1051/medsci/2018290.Balayage du génome et repérage des personnes à risque Des GWAS aux GPS Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS ; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Études cas-témoins, Maladie coronarienne, Maladie de Crohn, Analyse de mutations d'ADN, Bases de données génétiques, Prédisposition génétique à une maladie, Dépistage génétique, Étude d'association pangénomique, Séquençage nucléotidique à haut débit, Humains, Hérédité multifactorielle, Polymorphisme de nucléotide simple, Plan de recherche, épidémiologie, génétique, méthodes, tendances |

Après les premiers succès de la génétique médicale pour l’identification des gènes impliqués dans les affections héréditaires mendéliennes, la recherche des déterminants génétiques des maladies complexes a piétiné jusqu’à l’achèvement du programme Génome humain, la mise au point des puces à ADN (microarrays), et leur emploi pour des études d’association génétique balayant l’ensemble du génome (GWAS, genome-wide association studies). Les GWAS ont permis, à partir de 2005, de progresser dans la compréhension de ces affections et d’identifier, pour nombre d’entre elles, plusieurs gènes ou locus dont les variants augmentent ou diminuent le risque d’apparition de la pathologie considérée [1]. Il est néanmoins apparu progressivement que les résultats obtenus étaient incomplets : l’ensemble des locus identifiés ne rendait compte que d’une faible fraction de l’héritabilité de l’affection considérée, 10 à 20 % en général [2] (→), et chacun d’entre eux était associé à une très faible variation du risque, significative du point de vue statistique mais sans utilité clinique du point de vue de l’individu. On a alors tenté de combiner les informations sur les différents locus repérés afin d’obtenir un index de risque génétique global (multilocus genetic risk score), mais les résultats restaient insuffisants. Une étude publiée en 2010 montrait par exemple que la combinaison des 13 polymorphismes nucléotidiques (SNP, pour single nucleotide polymorphisms) alors connus pour le risque génétique de cardiopathie coronarienne permettait d’identifier les 20 % de la population présentant un risque accru d’environ 70 % - c’est scientifiquement significatif mais, comme l’indiquent les auteurs dans leur conclusion [3], « l’utilisation clinique potentielle de ce jeu de SNP reste à définir »1. (→) Voir la Chronique génomique de B. Jordan, m/s n° 5, mai 2010, page 541 |
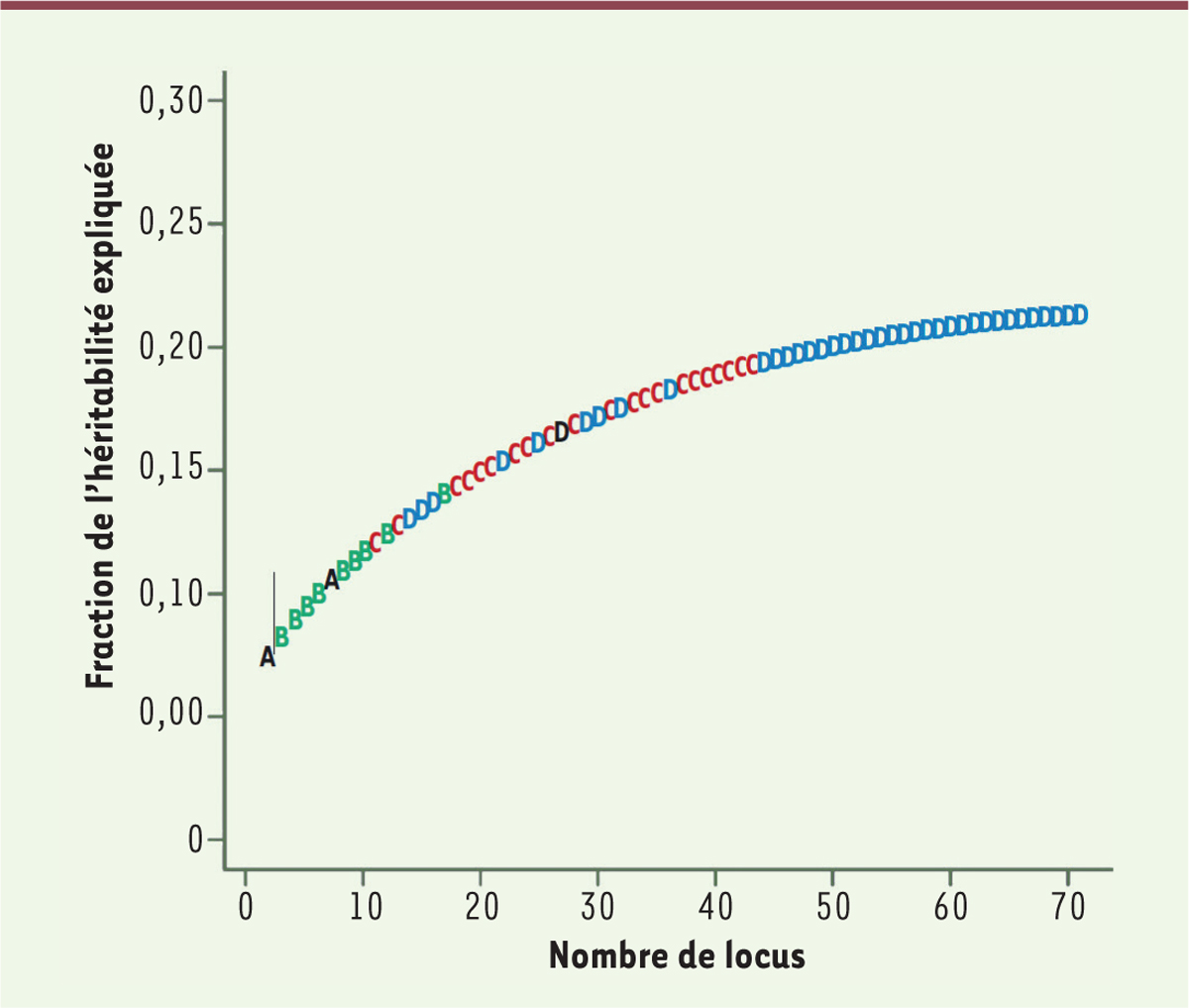
Sans que le principe des GWAS ait fondamentalement changé, leur efficacité s’est accrue grâce à plusieurs facteurs. Le plus important sans doute est l’existence de grandes bases de données librement accessibles portant sur des centaines de milliers de personnes et répertoriant pour chacune d’elles, un ensemble de données physiologiques et médicales ainsi qu’un profil génétique (établi grâce aux puces à ADN ou par séquençage). À l’heure actuelle, le meilleur exemple de ces systèmes est la UK Biobank Resource qui rassemble les données (et l’ADN) d’environ 500 000 participants, avec une caractérisation phénotypique étendue (dossier médical, imagerie de différents organes, tests sensoriels et cognitifs, style de vie et données socio-économiques, etc.) et un profil génétique établi grâce à un microarray spécialement conçu examinant plus de 800 000 SNP [4]. La deuxième avancée importante a consisté à augmenter considérablement la richesse des profils génétiques grâce à des techniques d’« imputation », qui permettent de passer des 800 000 variants directement examinés par la puce à ADN à plus de 92 millions de ces SNP. Il s’agit là d’utiliser l’existence du déséquilibre de liaison (l’association préférentielle de certains allèles pour des SNP proches dans le génome) pour transposer sur les ADN génotypés de la banque les données bien plus détaillées obtenues par le séquençage intégral dans le cadre du projet britannique UK 1000 genomes [5]. Cette extrapolation (ou plutôt interpolation) peut sembler hardie, mais elle a été effectuée systématiquement dans le cadre de la UK Biobank et a fait l’objet de plusieurs contrôles qui montrent son bien-fondé [4]. C’est évidemment un avantage considérable de disposer maintenant de profils comportant près de cent millions de SNP ! La dernière avancée, enfin, porte sur les techniques d’analyse des données : plus sophistiquées du point de vue statistique, elles ne sont maintenant plus centrées sur la mise en évidence de quelques SNP supplémentaires et de moins en moins significatifs (Figure 1), mais sur la définition d’« index polygéniques » (Genome-Wide Polygenic Scores, ou GPS - le choix de l’acronyme n’est pas innocent…) susceptibles de fournir une prédiction de risque qui soit significative [6] (→). (→) Voir la Chronique génomique de B. Jordan, m/s n° 3, mars 2011, page 323  | Figure 1.GWAS « classique », cas de la maladie de Crohn. Les locus successivement découverts sont figurés, dans l’ordre chronologique, par les lettres A (noir), B (vert), C (rouge), D (bleu). On voit qu’au fil du temps les locus nouvellement découverts expliquent une part de plus en plus faible de l’héritabilité, et que celle-ci semble plafonner aux alentours de 25 % (figure reprise de [ 6]). |
Pour l’essentiel, ces index polygéniques sont obtenus en faisant la somme des « allèles de risque » détectés dans le profil génétique d’une personne, chacun des termes étant affecté d’un coefficient qui reflète l’intensité du risque qui lui est associé. La mise au point de l’index (du « prédicteur ») consiste à choisir les locus qui en feront partie et à ajuster les coefficients qui leur sont associés. |
L’exemple d’un GPS pour la maladie coronarienne L’article qui fait l’objet de cette chronique [7] émane de plusieurs équipes états-uniennes et est fondé sur l’exploitation des données de la UK Biobank. L’objectif des auteurs était de voir s’il était possible de définir un index polygénique (ou GPS, j’utiliserai cet acronyme par la suite), dont l’impact clinique puisse être comparable à celui de mutations rares déjà connues. Pour la maladie coronarienne, il existe des mutations rares présentes dans 0,4 % de la population (hypercholestérolémie familiale), dont la présence à l’état hétérozygote multiplie par trois le risque de maladie coronarienne [8], et constitue une indication forte pour un traitement visant à réduire le taux de cholestérol. Il s’agit donc d’examiner si un GPS peut atteindre une spécificité équivalente. Les auteurs ont commencé par utiliser les données d’une méta-étude GWAS déjà publiée [9] (60 000 cas et 123 000 témoins au total) pour en déduire une trentaine de « prédicteurs », index polygéniques censés évaluer le risque génétique de maladie coronarienne. Ces prédicteurs sont ensuite appliqués sur un premier jeu de 120 000 membres de la UK Biobank (parmi lesquels la prévalence de maladie coronarienne est de 3,4 %) afin de choisir le plus performant d’entre eux (celui qui différencie le mieux les malades des témoins). Ce prédicteur est alors testé sur un jeu indépendant de 290 000 membres (prévalence 3 %) et s’avère avoir sur cet échantillon des performances équivalentes. On a ainsi défini un GPS (index polygénique) qui indique, pour chaque personne, un index de risque génétique de maladie coronarienne. On peut alors relever, pour chaque valeur de l’index exprimée sur une échelle de 1 à 100, la prévalence de maladie coronarienne dans la population correspondante, et l’on obtient la courbe de la Figure 2.
 | Figure 2.Prévalence de maladie coronarienne (ordonnée, en pour cent) en fonction de la valeur du GPS (index polygénique) (abscisse, échelle arbitraire de 0 à 100). Pour mémoire, la valeur moyenne de la prévalence dans l’ensemble de l’échantillon est de 3 %. (Vue partielle modifiée de la Figure 2 de [ 7]). |
Insistons sur le fait que dans cette figure, il s’agit bien de données expérimentales : par exemple, chez le groupe de personnes pour lesquelles le GPS a une valeur de 65, la prévalence relevée pour la maladie coronarienne est d’environ 3 %. On voit immédiatement sur ce graphique que les GPS élevés ont une valeur prédictive (et donc clinique) évidente : pour le dernier point à droite du graphique, la prévalence est de l’ordre de 11 %, soit près de quatre fois la valeur moyenne dans la population étudiée… Reste à savoir quel est l’effectif de ces groupes à haut risque, ce que la Figure 2 n’indique pas - mais les données sont disponibles puisqu’il suffit de compter le nombre de personnes dont la valeur du GPS tombe dans un intervalle donné. Les résultats sont éloquents : 8 % des personnes présentent un risque supérieur à 3 fois le risque moyen (risque relatif > 3). En d’autres termes, le GPS identifie 20 fois plus d’individus à haut risque (et donc justiciables d’un traitement par des statines) que la recherche de mutations d’hypercholestérolémie familiale (dont la fréquence déjà mentionnée est de 0,4 %). Si l’on met la barre plus haut, 2,3 % des personnes ont un risque relatif supérieur à 4, et 0,5 % un risque relatif supérieur à 5. Au total, il apparaît donc que ce GPS dérivé d’un GWAS « nouveau genre » est bel et bien capable de détecter des personnes présentant un risque cliniquement significatif de maladie coronarienne - et l’article présente des résultats très similaires pour la fibrillation atriale, le diabète de type 2, les maladies inflammatoires de l’intestin (ou MICI, dont fait partie la maladie de Crohn) et le cancer du sein. Bien que la question de l’« héritabilité manquante » ne soit pas discutée dans cette publication [7], on peut considérer que la mise au point des GPS contourne ce problème et montre que des prédictions fortes peuvent être obtenues grâce à la prise en compte d’un grand nombre de locus dans l’élaboration de ces index polygéniques. |
Un premier chiffre est susceptible de doucher l’enthousiasme que l’on peut ressentir à la vue de ces résultats : le GPS défini par Khera et al. [7] inclut un total de 6 630 150 SNP. Vous avez bien lu, plus de six millions de SNP, bien plus que n’en peut analyser un microarray de génotypage, généralement limité à un million de marqueurs environ. Faut-il pour autant séquencer intégralement l’ADN d’une personne pour évaluer son risque génétique à l’aide d’un GPS ? Il semble que non - bien que ce point ne soit guère développé dans les articles cités. Il est apparemment possible d’utiliser, là aussi, les techniques d’imputation, pour autant que l’on reste au sein du même type de population (afin que les données de déséquilibre de liaison restent valables). C’est d’ailleurs une limite générale à ces approches : elles supposent que les populations de référence (celle du UK 1000 genomes, de la UK Biobank) et celle que l’on analyse soient comparables - ce qui, en pratique, limite l’approche aux personnes d’origine européenne. L’extension à d’autres groupes humains a commencé, mais les données sont encore limitées. Par ailleurs, il est très vraisemblable que le jeu de six millions de SNP puisse être considérablement réduit sans perdre beaucoup d’informativité : Sekar Kathiresan, l’auteur senior de l’article étudié [7], considère que 6 000 SNP bien choisis pourraient suffire, et, dans une interview récente, prévoit une mise sur le marché de ce type de test dès 20192. En admettant que l’approche par les GPS confirme sa validité, ce qui est très probable (d’autant plus que l’obtention de profils génétiques, y compris par séquençage intégral, est en train de se généraliser), on peut penser qu’elle va soulever des questions éthiques. Le problème posé par leur restriction à une population d’origine européenne n’est probablement que transitoire. Par contre, cette transformation des GWAS en fournisseur d’outils de prédiction efficaces (les GPS) va de nouveau stimuler la tendance au déterminisme génétique, à oublier que le rôle de l’environnement et de l’histoire personnelle est généralement aussi, sinon plus, important que la configuration des gènes pour induire le destin d’un individu. On a déjà vu paraître un index polygénique concernant le succès scolaire (educational attainment) qui serait lié à un jeu de 1 271 SNP… [10] Certes, cet index ne rend compte que de 10 % de la variabilité au sein de la population, mais on imagine les interprétations auxquelles il pourrait donner lieu. Notons à cet égard que la mise en valeur de ces derniers résultats dans une revue de premier plan (Nature Genetics) contraste avec le peu d’écho d’une étude du National Bureau of Economic Research qui étudie l’interaction entre un tel index polygénique et les conditions socioéconomiques et conclut que « la pauvreté dans l’enfance limite le succès scolaire d’individus à haut potentiel »3 [11]. C’est une évidence que l’on a parfois tendance à perdre de vue. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Wellcome Trust Case Control Consortium. Genome-wide association study of CNVs in 16,000 cases of eight common diseases and 3,000 shared controls . Nature. 2010; ; 464 : :713.–720. 2.
Jordan B.. À la recherche de l’héritabilité perdue . Med Sci (Paris). 2010; ; 26 : :541.–543. 3.
Ripatti S, Tikkanen E, Orho-Melander M, et al. A multilocus genetic risk score for coronary heart disease: case-control and prospective cohort analyses . Lancet. 2010; ; 376 : :1393.–1400. 4.
Bycroft C, Freeman C, Petkova D, et al. The UK Biobank resource with deep phenotyping and genomic data . Nature. 2018; ; 562 : :203.–209. 5. The 1000 Genomes Project Consortium. A global reference for human genetic variation . Nature. 2015;; 526 : :68.–74. 6.
Jordan B.. Maladie de Crohn et GWAS, d’analyses en méta-analyses . Med Sci (Paris). 2011; ; 27 : :323.–325. 7.
Khera AV, Chaffin M, Aragam KG, et al. Genome-wide polygenic scores for common diseases identify individuals with risk equivalent to monogenic mutations . Nat Genet. 2018; ; 50 : :1219.–1224. 8.
Abul-Husn NS, Manickam K, Jones LK, et al. Genetic identification of familial hypercholesterolemia within a single U.S. health care system . Science. 2016; :354.. pii: aaf7000.. 9.
Nikpay M, Goel A, Won HH, et al. A comprehensive 1,000 Genomes-based genome-wide association meta-analysis of coronary artery disease . Nat Genet. 2015; ; 47 : :1121.–1130. 10.
Lee JJ, Wedow R, Okbay A, et al. Gene discovery and polygenic prediction from a genome-wide association study of educational attainment in 1.1 million individuals . Nat Genet. 2018; ; 50 : :1112.–1121. 11.
Papageorge NW, Thom K. Genes, education, and labor market outcomes: evidence from the health and retirement study . National Bureau of Economic Research Working Paper No. 25114. September. 2018 http://www.nber.org/papers/w25114. |