| dc.contributor.author | Des Georges, M | fr_FR |
| dc.contributor.author | Guittard, C | fr_FR |
| dc.contributor.author | Claustres, M | fr_FR |
| dc.date.accessioned | 2012-08-23T07:50:07Z | |
| dc.date.available | 2012-08-23T07:50:07Z | |
| dc.date.issued | 1998 | fr_FR |
| dc.identifier.citation | Des Georges, M ; Guittard, C ; Claustres, M, Les bases moléculaires de la mucoviscidose en France : plus de 300 mutations et 506 génotypes différents sont en cause., Med Sci (Paris), 1998, Vol. 14, N° 12; p.1413-21 | fr_FR |
| dc.identifier.issn | 1958-5381 | fr_FR |
| dc.identifier.uri | http://hdl.handle.net/10608/984 | |
| dc.description.abstract | Nous avons collecté auprès de 19 laboratoires impliqués
dans le diagnostic moléculaire de la mucoviscidose en
France les données alléliques et/ou génotypiques de près
de 3 595 patients. L’analyse de 7 190 allèles révèle l’existence
de 300 mutations différentes parmi les 6 700 allèles
mutés identifiés (taux global de détection = 93,18 %), totalisant
près de 40% des 770 mutations CFTR (cystic fibrosis
transmembrane conductance regulator) répertoriées à ce jour
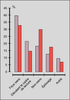
dans le monde. La délétion ΔF508 est présente sur
4 833 allèles (67,21 %), avec un gradient décroissant entre le
Nord (70 %) et le Sud (61 %) du pays, et une fréquence
spécifique de 81% dans la population celtique de Bretagne.
Viennent ensuite G542X (2,89 % ; 1-6,6 %), N1303K
(2,05%; 0,75-4,5 %), 1717 – 1G > A (1,33%; 0-2,7 %), et
2789 + 5G > A (1%; 0-2,8 %). Seules 9 autres mutations ont
une fréquence supérieure à 0,4 % (G551D, W1282X,
R553X, ΔI507, 2183AA > G, 1078delT, 711 + 1G > T,
R1162X, et Y1092X). 133 mutations ont été identifiées sur
un faible nombre de chromosomes (de 28 à 2), et 153 sont
présentes en un seul exemplaire. Ce spectre de mutations
CFTR, le plus large rapporté jusqu’ici pour un seul pays, est
à l’origine de 506 génotypes délétères différents. Cette
étude souligne l’hétérogénéité allélique de la mucoviscidose
en France et met en évidence des disparités régionales et
ethniques dans la distribution de certaines mutations. Ces
informations seront utiles pour améliorer la détection des
mutations et la précision du conseil génétique, ainsi que
pour orienter de futures stratégies thérapeutiques. | fr |
| dc.description.abstract | We have collated the results of cystic fibrosis (CF) mutation analysis conducted in 19 laboratories in France. 7190 CF allelcs have been analyzed, demonstrating a total of 300 different mutations so far observed and accounting for 93.18% of CF genes in patients living in France. This mutational spectrum represent 40% of the 770 mutations described worldwide. DeltaF508 is the most common at 67.21% of CF mutations (range 61-70 between Northern and Southern regions) including a unique frequency of 81% in the Celtic Brittany, followed by G542X (2.89%; range 1-6.6%), N1303K (2.05%; range 0.75-4.5%), 1717-1G > A (1.33 %; range 0-2.7%), and 2789 + 5G > A (1%; range 0-2.8%). Only 9 mutations had relative frequency > 0.4% (G551D, W1282X, R553X, Delta1507, 2183AA > G, 1078delT, 711 + 1G > T, R1162X and Y1092X. 133 mutations were found on a small number of CF alleles (from 28 to 2), and 153 were unique. There was a clear geographical and/or ethnic variation in the distribution of several mutations. Overall, at least 506 different genotypes were found responsible for CF. These data demonstrate a high allelic heterogeneity of CF in France and illustrate that the mutations present within a particular population need to be defined in order to provide meaningful carrier screening and testing for rare mutations in affected individuals. [References: 17] | en |
| dc.language.iso | fr | fr_FR |
| dc.publisher | Masson Périodiques, Paris | fr_FR |
| dc.rights | Article en libre accès | fr |
| dc.rights | Médecine/Sciences - Inserm - SRMS | fr |
| dc.source | M/S. Médecine sciences [revue papier, ISSN : 0767-0974], 1998, Vol. 14, N° 12; p.1413-21 | fr_FR |
| dc.title | Les bases moléculaires de la mucoviscidose en France : plus de 300 mutations et 506 génotypes différents sont en cause. | fr |
| dc.title.alternative | Molecular basis of cystic fibrosis in France: more than 300 different mutations and 506 genotypes are involved | fr_FR |
| dc.type | Article | fr_FR |
| dc.contributor.affiliation | Laboratoire de genetique moleculaire, IGH-Cnrs UPR 1142 et CHU, Institut de biologie, boulevard Henri-IV, 34060 Montpellier, France; Laboratoire de biochimie pediatrique, Hopital Debrousse, 29, rue Soeur-Bouvier, 69322 Lyon, France; Centre de biogenetique, ETSBO, 46, rue Felix-le-Dante, BP 454, 29275 Brest, France; Laboratoire de biochimie, Hopital Henri-Mondor, 51, avenue du Marechal-de-Lattre-de-Tassigny, 94010 Creteil, France; Laboratoire de biochimie et genetique moleculaire, Pavillon Cassini, Groupe hospitalier Cochin, 123, boulevard de Port-Royal, 75014 Paris, France; Laboratoire de biochimie et de biologie moleculaire, Hopital Trousseau, 24-26, rue Docteur-Arnold-Netter, 75012 Paris, France; Laboratoire de genetique medicale, Hopital Purpan, Pavillon Lefebvre, place du Docteur-Baylac, 35033 Rennes, France; Laboratoire de genetique moleculaire, CHU Pontchaillou, 2, rue Henri-le-Guillon, 35033 Rennes, France; Conformations cellulaires et moleculaires, Inserm U.314, CHR Maison-Blanche, 45, rue Cognacq-Jay, 51092 Reims, France; Laboratoire de biochimie, biologie moleculaire et enzymologie, Faculte de medecine, 28, place Henry-Dunant, 63001 Clermont-Ferrand, France; Service de genetique, CHU d'Angers, 4, rue Larrey, 49043 Angers, France; Biochimie de l'ADN, Hopital de la Tronche, BP 217, 38043 Grenoble, France; Biologie moleculaire appliquee, Centre de diagnostic prenatal, Hopital de la Timone, rue Saint-Pierre, 13383 Marseille, France; Laboratoire de Biochimie B, CHU George-Clemenceau, 14033 Caen, France; Biochimie B, Tour Lavoisier, Hopital Necker-Enfants Malades, 149, | - |
| dc.identifier.doi | 10.4267/10608/984 | |