| |
| Med Sci (Paris). 35(3): 245–251. doi: 10.1051/medsci/2019030.Pourquoi le taux de mutation n’est-il jamais égal à
zéro ? Didier Casane,1,2 Maxime Policarpo,1,2 and Patrick Laurenti2,3* 1Laboratoire Évolution, Génomes, Comportement, Écologie, UMR9191,
CNRS, IRD, Univ Paris-Sud, Université Paris-Saclay,
91198Gif-sur-Yvette,
France 2Université Paris-Diderot, UFR des sciences du vivant, Sorbonne
Paris Cité, Paris, France 3Laboratoire Interdisciplinaire des Énergies de Demain, UMR 8236
CNRS, Bâtiment Lamarck, 35, rue Hélène Brion75013Paris,
France |
« Why does the mutation rate not become reduced to zero? No answer seems
possible at present, other than the surmise that the nature of genes does
not permit such a reduction. In short, mutations are accidents, and
accidents will happen ».
Alfred Henry Sturtevant (1937)
« There seems to be enough evidence to show that mutation rates are under
genetic control. Thus, the mutation rate characteristic of each species must
be a product of past evolution like any other morphological or physiological
character ».
Motoo Kimura (1967)
« A potentially revealing hypothesis for mutation-rate evolution is that
natural selection primarily operates to improve replication fidelity, with
the ultimate limits to what can be achieved set by the power of random
genetic drift. This drift-barrier hypothesis is consistent with comparative
measures of mutation rates, provides a simple explanation for the existence
of error-prone polymerases and yields a formal counter-argument to the view
that selection fine-tunes gene-specific mutation rates ».
Michael Lynch (2016)
En 1937, quand Alfred Henry Sturtevant1 pose cette
question simple et pourtant extraordinairement importante : « Pourquoi le taux de
mutation n’atteint-il jamais zéro ? » [1], la génétique, en particulier grâce à l’utilisation intensive des
drosophiles comme modèle d’étude, a fait des progrès considérables depuis sa fondation
quelques décennies auparavant. Quelques faits sont déjà bien établis : des individus
mutants apparaissent de façon récurrente dans les populations, comme des mouches aux
yeux blancs dans une population de mouches avec les yeux rouges ; ces individus mutants
sont les descendants de parents porteurs de modifications (des mutations) dans des
cellules de leur lignée germinale ; enfin, la plupart des mutations qui modifient le
phénotype ont des effets délétères car ces mutants ont souvent une viabilité et/ou une
fécondité réduite, voire nulle (si la mutation est létale ou rend le porteur stérile).
Il est par ailleurs établi que le nombre de mutations qui se produisent par génération,
ou taux de mutation, est variable dans les différentes lignées de drosophiles [2-4]. Le taux de mutation étant un caractère
héréditaire et variable, il peut donc évoluer. Dans ce cadre, un taux de mutation réduit
représente un avantage qui devrait conduire à la sélection des individus
antimutateurs. En effet, les individus qui produisent le moins de
descendants mutants produiront plus de descendants viables et fertiles à la génération
suivante. Notons que la sélection ne se fait pas sur le phénotype de l’individu mutateur
mais sur le phénotype de ses descendants, une forme de sélection appelée parfois
sélection de second ordre. Progressivement, la sélection récurrente des individus les
moins mutateurs, qui apparaissent au cours du temps, devrait faire tendre le taux de
mutation vers zéro. Comme l’existence des mutations, à l’origine de la diversité
génétique, est la condition sine qua non de l’évolution des espèces,
l’évolution devrait donc, en théorie, s’arrêter faute de matière première ; or ce n’est
manifestement pas le cas. Sturtevant en conclut que la nature des gènes, inconnue en
1937, « ne permet pas une telle réduction » et que « les mutations sont des accidents et
que les accidents finissent toujours par survenir » [1]. Pour mieux comprendre le contrôle du flux de mutations et son évolution
au cours du temps, il faudra attendre le dernier tiers du XXe siècle. En
effet, jusque-là, les mutations n’étaient qu’indirectement observables par le biais de
leurs effets phénotypiques associés : modifications de couleur, de forme, de viabilité,
de fertilité. Les mutations n’étaient donc définies, au mieux, que par une association
entre une position sur un chromosome et un phénotype. Avec la découverte de méthodes de
séquençage de l’ADN, il devient possible d’observer des différences entre génomes
indépendamment de tout effet observable sur les organismes. L’accroissement récent des
capacités de séquençage rend possible la quantification des différences à l’échelle des
génomes entiers. Toutefois, si les mutations à petite échelle, comme le remplacement
d’un nucléotide par un autre, une insertion ou une délétion de quelques nucléotides,
sont facilement identifiables, les remaniements à grande échelle restent difficiles à
étudier. C’est pour cela que les analyses du taux de mutation et de son évolution sont
aujourd’hui essentiellement fondées sur l’identification de mutations ponctuelles
(changement d’un nucléotide en un autre ou polymorphisme nucléotidique – SNP en
anglais). Dans la suite de cet article, nous utiliserons le mot mutation dans le sens
étroit de mutation ponctuelle car la plupart des travaux sont focalisés sur ce type de
mutations, mais cela ne devrait pas modifier significativement la validité et la portée
générale des mécanismes évolutifs mis en évidence. Le nombre de mutations qui se produisent par unité de temps dépend en particulier des
complexes protéiques impliqués dans la réplication et la réparation de l’ADN. Des
mutations peuvent se produire dans les gènes qui codent ces protéines, il peut donc
exister une variabilité génétique interindividuelle des mécanismes de réplication et de
réparation de l’ADN, c’est-à-dire une variation interindividuelle du taux de mutation.
Dans les cas les plus extrêmes, des mutations dans les gènes de réparation de l’ADN sont
responsables de maladies génétiques extrêmement graves, comme le xeroderma
pigmentosum
2. Ces mutations ne sont pas maintenues au cours
des générations car leurs porteurs ont une espérance de vie et des capacités de
reproduction réduites. En théorie, à l’échelle de l’évolution, seules des mutations de
ces gènes avec des effets plus réduits sur le taux de mutation des autres gènes
devraient se maintenir dans les populations, mais nous verrons plus loin qu’il peut
exister des exceptions à cette règle. Les taux de mutation mesurés chez des organismes aussi divers que des virus à ARN, des
virus à ADN, des bactéries, des eucaryotes unicellulaires et pluricellulaires sont
relativement bas, mais ne sont jamais égaux à zéro et varient dans une gamme extrêmement
étendue : de 10-3 à 10-11 mutations par site par réplication
[5-7]. En d’autres termes, la probabilité qu’un
nucléotide soit remplacé par un autre peut être proche de 1 sur mille chez des virus à
ARN [7], mais être proche de 1 sur cent milliards
chez des paramécies [8]. Chez
l’homme, le taux de mutation par génération est d’environ 10-8, soit moins de
10-10 par réplication [9]. Les mutations qui n’ont pas d’effets sur le nombre de descendants sont invisibles du
point de vue de la sélection naturelle : ce sont des mutations neutres. Les effets des
mutations ponctuelles ont été caractérisés à de multiples reprises chez des organismes
aussi différents que des virus et l’homme, en passant par des bactéries et des levures
[10]. Toutes ces analyses
montrent que la plupart des mutations qui apparaissent sont neutres, qu’une petite
fraction est délétère et que les mutations avantageuses sont extrêmement rares. Très
récemment, cette observation classique a pu être analysée très finement grâce à une
technologie très sophistiquée qui permet l’analyse d’un très grand nombre de mutants
chez la bactérie Escherichia coli [11]. Les mutations qui modifient le phénotype ont
un effet le plus souvent légèrement délétère, beaucoup moins souvent franchement
délétère, rarement létal et quasi jamais avantageux. Cette observation, qui peut
paraître contre-intuitive, se comprend facilement si l’on considère que les systèmes
génétiques sont les résultats de processus d’optimisation qui se sont déroulés pendant
des millions, voire parfois des milliards d’années, et qu’il est donc aujourd’hui
beaucoup plus facile de les détériorer que de les améliorer [12] (→).
(→) Voir le Forum de D. Casane et P. Laurenti, m/s n° 12,
décembre 2014, page 1177
En 1967, Motoo Kimura, un des principaux théoriciens de la biologie évolutive, suggère
qu’il doit y avoir une forte sélection contre les mutations qui augmentent le taux de
mutation. Par ailleurs, il doit aussi exister une sélection contre un taux de mutation
trop bas, car dans un environnement à long terme instable, il est nécessaire qu’il
existe une diversité génétique (polymorphisme génétique) pour que l’adaptation puisse se
réaliser. Sans ce polymorphisme, l’extinction devient inévitable. La baisse du taux de
mutation à petite échelle évolutive impliquerait une sélection interindividuelle
(intragroupe), alors que son maintien serait favorisé à long terme par une sélection
intergroupe (entre populations ou espèces), de façon analogue aux mécanismes qu’on
suppose à l’œuvre pour le maintien de la reproduction sexuée qui présente aussi un
désavantage à court terme à l’échelle des individus, mais un avantage à long terme à
l’échelle des populations [12]. Le taux de
mutation observé correspondrait à un état d’équilibre entre deux forces de sélection qui
agissent à des niveaux d’organisation différents. Le point d’équilibre dépend de
l’environnement dans lequel vit une espèce. Si cet environnement est très stable, la
pression pour maintenir de la variabilité sera faible et le taux de mutation tendra vers
une valeur très basse sous l’effet de la sélection interindividuelle, alors que pour une
espèce vivant dans un environnement très instable, un taux de mutation élevé sera
maintenu. Si Kimura ne doute pas de l’existence de la sélection contre les allèles
mutateurs qui augmente le taux de mutation des génomes, il doute fortement de
l’existence d’une sélection contre les allèles antimutateurs qui mène vers un taux de
mutation trop bas. Il propose donc une autre explication aux taux de mutation observés :
ils correspondent aux taux de mutation les plus bas possibles, en tenant compte du fait
qu’il y a peut-être une limite biochimique à l’amélioration des systèmes de réplication
et de réparation qui ne peut être franchie. On peut aussi supposer un coût, en terme
énergétique par exemple, de la réduction du taux de mutation et que ce coût peut devenir
trop important pour pouvoir être supporté par un organisme. Au début des années 1970, il
n’était pas possible de tester toutes ces hypothèses. Depuis une dizaine d’années, Michael Lynch3, propose
de tenir compte d’un paramètre négligé jusqu’alors : la taille finie des populations
d’organismes vivants. En effet, les fréquences des allèles varient au cours du temps du
fait de leur valeur sélective, mais aussi du seul fait du hasard de l’échantillonnage
des gamètes à l’origine des individus qui constituent une nouvelle génération. Par
exemple, un allèle peut être présent avec une fréquence de 0,5 à une génération donnée,
puis être présent à une fréquence de 0,6 à la génération suivante, du seul fait de ce
processus d’échantillonnage. Cette variation aléatoire des fréquences, qu’on appelle la
dérive génétique, sera d’autant plus grande que la population est de petite taille. Dans
une très petite population, un allèle peut ainsi se fixer ou disparaître en quelques
générations sans qu’il ait un effet sur la fitness4. Dans les grandes populations, ces fluctuations stochastiques
sont de faibles amplitudes et le polymorphisme génétique peut se maintenir pendant un
grand nombre de générations. Les généticiens s’intéressent ainsi à ce qu’ils appellent
l’effectif efficace de la population (Ne) plutôt qu’au nombre d’individus qui
la constituent. Ne peut être considéré, en première approximation, comme le
nombre d’individus qui se reproduisent à chaque génération. L’effectif efficace est donc
souvent beaucoup plus petit que l’effectif réel d’individus. Une forte variance du
nombre de descendants parmi les individus qui se reproduisent et de fortes fluctuations
de la taille de la population au cours du temps réduisent aussi beaucoup la valeur de
Ne. Pour fixer les idées, on estime à 10 000 l’ordre de grandeur de
l’effectif efficace à long terme de l’espèce humaine dans son ensemble. L’effectif
efficace est un paramètre difficile à estimer, et on utilise souvent le polymorphisme
génétique neutre pour l’évaluer. En effet, les grandes variations de fréquences
alléliques dans les petites populations conduisent à une élimination plus rapide du
polymorphisme. En conséquence, s’il y a peu de polymorphisme, on estimera que
Ne est petit. À l’inverse, s’il y a beaucoup de polymorphisme, on
estimera Ne grand. Pour un allèle soumis à sélection, s’il y a peu de dérive
génétique (Ne grand), il se fixera (sa fréquence atteindra 1) presque
sûrement s’il est avantageux, et il sera presque sûrement éliminé s’il est délétère.
Mais ce même allèle peut se fixer alors qu’il est délétère ou être perdu alors qu’il est
avantageux, s’il y a de fortes fluctuations stochastiques des fréquences alléliques
(Ne petit) qui perturbent les effets de la sélection directionnelle. En résumé, les effets de la sélection naturelle peuvent être réduits à néant par la
dérive génétique d’autant plus que cette dernière est forte et que l’avantage (ou
désavantage) sélectif est faible (pour une explication plus détaillée voir [13], et une analogie
footballistique présentée dans l’Encadré « le football évolutionnaire
»). La dérive génétique est donc une barrière à la sélection naturelle quand cette
dernière est de faible ampleur. Lynch propose que c’est cette barrière qui s’oppose à la
réduction continue vers zéro du taux de mutation et il rejette l’hypothèse que le taux
de mutation soit le résultat de la sélection pour une valeur optimale. Il propose
également que les espèces qui ont le taux de mutation le plus faible sont celles qui ont
les effectifs efficaces les plus grands et donc chez lesquelles la sélection pour un
taux de mutation bas est la plus efficace [14]. Nous allons maintenant examiner un ensemble de résultats récents et très divers qui
permettent de tester quelques hypothèses et des attendus associés à ces différents
modèles pour expliquer l’existence des mutations et l’extrême variabilité de leur
fréquence d’apparition chez différents organismes |
Le taux de mutation a-t-il atteint la plus basse valeur possible ? Question à laquelle il est très difficile de répondre pour les organismes
multicellulaires, mais qui est plus facilement testable chez des organismes
unicellulaires en faisant des expériences de mutagenèse pour générer des allèles
antimutateurs qui réduisent le taux de mutation. C’est ainsi qu’il est possible
d’abaisser le taux de mutation chez la bactérie E. coli pourtant à
l’origine déjà très bas (~10-10) [15, 16]. Bien qu’il soit souhaitable de tester d’autres espèces, ce résultat
suggère néanmoins que le taux de mutation observé dans les populations naturelles
n’est pas le plus bas possible. |
Le coût de la fidélité limite-t-il la baisse du taux de mutation ? Question à nouveau difficile à aborder expérimentalement, mais qui a pu être testée
chez un virus à ARN [17].
Les virus à ARN sont connus pour avoir un taux de mutation extrêmement élevé, mais
ce n’est pas toujours le cas [17]. Dans le
cadre du modèle d’équilibre entre le fardeau de mutations (les nouvelles mutations
délétères à éliminer) et la nécessité de générer des mutations adaptatives, un taux
de mutation élevé est supposé être la conséquence d’un besoin d’adaptation fréquent
[18]. Ainsi, le taux
très élevé de mutation d’un virus comme le VIH (virus de l’immunodéficience
humaine), le menant à la limite du fardeau de mutation supportable, serait
nécessaire à son adaptation rapide et récurrente au système immunitaire de son hôte
et aux traitements médicaux [17]. L’équipe de
Furio et al. [17] montre que
l’augmentation de la fidélité de la réplication par mutation de l’ARN polymérase se
fait au prix d’un ralentissement du taux de réplication, induisant une réduction de
la valeur sélective. Les lignées les plus mutatrices ne présentent pas une meilleure
adaptabilité. Les auteurs en concluent que le taux de mutation est un équilibre
résultant de deux forces sélectives opposées, le fardeau de mutations favorisant les
taux de mutation faibles et la vitesse de réplication favorisant les taux de
mutation élevés. |
Le football évolutionnaire Dans une popula tion, les gènes existent sous différentes formes qu’on
appelle allèles. Si les individus porteurs d’un allèle donné produisent, en
moyenne, plus de descendants viables et fertiles que les porteurs d’autres
allèles, cet allèle aura une meilleure valeur adaptative (ou meilleure
fitness). En toute logique, un tel allèle finira
toujours par se fixer, c’est-à-dire qu’il sera le seul à se maintenir dans
la population, tandis que les autres allèles disparaitront. Pourtant, dans
les populations naturelles, ce destin n’est pas écrit, en particulier à
cause de la dérive génétique. Risquons une analogie footballistique pour
comprendre comment la dérive génétique, un processus stochastique, perturbe
un processus déterministe comme la sélection naturelle. Si un joueur pose un
ballon sur le point central d’un terrain de foot et qu’il le pousse d’un
mètre dans la direction du but adverse, et cela de façon répétée, le ballon
finira inévitablement sa course dans ce but. Supposons maintenant qu’avant
de pouvoir pousser à nouveau le ballon dans la bonne direction, un joueur
adverse contre le ballon et l’envoie dans n’importe quelle direction
(imaginons que le défenseur arrive parfois par les côtés, parfois par
derrière et parfois en face de l’attaquant). Si le contre ne pousse ainsi le
ballon que d’un centimètre avant qu’il ne soit poussé à nouveau d’un mètre
dans la bonne direction, le ballon finira tout de même sa course, et
rapidement, dans le but adverse. Mais, si le contre envoie le ballon dans
une direction quelconque et à une distance de quelques mètres avant qu’il ne
soit poussé à nouveau d’un mètre dans la bonne direction, il y aura alors de
fortes chances que ce ballon sorte du terrain sans atteindre le but de
l’adversaire. Il est même possible qu’il finisse sa course dans le but du
joueur attaquant ! Taper au hasard dans le ballon correspond à la dérive génétique, c’està- dire
aux variations aléatoires des fréquences alléliques entre deux générations.
Le déplacement directionnel correspond à la sélection. Si c’est un petit
déplacement aléatoire qui est réalisé par rapport au déplacement
directionnel, cela a peu d’effet et c’est l’effet de la sélection qui domine
; si le déplacement aléatoire est grand, l’évolution des fréquences
alléliques n’est plus prévisible et seule une probabilité peut être
attribuée à la fixation d’un allèle.
|
|
Quel est le taux de mutation le plus élevé possible ? Des taux de mutation très élevés ne sont observés que chez certains virus, mais
jamais chez les procaryotes, ni chez les eucaryotes. On peut faire l’hypothèse que
ces virus vivent dans des environnements beaucoup plus instables que les autres
virus et les organismes cellulaires. Non seulement c’est assez peu convaincant, mais
une autre explication est suggérée par l’observation d’une corrélation négative
entre la taille du génome et le taux de mutation [5]. Cette corrélation est encore plus évidente en considérant la taille
efficace du génome. La taille efficace du génome est la partie du génome soumise à
sélection, c’est-à-dire la part du génome où les mutations ne sont pas neutres ;
soit pratiquement la totalité du génome chez la plupart des virus et des
procaryotes, mais souvent seulement une petite partie du génome des eucaryotes qui
est composé essentiellement de séquences intergéniques. La taille efficace d’un
génome est souvent estimée, en première approximation, en additionnant la longueur
des séquences codant les protéines. Il est attendu que plus un génome contient de
séquences codantes, plus le nombre de sites soumis à sélection est important,
c’est-à-dire plus de sites qui, s’ils mutent, auront un effet négatif sur la valeur
sélective. Focalisons-nous maintenant sur les mutations létales pour faire un petit
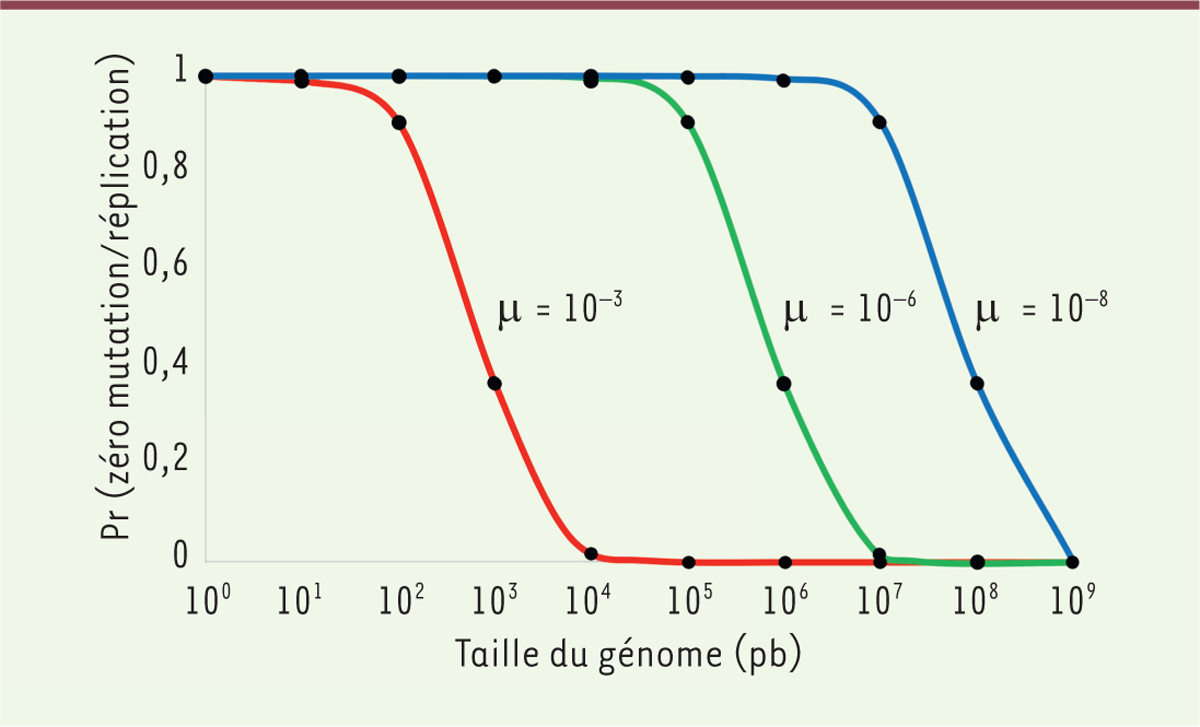
calcul simple. Si un génome contient 1 000 nucléotides invariables, qui ne peuvent
être mutés sans conséquence létale pour le porteur, alors la probabilité de
répliquer ce génome sans qu’il y ait une seule mutation à ces mille positions est
égal à (1-m)1 000 (m étant le taux de mutation). Cette probabilité peut
être calculée en fonction du nombre de sites invariables et pour différents taux de
mutation (Figure 1). Pour un
taux de mutation égal à 10-3, un génome contenant moins de mille sites
invariables (soit seulement quelques gènes) peut produire des copies sans mutation,
donc fonctionnelles. Pour ce même taux de mutation, un génome contenant plus de
10 000 sites invariables ne pourra pas produire de copies fonctionnelles. Ainsi, un
virus ne contenant que quelques gènes peut avoir un fort taux de mutation sans que
cela empêche la production de copies fonctionnelles du génome. Les procaryotes et
les eucaryotes contiennent entre quelques centaines et quelques dizaines de milliers
de gènes, ce qui implique nécessairement bien plus de 10 000 sites invariables et
donc l’impossibilité d’observer un tel taux de mutation. Par exemple, le génome
humain contient un peu plus de 20 000 gènes dont la longueur moyenne de la partie
codante est d’un peu plus de 1 000 paires de bases, soit au total plus de 20
millions de nucléotides constituant les séquences codant les protéines. Sans même
considérer les séquences non-codantes ultra-conservées qui sont aussi soumises à la
sélection, et en tenant compte que seule une fraction des séquences codantes ne peut
pas être modifiée sans conséquence létale, il existe plusieurs millions de positions
invariables parmi les 3,2 milliards de nucléotides par génome haploïde. Dans une
telle situation, seul un taux de mutation inférieur à 10-8 permet de
produire une forte proportion de génomes fonctionnels (Figure 1). Le taux de mutation maximal étant limité
par la taille efficace du génome, on peut en déduire qu’au cours de l’évolution,
l’augmentation de la taille efficace d’un génome ne peut se faire qu’en association
avec la réduction du taux de mutation. S’il s’avérait qu’il est très difficile
d’améliorer les taux de mutation les plus bas observés, alors ceci pourrait
expliquer que le nombre de gènes dans des génomes plafonne toujours à quelques
dizaines de milliers.
 | Figure 1. Probabilité d’une réplication sans mutation en fonction de
la taille du génome et du taux de mutation. μ :
taux de mutation ; pb : paire de bases ; Pr : probabilité. |
|
Quel est le taux de mutation le plus bas possible ? Les analyses théoriques décrites précédemment convergent pour nous amener à conclure
que chez des organismes dont le génome est constitué de plusieurs milliers de gènes
et dans un environnement relativement stable, le taux de mutation doit tendre vers
zéro mais rester au-dessus d’une limite imposée par les propriétés physico-chimiques
des complexes moléculaires qui répliquent et réparent l’ADN et par la nécessité de
produire des mutations à valeur adaptative. Lynch a proposé que s’il ne tend pas
vers zéro, c’est plutôt parce que la sélection pour des taux de mutation toujours
plus bas devient de plus en plus difficile car les différences de valeurs sélectives
deviennent de plus en plus faibles. Pour comprendre ce point, imaginons un taux de
mutation de 10-1 dans une population dans laquelle apparaît un individu
avec un taux de mutation dix fois plus faible, soit 10-2. La différence
des nombres de descendants viables produits par ce mutant par rapport aux autres
individus sera importante. Mais si le taux de mutation est de 10-8 dans
une population dans laquelle apparaît un individu avec un taux de mutation dix fois
plus faible, soit 10-9, cette différence sera très faible car dans cette
population, la plupart des descendants sont viables. En d’autres termes, plus un
système est optimisé, plus son amélioration par le mécanisme de sélection de
nouveaux allèles devient difficile dans une population de taille finie, car son
action est de plus en plus faible et donc d’autant plus perturbée par la dérive
génétique qui augmente ou réduit de façon aléatoire la fréquence des allèles
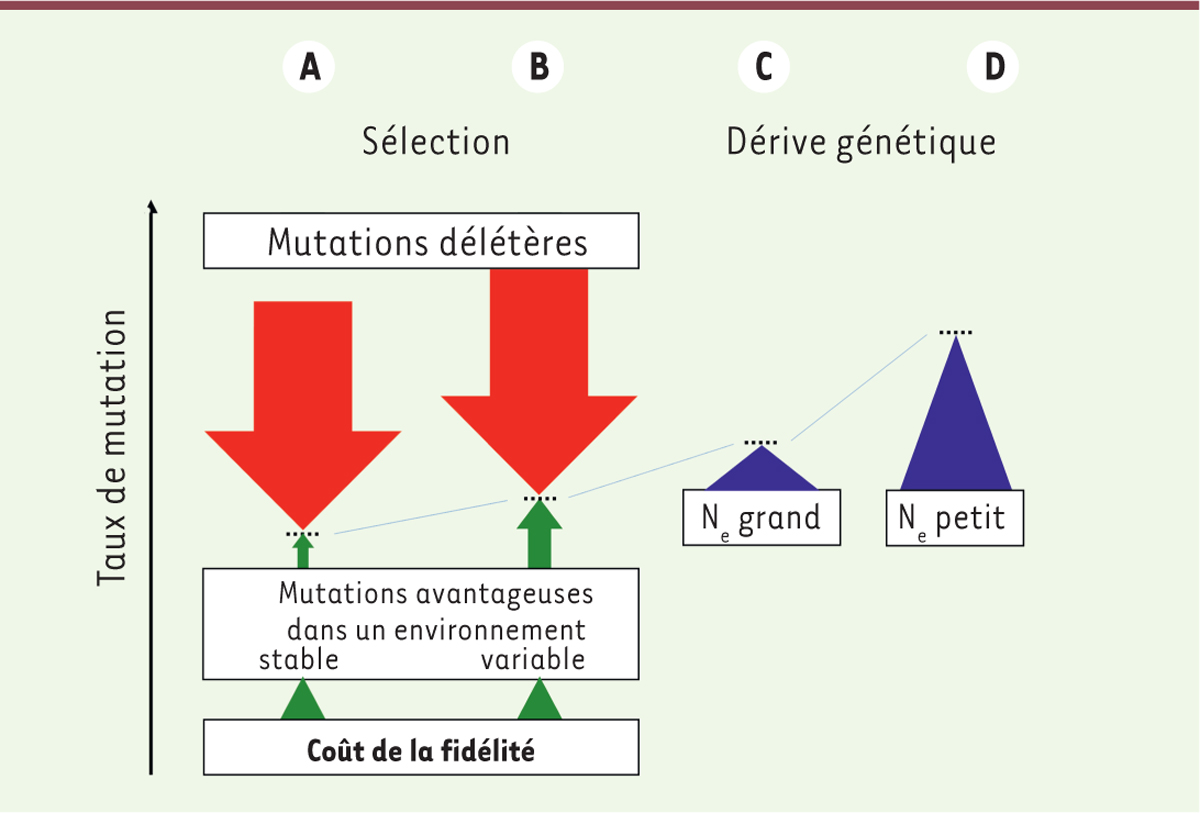
antimutateurs. Dans une population de taille efficace très grande, la dérive
génétique étant plus faible, la sélection devrait amener le taux de mutation vers
des valeurs plus petites (Figure
2). Cet attendu est largement vérifié, aussi bien pour les
mutations ponctuelles [9, 14, 19] que pour le taux d’insertion/délétion de courtes séquences
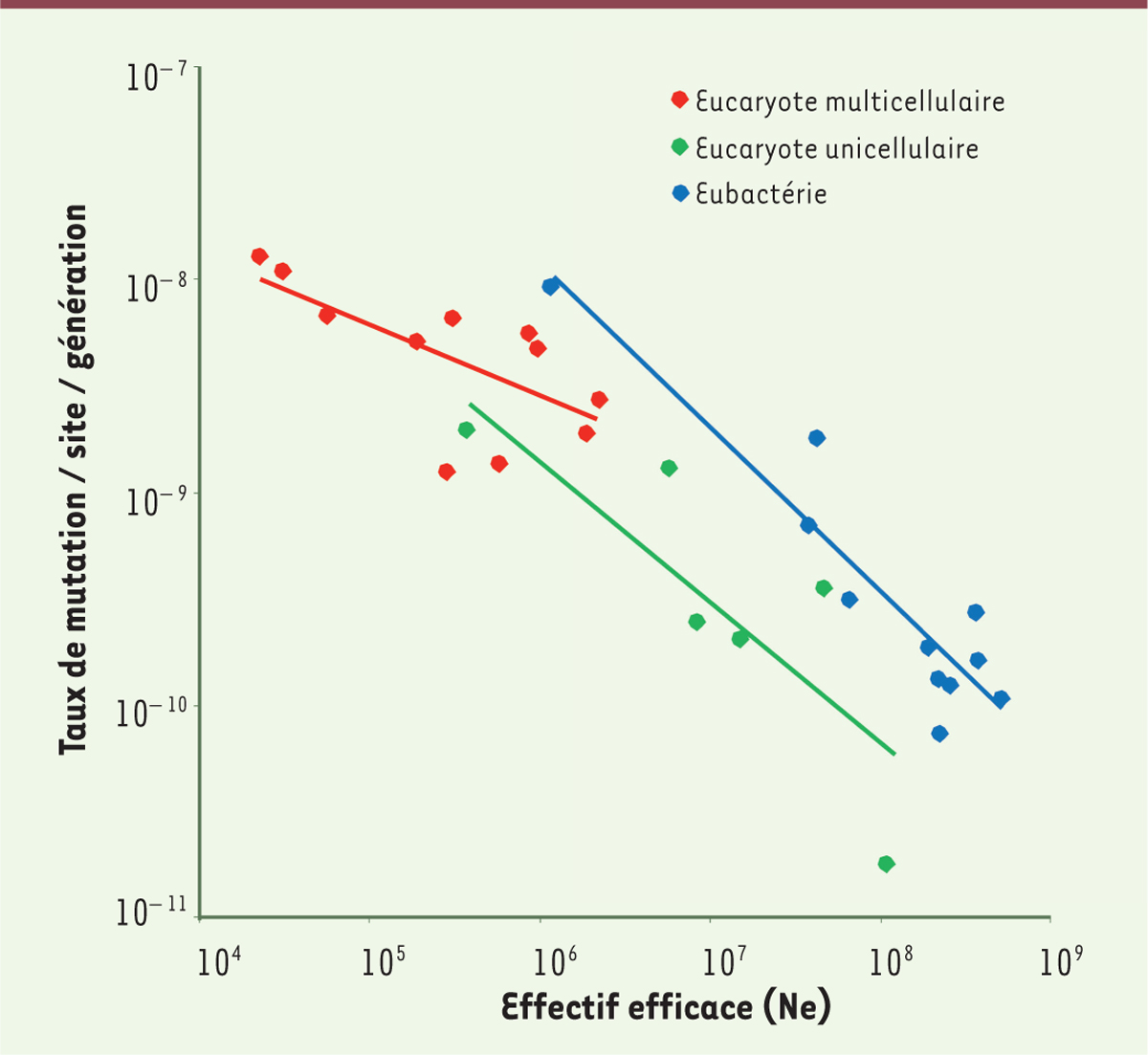
[20]. Ainsi, le taux de
mutation est corrélé négativement à la taille efficace des populations
(Figure 3) : les
bactéries ont des tailles efficaces souvent plus grandes que celles des eucaryotes
et des taux de mutation plus faibles ; les eucaryotes unicellulaires ont des tailles
efficaces souvent plus grandes que celles des pluricellulaires et des taux de
mutation plus faibles. |
Le taux de mutation est-il égal dans la lignée germinale et les lignées
somatiques ? Nous avons discuté des contraintes agissant sur l’évolution du taux de mutation, en
particulier la sélection naturelle qui favorise les individus antimutateurs au
travers d’une meilleure valeur adaptative de leurs descendants. C’est donc le taux
de mutation dans la lignée germinale qui est importante ici. Même si beaucoup de
mutations se produisent dans les lignées somatiques, c’est finalement peu important
car ces mutations disparaîtront toutes à la mort de l’individu. On peut donc
supposer que les lignées somatiques sont soumises à une sélection moins forte pour
réduire le taux de mutation, et c’est effectivement ce qui est observé [9]. |
Quel est le rôle du mode de reproduction sur l’évolvabilité 5 du taux de mutation ? Nous venons de décrire des arguments théoriques et expérimentaux qui suggèrent que le
taux de mutation dans une espèce donnée dépend principalement de 1) la taille
efficace de son génome qui définit le taux de mutation maximum et 2) l’effectif
efficace qui contrôle le taux de mutation minimum accessible, c’est-à-dire au-dessus
de la barrière de la dérive génétique. Il est attendu que le taux de mutation pourra
donc fluctuer autour du taux de mutation minimum accessible au gré des allèles
mutateurs et antimutateurs qui apparaîtront au cours du temps. Nous pouvons introduire maintenant un niveau de complexité supplémentaire en tenant
compte du fait que des espèces possèdent une reproduction sexuée (la plupart des
eucaryotes) et d’autres non (les procaryotes). Nous allons voir que cette différence
a des conséquences sur l’évolvabilité du taux de mutation. Chez les procaryotes, si
une cellule a un fort taux de mutation alors, dans des conditions où une mutation
est nécessaire pour s’adapter à l’environnement, il est probable que cette mutation
utile apparaisse chez un descendant de la cellule mutatrice. Sans reproduction
sexuée, il y a peu de recombinaisons des génomes et l’allèle muté se fixera dans la
population en association avec l’allèle mutateur (c’est l’effet auto-stop,
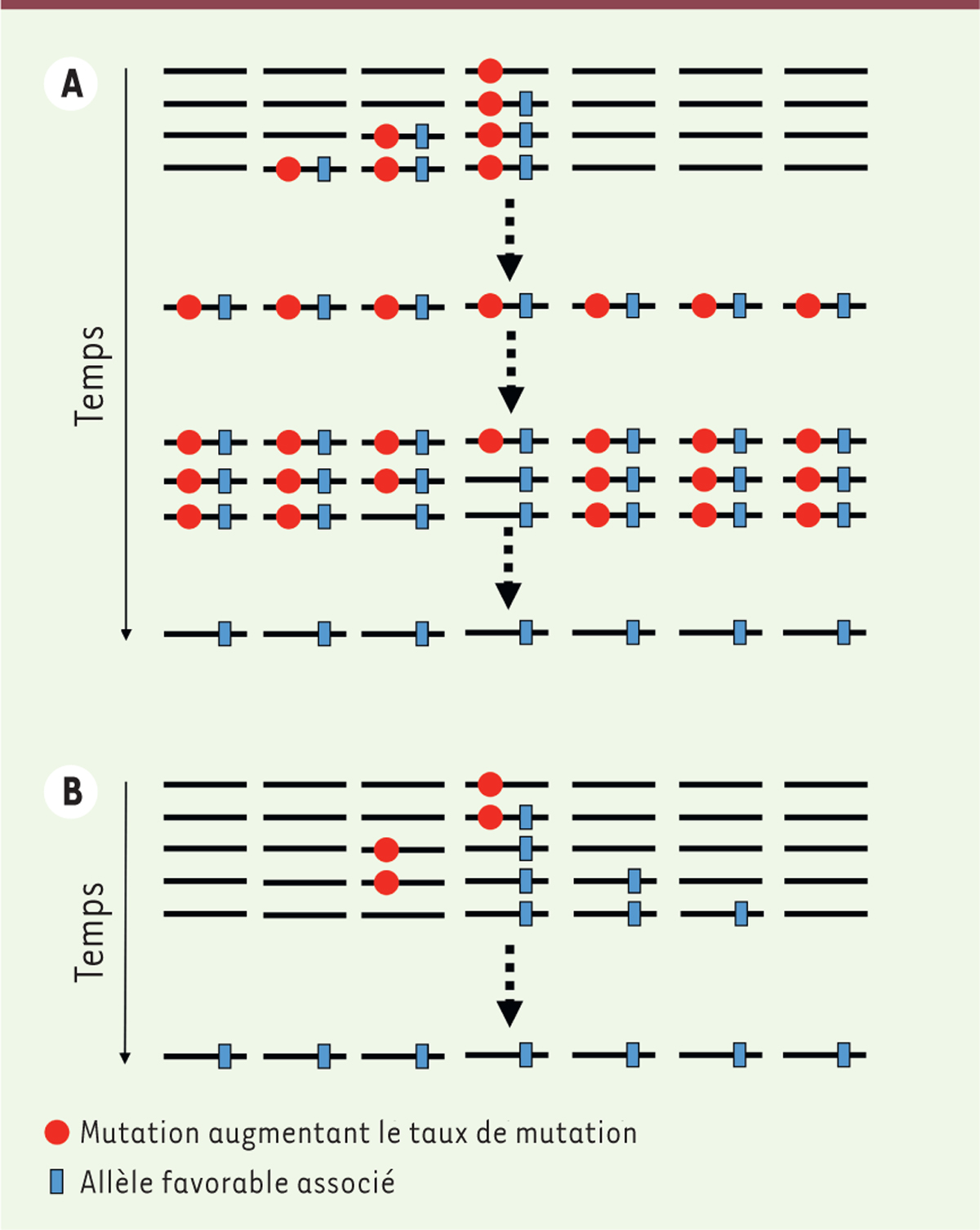
Figure 4. En parallèle
de son adaptation, la population aura acquis un fort taux de mutation. Si une
cellule, tout en gardant l’allèle utile, perd l’allèle mutateur, elle sera avantagée
car elle restera adaptée à l’environnement et elle réduira le fardeau de mutation dû
à l’allèle mutateur. Ainsi, dans des milieux instables, il peut se mettre en place
des cycles dans lesquels des populations de bactéries voient leur taux de mutation
augmenter fortement puis revenir vers des valeurs beaucoup plus basses. Bien décrit
sur le plan théorique, ce processus a été observé aussi bien dans des population
expérimentales que naturelles de bactéries E. coli [21-27]. Chez une espèce à reproduction sexuée, si
une mutation utile apparaît associée à un allèle mutateur, la recombinaison sépare
rapidement les deux allèles, il n’y a pas d’effet auto-stop et il est donc peu
probable de pouvoir identifier des populations mutatrices apparues par ce mécanisme
[28] (Figure 4).
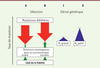 | Figure 2. Effets de la sélection et de la dérive génétique sur le
taux de mutation. A, B. sans dérive génétique,
le taux de mutation est un équilibre entre des forces sélectives
opposées favorisant les organismes mutateurs et antimutateurs.
C, D. la dérive génétique pousse
l’équilibre vers des valeurs plus grandes que celles attendues du seul
fait de la sélection. Ne : effectif efficace de la
population. |
 | Figure 3.Corrélation entre le taux de mutation et l’effectif
efficace (modifié d’après Lynch et
al. [ 14]). |
 | Figure 4.Effet du mode de reproduction sur le taux de mutation.
A. Dans une population à reproduction clonale,
du fait de la liaison totale des gènes, par effet auto-stop, l’allèle «
mutateur » se fixe dans la population avec l’allèle favorable.
L’élimination de l’allèle mutateur implique une réversion ou le
transfert horizontal d’un allèle non-mutateur.
B. Dans une population à reproduction
sexuée, la recombinaison dissocie rapidement l’allèle mutateur de
l’allèle favorable si la liaison n’est pas très forte, diminuant ainsi
l’effet auto-stop et la probabilité de fixation de l’allèle mutateur en
même temps que l’allèle favorable (modifié d’après Sniegowski et
al. [ 5]). |
|
La question « Pourquoi le taux de mutation n’est-il jamais égal à zéro » pourrait
sembler triviale au premier abord : s’il n’y avait pas de mutations, il n’y aurait
pas eu d’évolution et puisqu’il y a évolution, il y a donc bien eu des mutations,
fermez le ban ! Elle a pourtant été à l’origine d’une suite de réflexions théoriques
et de travaux expérimentaux d’une grande subtilité. Il reste beaucoup de questions
ouvertes concernant ce point très important de la biologie évolutive, mais le voile
se lève peu à peu. L’ensemble des approches disponibles aujourd’hui montre que la
production de mutations, matériel de base de l’évolution, est elle-même un processus
soumis aux mécanismes évolutifs qui dépendent de façon complexe de l’architecture
des génomes des différentes espèces ainsi que de la stabilité des environnements
dans lesquels elles vivent, et de leur démographie. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
Nous remercions chaleureusement nos collègues Jean-Luc Da Lage et Julien Fumey pour
leurs fort pertinents commentaires et suggestions au cours de l’élaboration de cet
article.
|
Footnotes |
1. Sturtevant
AH. Essays on
evolution. I. On the effects of selection on mutation rate .
Quart Rev. Biol.
1937; ; 12 :
:464.–467. 2. Demerec
M.. Genetic factors
stimulating mutability of the miniature gamma wing character of Drosophila
virilis . Proc Natl Acad Sci USA.
1929; ; 15 :
:834.–838. 3. Demerec
M.. Frequency of
spontaneous mutations in certain stocks of Drosophila
melanogaster . Genetics.
1937; ; 22 :
:469.–478. 4. Muller
HJ. The measurement
of gene mutation rate in Drosophila, its high variability, and its
dependence upon temperature . Genetics.
1928; ; 13 :
:279.–357. 5. Sniegowski
PD,
Gerrish
PJ,
Johnson
T,
Shaver
A. The evolution of
mutation rates: separating causes from consequences .
Bioessays.
2000; ; 22 :
:1057.–1066. 6. Drake
JW. A constant rate
of spontaneous mutation in DNA-based microbes . Proc
Natl Acad Sci USA.
1991; ; 88 :
:7160.–7164. 7. Drake
JW. Rates of
spontaneous mutation among RNA viruses . Proc Natl
Acad Sci USA.
1993; ; 90 :
:4171.–4175. 8. Sung
W,
Tucker
AE,
Doak
TG, et al.
Extraordinary genome stability in the ciliate Paramecium
tetraurelia . Proc Natl Acad Sci USA.
2012; ; 109 :
:19339.–19344. 9. Lynch
M.. Evolution of the
mutation rate . Trends Genet.
2010; ; 26 :
:345.–352. 10. Eyre-Walker
A,
Keightley
PD. The distribution
of fitness effects of new mutations . Nat Rev
Genet.
2007; ; 8 :
:610.–618. 11. Robert
L,
Ollion
J,
Robert
J, et al.
Mutation dynamics and fitness effects followed in single
cells . Science.
2018; ; 359 :
:1283.–1286. 12. Casane
D,
Laurenti
P. Syllogomanie
moléculaire: l’ADN non codant enrichit le jeu des possibles .
Med Sci (Paris).
2014; ; 30 :
:1177.–1183. 13. Charlesworth
B.. Effective
population size and patterns of molecular evolution and
variation . Nat Rev Genet.
2009; ; 10 : :195.. 14. Lynch
M,
Ackerman
MS,
Gout
JF, et al.
Genetic drift, selection and the evolution of the mutation
rate . Nat Rev Genet.
2016; ; 17 :
:704.–714. 15. Quinones
A,
Piechocki
R. Isolation and
characterization of Escherichia coli antimutators. A new strategy to study
the nature and origin of spontaneous mutations . Mol
Gen Genet.
1985; ; 201 :
:315.–322. 16. Loh
E,
Salk
JJ,
Loeb
LA. Optimization of
DNA polymerase mutation rates during bacterial evolution .
Proc Natl Acad Sci USA.
2010; ; 107 :
:1154.–1159. 17. Furio
V,
Moya
A,
Sanjuan
R. The cost of
replication fidelity in an RNA virus . Proc Natl Acad
Sci USA.
2005; ; 102 :
:10233.–10237. 18. Lloyd
SB,
Kent
SJ,
Winnall
WR. The High Cost of
Fidelity . AIDS Res Hum Retrovir.
2014; ; 30 :
:8.–16. 19. Lynch
M.. The lower bound
to the evolution of mutation rates . Genome Biol
Evol.
2011; ; 3 :
:1107.–1118. 20. Sung
W,
Ackerman
MS,
Dillon
MM, et al.
Evolution of the insertion-deletion mutation rate across the tree
of life . G3 (Bethesda).
2016;; 6 :
:2583.–91. 21. Giraud
A,
Matic
I,
Tenaillon
O, et al.
Costs and benefits of high mutation rates: adaptive evolution of
bacteria in the mouse gut . Science.
2001; ; 291 :
:2606.–2608. 22. Denamur
E,
Matic
I. Evolution of
mutation rates in bacteria . Mol Microbiol.
2006; ; 60 :
:820.–827. 23. Denamur
E,
Lecointre
G,
Darlu
P, et al.
Evolutionary implications of the frequent horizontal transfer of
mismatch repair genes . Cell.
2000; ; 103 :
:711.–721. 24. Labat
F,
Pradillon
O,
Garry
L, et al.
Mutator phenotype confers advantage in Escherichia coli chronic
urinary tract infection pathogenesis . FEMS Immunol
Med Microbiol.
2005; ; 44 :
:317.–321. 25. Taddei
F,
Radman
M,
MaynardSmith
J, et al.
Role of mutator alleles in adaptive evolution .
Nature.
1997; ; 387 :
:700.–702. 26. Tenaillon
O,
Toupance
B, Le
Nagard
H, et al.
Mutators, population size, adaptive landscape and the adaptation
of asexual populations of bacteria .
Genetics.
1999; ; 152 :
:485.–493. 27. Tanaka
MM,
Bergstrom
CT,
Levin
BR. The evolution of
mutator genes in bacterial populations: the roles of environmental change
and timing . Genetics.
2003; ; 164 :
:843.–854. 28. Johnson
T.
Beneficial mutations, hitchhiking and the evolution of mutation
rates in sexual populations . Genetics.
1999; ; 151 :
:1621.–1631. |