Édition de gènes : méthodes, intérêts, limitations dans l’usage et l’innovation
thérapeutique Une brève histoire de l’édition de gène… L’idée d’éditer les gènes (au sens anglo-saxon, on traduit par « modifier », en
français) pour modifier leur séquence nucléotidique date des années 1970,
lorsque Werner Arber découvrit les enzymes de restriction [ 2], des protéines qui servent à la
défense des bactéries contre les agents qui les infectent ( Figure 1). Ces enzymes clivent
l’ADN étranger en des sites précis, dits sites de restriction (4 à 12
nucléotides), qui sont absents de leur propre génome. Pour le bio-technicien, il
était ainsi devenu possible de couper une séquence d’ADN en un site précis puis
de rabouter les extrémités créées avec d’autres morceaux d’ADN coupés par le
même enzyme. Cette opération pouvait être réalisée dans un tube à essai, sur des
fragments d’ADN de petite taille. Elle n’était en revanche pas compatible avec
l’édition de génomes complets, en raison de la grande probabilité de présence de
multiples sites de restrictions dans les génomes de grande taille. Les
chercheurs ont alors cherché à obtenir une meilleure spécificité de
ré-appariement et ont pour cela exploité, dans un premier temps, le processus de
recombinaison homologue. Il s’agit d’un système cellulaire qui s’appuie sur
l’appariement de séquences longues permettant d’effectuer la réparation d’ADN
endommagé. Ce système permettait de réaliser l’édition de gène à l’échelle d’un
génome complet. Cependant, le processus de recombinaison présente des effets non
prévisibles. L’intégration d’un fragment d’ADN à l’intérieur d’un gène non ciblé
est en effet fréquente et potentiellement dangereuse pour l’individu. Le
repérage d’erreurs de réparation était à l’époque des années 1980-1990
particulièrement fastidieux car les technologies de séquençage n’avaient pas
encore atteint les grands débits que l’on connaît de nos jours.
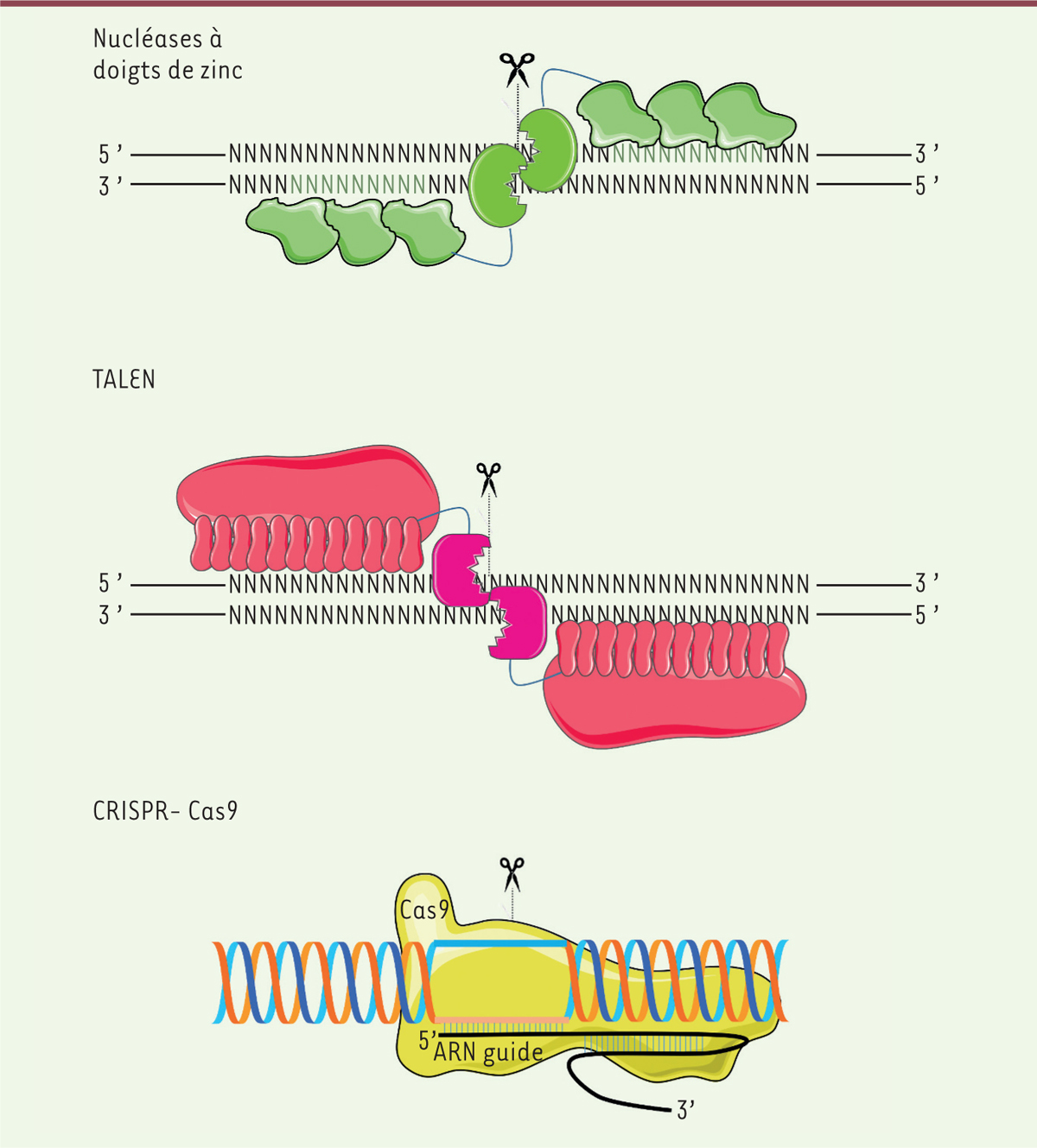
 | Figure 1. Édition de gènes. Les nucléases à doigt de zinc et les TALEN sont des
protéines contenant un domaine enzymatique qui clive l’ADN de
manière indépendante de la séquence nucléotidique, et plusieurs
domaines répétés de reconnaissance de l’ADN. Dans les protéines à
doigts de zinc, les séquences d’ADN reconnues par chaque domaine
sont de trois nucléotides, mais des combinaisons allant jusqu’à 8
domaines en tandem ont été observées. Dans les protéines TALEN, le
domaine de reconnaissance de l’ADN est composé d’un segment de 33 à
34 acides aminés dont les deux résidus en position 12 et 13 sont
variables et reconnaissent spécifiquement un nucléotide.
L’enchaînement répété de ces domaines (fréquemment plus de 12)
confère la spécificité de reconnaissance. Dans le système CRISPR-Cas
optimisé pour l’édition de gène, la reconnaissance de la séquence
nucléique clivée par l’enzyme Cas est assurée par la partie 5’ d’un
ARN guide. Cet ARN contient dans sa partie 3’ une séquence qui
assure l’interaction avec l’enzyme Cas et son activation. La
spécificité est assurée par le fait que l’enzyme Cas non liée à son
ARN guide n’a pas d’activité catalytique. |
Des approches plus spécifiques sont apparues à la fin des années 1990, avec
l’utilisation de nucléases complexes reconnaissant de grands fragments d’ADN :
les méganucléases [3,
4], les nucléases à
doigt de zinc (ZFN) [5],
puis, en 2009, les TALEN (transcription activator-like effector
nuclease). Les méganucléases sont des enzymes de restriction qui
reconnaissent et clivent des séquences nucléotidiques longues (20 paires de
bases et plus) et donc rares, voire uniques dans un génome. Ces enzymes sont
très efficaces et peu toxiques, mais leur mutagénèse à des fins d’édition de
gènes est complexe et difficilement généralisable. Leur emploi est donc très
limité. Les deux autres familles de nucléases (ZFN et TALEN) présentent des domaines
répétés de reconnaissance de l’ADN, ce qui leur confère une grande spécificité
de reconnaissance de la séquence à éditer. En revanche, la construction de
telles enzymes comme outils de manipulation génétique nécessite des stratégies
sophistiquées car il s’agit non seulement de protéines de très grande taille,
mais de molécules constituées de domaines répétés présentant entre eux de fortes
homologies. Quelques succès notoires ont été obtenus permettant de mener des
études cliniques chez des patients infectés par le VIH (virus de
l’immunodéficience humaine) et atteints de Sida (syndrome de l’immunodéficiences
acquise) à l’aide de nucléases à doigt de zinc [6], ou dans le traitement de leucémies avec
les TALEN [7, 8]. Très rapidement cette approche, techniquement lourde, a été supplantée par la
technique dite de CRISPR-Cas9 inspirée du système immunitaire des bactéries et
des archées [9]
(→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 11,
novembre 2016, page 1035
Afin de comprendre le mécanisme et l’utilisation de CRISPR-Cas9, voyons
brièvement ses caractéristiques : lorsqu’une bactérie est infectée par un virus
qui lui est spécifique, un bactériophage, elle se défend en fragmentant l’ADN
intrus et en mettant en mémoire cet évènement dans son propre ADN afin de mieux
répondre à une agression ultérieure. Pour cela, elle insère des fragments de 20
à 50 paires de bases (pb) de l’ADN intrus à l’intérieur d’une séquence répétée
de 29 nucléotides de son génome. Cet ensemble « séquence de l’intrus + séquence
répétée », est ensuite transcrit en un ARN, appelé crARN (pour CRISPR-ARN), dans
lequel la partie homologue à l’ADN de l’intrus sert à détecter sa présence, la
partie répétée recrutant une enzyme, Cas9, capable de cliver l’ADN ainsi
reconnu. Cette étape de liaison du crARN et de l’enzyme est réalisée grâce à un
intermédiaire, tracr-ARN (trans-activating CRISPR RNA) qui est
un transactivateur de Cas9. En se liant à Cas9, ce transactivateur amorce
l’activité nucléase de Cas9. Simultanément, il se lie à la séquence répétée du
crARN et guide l’enzyme vers l’ADN à reconnaître. La détection de l’ADN intrus
est associée à une liaison de la partie nucléase et à son activation, ce qui
conduit à sa dégradation. De nombreux perfectionnements de ce système ont été
développés pour un usage biotechnologique et thérapeutique, et plus précisément,
pour l’inactivation ou l’induction de gènes ou la mutagenèse in
situ (voir Encadré). |
L’édition de gènes avec CRISPR-Cas9 Dans sa version initiale, la mise en œuvre de la technique CRISPR-Cas9
nécessitait l’introduction de trois éléments distincts dans la cellule :
un ADN codant le crARN guidant Cas sur le site de clivage, un ADN codant
l’ARN tracrARN et un ADN codant Cas9. Très rapidement, les éléments
crARN et tracrARN ont été fusionnés en un seul appelé guide
(single guide-RNA ou sg-ARN), réduisant le nombre
de composants du système à deux. Pour obtenir le clivage spécifique
d’une séquence d’ADN, il suffisait alors d’intégrer deux éléments à une
cellule, deux fragments d’ADN (ou d’ARN) ou un complexe préformé
riboprotéique contenant l’ARN guide et Cas9. Une des forces du système réside dans le fait que Cas9 ne clive l’ADN que
lorsqu’elle est fixée à l’ADN cible via son ARN guide.
On peut aussi altérer l’activité de Cas9 pour lui faire perdre sa
capacité de clivage (dCas9, pour dead Cas9). Il en
résulte une entrave à la progression des polymérases, autrement dit une
inhibition de réplication ou de transcription. Certains auteurs ont
fusionné un activateur de transcription à dCas9, transformant ainsi
CRISPR Cas en activateur de l’expression de gènes. Ces innovations n’ont
pas encore débouché sur des applications thérapeutiques. Notons
toutefois dès à présent que tout comme dans le cas des nucléases à
doigts de zinc et des TALEN, l’usage de CrispR Cas se heurte au problème
de la spécificité de reconnaissance des séquences ciblées par les ARN
guides, ce qui oblige les utilisateurs à un examen critique des
résultats obtenus, et a motivé l’optimisation du système. L’ingénierie de Cas9 a permis d’identifier une mutation qui conduit à un
clivage simple brin (activité nickase, enzyme Cas9n). Dès lors, le
clivage complet de l’ADN nécessite deux exemplaires du complexe sg-ARN x
Cas9n, chaque exemplaire reconnaissant une séquence complémentaire, pour
obtenir une coupure double brin. Cela permet d’améliorer la spécificité
du système sans totalement éliminer des effets non ciblés.
|
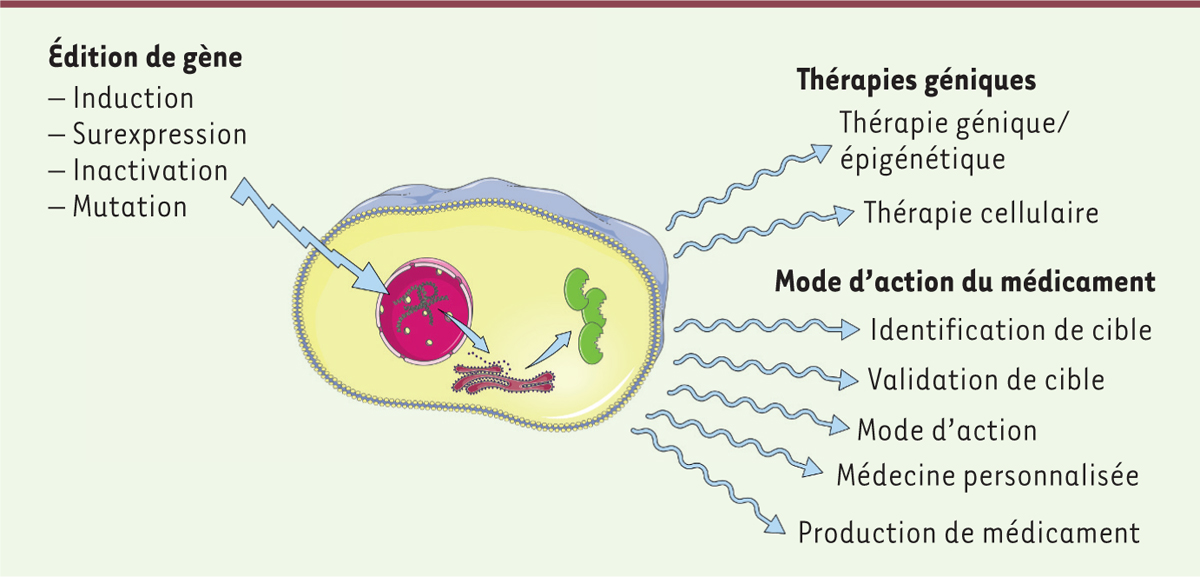
Édition de gène dans le domaine thérapeutique Nous considèrerons ici les applications dans un sens large : celles qui
utilisent les techniques d’édition dans le domaine des maladies génétiques,
mais aussi celles qui les exploitent dans une démarche d’identification et
de validation de cibles thérapeutiques ( Figure 2).
 | Figure 2. Édition de gènes et innovation thérapeutique. |
Thérapies cellulaire et génique Cette approche est utilisée de manière très prometteuse dans plusieurs
cas. Le premier groupe d’applications qui vient immédiatement à l’esprit
est celui qui consiste à rétablir l’expression de gènes défectueux. La
toute première thérapie d’édition génique chez l’homme a été développée
aux États-Unis en 2017. Elle a utilisé une nucléase à doigts de zinc
pour insérer à l’intérieur du gène de l’albumine le gène qui code une
protéine absente chez les patients. Dans le cas de patients atteints du
syndrome de Hunter 1, le gène inséré
est celui de l’enzyme iduronate-2-sulfatase, dont l’absence cause un
maladie lysosomale dont les symptômes sont ceux d’une maladie
neurodégénérative [ 10]. Les outils génétiques sont introduits grâce à un virus
atténué qui est injecté directement dans le foie du patient. Cette
thérapie est donc réalisée directement chez le patient, sans passer par
une étape de traitement ex vivo de ses cellules. Elle
lui fait donc courir un risque important, car toute erreur ne sera
détectable qu’ a posteriori. La prise de conscience de ce risque a conduit les chercheurs à développer
des approches incluant une étape ex vivo afin de
pouvoir contrôler la survenue d’effets indésirables [11]. Ainsi, la société
CrispR Therapeutics mène une étude clinique de
phase II sur l’efficacité et la sécurité d’usage de cellules autologues
modifiées capable de produire de l’hémoglobine fœtale chez des adultes
atteints de beta thalassémie [12]. Ce protocole revêt un caractère générique : il
peut en effet être appliqué à un nombre important de pathologies et
permet d’effectuer les contrôles nécessaires (par exemple de ciblage)
préalablement à l’infusion des cellules modifiées au patient. Ces corrections de phénotypes obtenues par édition de gènes ne peuvent
pas, en principe, être mimées par des médicaments, en particulier
lorsque ceux-ci sont de petites molécules. Elles s’avèrent donc être des
éléments très importants de l’arsenal thérapeutique. Il faut toutefois
noter que les effets à long terme de ces thérapies restent inconnus, que
l’effet est « théoriquement » irréversible, ce qui interdit toute
approche d’essai-erreur, que leur coût sera toujours important en raison
du caractère personnalisé du traitement, et que la généralisation à des
cohortes importantes présentera toujours un risque non négligeable
d’effets indésirables liés à la variation interindividuelle. À titre
d’exemple, de nombreuses études cliniques se mettent actuellement en
place dans le monde entier, principalement aux États-Unis et en Chine,
pour des applications dans le domaine de l’immunomodulation du cancer.
Elles se posent ainsi en concurrentes des méthodes traditionnelles qui
utilisent des anticorps ou des médicaments. Deux problèmes additionnels
se posent. Ils relèvent tous deux du domaine de l’immunité : (1) l’usage
de vecteurs d’origine virale pour pratiquer cette thérapie est
nécessaire2,, ce qui limitera le
nombre d’administrations, en particulier en raison du déclenchement
possible d’une réponse immunitaire vis-à-vis du vecteur qui aura pour
conséquence son élimination rapide, et donc une inefficacité du
traitement ; (2) la seconde préoccupation est celle de l’usage des
protéines Cas9 [12, 13]. Ces
protéines sont d’origine bactérienne et 70 % de la population possède
des anticorps dirigés contre les protéines Cas9 qui sont utilisées en
laboratoire pour l’édition des gènes [14]. Mais plus préoccupant encore,
les lymphocytes T sont susceptibles de répondre à Cas9. Ainsi, des
lymphocytes T CD8 et T CD4 spécifiques de Cas9 peuvent être stimulés par
l’enzyme [15].
Le risque pour le patient est néanmoins limité dans le cas d’un
traitement comportant une seule injection, mais l’histoire du patient
devenant déterminante, une deuxième intervention peut s’avérer
impossible. Dans tous les cas, l’usage chez l’humain des méthodes d’édition des gènes
place la question de la réponse immunitaire au cœur des préoccupations,
car les « objets » biologiques utilisés (ADN, vecteurs viraux,
protéines, etc.) sont de grande taille, donc potentiellement
immunogènes, et donc neutralisés et/ou détruits par le système
immunitaire. L’ingénierie biotechnologique va sans doute améliorer ces
outils d’édition de manière considérable. Il est donc raisonnable
d’attendre des succès, même à brève échéance, mais la sélection des
patients susceptibles d’être traités sera sans doute inévitable, et les
thérapies seront vraisemblablement très onéreuses en raison des
batteries de tests compagnons qu’il faudra associer pour sécuriser leur
emploi [16]. Identification, validation et traitement de cibles
thérapeutiques L’édition de gène trouve des applications nombreuses dans le domaine de
la recherche fondamentale, connu sous le nom de génomique
fonctionnelle. Par la simplicité de sa mise en œuvre, elle
révolutionne les méthodes d’excision/insertion de gène ou de mutagénèse
in situ, et surtout, elle permet l’introduction de
modifications ciblées dans des espèces animales autres que la souris qui
a longtemps été le seul organisme accessible en raison de l’emploi de
cellules souches embryonnaires. De nouvelles perspectives se sont donc
offertes. Dans la recherche sur le médicament, l’ère de la génomique et
du séquençage à haut débit a ainsi permis le développement de nouvelles
stratégies fondées sur la correction de phénotypes, sans a
priori sur les mécanismes par lesquels ces corrections
s’effectuaient. On a nommé cette démarche criblage
phénotypique, ou chimiogénomique directe
[ 17]. Elle
a été rendue accessible grâce aux évolutions dans la miniaturisation des
essais [ 18].
Bien que cette approche soit utilisée depuis plus de vingt ans, elle se
heurte à une difficulté majeure : l’identification de la cible de la
molécule bioactive. En effet, le criblage phénotypique s’appuie sur des
cellules, des organes ou des animaux modèles présentant le phénotype que
l’on veut modifier (par exemple, la production d’un métabolite ou
l’arrêt de sa production) que l’on expose à des petites molécules parmi
lesquelles on recherche les composés qui provoquent le phénotype désiré.
En cas de succès, les molécules identifiées sont appelées « touches »
(ou hits en anglais) par référence à la pêche à la
ligne. Si le processus étudié relève d’un enjeu de santé, les touches
peuvent devenir des amorces (ou leads) pour le
développement de nouveaux médicaments. Cette approche phénotypique s’avère performante pour perturber des voies
de signalisation ou des circuits métaboliques, mais les molécules
identifiées peuvent agir à plusieurs niveaux dans chacun de ces
circuits, et conduire, en fait, au même phénotype par des mécanismes
différents [19]. Le fait de connaître la, ou les, cible(s) de ces molécules
devient donc un enjeu pour le chimiste, qui doit les optimiser, et pour
le biologiste, qui doit pouvoir expliquer leur mécanisme d’action.
L’édition de gène intervient ici de plusieurs façons : les technologies
d’édition à grande échelle permettent de cibler tout ou partie du
transcriptome d’une espèce de façon massivement parallèle pour induire
ou bloquer l’expression de gènes, ou pour modifier leur séquence. Voyons
ces applications. Surexpression de [20-22] Cette technique consiste à augmenter l’activité d’un gène donné afin de
détecter si le surcroît d’activité confère une résistance à un
traitement par des inhibiteurs ( versus une
sensibilisation pour ces mêmes activateurs) de l’activité de la protéine
codée par le gène d’intérêt. Le concept exploité repose sur la notion
que la surexpression du gène de la protéine cible diminue l’efficacité
du médicament, pour une même dose de ce dernier. Cette méthode a d’abord
été validée par des techniques de transfection de banques de plus de 10
000 ADN codants « pleine longueur » au sein desquelles on parvenait à
identifier les gènes de résistance aux agents cytotoxiques
anticancéreux. Les techniques d’édition de gène ont supplanté cette
approche en raison de leur plus grande simplicité de mise en œuvre et du
nombre plus important de gènes ciblés dans une même campagne [ 21, 23]. Dans cette
approche, on introduit des mutations dans le gène codant Cas9 pour lui
faire perdre sa capacité à cliver l’ADN ; on fusionne ce gène mutant
(appelé dCas9) avec des domaines d’activation de la transcription [ 21]. Il en résulte une augmentation
sensible de l’expression du gène ciblé. Des collections de plusieurs
dizaines de milliers d’ARN guides sont disponibles pour ce type
d’étude. Réduction d’expression ou délétion de cible Il existe maintenant plusieurs exemples d’utilisation de l’édition de
gènes pour l’identification de la cible de médicaments. Un exemple
frappant est celui du repositionnement de médicaments dans des
indications thérapeutiques nouvelles. Le criblage d’une collection de
médicaments sur des lignées cellulaires d’adénocarcinome de l’œsophage a
ainsi identifié des glycosides cardiotoniques (ouabaïne, digoxine et
digitoxine) comme inhibiteurs de la prolifération cellulaire [ 24]. Le
séquençage des ARN des cellules traitées par ces glycosides a montré que
l’expression de la kinase MKK6 ( mitogen-activated protein kinase
kinase 6) était réduite, diminuant ainsi l’expression du
facteur de transcription Sox9 ( SRY-Box 9). La baisse
d’expression de MKK6 a été reproduite, à l’aide d’ARN interférant. Un
ralentissement de la prolifération des cellules cancéreuses auquel était
associée une baisse du taux d’expression de Sox9 a été observé, ce qui
mime l’effet des glycosides. La suppression du gène codant Sox9, par la
technique Crispr-Cas9, dans des cellules cancéreuses, a montré un
ralentissement de la croissance tumorale, même en l’absence de
traitement, confirmant le mécanisme d’action de ces agents
cardiotoniques. Il s’agit ici d’un exemple parmi d’autres de validation
de cible in vitro. Le défi reste majeur lors du passage
de l’ in vitro à l’animal entier, voire à l’humain, chez
qui il faut pouvoir démontrer « l’engagement » de la cible. Là encore,
les techniques d’édition de gènes ont joué un rôle important pour
démontrer, par exemple, le mécanisme d’action de la curcumine. La
curcumine est une substance naturelle utilisée comme complément
alimentaire et supposée exercer des effets anti-inflammatoires et
anti-oxydants, mais sa cible n’est pas connue. Des auteurs chinois
[ 25] ont
identifié in vitro la cible de la curcumine. Il s’agit
de la kinase de tyrosine DYRK2 ( dual-specificity tyrosine
phosphorylation-regulated kinase 2), un activateur du
protéasome 26S, que la curcumine bloque spécifiquement, parmi 140
kinases testées. La curcumine, en association avec le carfilzomib,
réduit en fait la prolifération de cellules de cancer mammaire et
diminue leur caractère invasif in vitro. Elle réduit
aussi la croissance de tumeurs mammaires humaines implantées chez des
souris imunodéficientes. La validation de DYRK2 comme cible de la
curcumine in vivo a été réalisée en montrant que la
suppression du gène qui la code dans les tumeurs mime l’effet d’un
traitement par la curcumine. Il existe des exemples naturels de délétion de gènes qui ont été utilisés
comme méthode de validation thérapeutique d’une cible. Un exemple récent
est celui de la protéine PCSK9 (proprotéine convertase
subtilisine/kexine de type 9), une protéine qui se fixe au récepteur des
lipoprotéines de faible densité (LDL-R) et provoque sa dégradation.
Cette réduction du nombre de récepteurs LDL-R réduit la dégradation des
lipoprotéines de faible densité conjuguée au cholestérol
(LDL-cholestérol) et provoque une augmentation de leur taux ; en
inhibant la dégradation des LDL-R, les inhibiteurs de PCSK9 réduisent
les taux circulants de cholestérol. Cette baisse du taux circulant de
cholestérol a été aussi observée chez des sujets humains dépourvus de
PCSK9 chez qui les accidents cardiaques sont statistiquement moins
fréquents que dans la population. Inversement, certains humains
surexpriment naturellement PCSK9 et présentent des taux chroniquement
élevés de cholestérol. L’idée a donc été proposée d’associer le fort
taux de cholestérol au risque cardiaque accru, ce qui a motivé une
recherche très compétitive d’inhibiteurs de PCSK9. Deux compagnies
pharmaceutiques, Sanofi et Amgen se
sont affrontées sur le terrain de la production d’anticorps
thérapeutiques neutralisant PCSK9 dans une étude clinique de trois ans
avec plusieurs milliers de patients. Malheureusement, les résultats de
l’étude ne sont pas probants, l’anticorps n’est pas efficace chez
l’homme, bien qu’il bloque effectivement l’activité de PCSK9 : la baisse
de risque cardiovasculaire n’est que de 15 % comparé au placebo [26]. Les raisons
de cet absence de lien entre l’inhibition de l’enzyme PCSK9 et le risque
cardiovasculaire ne sont pas comprises aujourd’hui. Il se pourrait que
chez les sujets humains chez qui l’enzyme fait défaut, ou inversement
est suractive, des mécanismes de compensation se mettent en place,
mécanismes que le traitement par anticorps ne permet pas de
stimuler. Production de molécules thérapeutiques Les produits naturels d’origine végétale ou bactérienne sont reconnus
comme source de composés biologiquement actifs et potentiellement
thérapeutiques. Le genre Streptomyces est réputé faire
partie de ce groupe de bactéries produisant des antibiotiques d’intérêt
majeur. Il contient plus de 50 clusters de gènes biosynthétiques, dont
une grande partie est peu ou pas exprimée. Les méthodes d’édition des
génomes procaryotes ont fait des progrès notoires au cours des années
récentes [ 27],
rendant possible l’activation de ces clusters et la production de
nouvelles molécules. L’insertion d’un promoteur dans un cluster, prédit
comme tel mais silencieux, a conduit à la production de macrolactames,
des antifongiques naturels [ 28]. D’autres approches de surexpression de
clusters dans des hôtes non naturels permettent de cribler leur capacité
de production ou d’identifier les métabolites produits [ 29]. Création de modèles de pathologies et découverte de
médicaments La création de modèles prédictifs de pathologies humaines est un domaine
dans lequel les attentes des laboratoires publics et privés sont
considérables. L’avènement des technologies utilisant Crispr-Cas9 a
permis de s’affranchir de l’emploi de cellules souches embryonnaires ce
qui, à son tour, a nettement accéléré le processus de création de
modèles animaux, et a permis l’obtention de modèles à partir de
nombreuses espèces animales [ 30]. Ces modèles sont très puissants pour l’étude
de processus biologiques fortement conservés au cours de l’évolution,
mais leur caractère prédictif est nettement moins performant dans les
modèles de pathologies humaines. Ils peuvent conduire à des taux
importants d’attrition dans le développement de médicaments [ 31– 33]. Les
développements récents de culture cellulaire en 3 dimensions ont conduit
les chercheurs à s’intéresser à de nouveaux systèmes modèles obtenus à
partir de matériel humain, capables de mimer l’organisation des tissus :
les organoïdes, ou mini organes. Ils peuvent être obtenus à partir de
cellules d’origine humaine, de sujets sains ou de patients, ce qui
permet de tester des molécules thérapeutiques sur des modèles de tissus,
voire de tissus connectés entre eux [ 34]. La possibilité de créer des
organoïdes humains nourrit l’espoir de tests plus exploitables pour les
mesures de toxicologie, de pharmacocinétique et d’efficacité des
molécules. Les illustrations de ce type d’approche se multiplient avec un taux de
réussite encore très variable. Les conditions opératoires de production
des organoïdes ne sont pas encore parfaitement maitrisées, mais les
progrès sont réels et, dans ce cas aussi, les méthodes d’édition de
gènes sont exploitées. L’introduction de marqueurs dans les cellules
souches permettent de mieux contrôler leur différenciation et leur
croissance, ainsi que la formation des cultures en trois dimensions. On
obtient ainsi des modèles de rein [35], de foie [36, 37], ou de tube digestif [38], qui
expriment des marqueurs de différenciation et d’environnement de plus en
plus semblables à ceux détectés dans l’organe in vivo.
Des organoïdes de cerveau ont ainsi pu servir à étudier les mécanismes
d’infection par le virus Zika qui provoque une microcéphalie chez le
nouveau-né, et à tester des molécules thérapeutiques [39]. Une expérience menée
aux Pays-Bas montre que 90 % des biopsies de cancer colorectal peuvent
être cultivées sous forme d’organoïdes [38]. On obtient ainsi pour chaque patient un organoïde
cancéreux et un organoïde sain en fonction du site de prélèvement. Sur
ces tissus modèles, on observe une excellente corrélation entre la
réponse du patient et celle de l’organoïde à un traitement donné. Ainsi,
les cellules p53 mutées (mutation du gène suppresseur
de tumeur TP53) sont insensibles à la nutline, un
activateur de p53 sauvage [40]. La réponse au 5-fluorouracile, qui bloque la
réplication de l’ADN et déclenche l’apoptose [41], n’est pas prévisible à partir
du génotype, mais la corrélation entre la réponse du patient et la
réponse de l’organoïde est excellente. Cela ouvre des perspectives
intéressantes tant pour ce qui relève de l’efficacité des traitements
que de la qualité de vie des patients qui bénéficieront des traitements
les mieux adaptés. Bien que cet exemple ne soit pas une illustration
directe de l’apport de l’édition de gène dans la découverte des
médicaments, il en a ouvert la voie. Un second niveau d’innovation du
domaine des organoïdes est celui de l’utilisation des cellules souches
reprogrammées. Ces cellules sont obtenues directement à partir de
cellules de patients et redifférenciées pour obtenir théoriquement
n’importe quel organe [42, 43]. Elles servent aussi à produire différents modèles,
comme par exemple un modèle de glioblastome [44] par introduction de mutations
oncogéniques identifiées chez des patients. Dans ces modèles, on réalise
des traitements avec des anticancéreux et on montre une corrélation
entre l’efficacité des candidats médicaments et la nature des
aberrations génétiques [44]. Ces
nouveaux systèmes modèles font ainsi preuve d’une meilleure
prédictibilité en termes thérapeutiques, et s’avèrent aussi plus
pertinents pour l’étude des mécanismes mis en jeu dans la
physiopathologie. |
1. Hess
GT,
Tycko
J,
Yao
D,
Bassik
MC. Methods and
applications of CRISPR-mediated base editing in eukaryotic
genomes . Mol Cell.
2017; ; 68 :
:26.–43. 2. Arber
W.. Restriction
endonucleases . Angew Chem Int Ed Engl.
1978; ; 17 :
:73.–79. 3. Redondo
P,
Prieto
J,
Munoz
IG, et al.
Molecular basis of xeroderma pigmentosum group C DNA recognition
by engineered meganucleases . Nature.
2008; ; 456 :
:107.–111. 4. Stoddard
BL. Homing
endonucleases : from microbial genetic invaders to reagents for targeted DNA
modification . Structure.
2011; ; 19 :
:7.–15. 5. Urnov
FD,
Miller
JC,
Lee
YL, et al.
Highly efficient endogenous human gene correction using designed
zinc-finger nucleases . Nature.
2005; ; 435 :
:646.–651. 6. Tebas
P,
Stein
D,
Tang
WW, et al.
Gene editing of CCR5 in autologous CD4 T cells of persons
infected with HIV . N Engl J Med.
2014; ; 370 :
:901.–910. 7. Sunderland
M,
Peggs
Z. succesful
translation and future prospects of TALEN editing for leukemia
patients . Expert Opin Biol Ther.
2018; ; 18 :
:725.–726. 9. Jordan
B.. Les débuts de
CRISPR en thérapie génique . Med Sci (Paris).
2016; ; 32 :
:1035.–1037. 10. Sharma
R,
Anguela
XM,
Doyon
Y, et al.
In vivo genome editing of the albumin locus as a platform for
protein replacement therapy . Blood.
2015; ; 126 :
:1777.–1784. 11. Hirsch
T,
Rothoeft
T,
Teig
N, et al.
Regeneration of the entire human epidermis using transgenic stem
cells . Nature.
2017; ; 551 :
:327.–332. 12. Lee
C,
Bao
G,
Porteus
MH, et al. Gene
editing with Crispr-Cas9 for treating
beta-hemoglobinopathies . Blood.
2015;; 126. 13. Charlesworth
C,
Desphande
P,
Dever
D, et al. Identification of
pre-existing adaptive immunity to Cas9 proteins in humans .
BioXriv 2018. https://doi.org/10.1101/243345.. 14. Simhadri
VL,
McGill
J,
McMahon
S, et al.
Prevalence of pre-existing antibodies to CRISPR-associated
nuclease Cas9 in the USA population . Mol Ther
Methods Clin Dev.
2018; ; 10 :
:105.–112. 15. Crudele
JM,
Chamberlain
JS. Cas9 immunity
creates challenges for CRISPR gene editing therapies .
Nat Commun.
2018; ; 9 : :3497.. 16. Chen
Y,
Zhang
Y. Application of
the CRISPR/Cas9 system to drug resistance in breast cancer .
Adv Sci (Weinh).
2018; ; 5 :
:1700964.. 17. Bredel
M,
Jacoby
E. Chemogenomics :
an emerging strategy for rapid target and drug discovery .
Nat Rev Genet.
2004; ; 5 :
:262.–275. 18. la Villa
P.. miniaturisation
pour la découverte de candidats médicaments .
L’Actualité Chimique.
2017; :67.–70. 19. Ks
N. les modificateurs
de la réponse biologique pour réduire l’inflammation : pleins feux sur les
rusques d’infection . Pediatr Child Health.
2012; ; 17 :
:151.–154. 20. Arnoldo
A,
Kittanakom
S,
Heisler
LE, et al.
A genome scale overexpression screen to reveal drug activity in
human cells . Genome Med.
2014; ; 6 : :32.. 21. Konermann
S,
Brigham
MD,
Trevino
AE, et al.
Genome-scale transcriptional activation by an engineered
CRISPR-Cas9 complex . Nature.
2015; ; 517 :
:583.–588. 22. Rusk
N.. CRISPR
gain-of-function screens . Nat Methods.
2015; ; 12 :
:102.–103. 23. le Sage
C,
Lawo
S,
Panicker
P, et al.
Dual direction CRISPR transcriptional regulation screening
uncovers gene networks driving drug resistance . Sci
Rep.
2017; ; 7 : :17693.. 24. Lin
S,
Liu
K,
Zhang
Y, et al.
Pharmacological targeting of p38 MAP-kinase 6 (MAP2K6) inhibits
the growth of esophageal adenocarcinoma . Cell
Signal.
2018; ; 51 :
:222.–232. 25. Banerjee
S,
Ji
C,
Mayfield
JE, et al.
Ancient drug curcumin impedes 26S proteasome activity by direct
inhibition of dual-specificity tyrosine-regulated kinase 2 .
Proc Natl Acad Sci USA.
2018; ; 115 :
:8155.–8160. 26. Sabatine
MS,
Giugliano
RP,
Keech
AC, et al.
Evolocumab and clinical outcomes in patients with cardiovascular
disease . N Engl J Med.
2017; ; 376 :
:1713.–1722. 27. Tao
W,
Yang
A,
Deng
Z,
Sun
Y. CRISPR/Cas9-Based
editing of streptomyces for discovery, characterization, and production of
natural products . Front Microbiol.
2018; ; 9 : :1660.. 28. Zhang
MM,
Wong
FT,
Wang
Y, et al.
CRISPR-Cas9 strategy for activation of silent Streptomyces
biosynthetic gene clusters . Nat Chem Biol.
2017. doi: 10.1038/nchembio.2341.
29. Li
L,
Zheng
G,
Chen
J, et al.
Multiplexed site-specific genome engineering for overproducing
bioactive secondary metabolites in actinomycetes .
Metab Eng.
2017; ; 40 :
:80.–92. 30. Birling
MC,
Herault
Y,
Pavlovic
G. Modeling human
disease in rodents by CRISPR/Cas9 genome editing .
Mamm Genome.
2017; ; 28 :
:291.–301. 31. Bhatia
S,
Daschkey
S,
Lang
F, et al.
Mouse models for pre-clinical drug testing in
leukemia . Expert Opin Drug Discov.
2016; ; 11 :
:1081.–1091. 32. hutchinson
L,
Kirk
R.
high drug attrition rates- where are we going wrong
?
Nat Rev Clin Oncol.
2011;; 8. 33. Perlman
RL. Mouse models of
human disease : an evolutionary perspective . Evol
Med Public Health.
2016; ; 2016 :
:170.–176. 34. Takahashi
T.. Organoids for
drug discovery and personalized medicine . Annu Rev
Pharmacol Toxicol.
2019; ; 59 :
:447.–462. 35. Borestrom
C,
Jonebring
A,
Guo
J, et al.
A CRISP(e)R view on kidney organoids allows generation of an
induced pluripotent stem cell-derived kidney model for drug
discovery . Kidney Int.
2018; ; 94 :
:1099.–1110. 36. Clayton
NP,
Burwell
A,
Jensen
H, et al.
Preparation of three-dimensional (3-D) human liver (HepaRG)
cultures for histochemical and immunohistochemical staining and light
microscopic evaluation . Toxicol Pathol.
2018; ; 46 :
:653.–659. 37. Higuchi
Y,
Kawai
K,
Kanaki
T, et al.
Functional polymer-dependent 3D culture accelerates the
differentiation of HepaRG cells into mature hepatocytes .
Hepatol Res.
2016; ; 46 :
:1045.–1057. 38. Drost
J,
Clevers
H. Organoids in
cancer research . Nat Rev Cancer.
2018; ; 18 :
:407.–418. 39. Watanabe
M,
Buth
JE,
Vishlaghi
N, et al.
Self-organized cerebral organoids with human-specific features
predict effective drugs to combat Zika virus infection .
Cell Rep.
2017; ; 21 :
:517.–532. 40. Vassilev
LT,
Vu
BT,
Graves
B, et al.
In vivo activation of the p53 pathway by small-molecule
antagonists of MDM2 . Science.
2004; ; 303 :
:844.–848. 41. Boccardo
F,
Canobbio
L I. 5-fluorouracil,
twenty-five years later . An appraisal.
Chemioterapia.
1983; ; 2 :
:88.–96. 42. Auclair
G,
Weber
M. Mechanisms of DNA
methylation and demethylation in mammals .
Biochimie.
2012; ; 94 :
:2202.–2211. 43. Holtzman
L,
Gersbach
CA. Editing the
epigenome : reshaping the genomic landscape . Annu
Rev Genomics Hum Genet.
2018; ; 19 :
:43.–71. 44. Bian
S,
Repic
M,
Guo
Z, et al.
Genetically engineered cerebral organoids model brain tumor
formation . Nat Methods.
2018; ; 15 :
:631.–639. |