| |
| Med Sci (Paris). 35(5): 483–485. doi: 10.1051/medsci/2019080.Extension du domaine du codage : l’ADN
hachimoji Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS ;
CoReBio PACA, case 901, Parc scientifique de Luminy,
13288Marseille Cedex 09,
France MeSH keywords: Adénine, Cytosine, ADN, Guanine, Liaison hydrogène, Structure moléculaire, Nucléotides, Oligonucléotides, Biologie synthétique, Thymine, analogues et dérivés, composition chimique, synthèse chimique |

Nous avions rapporté il y a un an le très joli travail réalisé au département de chimie
du Scripps Research Institute (La Jolla, Californie), associé à
l’entreprise Synthorx, qui avait réussi à synthétiser deux nouvelles
bases analogues aux classiques C, T, A et G et qui pouvaient former dans l’ADN une
nouvelle paire de bases. Ces chercheurs avaient même réussi à faire répliquer cet ADN
par une bactérie, à assurer (grâce à diverses manipulations de haute volée) sa
transcription et sa traduction et ainsi à insérer dans une protéine un acide aminé
anormal (ne faisant pas partie des vingt acides aminés classiques) [1, 2] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 2, février
2018, page 179
Bien que très intéressant et novateur, ce travail comportait une limite : les deux bases
artificielles ne forment pas de liaisons hydrogène au sein de la double hélice de l’ADN
(comme le font G et C, d’une part, A et T, d’autre part) et leur association repose sur
la compatibilité de leurs formes. Du coup, une paire artificielle doit être entourée de
bases naturelles pour assurer la stabilité de la double hélice, ce qui limite sévèrement
la gamme des séquences réalisables. Un article récemment publié dans la revue
Science par un ensemble de laboratoires et d’entreprises démontre,
lui, la possibilité de concevoir des bases alternatives qui peuvent s’associer par des
liaisons hydrogène et respectent ainsi la structure de la double hélice [3]. Ces auteurs obtiennent ainsi un système
baptisé « hachimoji » comprenant huit (« hachi » en japonais) lettres (« moji ») — les
quatre habituelles plus quatre nouvelles — et montrent qu’il possède les propriétés
minimales nécessaires à un système biologique capable de porter de l’information. |
De nouvelles « lettres » respectant la structure en double hélice Au terme d’un important travail de chimie, les auteurs ont obtenu quatre nouvelles
bases dont la structure est assez proche (mais différente) des bases classiques : P
et Z (qui peuvent s’apparier par trois liaisons hydrogène), et S et B (trois
liaisons également). Elles sont représentées dans la Figure 1 sous forme de déoxynucléotides, tels
qu’ils existent dans l’ADN.
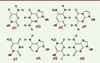 | Figure 1.
Les deux couples de bases classiques (C-G et T-A), à
gauche, et les deux nouveaux couples Z-P, et
S-B. Les liaisons hydrogène se forment entre atomes
donneurs (H) et accepteurs (O ou N). dR désigne la chaîne de
désoxyribose à laquelle est attachée chaque base. On note que le couple
S-B peut former trois liaisons hydrogène au lieu de deux dans le couple
T-A (extrait partiel et remanié de la figure 1 de [ 3]). |
A priori, compte tenu de l’encombrement de ces molécules (très
proche de celui des bases classiques) et de leur capacité à former des liaisons
hydrogène, on peut s’attendre à ce qu’elles s’incorporent dans la double hélice de
l’ADN sans la déformer. Ce point est examiné tout d’abord en synthétisant des
oligonucléotides auto-complémentaires contenant deux (par exemple GCPTAZGC) ou
quatre (par exemple CSZATPBG) de ces nouveaux nucléotides. Chacun de ces
oligonucléotides peut former un duplex avec lui-même1,, et l’on peut alors établir une courbe de fusion et mesurer la
température de fusion (Tm) qui indique le degré de stabilité de la double hélice
précédemment formée. Les Tm obtenus pour une série de presque cent duplex hachimoji
sont conformes à la prédiction pour une double hélice, indiquant donc que cette
structure s’est bien formée et qu’elle est aussi stable qu’une double hélice «
naturelle ». Pour aller plus loin et observer directement la double hélice, les
auteurs ont utilisé une astuce, la possibilité de cristalliser facilement des
complexes entre un segment d’ADN double brin et un fragment d’une protéine virale,
complexes dans lesquels chaque extrémité du duplex est liée à un fragment
protéique2, l’essentiel de l’ADN restant
libre d’adopter sa structure en double hélice [4]. Ils ont pour cela synthétisé trois
oligonucléotides auto-complémentaires longs de 16 bases et contenant quatre
nouvelles bases. Après formation des duplex, association avec les fragments
protéiques et cristallisation, la structure obtenue est bien une double hélice dont
les paramètres sont conformes à ceux d’une structure naturelle. La
Figure 2 montre la structure
obtenue pour la double hélice formée par l’oligonucléotide CTTATPPSBZZATAAG apparié
à lui-même.
 | Figure 2.Structure tridimensionnelle par cristallographie aux rayons
X du duplex formé par l’oligonucléotide auto-complémentaire
CTTATPPSBZZATAAG. Cette double hélice correspond
précisément à la forme B de l’ADN (extrait partiel et remanié de la
figure 3 de [ 3]). |
On voit donc que ces quatre nouvelles bases peuvent être incorporées dans l’ADN sans
perturber la structure en double hélice, respectant ainsi une propriété fondamentale
de l’ADN, celle de pouvoir contenir n’importe quelle suite de bases sans que cela ne
perturbe sa structure tridimensionnelle, propriété essentielle pour une molécule
devant coder de l’information. |
L’ADN hachimoji peut être transcrit Pour être mise en œuvre, l’information contenue dans l’ADN doit être transcrite en
ARN messager pour enfin être traduite en protéine. L’étape suivante a donc
logiquement consisté à obtenir la transcription de l’ADN hachimoji. En fait, la
classique ARN polymérase du bactériophage T7 s’est avérée capable de transcrire dP,
dS et dZ (déoxyribonucléotides dans l’ADN) en leurs ribonucléotides complémentaires
(Z, B et P), seule la transcription de dB (en S) ne fonctionnait pas. Mais il existe
des variants de cette enzyme obtenus dans le cadre de projet d’amélioration menés
par des entreprises de biotechnologie, et l’un d’eux, l’ARN polymérase T7 dite FAL,
est capable d’effectuer cette opération. Il est donc possible grâce à ce variant de
transcrire un ADN hachimoji en ARN – lui aussi hachimoji, c’est-à-dire incluant les
bases anormales Z, B, P et S sous forme, bien sûr, de ribonucléotides. L’article [3] se termine sur la synthèse d’un
oligonucléotide 84-mer contenant quatre nouvelles bases et la démonstration que cet
oligonucléotide conserve sa capacité à émettre de la fluorescence après liaison d’un
composé dérivé de l’imidazolinone, lorsque les nouvelles bases sont situées loin du
site de liaison de ce produit, et les perd lorsqu’elles sont à proximité. C’est un
premier pas (encore modeste) vers des applications fonctionnelles de ce système. |
Le chemin qui reste, les possibilités déjà ouvertes Si l’on compare ce travail à celui publié il y a plus d’un an [1], il est clair qu’il va beaucoup moins loin sur le plan
fonctionnel : l’ADN hachimoji peut certes être transcrit, mais on n’a pas de données
précises sur la fidélité de cette transcription. La structure de cet ADN est bien en
double hélice, mais sa réplication (in vitro ou in
vivo) n’a pas été démontrée. Et avant d’envisager la synthèse de
nouvelles protéines grâce à ce code étendu à huit lettres, il faudrait mettre en
place tous les intermédiaires indispensables à la traduction : ARN de transfert
reconnaissant de nouveaux codons, enzymes capables de charger ces ARN avec les
acides aminés « exotiques » que l’on voudra incorporer dans les protéines… L’examen
des travaux précédents [1, 2] montre l’ampleur de la tâche. Il n’en reste pas moins que l’ADN hachimoji a déjà de nombreuses applications
potentielles. Le codage par huit bases différentes augmente considérablement la
quantité d’information que peut porter un segment d’ADN (un décamère hachimoji a
810 séquences possibles au lieu de 410 pour un décamère
classique, soit 1 024 fois plus). Cela peut être très utile pour différents systèmes
dits de barcoding ou d’étiquetage combinatoire [5], de plus en plus employés dans le
secteur du diagnostic, qui utilisent la séquence de petits segments d’ADN pour
caractériser différentes fractions ou pour afficher le résultat final. Pour les
projets de stockage de l’information dans l’ADN déjà évoqués dans ces chroniques
[6] (→), un
code à huit lettres présente lui aussi des avantages évidents. Un autre domaine de
recherche très actif, celui des nanostructures à base d’ADN [7], pourrait également bénéficier de ces
nouvelles possibilités. Une entreprise, Firebird Biomolecular
Sciences3 se charge d’ailleurs de
commercialiser ces applications, et près de la moitié des auteurs du présent article
sont impliqués dans cette firme.
(→) Voir la Chronique génomique de B. Jordan, m/s n° 6-7,
juin-juillet 2018, page 622
Sur un plan plus philosophique, et comme déjà mentionné précédemment [2], ce travail confirme que les bases C, A, T et
G utilisées par tous les organismes terrestres ne sont pas les seules possibles.
Leur ubiquité dans notre monde vivant reflète simplement le fait que nous descendons
tous du même organisme qui s’est trouvé fonctionner avec ces éléments : le hasard de
la constitution du premier « réplicateur » s’est combiné avec la nécessité de la
sélection naturelle pour aboutir aux organismes d’hier et d’aujourd’hui. Mais nous
aurions bien tort, dans nos tentatives de découvrir des traces de vie sur Mars, sur
le satellite Europe (une lune de Jupiter) et peut-être, plus tard, sur des planètes
extrasolaires, de cibler trop précisément nos analyses sur des molécules semblables
à celles que nous connaissons. Même si une vie apparue ailleurs est fondée sur la
chimie du carbone, de l’oxygène et de l’azote, même si elle utilise une molécule
d’ADN comme mémoire génétique, il n’y a aucune raison pour s’attendre à ce que les
bases alignées le long de cette molécule soient nos habituels C, A, T ou G… |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1. Zhang
Y,
Ptacin
JL,
Fischer
EC, et al.
A semi-synthetic organism that stores and retrieves increased
genetic information . Nature .
2017; ; 551 :
:644.–647. 2. Jordan
B.. Bases
alternatives et organismes synthétiques . Med Sci
(Paris) . 2018; ; 34 :
:179.–182. 3. Hoshika,
Leal
NA,
Kim
MJ, et al.
Hachimoji DNA and RNA: a genetic system with eight building
blocks . Science.
2019;; 363 :
:884.–7. 4. Coté
ML,
Yohannan
SJ,
Georgiadis
MM. Use of an
N-terminal fragment from moloney murine leukemia virus reverse transcriptase
to facilitate crystallization and analysis of a pseudo-16-mer DNA molecule
containing G-A mispairs . Acta Crystallogr D Biol
Crystallogr . 2000; ; 56 :
:1120.–1131. 5. Tsang
HF,
Xue
VW,
Koh
SP, et al.
NanoString, a novel digital color-coded barcode technology:
current and future applications in molecular diagnostics .
Expert Rev Mol Diagn . 2017; ;
17 : :95.–103. 6. Jordan
B.. L’ADN comme
mémoire informatique ? . Med Sci (Paris) .
2018; ; 34 :
:622.–625. 7. Linko
V,
Ora
A,
Kostiainen
MA. DNA
nanostructures as smart drug-delivery vehicles and molecular
devices . Trends Biotechnol .
2015; ; 33 :
:586.–594. |