| |
| Med Sci (Paris). 35(6-7): 515–518. doi: 10.1051/medsci/2019108.Identification de cibles thérapeutiques et
repositionnement de médicaments par analyses de réseaux géniques Andrée Delahaye-Duriez,1,2,3 Clémence Réda,1,4 and Pierre Gressens1 1NeuroDiderot, UMR 1141, Inserm, Université de Paris,
Sorbonne Paris Cité, Hôpital Robert Debré, 48, boulevard Sérurier, 75019Paris,
France. 2Université Paris 13, Sorbonne Paris Cité, UFR de santé,
médecine et biologie humaine, 93000Bobigny,
France. 3Service histologie-embryologie-cytogénétique-biologie de la
reproduction-CECOS, Hôpital Jean Verdier, AP-HP,
93140Bondy,
France. 4École Normale Supérieure Paris-Saclay,
94230Cachan,
France. MeSH keywords: Découverte de médicament, Repositionnement des médicaments, Régulation de l'expression des gènes, Réseaux de régulation génique, Humains, Récepteurs de surface cellulaire, méthodes, analyse |
Développement et validation de thérapies Malgré les avancées technologiques et l’amélioration des connaissances sur les
maladies humaines, les progrès thérapeutiques restent lents. L’identification et la
validation de cibles thérapeutiques demeurent des étapes critiques du processus de
découverte de médicaments. Le développement de nouvelles molécules est long et
coûteux. La validation de l’efficacité des molécules candidates et les démarches
nécessaires pour obtenir une mise sur le marché représentent des investissements
importants pour l’industrie pharmaceutique. Une alternative efficace est le
repositionnement de médicaments ou drug repurposing. Le
repositionnement de médicament consiste à utiliser des molécules déjà présentes sur
le marché pour les appliquer à d’autres maladies qui ne relèvent pas de l’indication
médicale d’origine [1]
(→).
(→) Voir l’Éditorial de C. Martinat et M. Peschanski, m/s n°
12, décembre 2018, page 1019
|
Identification des réseaux de régulation génique perturbés en pathologie La caractérisation d’une maladie en termes de perturbations de réseaux de régulation
génique, ou GRN (gene regulatory network) offre des opportunités
considérables pour accélérer l’identification de cibles thérapeutiques et guider le
repositionnement de médicaments. Les GRN sont des réseaux complexes de gènes, de
protéines, et de petites molécules dont les interactions contrôlent les fonctions
biologiques. Grâce aux efforts concertés de la génétique, de la biologie
moléculaire, de la biochimie et de la physiologie, des quantités énormes de données
portant sur les différents composants de ces réseaux sont maintenant disponibles.
Les domaines de la génomique fonctionnelle et de la biologie des systèmes
développent et utilisent des modèles mathématiques afin d’étudier et de prédire le
comportement de ces GRN. Une des approches pour les identifier est d’étudier la
co-expression des gènes. Si l’on dispose de suffisamment de profils d’expression
recueillis à partir de plusieurs échantillons et selon plusieurs conditions
expérimentales, on peut alors évaluer la similarité de l’expression de chaque paire
de gènes et construire un réseau génique à partir duquel il sera possible de
regrouper les gènes présentant un profil d’expression similaire et ainsi construire
des modules de gènes co-exprimés (Figure
1). Ces modules sont intéressants sur le plan biologique.
Ils sont en effet fonctionnellement liés et codent des protéines qui, pour la
plupart, interagissent entre elles. Ils reflètent également les GRN en révélant leur
contrôle et les liens de régulation qui les régissent [2]. L’intégration avec des données de
susceptibilité génétique ou des informations phénotypiques permet de connecter des
modules de gènes co-exprimés ou des GRN à des états pathologiques. Ainsi, on peut
identifier des voies de transmission de signaux cellulaires impliqués dans une
maladie et leurs régulateurs comme de nouvelles cibles thérapeutiques [3]. Il existe plusieurs
exemples fructueux dans lesquels l’application d’approches de cartographie
d’expression de traits quantitatifs d’expression (expression quantitative
trait loci, eQTL) à des modules de gènes co-exprimés associés à une
maladie a permis l’identification de régulateurs clés comme celle de possibles
cibles thérapeutiques [4-6]. Pourtant,
les régulateurs identifiés ne constituent pas toujours des cibles facilement
exploitables d’un point de vue thérapeutique. C’est pourquoi d’autres approches
intègrent les régulations liées spécifiquement aux protéine kinases, comme X2K
(programme eXpression2kinase) [7] ou aux récepteurs membranaires [8].
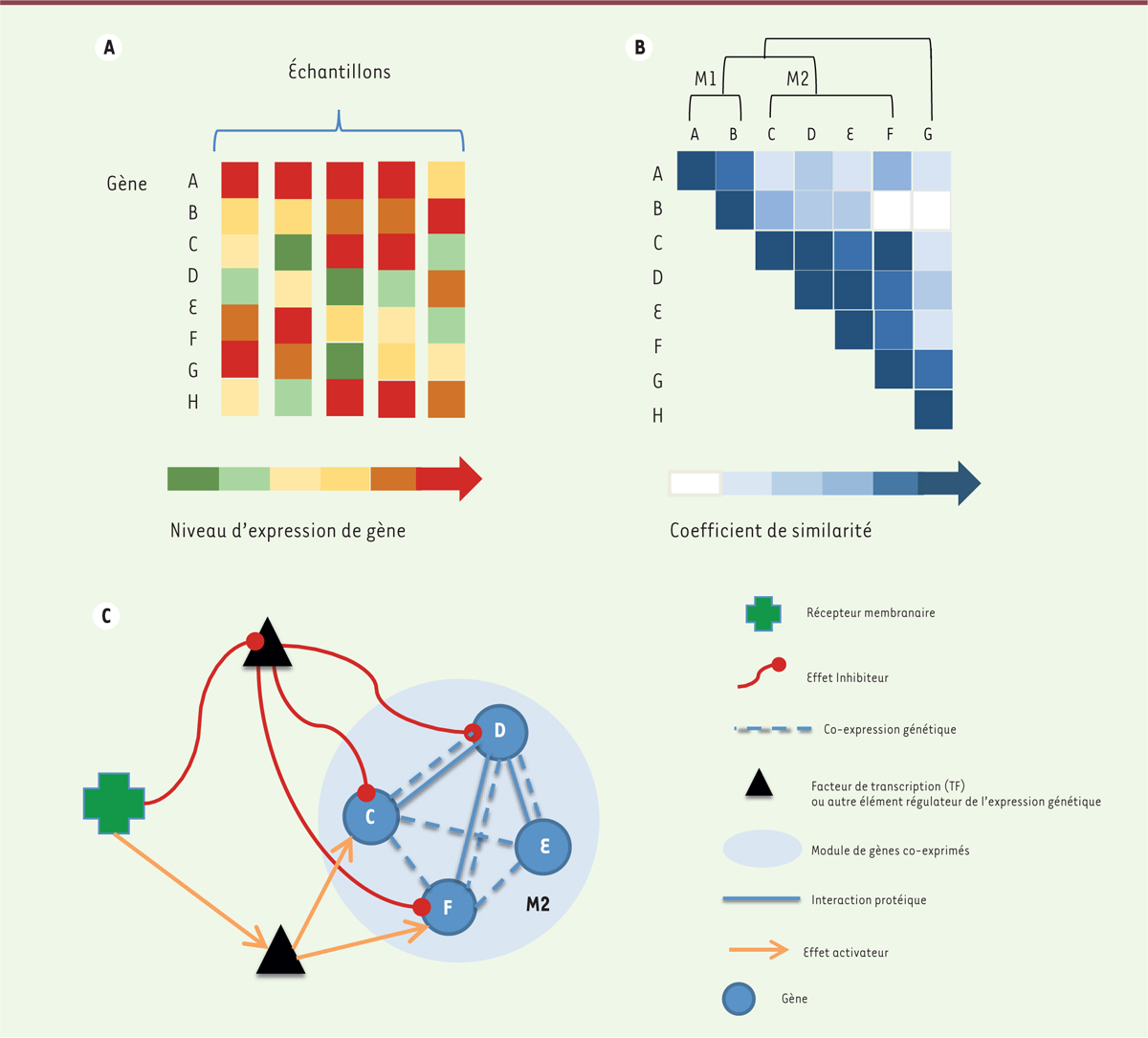
 | Figure 1. Du profil d’expression au réseau de régulation génique.
A. Les profils d’expression pour
chaque gène considéré (ici, on considère 7 gènes notés A, B, C, D, E, F
et G) sont construits à partir de données transcriptomiques issues de
différents échantillons, en concaténant les niveaux d’expression du gène
considéré pour chaque condition testée.
B..On peut inférer les modules de
coexpression de gènes en calculant la distance entre les profils
d’expression de chaque paire de gènes d’intérêt et en appliquant une
méthode de classification non supervisée afin de les grouper en modules
de gènes ayant des niveaux d’expression similaires sous les mêmes
conditions (ici, deux modules sont inférés, M1 comportant les gènes A et
B, et M2 contenant les gènes C, D, E et F).
C. Réseau de régulation génique ou GRN
(gene regulatory network) considérant les éléments
régulateurs de l’expression des gènes du module M2, par exemple, les
facteurs de transcription (TF), et un récepteur membranaire. Les
interactions de régulation (activatrices ou inhibitrices de
l’expression) sont soit inférées par corrélation entre les niveaux
d’expression, soit prouvées expérimentalement (et une relation causale
est alors établie). |
|
Exemple d’identification d’un récepteur membranaire comme cible
thérapeutique Dans un travail interdisciplinaire coordonné par Michael Johnson et Enrico Petretto,
en collaboration avec UCB Pharma et Clarivates
Analytics, une série d’analyses appelée CRAFT (causal reasoning
analytical framework for target discovery) a été développée de façon à
sélectionner les récepteurs membranaires participant à la régulation de l’expression
génique dans la direction opposée à celle observée en pathologie [8]. Comme preuve de concept, CRAFT a été
appliqué à l’épilepsie et a permis d’identifier CSF1R (colony-stimulating
factor 1 receptor) comme une cible thérapeutique. Dans un modèle murin
d’épilepsie du lobe temporal (TLE, temporal lobe epilepsy) induite
par l’injection de pilocarpine, les réseaux de transcription dans les hippocampes
ont été analysés et des modules de gènes co-exprimés associés à la maladie
épileptique ont été isolés. Douze modules se sont révélés être différemment
co-exprimés de manière significative entre des hippocampes épileptiques et des
hippocampes témoins (100 échantillons dans les deux cas). Pour hiérarchiser
davantage les modules en fonction de leur relation à l’épilepsie, la corrélation
entre l’expression de chaque module et la fréquence des crises a été explorée. Parmi
les 9 modules dont l’expression était reliée aux crises, le module 18 s’est révélé
être le plus significativement corrélé aux convulsions. Ce module était
particulièrement enrichi en gènes connus pour coder des protéines impliquées dans
les processus inflammatoires ainsi qu’en gènes marqueurs des cellules microgliales.
Sept modules, dont ce module 18, sont conservés chez l’homme. Ils sont également
co-exprimés de manière significativement différente dans les pièces chirurgicales
d’hippocampes de patients atteints d’épilepsie du lobe temporal par rapport aux
données obtenues à partir de prélèvements post-mortem sur des hippocampes
d’individus dépourvus d’antécédent psychiatrique ou neurologique. La suite
d’analyses CRAFT a été appliquée aux 7 modules candidats sélectionnés. CRAFT permet
de modéliser les différents scénarios de régulation induite par des récepteurs
membranaires sur des facteurs de transcription et leurs gènes cibles. Des tests
statistiques ont permis d’ordonner ces récepteurs membranaires selon leur influence
sur les gènes du module testé, dans une direction opposée à celle observée dans les
hippocampes épileptiques. CSF1R (colony-stimulating factor 1
receptor) a été ainsi prédit comme un régulateur de deux des sept
modules candidats (dont le module 18). Il a alors été émis l’hypothèse que le
blocage de ce récepteur pourrait être une cible thérapeutique dans le traitement de
l’épilepsie. La disponibilité du PLX3397 (Pexidartinib), un
inhibiteur connu de CSF1R, a permis de tester expérimentalement cette hypothèse. Le
blocage de CSF1R a atténué les crises épileptiques dans trois modèles précliniques
d’épilepsie, ce qui a permis de confirmer qu’il était une cible thérapeutique
potentielle pour lutter contre l’épilepsie acquise. CSF1R est un récepteur
membranaire exprimé par les cellules de la lignée myéloïde, notamment les monocytes,
les macrophages et la microglie. Il a été suggéré que la survie des cellules
microgliales dépendait de la signalisation induite par CSF1R, un traitement prolongé
à fortes doses de PLX3397 (c’est-à-dire 5 à 6 fois supérieure à celle utilisée dans
le travail de Srivastava et al. [8]) entraînant une réduction du nombre de cellules microgliales dans le
cerveau de souris naïves [9].
Aux doses faibles de PLX3397, telles qu’utilisées par Srivastava et
al. [8], il n’y a pas de mort
cellulaire des cellules microgliales, mais les changements d’expression des gènes du
module 18 associés à l’épilepsie sont inversés. L’efficacité antiépileptique de
PLX3397 a été démontrée dans deux modèles murins d’épilepsie du lobe temporal : le
modèle utilisant la pilocarpine et celui impliquant le kainate1,. Dans les deux modèles, PLX3397 a significativement réduit la
fréquence ou la durée des crises spontanées. En revanche, PLX3397 n’a pas montré
d’efficacité dans trois modèles de crises aiguës chez les souris non épileptiques :
le modèle de crise par électrochoc maximal, le modèle de crise à 6 Hz2, et le modèle au PTZ (pentylènetétrazole)3. Ces données distinguent clairement le PLX3397
de tous les médicaments antiépileptiques/anticonvulsivants actuellement utilisés,
qui sont généralement efficaces dans de tels modèles de crises épileptiques. Cette
étude démontre donc que la restauration de l’expression des modules liés à la
maladie vers un état sain permet de prédire l’efficacité thérapeutique et ouvre le
pas à une accélération de l’identification et de la validation de nouvelles cibles
thérapeutiques non seulement pour le traitement de l’épilepsie, mais également pour
celui d’autres maladies. |
Application pour le repositionnement de médicaments La quantification de l’inversion des changements d’expression génique associés à un
état pathologique peut être exploitée pour prédire l’efficacité des médicaments. Les
composés thérapeutiques sont alors jugés non pas par leur affinité de liaison avec
une protéine particulière, mais par leur capacité à induire une réponse
transcriptionnelle anti-corrélée au programme de régulation de l’expression génique
qui sous-tend l’état pathologique ou l’exposition à un facteur de risque. Les
approches fondées sur ce paradigme de l’inversion de signature bénéficient de bases
de données publiques de profils d’expression des gènes de lignées cellulaires
traitées avec des composés chimiques, comme CMap (connectivity map)
[10]. Par exemple,
parmi les nombreux médicaments référencés dans cette base de données, l’acide
valproïque, un médicament antiépileptique largement utilisé, a été prédit comme le
meilleur pour inverser la signature d’expression génique d’un module associé à
l’épilepsie [11]. Dans la
continuité de CMap, la base de données LINCS (library of integrated
network-based cellular signatures, http://www.lincsproject.org)
des NIH (National institutes of health, États-Unis) s’enrichit
considérablement et contient aujourd’hui des profils d’expression pour plus de
20 000 molécules [12,13]. Ces données en accès
libre représentent une énorme ressource pour effectuer des criblages in
silico de molécules. La création de méthodes d’exploitation
systématique de ces données pour générer de nouvelles hypothèses d’efficacité de
médicaments est un défi qui nécessite la collaboration de chercheurs spécialisés en
mathématiques, en informatique et en biologie. Ces recherches préparent la médecine
de précision de demain qui aura, en plus, le défi d’intégrer les données
individuelles des patients pour adapter leur traitement de façon personnalisée. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
Footnotes |
1. Martinat
C,
Peschanski
M. L’heure est venue
du (re)positionnement thérapeutique des maladies
ultra-rares . Med Sci (Paris).
2018; ; 34 :
:1019.–1021. 2. Weirauch
MT,
Dehmer
M,
Emmert-Streib
F,
Salvador
A,
Graber
A. Gene coexpression
networks for the analysis of DNA microarray data .
Applied statistics for network biology: methods in systems
biology.
2011 ; New York:
Wiley;
:215.. 3. Califano
A,
Butte
AJ,
Friend
S, et al.
Leveraging models of cell regulation and GWAS data in integrative
network-based association studies . Nat
Genet.
2012; ; 44 :
:841.–847. 4. Heinig
M,
Petretto
E,
Wallace
C, et al.
A trans-acting locus regulates an anti-viral expression network
and type 1 diabetes risk . Nature.
2010; ; 467 :
:460.–464. 5. Kang
H,
Kerloc’h
A,
Rotival
M, et al.
Kcnn4 is a regulator of macrophage multinucleation in bone
homeostasis and inflammatory disease . Cell
Rep.
2014; ; 8 :
:1210.–1214. 6. Johnson
MR,
Behmoaras
J,
Bottolo
L, et al.
Systems genetics identifies Sestrin 3 as a regulator of a
proconvulsant gene network in human epileptic hippocampus .
Nat Commun.
2015; ; 6 : :6031.. 7. Clarke
DJB,
Kuleshov
MV,
Schilder
BM, et al.
eXpression2Kinases (X2K) web: linking expression signatures to
upstream cell signaling networks . Nucleic Acids
Res.
2018; ; 46 :
:W171.–W179. 8. Srivastava
PK, van
Eyll
J,
Godard
P, et al.
A systems-level framework for drug discovery identifies Csf1R as
an anti-epileptic drug target . Nat Commun.
2018; ; 9 : :3561.. 9. Elmore
MRP,
Najafi
AR,
Koike
MA, et al.
Colony-stimulating factor 1 receptor signaling is necessary for
microglia viability, unmasking a microglia progenitor cell in the adult
brain . Neuron.
2014; ; 82 :
:380.–397. 10. Lamb
J,
Crawford
ED,
Peck
D, et al.
The connectivity map: using gene-expression signatures to connect
small molecules, genes, and disease .
Science.
2006; ; 313 :
:1929.–1935. 11. Delahaye-Duriez
A,
Srivastava
P,
Shkura
K, et al.
Rare and common epilepsies converge on a shared gene regulatory
network providing opportunities for novel antiepileptic drug
discovery . Genome Biol.
2016; ; 17 : :245.. 12. Subramanian
A,
Narayan
R,
Corsello
SM, et al.
A next generation connectivity map: L1000 platform and the first
1,000,000 profiles . Cell.
2017; ; 171 : (1437–52)
:e17.. 13. Keenan
AB,
Jenkins
SL,
Jagodnik
KM, et al.
The library of integrated network-based cellular signatures NIH
program: system-level cataloging of human cells response to
perturbations . Cell Syst.
2018; ; 6 :
:13.–24. |