| |
| Med Sci (Paris). 35(6-7): 589–591. doi: 10.1051/medsci/2019099.Slide-seq : L'analyse d'expression à haute résolution
spatiale Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université /EFS/CNRS ; CoReBio
PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09,
France. MeSH keywords: Animaux, Analyse de profil d'expression de gènes, Hétérogénéité génétique, Séquençage nucléotidique à haut débit, Humains, Souris, Séquençage par oligonucléotides en batterie, Analyse de séquence d'ADN, Analyse sur cellule unique, Distribution tissulaire, Fixation tissulaire, méthodes, tendances |

La mise au point des puces à ADN (ou microarrays) puis, plus tard, l’emploi du séquençage
à haut débit ont permis de multiples avancées dans la connaissance de l’expression des
gènes. Mais l’étude détaillée des gènes actifs au sein des différentes régions d’un
tissu ou d’un organe restait peu accessible puisque l’analyse portait sur l’ensemble des
ARN messagers obtenus par extraction à partir de l’échantillon. La microdissection d’un
tissu déposé sur une lame fournissait une réponse partielle à ce besoin de localisation,
de même que l’analyse - assez acrobatique - sur cellules uniques. Une première avancée
importante est apparue récemment avec la technique de « transcriptomique spatiale »
[1], mais la résolution
laissait encore à désirer. Une publication toute récente [2] montre qu’il est maintenant possible d’analyser
l’expression des gènes au sein d’un tissu avec une grande finesse. Il s’agit d’une très
importante évolution technique qui va certainement être appliquée dans de nombreux
domaines. |
2016 : une première approche Dans un article paru dans la revue Science à l’été 2016, un ensemble de laboratoires
suédois présente une technique permettant d’analyser l’expression des gènes en
quelques centaines de points au sein d’une coupe de tissus [1]. En fait, ces auteurs ont réalisé des microarrays de petite
taille (6 × 6 mm2) comportant un millier de plots, chacun d’eux portant un
oligonucléotide qui se termine par une série de T (permettant la fixation des ARN
messagers (ARNm) via leur séquence polyA terminale) et incluant un code-barres
unique long d’une dizaine de nucléotides et différent pour chaque plot du
microarray. Une coupe de tissu est déposée sur le microarray, traitée de manière à
permettre la fixation des ARNm aux oligonucléotides ; l’ensemble est ensuite incubé
avec la transcriptase inverse de manière à synthétiser l’ADN complémentaire (ADNc)
correspondant à chaque ARNm (et portant le code-barres du plot au niveau duquel se
trouvaient ces ARNm). L’ensemble est alors détaché du support et séquencé. Le
code-barres associé à chaque plot permet de relier chacune de ces séquences à un
point précis du microarray : on a ainsi, pour chaque point de l’échantillon
correspondant à un plot, un profil d’expression répertoriant les gènes exprimés dans
le tissu à cet endroit. Bien entendu, cette méthode a nécessité de nombreuses mises
au point pour assurer la fixation des ARNm aux oligonucléotides des plots et le bon
déroulement des étapes suivantes, mais le résultat est convaincant, comme le montre
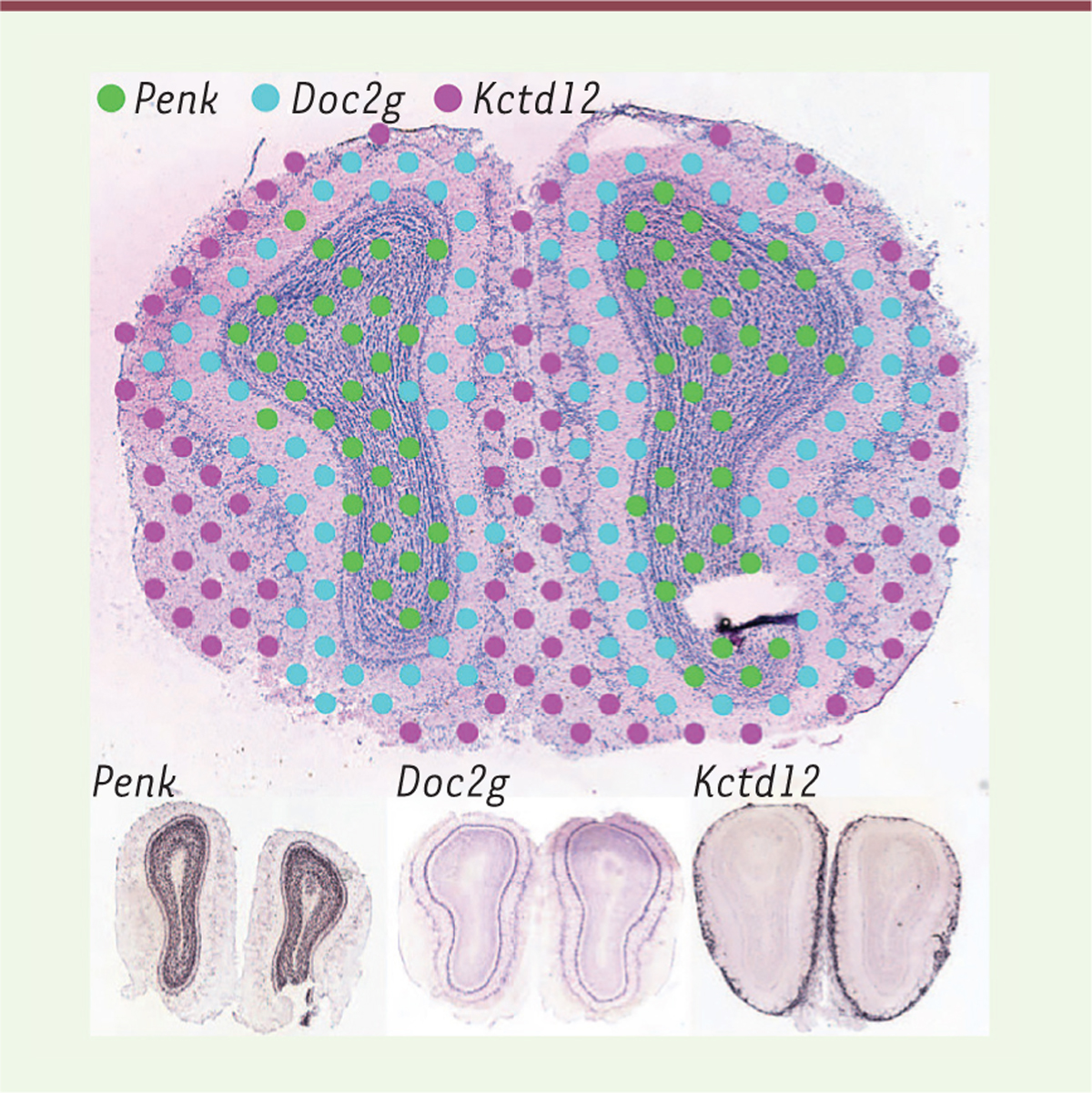
la Figure 1. La limite de ce procédé est liée à
la résolution du microarray : ses plots sont espacés de 200 µM, ce qui donne une
résolution relativement grossière par rapport à celle de l’hybridation in situ,
comme on peut le voir sur la Figure 1 (comparer
les spots en haut au marquage en bas). Notons néanmoins que, pour chaque plot, on a
une information sur l’ensemble des gènes exprimés, alors que l’hybridation in situ
n’examine qu’un seul gène à la fois.
 | Figure 1.Localisation des transcrits par « transcriptomique spatiale ». En haut :
coupe de bulbe olfactif de souris montrant la localisation des plots du
microarray, colorés selon le code indiqué en haut
si l’expression du gène correspondant est importante dans le profil
d’expression obtenu. En bas : hybridation in situ sur
des coupes similaires avec chacun de ces trois gènes. On voit que les
localisations correspondent, avec une résolution bien meilleure en
hybridation in situ – mais une information limitée à un
seul gène par coupe (extrait partiel et remanié de la figure 2 de [ 1]). |
|
2019 : une résolution quasi-cellulaire Fin mars 2019, plusieurs équipes états-uniennes du MIT (Massachusetts institute of
technology) et du Broad Institute of Harvard (Cambridge, MA) publie une technique
baptisée Slide-seq [2], qui permet une
visualisation beaucoup plus fine, quasiment à l’échelle cellulaire, de l’expression
des gènes dans une coupe de tissu. Dans ses grandes lignes, l’approche ressemble à
la précédente, mais le microarray (1 000 plots avec un pas de 200 µM) est remplacé
par un « palet » (puck, comme le palet de hockey sur glace) de trois millimètres de
diamètre portant des dizaines de milliers de billes (d’un diamètre de 10 µM),
chacune étant, comme précédemment, couplée à un oligonucléotide comportant une
séquence dT et un code-barres. Le mélange des billes est déposé et fixé sur la
surface du palet ; le palet entier est alors séquencé par la technique SOLiD1 qui permet d’identifier quel code-barres porte
quelle bille dans le palet. À l’issue de ce « décodage », on a donc un palet portant
un très grand nombre de billes codées (environ 70 000), dont les positions sont
connues, et qui va permettre d’interroger avec une grande finesse une coupe de
tissus. La suite ressemble donc à ce qui a été décrit précédemment : une coupe de tissu
(coupe congelée de 10 µM d’épaisseur) est déposée sur le palet ; la capture des ARNm
présents à chaque endroit est suivie d’une transcription inverse (incluant le
code-barres). Après décrochage de l’ensemble des produits de transcription inverse,
un séquençage à forte redondance du mélange, suivi d’un repérage spatial grâce au
code-barres, permet d’accéder au profil d’expression en chaque point de
l’échantillon. La grande différence est que, maintenant, la résolution est de
l’ordre de 10 µM, soit quasiment au niveau cellulaire, et que l’on mesure plusieurs
milliers de points sur la surface de l’échantillon. Les auteurs montrent comment ils
peuvent identifier divers types cellulaires à travers leurs profils d’expression, et
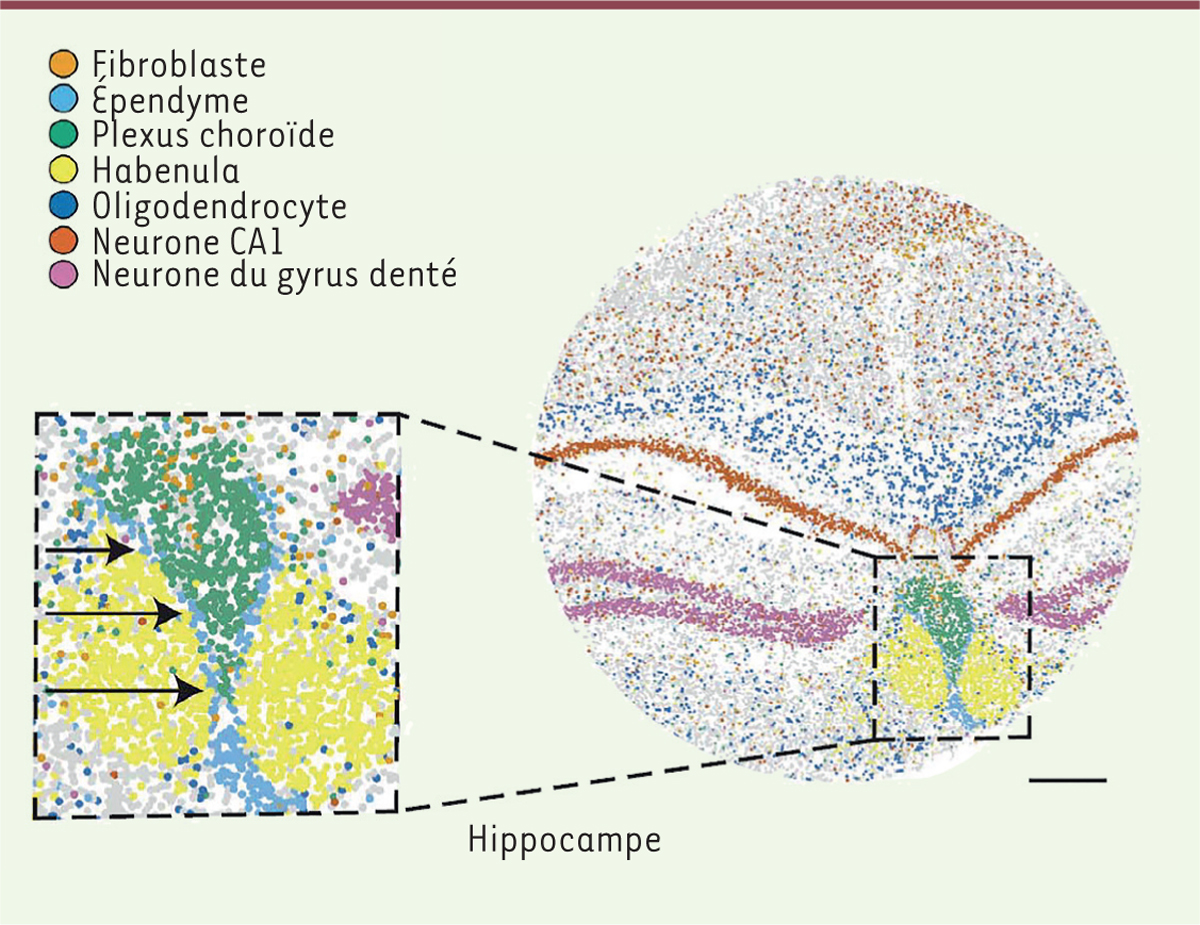
présentent leurs résultats en utilisant ces identifications. La Figure 2 montre par exemple l’analyse d’une coupe coronale (ou
coupe frontale) de l’hippocampe de souris, les points colorés correspondant à des
profils d’expression spécifiques, de type fibroblastique, épendymaire, choroïdal,
etc. On voit que ces images sont très détaillées et bien nettes.
 | Figure 2. Image d’une coupe coronale d’hippocampe de souris obtenue par la
technique Slide-seq. Les plots (environ 70 000 dans un « palet ») ont
été colorés en fonction du profil d’expression obtenu, qui indique le
type cellulaire correspondant, selon le code en haut de la figure
(fibroblaste, épendyme, plexus choroïde, habenula, oligodendrocyte,
neurone CA1, neurone du gyrus denté [désignations anatomiques de
différentes régions de l’hippocampe et des types cellulaires
correspondants]). On voit que l’image obtenue est d’une grande finesse
(les flèches dans l’agrandissement indiquent une couche monocellulaire).
Le palet a un diamètre de 3 mm, la barre horizontale correspond à 500 μM
(extrait partiel et remanié de la figure 1 de [2]). |
|
Une méthode très puissante, de nombreuses applications La technique Slide-seq représente donc un progrès considérable, il suffit de comparer
les deux figures pour s’en persuader. Elle apporte la possibilité de déterminer le
profil d’expression en chaque point d’une coupe de tissu, avec une résolution qui
approche l’échelle cellulaire. Le fait que l’on retrouve, à travers l’analyse de ces
profils d’expression, les morphologies connues pour les organes étudiés (voir la
Figure 2) montre que la diffusion latérale
des ARNm, possible au cours de la procédure, n’est pas un problème. Les abondantes
données obtenues permettent des analyses sophistiquées, dont l’article [2] donne quelques exemples, notamment de
nouvelles localisations de types cellulaires dans différentes régions du cerveau
grâce à la caractérisation des cellules par leur profil d’expression, et à leur
localisation par la technique Slide-seq. Les auteurs montrent aussi qu’ils peuvent
étudier la réponse à une blessure au niveau des changements locaux dans le profil
d’expression des cellules concernées. Nul doute que de nombreuses autres
applications vont émerger, notamment en cancérologie, où la caractérisation
détaillée des tumeurs (dont l’hétérogénéité spatiale est maintenant reconnue) et des
tissus environnants revêt une grande importance. Au niveau technique, les auteurs
indiquent que la majeure partie du coût de l’analyse correspond au séquençage des
ADNc obtenus à la fin de la procédure, soit de 200 à 500 dollars américains par
palet2, et que ce coût va diminuer avec le
progrès technologique. La fabrication de ces palets, et surtout leur décodage
(identification des billes via leur code-barres) ne sont pas à la portée de tous les
laboratoires, mais on peut parier qu’il se trouvera rapidement une start-up
astucieuse pour se charger de leur commercialisation, puisqu’il s’agit d’outils
génériques (un type de palet par organisme). On peut donc s’attendre à une floraison
de résultats, qui vont ouvrir un nouveau chapitre dans l’étude fine des tissus
normaux et pathologiques. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1. Ståhl
PL,
Salmén
F,
Vickovic
S, et al.
Visualization and analysis of gene expression in tissue
sections by spatial transcriptomics .
Science.
2016; ; 353 :
:78.-82. 2. Rodriques
SG,
Stickels
RR,
Goeva
A, et al. Slide-seq:
a scalable technology for measuring genome-wide expression at high spatial
resolution . Science.
2019; ; 363 :
:1463.-7. |