| |
| Med Sci (Paris). 35(8-9): 613–615. doi: 10.1051/medsci/2019117.Un nouveau mécanisme de résistance aux
antibiotiques Le recyclage des ribosomes Mélodie Duval1,2,3 and Pascale Cossart1,2,3* 1Département de biologie cellulaire et infection, Unité des
interactions bactéries-cellules, Institut Pasteur,
25-28, rue du Docteur
Roux, 75015Paris,
France. 2Inserm, U604, F-75015Paris,
France. 3INRA, unité sous contrat H2020, F-75015Paris,
France. MeSH keywords: Protéines bactériennes, Résistance microbienne aux médicaments, Érythromycine, Protéines G, Régulation de l'expression des gènes bactériens, Lincomycine, Listeria monocytogenes, Biosynthèse des protéines, Ribosomes, Transcription génétique, génétique, physiologie, pharmacologie, effets des médicaments et substances chimiques, métabolisme |
L’enjeu de la résistance aux antibiotiques Chez les bactéries, résister aux antibiotiques est un enjeu majeur pour leur survie.
Si ces agents anti-bactériens sont depuis les années 1950 largement utilisés dans
des stratégies thérapeutiques, leur origine est bien plus ancienne. En effet, les
microbes qui les produisent (les champignons, les bactéries) en font usage depuis
toujours afin de coloniser des niches écologiques en inhibant, voire en tuant, leurs
compétiteurs. Afin de résister à ces antibiotiques, les bactéries possèdent
initialement, ou ont acquis par transfert, de nombreux gènes leur permettant de s’en
protéger. Leurs capacités de résistance sont ainsi en fait soit intrinsèques,
c’est-à-dire dues à des propriétés qui leur sont spécifiques, comme la présence
d’une membrane externe qui les protège, soit acquises, provenant d’autres bactéries
via des éléments génétiques mobiles et transmissibles, tels que
les plasmides ou les transposons. Les mécanismes de résistance les plus répandus permettent aux bactéries soit
d’expulser les antibiotiques dès leur pénétration, soit d’en empêcher l’entrée.
Elles peuvent aussi les dégrader ou les modifier, les rendant ainsi inefficaces.
Elles peuvent enfin transformer la cible de l’antibiotique, de sorte que celui-ci ne
puisse plus agir (Figure
1).
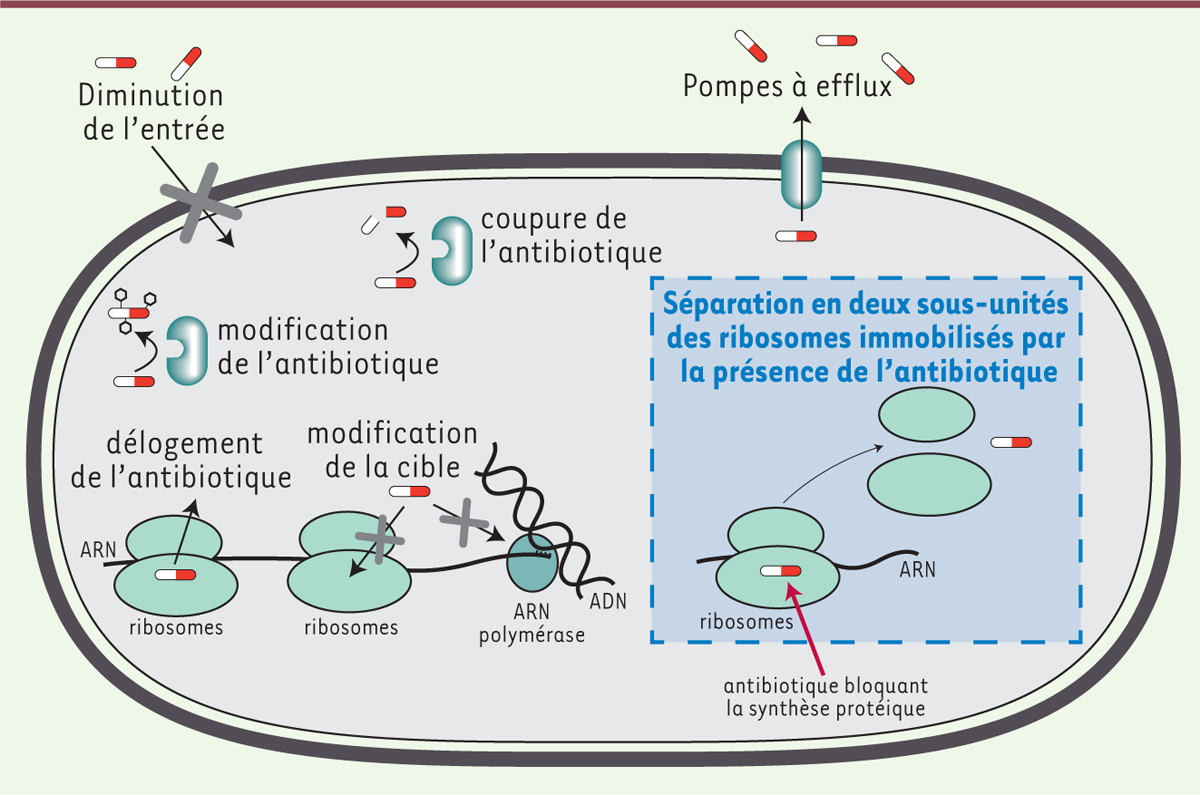
 | Figure 1. Différents types de résistance aux
antibiotiques. Les mécanismes de résistance chez les
bactéries sont variés. Les principaux permettent à la bactérie
d’expulser l’antibiotique ou d’empêcher son entrée, de le dégrader ou le
modifier afin de le rendre inactif, de le déloger ou de modifier la
machinerie cible afin de l’empêcher d’agir. Nous décrivons un nouveau
mécanisme de résistance par lequel les ribosomes bloqués sont dissociés
en deux sous-unités libres, permettant ainsi le redémarrage de la
synthèse protéique. |
|
Un nouveau mécanisme de résistance Notre laboratoire étudie depuis de nombreuses années, la bactérie Listeria
monocytogenes responsable de la listériose, et devenue un modèle pour
la recherche en biologie des infections [1]. Cette bactérie pathogène possède une extraordinaire capacité
à s’adapter aussi bien aux stress de l’environnement, ce qui lui permet, par
exemple, de survivre et de se multiplier sur le sol, qu’aux différents traitements
qu’elle rencontre dans la chaîne alimentaire (addition de sel, congélation, etc.).
Cette adaptabilité résulte d’un arsenal de gènes qu’elle possède, et dont elle
régule finement l’expression, grâce à différents mécanismes comme les
thermoswitchs
1, qui lui permettent de détecter par la
température son entrée dans un organisme, et ainsi d’exprimer des gènes critiques
pour sa virulence [2]. Nos collaborateurs de l’Institut Weizmann (Israël), avec qui nous avions identifié
les sites de démarrage de l’initiation de transcription de tous les gènes de
Listeria exprimés après sa croissance dans différentes
conditions de culture [3],
ont récemment développé une technique, appelée «term-seq»,
permettant d’identifier précisément, non seulement les sites de fin de
transcription, mais aussi la quantité de transcrits produits selon les conditions de
culture. En effet, la transcription de certains gènes, après son démarrage, peut
soit s’arrêter très rapidement, le gène n’est alors pas exprimé, soit continuer
jusqu’à transcription complète du gène (Figure
2). La technique «term-seq» permet
d’identifier les gènes qui sont soumis à cette régulation.
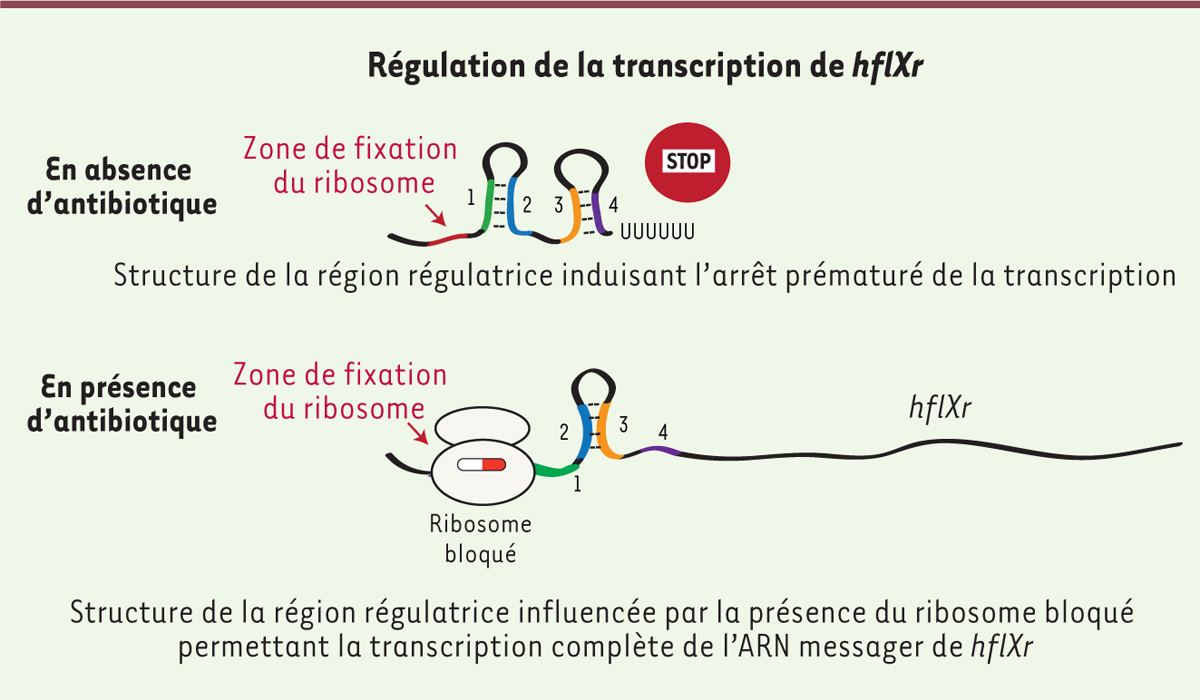
 | Figure 2. Mécanisme d’expression du nouveau gène de résistance
hflXr. L’induction spécifique de hflXr (r pour
résistance) en présence d’antibiotique s’explique par une modulation de
sa transcription via un mécanisme appelé atténuation, par lequel une
région régulatrice située en amont du gène peut adopter deux structures
distinctes : l’une qui induit l’arrêt de la transcription (aboutissant à
un ARN messager court), et une autre qui permet la transcription
complète de l’ARN messager du gène. La région régulatrice contient une
petite phase ouverte de lecture qui agit comme un senseur de la présence
de l’antibiotique : en sa présence, les ribosomes s’y fixent et s’y
arrêtent, empêchant la formation du « terminateur ». En son absence, le
terminateur se forme et la transcription s’arrête. |
Nous avons étudié par cette méthode, le comportement de Listeria en
l’absence ou en la présence de deux antibiotiques, la lincomycine et l’érythromycine
[4]. Ces antibiotiques
agissent sur les ribosomes, une machinerie essentielle à la croissance des
bactéries, et altèrent ainsi la synthèse protéique. Les résultats que nous avons
obtenus montrent que plusieurs gènes sont spécifiquement exprimés en présence de ces
antibiotiques: en leur absence, un court transcrit est détecté, mais le gène
lui-même n’est pas transcrit; en leur présence, un long transcrit est produit et le
gène est alors exprimé. L’un des gènes soumis à cette régulation, le gène hflX, est
particulièrement intéressant. Il avait été étudié chez deux bactéries,
Escherichia coli et Staphylococcus aureus, et
le mode d’action de la protéine produite par ce gène, identifié: la protéine HflX
permet en effet le recyclage des ribosomes qui ont été bloqués par un choc thermique
[5, 6]. HflX dissocie le ribosome en deux
sous-unités libres, qui peuvent se réassembler et réamorcer la synthèse protéique.
Il était donc tentant de penser que chez Listeria monocytogenes ce
gène puisse être un équivalent qui serait actif en présence des antibiotiques qui
bloquent le ribosome. Afin d’étudier si le gène hflX était impliqué dans la résistance aux
antibiotiques de L. monocytogenes, nous l’avons éliminé du génome
de la bactérie, et avons exposé les bactéries à différentes doses d’antibiotiques.
Les bactéries dont le gène hflX avait été supprimé se sont révélées
plus sensibles à l’action de l’antibiotique, démontrant l’implication de HflX dans
la résistance de la bactérie aux antibiotiques. Nous avons renommé
hflXr, ce gène chez L. monocytogenes, «r»
signifiant résistance. Afin de tester si, comme chez S. aureus et E. coli,
hflXr de Listeria participait au recyclage des
ribosomes, nous avons analysé la répartition des sous-unités des ribosomes chez des
bactéries possédant ou non le gène hflXr lorsqu’elles croissent en
présence d’antibiotiques. Une augmentation du nombre de ribosomes assemblés a été
observée dans les bactéries dont le gène hflXr avait été éliminé,
signe d’un dysfonctionnement du recyclage ribosomique. Ainsi, comme chez les autres
bactéries, hflXr semblait impliqué dans le recyclage des ribosomes.
Un tel mécanisme de recyclage, bien connu lors des chocs thermiques, n’avait
jusqu’alors jamais été montré comme jouant un rôle dans la résistance aux
antibiotiques. Ce mécanisme permet le recyclage de sous-unités ribosomiques
fonctionnelles, pourvu qu’un autre mécanisme chasse l’antibiotique de son site
[4]. |
Un gène parfaitement régulé Afin de comprendre les mécanismes permettant l’induction de l’expression du gène
hflXr en présence d’antibiotiques (autrement dit, pourquoi on
observe un transcrit court en absence d’antibiotique et un transcrit long en
présence d’antibiotique), nous avons analysé la région située en amont du gène.
Cette région présente une petite phase de lecture ouverte (ou ORF pour open
reading frame) qui code un peptide. En induisant des mutations dans
cette région du gène, nous avons pu montrer qu’en présence d’antibiotique, cette
séquence n’était pas traduite, les ribosomes effectuant une pause au niveau de cette
région, ce qui empêche la formation d’un «terminateur de transcription». En
l’absence d’antibiotique, cette petite ORF est traduite, et le terminateur de
transcription peut se former, empêchant l’expression du gène hflXr
[7] (→).
(→) Voir la Synthèse de J. Manry et L. Quintana-Murci, m/s
n°12, décembre2012, page1095
Soulignons que deux études de métagénomique fonctionnelle du sol au voisinage d’une
ferme utilisant des antibiotiques, ou près d’une usine fabriquant des antibiotiques,
ont montré la présence de gènes de résistance chez Simkania
negevensis et chez Emergensia timonensis, dont le gène
hflX [8,
9], ce qui renforce sous
un autre angle la pertinence de notre étude sur hflXr. |
Cette étude a donc permis de mettre en évidence un nouveau mécanisme de résistance
aux antibiotiques potentiellement répandu chez de nombreuses bactéries, qui est
fondé sur le recyclage des ribosomes grâce au gène hflXr. Les
niveaux de résistance apportés par ce gène ne permettent pas aux bactéries de
survivre à des concentrations élevées d’antibiotiques, telles que celles utilisées
en thérapeutique, mais nous pensons que ce gène confère à la bactérie qui en est
pourvue un avantage au sein des communautés microbiennes colonisant le sol. Cette
étude montre également comment les bactéries peuvent utiliser un mécanisme de
réponse au stress, tel que le recyclage des ribosomes, dans des contextes
différents: la réponse aux chocs thermiques ou la lutte contre les antibiotiques.
Cette découverte nous révèle aussi que la diversité des mécanismes permettant aux
bactéries de lutter contre les antibiotiques est encore plus grande que ce que l’on
pensait. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
Notre laboratoire est financé par les organismes suivants : European Research Council
(ERC) Advanced Grant BacCellEpi (670823), Agence Nationale de la Recherche
Investissement d’Avenir Programme (10-LABX-62-IBEID), Fondation le Roch les
Mousquetaires, l’Institut Pasteur, l’Inserm et l’INRA.
|
Footnotes |
1.
Cossart
P
,
Lebreton
A
. A trip in the New Microbiology with the bacterial
pathogen Listeria monocytogenes . FEBS Lett.
2014; ; 588 :
:2437.–2445. 2.
Radoshevich
L
,
Cossart
P
. Listeria monocytogenes: towards a complete
picture of its physiology and pathogenesis . Nat Rev
Microbiol.
2018; ; 16 :
:32.–46. 3.
Wurtzel
O
,
Sesto
N
,
Mellin
J
, et al.
Comparative transcriptomics of pathogenic and non-pathogenic
Listeria species . Mol Syst Biol.
2012; ; 8 : :583.. 4.
Duval
M
,
Dar
D
,
Carvalho
F
, et al.
HflXr, a homolog of a ribosome-splitting factor, mediates
antibiotic resistance . Proc Natl Acad Sci
USA.
2018; ; 115 :
:13359.–13364. 5.
Zhang
Y
,
Mandava
CS
,
Cao
W
, et al.
HflX is a ribosome-splitting factor rescuing stalled ribosomes
under stress conditions . Nat Struct Mol
Biol.
2015; ; 22 :
:906.–913. 6.
Coatham
ML
,
Brandon
HE
,
Fischer
JJ
, et al.
The conserved GTPase HflX is a ribosome splitting factor that
binds to the E-site of the bacterial ribosome .
Nucleic Acids Res.
2016; ; 44 :
:1952.–1961. 7.
Macé
K
,
Giudice
E
,
Gillet
R
. La synthèse des protéines par le ribosome : un
chemin semé d’embuches . Med Sci (Paris).
2015; ; 31 :
:282.–290. 8.
González-Plaza
JJ
,
Šimatovic
A
,
Milakovic
M
, et al.
Functional repertoire of antibiotic resistance genes in
antibiotic manufacturing effluents and receiving freshwater
sediments . Front Microbiol.
2018; ; 8 :
:1.–13. 9.
Lau
CHF
,
Van Engelen
K
,
Gordon
S
, et al.
Novel antibiotic resistant gene from agricultural soil exposed to
antibiotics widely used in human medicine and animal
farming . Appl Environ Microbiol.
2017; ; 83 :
:1.–18. |