| |
| Med Sci (Paris). 35(10): 753–760. doi: 10.1051/medsci/2019153.Chromosomes synthétiques Réécrire le code de la vie Vincent Baby,1,2 Fabien Labroussaa,3 Carole Lartigue,1,2 and Sébastien Rodrigue4* 1INRA, UMR 1332 de biologie du fruit et pathologie,
71 avenue E. Bourlaux, 33140Villenave d’Ornon,
France 2Univ. Bordeaux, UMR 1332 de biologie du fruit et
pathologie, 71 avenue E.
Bourlaux33140Villenave d’Ornon,
France 3Institute of veterinary bacteriology of Bern, Vetsuisse Faculty,
University of Bern, 3001Berne, Suisse 4Département de biologie, Université de Sherbrooke,
2500 boulevard de l’Université,
J1K 2R1Sherbrooke, Québec, Canada |
Au cours des 20 dernières années, le séquençage d’ADN s’est fortement démocratisé et son coût a
considérablement diminué, passant de 100 millions de dollars par génome en 2001 à moins de 1 000
dollars aujourd’hui. Cette technologie, très largement répandue dans les laboratoires médicaux et
de recherche, s’est même introduite dans les foyers en permettant aux particuliers d’obtenir la
séquence de leurs propres génomes. Bien que les coûts actuels soient encore élevés, la synthèse
d’ADN est appelée à connaître un essor similaire et devenir dans un futur proche un outil
incontournable dans le domaine des biotechnologies. Les récentes innovations technologiques
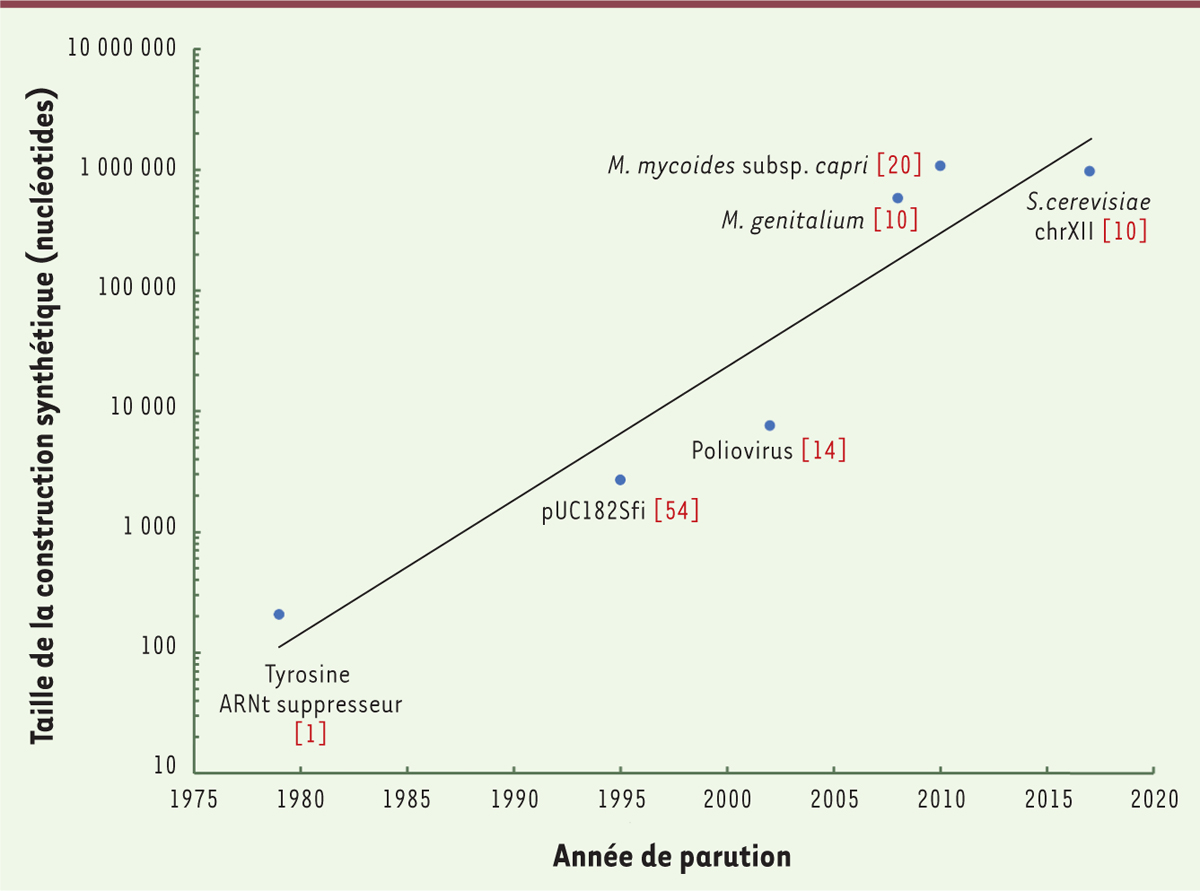
permettent de générer des molécules d’ADN toujours plus grandes et plus nombreuses. En 1979, le
premier gène synthétique était constitué de 207 paires de bases (pb) [1]. Depuis, des fragments d’ADN de plus d’un million de pb (Mpb)
ont été synthétisés et assemblés atteignant, pour certains, la taille de chromosomes complets
[2,3] (Figure 1). Ces
avancées marquent les débuts de l’ère de la « génomique synthétique », une discipline émergente
de la biologie se situant à l’interface entre science et ingénierie et dont l’un des objectifs
est de créer des organismes dont les génomes auront été complètement prédéterminés.
 | Figure 1. Évolution de la capacité de synthèse de génomes entiers en fonction du
temps. Les techniques de synthèse et d’assemblage permettent de produire des
constructions d’ADN de plus en plus grandes, qui peuvent maintenant atteindre plusieurs
millions de nucléotides. |
Après avoir résumé les techniques permettant de synthétiser et d’assembler de grandes molécules
d’ADN, nous présenterons dans cette revue les résultats marquants de cette nouvelle discipline,
la réglementation actuelle, qui incite à la vigilance, les aspects éthiques de cette recherche,
ainsi que les développements attendus pour les prochaines années. |
Techniques de synthèse et d’assemblage de fragments d’ADN Bien que de nouvelles technologies de synthèse de molécules fondées sur l’utilisation
d’enzymes soient en cours de développement et suscitent beaucoup d’intérêt [4], il n’existe pas encore de méthode permettant de
créer chimiquement de longues molécules d’ADN sans accumuler un nombre important de mutations
[5]. Les techniques actuelles reposent
donc sur un assemblage progressif de courts fragments d’ADN simple-brin, les oligonucléotides,
pour lesquels le taux d’erreur demeure relativement faible pour des tailles atteignant jusqu’à
200 nucléotides. La production de ces oligonucléotides par l’approche traditionnelle de la
chimie des phosphoramidites1 stagne depuis quelques années
à un coût d’environ 0,05-0,15 dollar par nucléotide en fonction de la quantité produite.
Cependant, l’introduction récente de puces à ADN permet d’augmenter considérablement le nombre
de molécules produites et de diminuer significativement le prix des oligonucléotides à moins de
0,01 dollar par nucléotide. Ces puces servent de support physique sur lequel un grand nombre
d’oligonucléotides de séquences différentes peuvent être synthétisés en parallèle, augmentant
ainsi le rendement de la production. Ces courtes molécules d’ADN simple-brin peuvent ensuite
être assemblées en utilisant des méthodes souvent apparentées à la réaction en chaîne par
polymérase (polymerase chain reaction ou PCR) afin d’obtenir des fragments
d’ADN pouvant atteindre quelques centaines à quelques milliers de pb (Figure 2). Plusieurs entreprises proposent de
synthétiser ces fragments à un coût inférieur à 100 dollars par millier de pb (kpb), incitant
ainsi les clients à commander des séquences d’ADN « prêtes à l’emploi » plutôt que de les
amplifier par PCR puis, si nécessaire, de les assembler pour obtenir le produit désiré. Malgré
des avantages certains en termes de gain de temps et d’argent, certaines contraintes demeurent.
En effet, de nombreuses séquences restent encore difficiles à synthétiser à l’heure actuelle,
notamment les séquences ayant un contenu en bases G et C très faible ou très élevé, ou encore
des séquences présentant des structures secondaires, des répétitions ou des homopolymères.
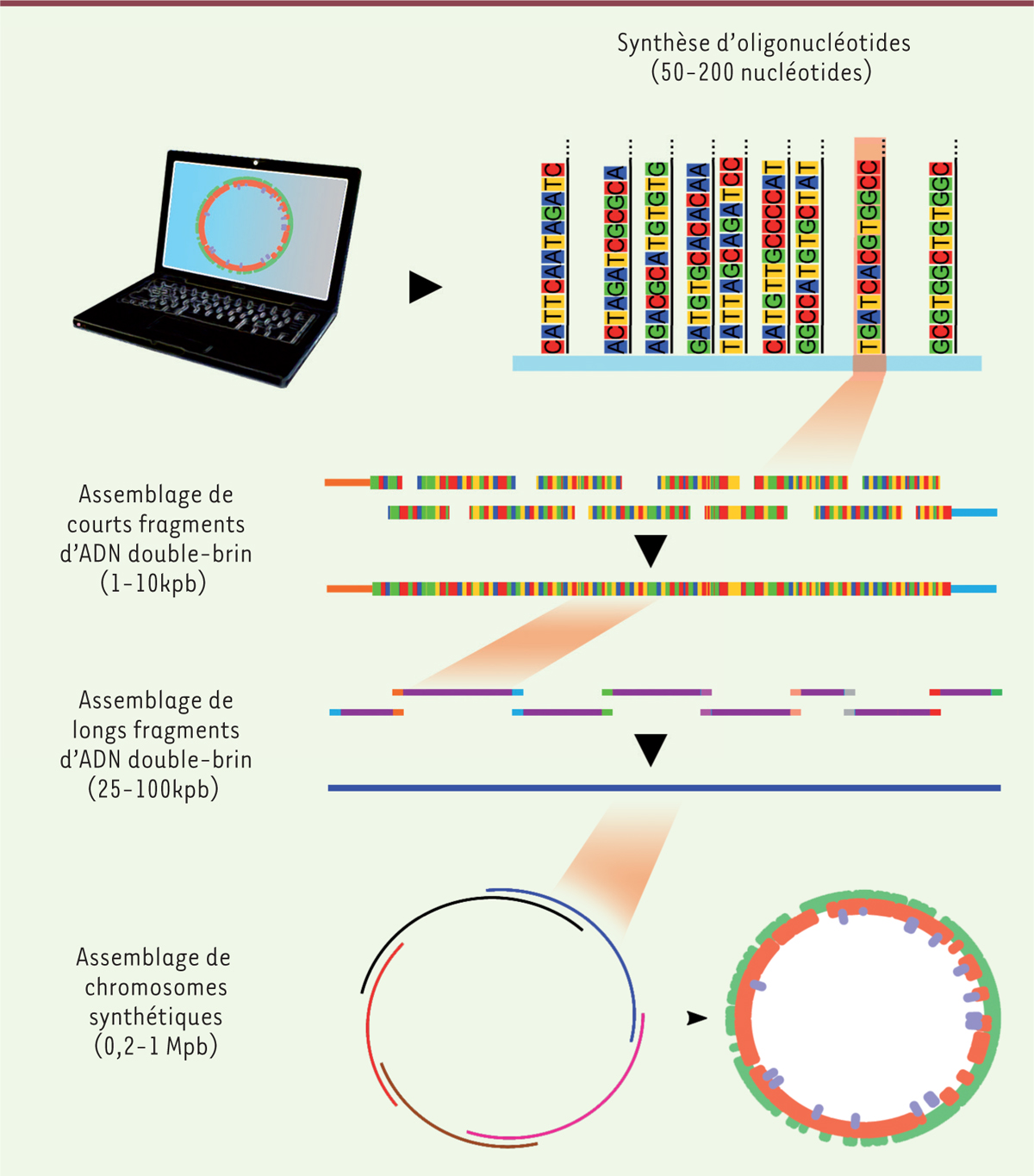
 | Figure 2. Résumé des étapes de conception et de construction d’un génome
synthétique. Des oligonucléotides synthétisés chimiquement sont
habituellement assemblés in vitro en éléments pouvant atteindre quelques milliers de paires
de bases (pb). Ceux-ci peuvent ensuite être combinés in vivo ou in vitro pour former des
fragments pouvant aller jusqu’à environ 100 kpb. Finalement, un assemblage de ces fragments
peut être complété in vivo pour obtenir des chromosomes complets de l’ordre du Mpb. |
Les fragments ainsi obtenus (comprenant entre 0,3 et 10 kpb) lors d’une synthèse initiale
(Figure 2) peuvent à leur tour être
combinés par assemblages itératifs en faisant appel à des techniques telles que l’assemblage de
Gibson2 [6], le Golden Gate3 [7] ou la recombinaison in
vivo4 [8]. Des molécules d’ADN dont la taille finale varie de quelques centaines de pb
jusqu’à plusieurs mégabases [9] peuvent
ainsi être assemblées (Figure 2). Alors
que des assemblages de taille inférieure ou égale à 10 kpb peuvent facilement être conservés en
solution ou amplifiés de nouveau par PCR, si nécessaire, les assemblages de plus grandes tailles
doivent être maintenus dans un vecteur. Ces vecteurs contiennent généralement une origine de
réplication à copie unique, un système de partitionnement, et au moins un marqueur de sélection.
Ces éléments permettent d’assurer la pérennité des fragments d’ADN clonés chez l’organisme hôte
choisi. Par exemple, les BAC (bacterial artificial chromosome) sont utilisés
chez les bactéries comme Escherichia coli, les YAC (yeast artificial
chromosome) chez la levure Saccharomyces cerevisiae, les HAC
(human artificial chromosome) dans des cellules humaines et les PAC
(plant artificial chromosome) chez les plantes. Le développement de protocoles
de transformation efficaces permettant d’introduire ces longues molécules d’ADN dans un
organisme d’intérêt représente aussi un aspect essentiel pour l’utilisation de tels chromosomes.
L’électroporation a d’abord été la méthode privilégiée, mais elle s’avère difficile pour des
constructions dépassant 0,5 Mpb [10].
La transformation de sphéroplastes5 [11], la transplantation de génomes [12] et la transformation par fusion cellulaire [13] dans la levure ont par la suite été
développées. Ces méthodes permettent la transformation des cellules avec des molécules d’ADN
plus grandes, certaines pouvant même dépasser 1,5 Mpb. |
Les génomes synthétiques : des virus aux cellules eucaryotes Plus la taille d’une molécule d’ADN à assembler est grande, plus sa construction et son
introduction dans un organisme à modifier deviennent complexes. Il n’est donc pas surprenant que
les génomes viraux de petites tailles (5 à 30 kpb), aient été les premiers à être
synthétisés. Le premier génome entièrement synthétique fût celui du poliovirus, l’agent responsable de la
poliomyélite, en 2002 [14]. Ce génome
de 7 740 nucléotides a été synthétisé en trois fragments issus d’oligonucléotides synthétiques.
Les fragments ont ensuite été clonés dans des plasmides qui ont enfin été combinés par digestion
et ligation enzymatiques. La transcription de cet ADN synthétique en ARN viral a généré des
poliovirus infectieux après transfection [14]. En 2003,
l’équipe de John Craig Venter a développé une méthode d’assemblage de fragments d’ADN qui a
permis de reconstruire le génome du bactériophage ΦX174 en moins de 15 jours [15]. Chacun des deux brins d’ADN du virus ont été
synthétisés en courts oligonucléotides de 42 bases qui ont été assemblés dans une réaction
inspirée de la PCR pour obtenir un seul chromosome linéaire de 5 386 pb. Ces deux expériences
ont permis d’établir les bases des futurs assemblages de génomes. Depuis, plusieurs autres virus
ont été synthétisés, comme le génome du bactériophage T7, celui du virus de la grippe espagnole
de 1918 ou encore du virus de l’immunodéficience humaine (VIH) (Tableau I). À l’heure actuelle, la synthèse de génomes viraux, que ce
soit pour des virus à ADN ou à ARN, des virus pathogènes ou non-pathogènes, et quelles que
soient leurs tailles (quelques milliers à plusieurs centaines de nucléotides), pose relativement
peu de difficultés. Elle fournit un outil puissant pour étudier la fonction des gènes et mieux
appréhender les mécanismes gouvernant la pathogénicité des virus. Il est aussi possible de
recoder entièrement ces génomes viraux en jouant, par exemple, sur la dégénérescence des codons.
Ces altérations permettent d’envisager des stratégies de lutte totalement inédites. Par exemple,
des vaccins candidats, à base de virus atténués ou après modification de leurs spécificité
d’hôtes, sont actuellement à l’étude [16–19].
Tableau I
| |
Génome synthétisé |
Taille (kpb) |
Référence |
| Virus |
Poliovirus PV1(M) |
7,6 |
[14] |
| Bactériophage ΦX174 |
5,4 |
[15] |
| Virus de la grippe espagnole de 1918 |
~13,0 |
[48] |
| HERV-K(HML-2) |
9,5 |
[56] |
| Bat SARS-like-SCoV |
29,7 |
[57] |
| VIH-1-C |
11,0 |
[58] |
| Bactériophage G4 |
5,6 |
[59] |
| Virus de l’hépatite C sous-type 1a |
9,6 |
[60] |
| Bactériophage S13-like |
5,4 |
[61] |
| CHIKV |
11,9 |
[62] |
| Virus de la mosaïque du tabac |
6,4 |
[63] |
| GALV-Nf |
4,7 |
[64] |
| PRRSV |
15,4 |
[64] |
| AcMNPV |
145,3 |
[65] |
| Virus de la variole équine |
212,8 |
[49] |
|
| Procaryotes |
Mycoplasma genitalium
|
583,0 |
[8, 10] |
|
Mycoplasma mycoides subsp. capri
|
1077,9 |
[20] |
| JCVI-syn3.0 |
531,5 |
[3] |
|
| Eucaryotes et organelles |
ADNm de souris |
16,3 |
[28] |
|
Saccharomyces cerevisiae chrIII
|
272,8 |
[33] |
|
Saccharomyces cerevisiae chrII
|
770,0 |
[34] |
|
Saccharomyces cerevisiae chrX
|
707,4 |
[37] |
|
Saccharomyces cerevisiae chrXII
|
976,0 |
[2] |
|
Saccharomyces cerevisiae chrV
|
536,0 |
[35] |
|
Saccharomyces cerevisiae chrVI
|
242,7 |
[36] |
Chromosomes synthétiques publiés à ce jour. |
Quelques années après la synthèse du génome du phage ΦX174, l’équipe de Venter a publié la
synthèse complète d’un premier génome bactérien, celui de Mycoplasma genitalium
[10]. Cette bactérie, qui possède le plus petit génome
naturel permettant à une cellule de croître de manière autonome, appartient à la classe des
Mollicutes, dont les membres sont caractérisés par leur petit génome et
l’absence de paroi cellulaire. Le génome de M. genitalium a pu être assemblé de
manière progressive en utilisant une approche hiérarchique : des petits fragments d’ADN
d’environ 5 à 7 kpb ont d’abord été assemblés in vitro en fragments de plus en
plus grands (jusqu’à environ 144 kpb), correspondant à des quarts de génomes, et propagés dans
la bactérie E. coli. Les quarts de génomes ont ensuite été assemblés par
recombinaison homologue chez la levure S. cerevisiae pour obtenir un chromosome
complet de 580 kpb. Cette méthode a par la suite été améliorée pour reconstruire le génome de
Mycoplasma mycoides sous-espèce capri, en utilisant
uniquement la levure S. cerevisiae [20].
Ce génome a pu être transplanté dans la bactérie Mycoplasma capricolum
sous-espèce capricolum, pour produire la première bactérie contrôlée par un
génome synthétique. La poursuite de ces travaux a permis, en 2016, la création d’une bactérie au
génome minimal (JCVI-syn3.0) duquel la majorité des gènes non-essentiels ont été retirés [3]. De nombreux projets visant à apporter des modifications
extensives au génome naturel d’autres bactéries d’intérêt sont en cours, comme en témoigne le
récent développement de deux souches d’E. coli n’utilisant respectivement que
59 et 57 codons des 64 habituellement retrouvés chez tous les autres organismes [21, 22]. Le projet Minibacillus, à l’image de JCVI-syn3.0, a,
lui, pour objectif d’obtenir une souche minimale de la bactérie modèle Bacillus
subtilis [23, 24] pour la caractériser entièrement et
déterminer le rôle précis de chaque gène essentiel. Des projets de réductions des génomes de
Streptomyces avermitilis [25, 26] et Pseudomonas
putida [27] sont également en
cours. À la limite entre l’eucaryote et le procaryote, un chromosome mitochondrial de souris a déjà
été synthétisé entièrement à partir d’oligonucléotides en seulement 5 jours [28]. En revanche, un tel génome n’a pas encore été introduit
dans une mitochondrie vivante puisque les techniques de transformation de cette organelle
restent difficiles à mettre en œuvre [29]. La suite logique consiste à générer les chromosomes d’un organisme eucaryote, ce qui a été
initié il y a quelques années dans le cadre du projet Saccharomyces cerevisiae
2.0 (Sc2.0). Plusieurs laboratoires à travers le monde ont uni leurs efforts dans le but de
synthétiser les 16 chromosomes de cette levure [30]. Le projet Sc2.0 ne vise pas seulement à recopier la séquence du génome.
Certaines modifications, comme la délétion de plusieurs introns non-essentiels et le
remplacement de tous les codons d’arrêt de la traduction (codons STOP) UAG par un équivalent
UAA, ont aussi été apportées. L’insertion d’un site de recombinaison loxP à la
suite de chaque gène non-essentiel permet également l’utilisation de la méthode SCRaMbLE
(synthetic chromosome recombination and modification by loxP-mediated
evolution) [31] visant à
mélanger aléatoirement l’ordre des gènes afin d’obtenir puis d’étudier de nouvelles
organisations génomiques supportant la vie. À la différence des projets précédents, les
chromosomes de la levure Sc2.0 ne sont pas réassemblés pour ensuite être introduits
intégralement dans la cellule. Des fragments de chromosomes natifs sont en effet remplacés
progressivement par leurs équivalents modifiés, d’une taille de 30 à 60 kpb. La modification du
génome est donc plus lente puisque la reconstruction d’un seul chromosome peut prendre plus
d’une trentaine de cycles de remplacement nécessitant chacun des étapes de vérification portant
autant sur la viabilité de la souche que sur la validité de la séquence introduite [2]. La reconstruction du génome Sc2.0 a débuté il y a environ
10 ans par la synthèse d’un des bras des chromosomes VI et IX de la levure [32] et s’est poursuivie avec la publication de la
synthèse complète du chromosome III trois ans plus tard [33]. Par la suite, le consortium a annoncé la synthèse des chromosomes II [34], V [35], VI [36], X [37] et XII
[2] dans un numéro spécial de Science.
La reconstruction du génome de la levure se poursuit et culminera avec le rassemblement des
différents chromosomes ainsi synthétisés dans une seule et même souche, ce qui devrait permettre
d’obtenir le premier organisme eucaryote portant un génome synthétique. Les chromosomes artificiels sont également des outils convoités par des chercheurs étudiant
les plantes. Sans pouvoir actuellement synthétiser des chromosomes complets de
novo, des chromosomes natifs peuvent être tronqués en mini-chromosomes qui peuvent
être modifiés. Cet outil a déjà été utilisé chez plusieurs organismes tels que le maïs [38], Arabidopsis thaliana
(arabette des dames) [39,40], le riz [41] et l’orge [42]. Des avancées importantes ont également été rapportées dans la
construction et la manipulation de chromosomes de chloroplastes6, beaucoup plus faciles à assembler et à modifier que les chromosomes nucléaires
[43]. Il est également possible de les
transformer en utilisant des méthodes comme la biolistique7
[44]. Le consortium international Genome Project-Write (GP-write), formé en 2016
dans le but de promouvoir le développement des technologies permettant la synthèse et
l’initialisation des génomes, propose de synthétiser un génome humain modifié et de l’introduire
dans une lignée cellulaire [45]
(→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 10, octobre 2016,
page 898
Plusieurs laboratoires, par exemple aux États-Unis, en Chine, en Angleterre, au Canada et au
Japon, ont signifié leur intérêt à y participer [46]. Plusieurs aspects, tant technologiques qu’éthiques, devront cependant
être davantage développés avant d’entreprendre cette tâche colossale. Actuellement, une douzaine
de projets pilotes sont en cours, dont un portant sur l’ingénierie de lignées cellulaires afin
de les rendre plus sûres et d’améliorer leurs propriétés pour des applications biotechnologiques
[47] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 8-9, aoûit-septembre
2018, page 749
Il n’est cependant pas question de modifier des organes ou des cellules germinales dans le
cadre de ce projet. |
Réglementation et considérations éthiques La capacité à synthétiser des chromosomes et des génomes provoque certains débats. La synthèse
du virus de la grippe espagnole de 1918 [48] et la
récente création d’une version chimérique du virus de la variole équine [49] ont soulevé plusieurs questions sur le développement accidentel ou
délibéré d’organismes potentiellement dangereux [52] (→).
(→) Voir le Forum de J.N. Tourier, m/s n° 2, février 2019, page
181
Bien que l’utilisation d’organismes pathogènes et de toxines soit bien réglementée, il y a
encore peu de règles législatives déterminant les séquences de nucléotides qui peuvent être
synthétisées. Face aux inquiétudes, le Department of health and human services
(HHS) américain a publié en 2009 un document intitulé Screening framework guidance for
providers of synthetic double-stranded DNA. Ce document décrit une procédure
volontaire à appliquer par les compagnies offrant des services de synthèse d’ADN pour contrôler
l’utilisation de séquences à des fins malveillantes. Les grandes lignes de cette approche
consistent à établir l’identité des acheteurs et à déterminer si les séquences qu’ils commandent
peuvent représenter une menace. Cette stratégie d’autogouvernance par les compagnies a été
adoptée pour permettre aux chercheurs ayant les équipements et la connaissance requise pour
manipuler des organismes potentiellement dangereux, d’effectuer leur recherche sans contraintes
majeures. Plusieurs compagnies se sont concertées pour fonder le consortium
International gene synthesis consortium (IGSC) dont les membres doivent suivre
les recommandations du HHS. Ces entreprises représentent 80 % des fournisseurs de séquences dans
le monde, assurant ainsi un certain niveau de contrôle du risque biologique. Les risques
associés à la biologie de synthèse ont également été évalués en décembre 2010 par la
Presidential commission for the study of bioethical issues (PCSBI). Dans un
rapport publié sous le titre New directions : the ethics of synthetic biology and
emerging technologies, il est conclu que les capacités actuelles de la biologie de
synthèse ne représentent pas un danger en fonction des connaissances scientifiques. Il
recommande toutefois une réévaluation de ces capacités au fil des années pour légiférer si un
risque plus concret se manifeste. La génomique synthétique possède donc un potentiel important
pour mieux comprendre la vie ou pour de nouvelles applications biotechnologiques. Il reste
néanmoins essentiel de demeurer vigilant pour éviter les usages éventuellement dangereux ou
malicieux. La synthèse de chromosomes pose également des questions d’ordre éthique. Dans un avenir
prochain, la synthèse de chromosomes humains sera techniquement possible et, combinée aux
avancées d’ingénierie de génome, des modifications majeures du génome humain pourront être
effectuées. Il sera par exemple possible de rendre résitantes des cellules contre certaines
maladies ou de leur ajouter de nouvelles voies métaboliques. Bien que certaines de ces
applications puissent être bénéfiques dans le cadre de la recherche, il est important de se
demander si de telles pratiques sont souhaitables en médecine. En 1997, lors de la
29e session de la conférence générale de l’Organisation des Nations unies pour
l’éducation, la science et la culture (UNESCO), la Déclaration universelle sur le génome humain
et les droits de l’homme a été adoptée. En plus d’interdire le clonage humain destiné à la
reproduction, cette déclaration vise aussi à protéger la dignité du génome humain. Durant cette
même conférence, la Déclaration sur les responsabilités des générations présentes envers les
générations futures a aussi statué que le génome humain doit être protégé et sa biodiversité
sauvegardée. Ces déclarations ne demandent pas de limiter la recherche, mais plutôt de l’exercer
avec prudence. L’utilisation de sujets humains n’est pas une option à considérer, même si
certaines modifications pourraient sembler avantageuses à court terme. En effet, une telle
pratique pourrait diminuer la diversité génétique humaine et ainsi affecter l’adaptabilité de
notre espèce à long terme. |
La génomique synthétique : promesses, déploiements et applications futures Bien que les techniques de synthèse de chromosomes permettent, en principe, la création de
nouvelles séquences génomiques inédites, les projets rapportés à ce jour demeurent très
fortement inspirés des organismes retrouvés dans la nature. Les séquences, la composition en
gènes et l’architecture des chromosomes n’ont en effet pas encore fait l’objet d’un grand nombre
de modifications qui, collectivement, permettront de faire émerger les principes fondamentaux
régissant l’ingénierie des génomes. Lorsque ces principes de base seront mieux compris, des
efforts de conception rationnelle pourront plus facilement être entrepris, en intégrant des
outils informatiques de modélisation cellulaire et de conception assistée par ordinateur. Ces
outils permettront non seulement de prédire efficacement les phénotypes attendus, mais aussi
d’établir l’architecture détaillée des chromosomes. Ces génomes pourront être organisés en
modules regroupant des fonctions communes, qui seront combinés selon les besoins spécifiques de
l’organisme désiré. Les cellules produites pourront ainsi contenir des gènes recodés, utilisant
des codons synonymes ou n’existant pas dans la nature (Figure 3), ou inclure diverses machineries cellulaires spécialisées ou des
combinaisons de voies métaboliques actuellement inexistantes. Une variété d’applications
pourront alors être envisagées comme la production de molécules d’intérêt, la bioremédiation, le
traitement de maladies ou encore la création de vaccins plus efficaces.
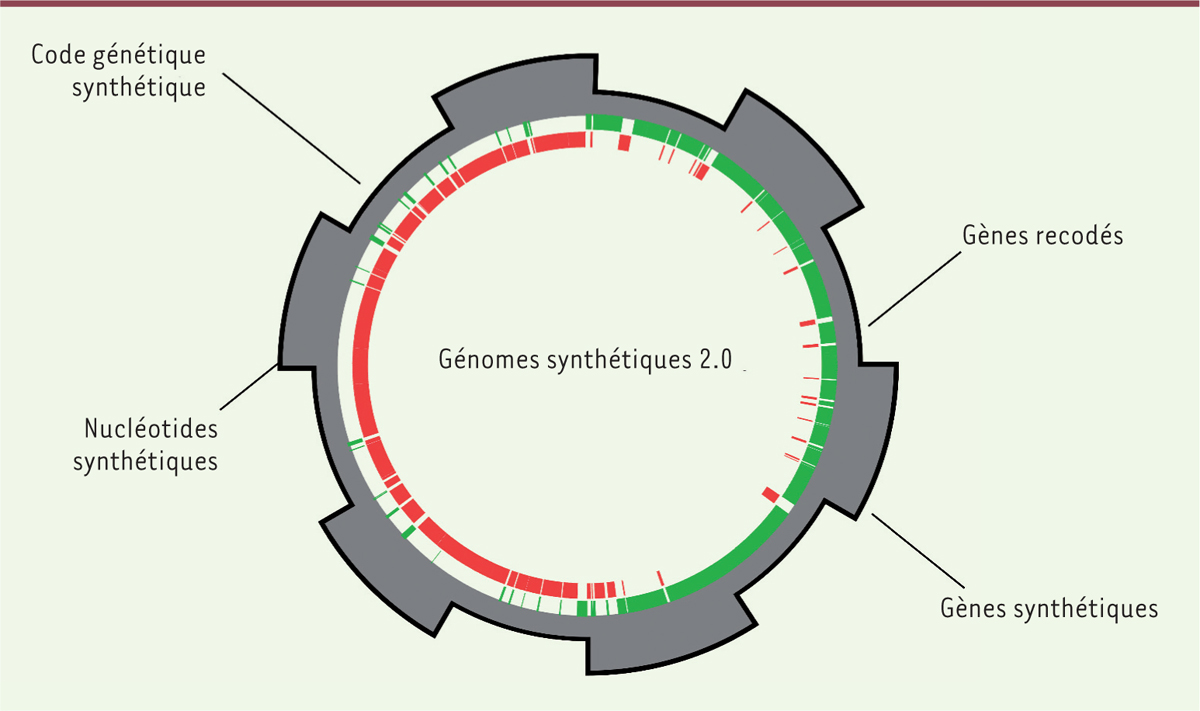
 | Figure 3. Les génomes synthétiques du futur. Les progrès de la
génomique synthétique mèneront à des génomes ayant des propriétés différentes de celles
observées dans la nature. Le développement de nucléotides synthétiques et l’utilisation de
codes génétiques alternatifs permettront de créer des cellules artificielles dont le matériel
génétique ne correspond à aucun autre organisme retrouvé dans la nature. |
Un autre axe en développement vise à créer des organismes utilisant des nucléotides
non-canoniques dans leur génome (Figure
3). Ceci permettrait non seulement de distinguer les organismes synthétiques des
organismes naturels, mais également de leur conférer des caractéristiques intéressantes [50] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 2, février 2018,
page 179
Un avantage de cet ADN artificiel est qu’il impose une dépendance à des nucléotides n’existant
pas naturellement, limitant ainsi la dissémination dans l’environnement des organismes
génétiquement modifiés. L’utilisation de nouvelles bases azotées pour la synthèse de l’ADN
[53] (→) implique
l’écriture de nouveaux codes génétiques, ce qui permettra d’immuniser l’organisme contre les
virus utilisant des codes naturels. Cette approche permettra également de limiter l’expression
de gènes acquis lors de potentiels évènements d’échange de matériel génétique par transfert
horizontal entre organismes naturels et synthétiques. L’incompatibilité entre les codes naturels
et synthétiques serait à l’origine d’erreurs de traduction des protéines, en causant des
interruptions précoces ou en menant à l’incorporation d’acides aminés inappropriés. Des acides
aminés non-naturels pourraient être incorporés par certains de ces codons non-utilisés ou
synthétiques, offrant ainsi des capacités structurales ou enzymatiques améliorées pour diverses
protéines. Une souche de la bactérie E. coli possédant des nucléotides
non-naturels a déjà été produite [51]
et des souches utilisant un code génétique réduit sont disponibles [21, 22].
(→) Voir la Chronique génomique de B. Jordan, m/s n° 5, mai 2019, page
483
|
Les avancées technologiques spectaculaires des dernières années nous permettent d’assister aux
prémices de l’ère de la génomique synthétique. Les défis sont encore nombreux avant d’atteindre
le plein potentiel de cette discipline émergente, mais il est probable qu’elle transformera la
recherche fondamentale et appliquée dans un futur proche [66] (→). La capacité de synthèse massive d’ADN
promet également d’accélérer les recherches en sciences biologiques en plus de constituer un
outil important en biologie de synthèse pour répondre aux multiples obstacles auxquels
l’humanité devra faire face au cours de ce siècle.
(→) Voir la Synthèse de F. Labroussaa et al., page 761 de ce
numéro
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées
dans cet article.
|
Footnotes |
1. Sekiya
T, Takeya
T, Brown
EL, et al.
Total synthesis of a tyrosine suppressor transfer RNA gene XVI. Enzymatic
joinings to form the total 207-base pair-long DNA . J Biol
Chem.
1979; ; 254 :
:5787.–801. 2. Zhang
W, Zhao
G, Luo
Z, et al.
Engineering the ribosomal DNA in a megabase synthetic
chromosome . Science.
2017; ; 355 : :eaaf3981.. 3. Hutchison
CA
III, Chuang
R-Y, Noskov
VN, et al.
Design and synthesis of a minimal bacterial genome .
Science.
2016; ; 351 :
:1414.–26. 4. Palluk
S, Arlow
DH, De
Rond T, et al.
De novo DNA synthesis using polymerasenucleotide
conjugates . Nat Biotechnol.
2018; ; 36 :
:645.–50. 5. Kosuri
S, Church
GM. Large-scale de novo DNA synthesis
: Technologies and applications . Nat Methods.
2014; ; 11 :
:499.–507. 6. Gibson
DG, Young
L, Chuang
R-Y, et al.
Enzymatic assembly of DNA molecules up to several hundred
kilobases . Nat Methods.
2009; ; 6 : :343.–5. 7. Engler
C, Kandzia
R, Marillonnet
S . A one pot, one step, precision
cloning method with high throughput capability . PLoS One.
2008; ; 3 : :e3647.. 8. Gibson
DG, Benders
GA, Axelrod
KC, et al.
One-step assembly in yeast of 25 overlapping DNA fragments to form a complete
synthetic Mycoplasma genitalium genome . Proc Natl
Acad Sci U S A.
2008; ; 105 :
:20404.–9. 9. Kuroiwa
Y, Tomizuka
K, Shinohara
T, et al.
Manipulation of human minichromosomes to carry greater than megabase-sized
chromosome inserts . Nat Biotechnol.
2000; ; 18 :
:1086.–90. 10. Gibson
DG, Benders
GA, Andrews-Pfannkoch
C, et al.
Complete chemical synthesis, assembly, and cloning of a Mycoplasma
genitalium genome . Science.
2008; ; 319 :
:1215.–20. 11. Lartigue
C, Vashee
S, Algire
MA, et al.
Creating bacterial strains from genomes that have been cloned and engineered in
yeast . Science.
2009; ; 325 :
:1693.–6. 12. Lartigue
C, Glass
JI, Alperovich
N, et al.
Genome transplantation in bacteria : changing one species to
another . Science.
2007; ; 317 :
:632.–8. 13. Karas
BJ, Jablanovic
J, Irvine
E, et al.
Transferring whole genomes from bacteria to yeast spheroplasts using entire
bacterial cells to reduce DNA shearing . Nat
Protoc.
2014; ; 9 : :743.–50. 14. Cello
J, Paul
AV, Wimmer
E . Chemical synthesis of poliovirus
cDNA : Generation of infectious virus in the absence of natural template .
Science.
2002; ; 297 :
:1016.–8. 15. Smith
HO, Hutchison
C a, Pfannkoch
C, et al.
Generating a synthetic genome by whole genome assembly : phiX174 bacteriophage
from synthetic oligonucleotides . Proc Natl Acad Sci
USA.
2003; ; 100 :
:15440.–5. 16. Brown
M, Lleras
R, Mehedi
M, et al.
Genetic stability of genome-scale deoptimized RNA virus vaccine candidates under
selective pressure . Proc Natl Acad Sci USA.
2017; ; 114 :
:E386.–95. 17. Le
Nouen C, Luongo
C, Yang
L, et al.
Attenuation of human respiratory syncytial virus by genome-scale codon-pair
deoptimization . Proc Natl Acad Sci USA.
2014; ; 111 :
:13169.–74. 18. Yurovsky
A, Mueller
S, Ward
CB, et al.
Large-scale recoding of an arbovirus genome to rebalance its insect versus
mammalian preference . Proc Natl Acad Sci USA.
2015; ; 112 :
:4749.–54. 19. Wimmer
E, Paul
AV. Synthetic poliovirus and other
designer viruses : What have we learned from them?
Annu Rev Microbiol.
2010; ; 65 :
:583.–609. 20. Gibson
DG, Glass
JI, Lartigue
C, et al.
Creation of a bacterial cell controlled by a chemically synthesized
genome . Science.
2010; ; 329 : :52.–6. 21. Fredens
J, Wang
K, de la Torre
D de, et al.
Total synthesis of Escherichia coli with a recoded
genome . Nature.
2019; ; 569 :
:514.–8. 22. Ostrov
N, Landon
M, Guell
M, et al.
Design, synthesis, and testing toward a 57-codon genome .
Science.
2016; ; 353 :
:819.–22. 23. Reuß
DR, Altenbuchner
J, Mäder
U, et al. . Large-scale
reduction of the Bacillus subtilis genome : Consequences for the transcriptional network,
resource allocation, and metabolism . Genome
Res.
2017; ; 27 :
:289.–99. 24. Reuß
DR, Commichau
FM, Gundlach
J, et al.
The blueprint of a minimal cell : MiniBacillus .
Microbiol Mol Biol Rev.
2016; ; 80 :
:955.–87. 25. Ikeda
H, Satoshi
KS. Genome mining of the
Streptomyces avermitilis genome and development of genome-minimized hosts
for heterologous expression of biosynthetic gene clusters . J
Ind Microbiol Biotechnol.
2014; ; 41 :
:233.–50. 26. Komatsu
M, Uchiyama
T, Omura
S, et al.
Genome-minimized Streptomyces host for the heterologous
expression of secondary metabolism . Proc Natl Acad Sci
USA.
2010; ; 107 :
:2646.–51. 27. Leprince
A, De
Lorenzo V, Völler
P, et al.
Random and cyclical deletion of large DNA segments in the genome of
Pseudomonas putida . Environ
Microbiol.
2012; ; 14 :
:1444.–53. 28. Gibson
DG, Smith
HO, Hutchison
CA, et al.
Chemical synthesis of the mouse mitochondrial genome .
Nat Methods.
2010; ; 7 : :901.–3. 29. Lightowlers
RN. Mitochondrial transformation :
time for concerted action . EMBO Rep.
2011; ; 12 : :480.–1. 30. Richardson
SM, Mitchell
LA, Stracquadanio
G, et al.
Design of a synthetic yeast genome .
Science.
2017; ; 355 :
:1040.–4. 31. Dymond
J, Boeke
J. The saccharomyces
cerevisiae SCRaMbLE system and genome minimization .
Bioeng Bugs.
2012; ; 3 : :168.–71. 32. Dymond
JS, Richardson
SM, Coombes
CE, et al.
Synthetic chromosome arms function in yeast and generate phenotypic diversity by
design . Nature.
2011; ; 477 :
:471.–6. 33. Annaluru
N, Muller
H, Mitchell
LA, et al.
Total synthesis of a functional designer eukaryotic chromosome .
Science.
2014; ; 344 : :55.–8. 34. Shen
Y, Wang
Y, Chen
T, et al.
Deep functional analysis of synII, a 770-kilobase synthetic yeast
chromosome . Science.
2017; ; 355 : :eaaf4791.. 35. Xie
Z-X, Li
B-Z, Mitchell
LA, et al.
“Perfect” designer chromosome V and behavior of a ring
derivative . Science.
2017; ; 355 : :eaaf4704.. 36. Mitchell
LA, Wang
A, Stracquadanio
G, et al.
Synthesis, debugging, and effects of synthetic chromosome consolidation : synVI
and beyond . Science.
2017; ; 355 : :eaaf4831.. 37. Wu
Y, Li
BZ, Zhao
M, et al.
Bug mapping and fitness testing of chemically synthesized chromosome
X . Science.
2017; ; 355 : :eaaf4706.. 38. Yu
W, Han
F, Gao
Z, et al.
Construction and behavior of engineered minichromosomes in
maize . Proc Natl Acad Sci USA.
2007; ; 104 :
:8924.–9. 39. Nelson
AD, Lamb
JC, Kobrossly
PS, et al.
Parameters affecting telomere-mediated chromosomal truncation in
Arabidopsis . Plant Cell.
2011; ; 23 :
:2263.–72. 40. Teo
CH, Ma
L, Kapusi
E, et al.
Induction of telomere-mediated chromosomal truncation and stability of truncated
chromosomes in Arabidopsis thaliana . Plant
J.
2011; ; 68 : :28.–39. 41. Xu
C, Cheng
Z, Yu
W
Construction of rice mini-chromosomes by telomere-mediated chromosomal
truncation . Plant J.
2012; ; 70 :
:1070.–9. 42. Kapusi
E, Ma
L, Teo
CH, et al.
Telomere-mediated truncation of barley chromosomes .
Chromosoma.
2012; ; 121 :
:181.–90. 43. Boehm
CR, Bock
R. Recent advances and current
challenges in synthetic biology of the plastid genetic system and metabolism .
Plant Physiol.
2019; ; 179 :
:794.–802. 44. Vafaee
Y, Staniek
A, Mancheno-Solano
M, et al.
A modular cloning toolbox for the generation of chloroplast transformation
vectors . PLoS One.
2014; ; 10 : :e110222.. 45. Jordan
B. HGP-write : après la lecture,
l’écriture ?
Med/Sci (Paris).
2016; ; 32 :
:898.–901. 47. Jordan
B. De HGP-write aux
super-cellules. Med/Sci (Paris).
2018; ; 34 :
:749.–51. 48. Tumpey
TM, Basler
CF, Aguilar
PV, et al.
Characterization of the reconstructed 1918 Spanish influenza pandemic
virus . Science.
2005; ; 310 :
:77.–80. 49. Noyce
RS, Lederman
S, Evans
DH. Construction of an infectious
horsepox virus vaccine from chemically synthesized DNA fragments . PLoS
One.
2018; ; 13 : :e0188453.. 50. Jordan
B. Bases alternatives et organismes
synthétiques . Med/Sci (Paris).
2018; ; 34 :
:179.–82. 51. Zhang
Y, Lamb
BM, Feldman
AW, et al.
A semisynthetic organism engineered for the stable expansion of the genetic
alphabet . Proc Natl Acad Sci USA.
2017; ; 114 :
:1317.–22. 52. Tournier
JN. L’éradication des maladies
infectieuses virales mise en danger par les avancées de la biologie
synthétique . Med Sci (Paris).
2019; ; 35 : :181.–6. 53. Jordan
B. Extension du domaine du codage :
l’ADN hachimoji . Med Sci (Paris).
2019; ; 35 : :483.–5. 54. Stemmer
WPC, Crameri
A, Ha
KD, et al.
Single-step assembly of a gene and entire plasmid from large numbers of
oligodeoxyribonucleotides . Gene.
1995; ; 164((49–53)) : :55..
55. Lee
YN, Bieniasz
PD. Reconstitution of an infectious
human endogenous retrovirus . PLOS Pathog.
2007; ; 3 : :e10.. 56. Becker
MM, Graham
RL, Donaldson
EF, et al.
Synthetic recombinant bat SARS-like coronavirus is infectious in cultured cells
and in mice . Proc Natl Acad Sci USA.
2008; ; 105 :
:19944.–9. 57. Nauwelaers
D, Van Houtte
M, Winters
B, et al.
A synthetic HIV-1 subtype C bakbone generates comparable PR and RT resistance
profiles to a subtype B backbone in a recombinant virus assay .
PLOS One.
2011; ; 6 : :e19643.. 58. Yang
R, Han
Y, Ye
Y, et al.
Chemical synthesis of bateriophage G4 . PloS
One.
2011; ; 6 : :e27062.. 59. Munshaw
S, Bailey
JR, Liu
L, et al.
Computational reconstruction of Bole1a, a representative synthetic hepatitis C
virus subtype 1a genome . J Virol.
2012; ; 86 :
:5915.–21. 60. Liu
Y, Han
Y, Huang
W, et al.
Whole-genome synthesis and characterization of viable S13-like
bacteriophages . PLoS One.
2012; ; 7 : :e41124.. 61. Scholte
FEM, Tas
A, Martina
BEE, et al.
Characterization of synthetic Chikungunya virus based on the consensus sequence
of recent E1–226V isolates . PLOS One.
2013; ; 8 : :e71047.. 62. Cooper
B. Proof by synthesis of Tobacco
mosaic virus . Genome Res.
2014; ; 15 : :R67.. 63. Lovato
A, Faoro
F, Gambino
G, et al.
Construction of a synthetic infectious cDNA clone of Grapevine Algerian latent
virus (GALV-Nf) and its biological activity in Nicotiana benthamiana and
grapevine plants . Virol J.
2014; ; 11 : :186.. 64. Vu
HLX, Ma
F, Laegreid
WW, et al.
A synthetic porcine reproductive and respiratory syndrome virus strain confers
unprecedented levels of heterologous protection . J
Virol.
2015; ; 89 :
:12070.–83. 65. Shang
Y, Wang
M, Xiao
G, et al.
Construction and rescue of a functional synthetic baculovirus .
ACS Synth Biol.
2017; ; 6 :
:1393.–402. 66. Labroussaa
F, Baby
V, Rodrigue
S, Lartigue
C. La transplantation des génomes :
redonner vie à des génomes bactériens naturels ou synthétiques .
Med Sci (Paris).
2019; ; 35 :
:761.–70. |