| |
| Med Sci (Paris). 35(11): 821–841. doi: 10.1051/medsci/2019160.Zelda, le maestro du réveil du génome
zygotique Jeremy Dufourt,1* Maëlle Bellec,1 Olivier Messina,1 Antonio Trullo,1 Cyril Favard,2 Ovidiu Radulescu,3 and Mounia Lagha1** 1Institut de génétique moléculaire de Montpellier, université
de Montpellier, CNRS-UMR 5535, 1919 route de Mende, 34293Montpellier Cedex 5,
France 2Institut de recherche en infectiologie de Montpellier, CNRS,
université de Montpellier UMR 9004, 1919 route de Mende, 34293Montpellier Cedex 5,
France 3LPHI, UMR CNRS 5235, université de
Montpellier, place E.
Bataillon - Bât. 24 cc 107, 34095Montpellier, Cedex 5,
France MeSH keywords: Animaux, Cycle cellulaire, Chromatine, Protéines de Drosophila, Développement embryonnaire, Éléments activateurs (génétique), Génome, Mitose, Protéines nucléaires, Organogenèse, ARN messager, Activation de la transcription, Zygote, physiologie, métabolisme, croissance et développement |
Chez de nombreux animaux, bien que la fertilisation aboutisse rapidement à la fusion des
génomes maternels et paternels, l’activation du génome zygotique ne débute pas dès les
premières heures du développement. En effet, il existe un stock d’ARN et de protéines
dans le gamète maternel qui va régir l’embryogenèse précoce. Durant cette première
période critique du développement, les cellules totipotentes issues de l’œuf fécondé
vont rapidement se différencier pour pouvoir ensuite former les trois feuillets
embryonnaires fondamentaux : ectoderme, endoderme, et mésoderme. |
La transition materno-zygotique Chez tous les métazoaires, le passage graduel du contrôle maternel au contrôle
zygotique du développement embryonnaire se déroule lors d’un évènement appelé
transition materno-zygotique (MZT), durant laquelle, de manière concomitante à la
dégradation des ARN et protéines provenant du gamète maternel, le génome de
l’embryon est activé. Cette activation est caractérisée par une vague progressive de
transcription, qui coïncide avec le ralentissement des cycles cellulaires dans de
nombreuses espèces. Le démarrage de cette vague de transcription ainsi que le nombre
de cycles de division qu’elle accompagne varient considérablement d’une espèce à
l’autre. Cependant, pour chaque espèce, le déroulement temporel de la MZT est précis
et très reproductible d’un embryon à l’autre. Chez les espèces au développement
rapide, telles que le nématode (Caenorhabditis elegans), le xénope
(Xenopus laevis), le poisson zèbre (Danio
rerio) et la drosophile (Drosophila melanogaster), la
MZT s’achève quelques heures seulement après la fécondation, et est suivie du
premier évènement de la morphogénèse : la gastrulation. En revanche, chez les
espèces à développement plus lent, telles que la souris (Mus
musculus) et l’homme (Homo sapiens), la MZT peut durer
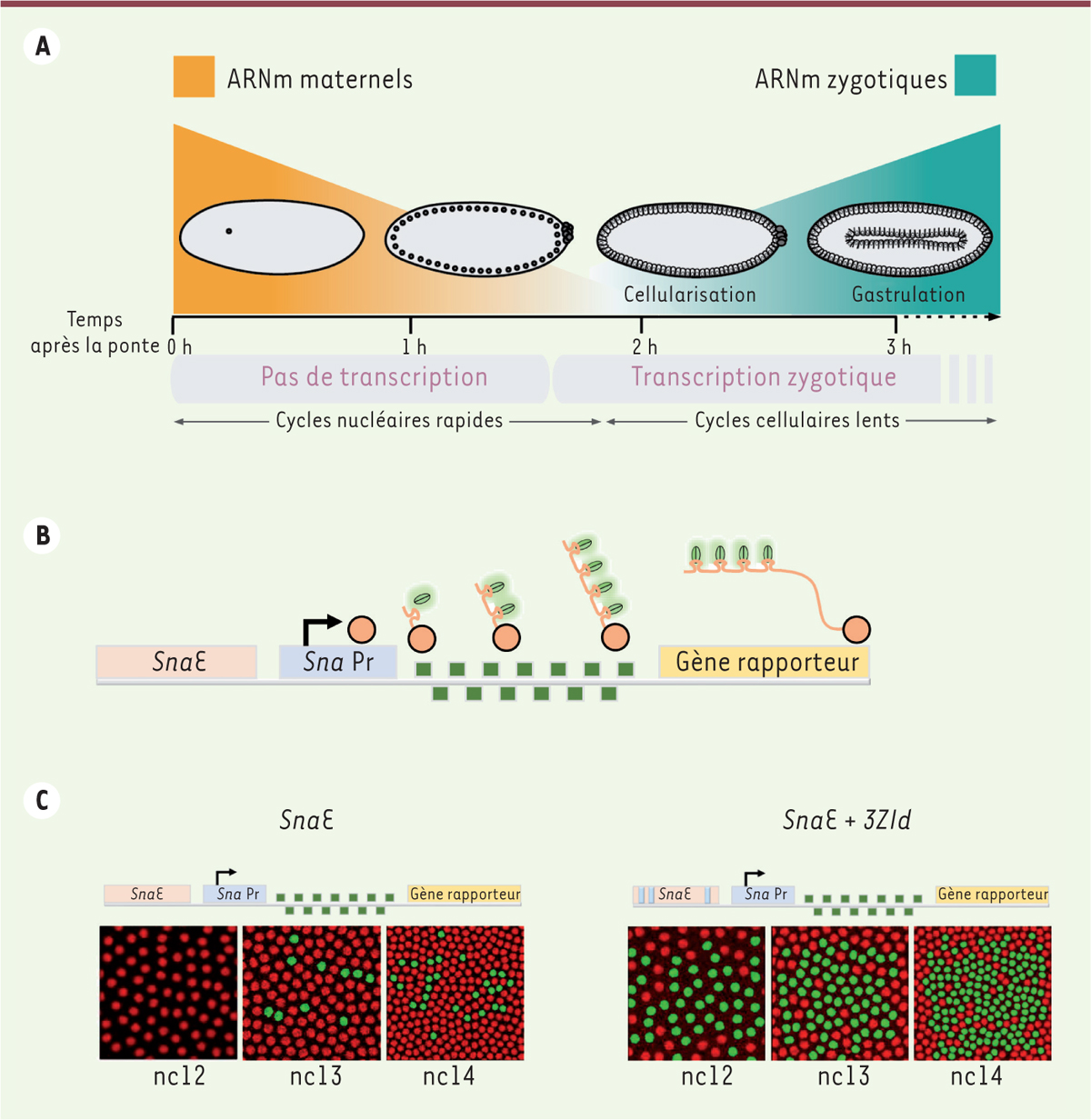
plusieurs jours [1]. Chez la drosophile, l’embryogénèse commence par 14 cycles nucléaires (nc) rapides au
sein d’un syncytium, contrôlés principalement par l’apport en ARN et protéines du
gamète maternel. Environ deux heures après la fécondation, le génome zygotique
s’active transcriptionnellement et le syncytium se sépare en cellules individuelles
(cellularisation) comportant chacune un seul noyau (Figure 1A). Le réveil transcriptionnel du génome
zygotique nécessite des facteurs de transcription spécifiques, dont la protéine
Zelda, qui joue un rôle essentiel dans ce processus [2]. Zelda possède plusieurs caractéristiques des
facteurs de transcription dits « pionniers » : 1) sa liaison aux éléments
cis-régulateurs avant l’activation des gènes, établissant une
pré-compétence pour l’adoption de certains destins cellulaires, et 2) sa capacité à
ouvrir la chromatine pour faciliter la liaison de facteurs de transcription
classiques. De plus, la distribution homogène de Zelda dans l’embryon permet de
potentialiser l’action locale de morphogènes (par exemple, bicoid
et dorsal) dans différentes régions de l’embryon [3–5] : la réponse de gènes-cibles à la concentration de morphogène
est renforcée par la liaison de Zelda à ces gènes. Cependant, le rôle de ce facteur
« pionnier » dans le contrôle temporel de l’activation transcriptionnelle n’a été
élucidé que très récemment.
 | Figure 1 A. Représentation schématique de la
transition materno-zygotique (MZT) chez la drosophile.
B. Schéma du système
MS2/MS2 coat protein (MCP) couplée à la
green fluorescent protein (GFP) permettant de
visualiser la transcription génique en temps réel.
C. Représentation schématique des
deux transgènes comportant soit la version tronquée de
l’enhancer distal de sna
(SnaE) ne comportant aucun site de liaison à Zelda,
soit cet enhancer avec la présence additionnelle de
trois sites de liaison à Zelda (SnaE + 3Zld), et
imagerie en temps réel de leur activité de transcription au cours de
trois cycles nucléaires (nc12, nc13, nc14). Chaque
enhancer (rectangle orange) contrôle le promoteur
minimal de sna (SnaPr, rectangle bleu), qui permet la
transcription d’un gène rapporteur (rectangle jaune) comportant 24
répétitions du motif de fixation de la protéine MCP (carrés verts) dans
sa partie 5’non codante. Les noyaux qui présentent une activité
transcriptionnelle sont détectés grâce à la fluorescence de la GFT et
colorés en vert. Ils sont beaucoup plus nombreux lorsque
l’enhancer distal contient des sites de liaison à
Zelda qu’en leur absence. |
|
Zelda favorise la coordination temporelle de la transcription Dès le début de la MZT, le gène snail (sna) est
transcrit dans les cellules du futur mésoderme de l’embryon de drosophile. Il code
un facteur de transcription qui va permettre la spécification du mésoderme et
contrôler la transition épithélio-mésenchymateuse lors de la gastrulation. Dès les
premiers cycles nucléaires, la protéine Zelda se lie à deux
enhancers (distal et proximal) de sna, qui
contrôlent spatialement et temporellement son expression. Pour tenter de comprendre le rôle de Zelda dans l’activation transcriptionnelle au
cours de la MZT, nous avons mesuré les temps d’activation de la transcription, dans
les cellules du mésoderme en formation, de plusieurs transgènes ayant une version
tronquée de l’enhancer distal de sna
(snaE) et possédant un nombre croissant de sites de fixation de
la protéine Zelda. Le suivi de l’activation de la transcription dans des embryons
vivants est possible grâce à l’ajout, dans une partie non-codante du transgène
rapporteur, de séquences de fixation de la protéine du bactériophage MS2 à l’ARN
[6]. Lors de la
transcription, ces étiquettes forment des « tiges-boucles » avec l’ARNm, auxquelles
va rapidement se lier une protéine de fusion fluorescente (MS2 coat
protein couplée à la green fluorescent protein), ce
qui permet de visualiser ces nouveaux transcrits (Figure 1B). Nous avons montré que la protéine Zelda
accélérait la coordination de l’activation de la transcription (synchronie) de
sna au sein du mésoderme en formation (Figure 1C). |
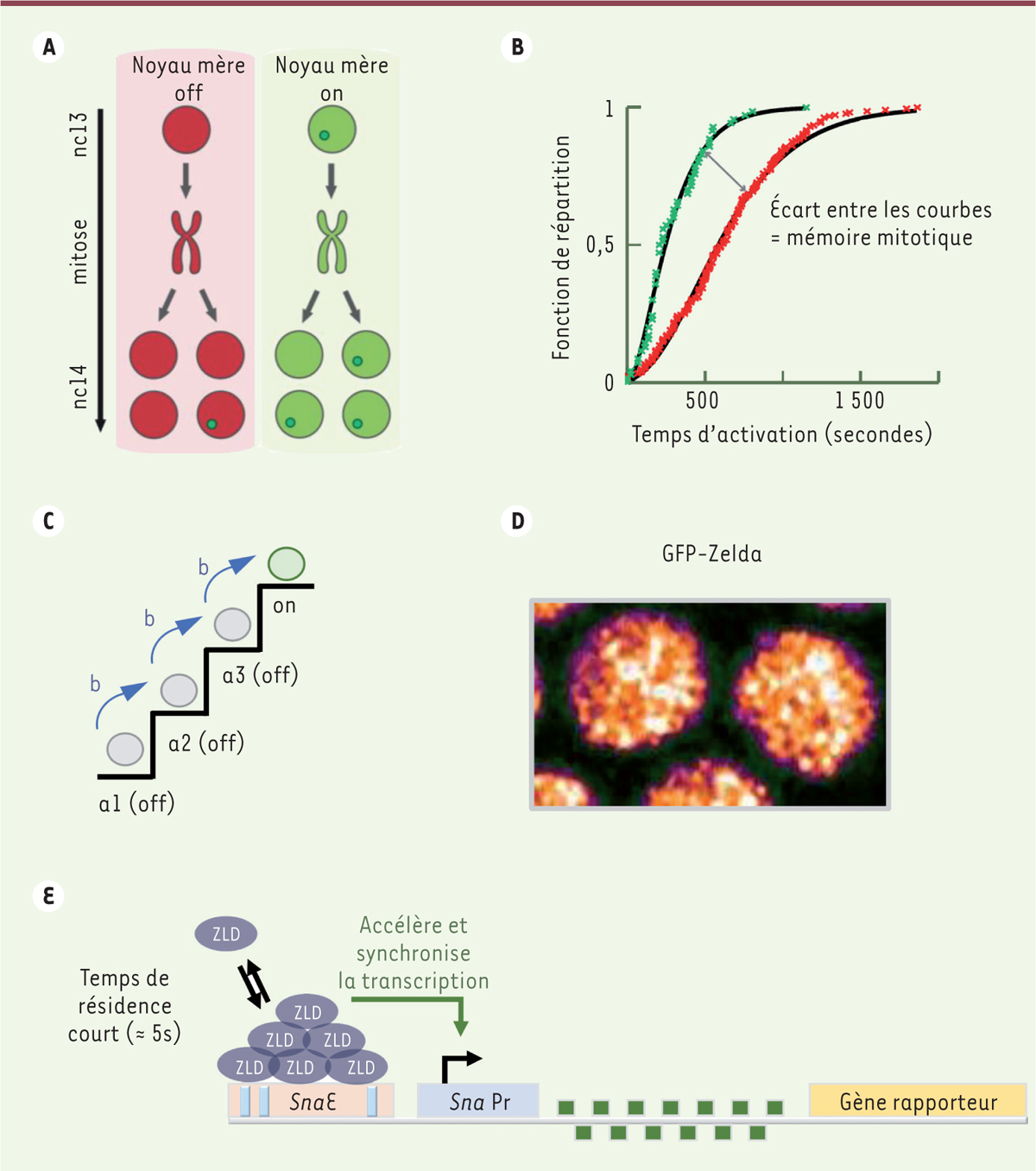
Zelda masque la mémoire transcriptionnelle La possibilité de suivre l’activation de la transcription dans des embryons vivants
permet aussi de déterminer l’effet de Zelda sur l’activité transcriptionnelle au
cours de multiples cycles nucléaires. Nous avons ainsi récemment documenté
l’existence d’une mémoire mitotique transcriptionnelle, en montrant que le statut
transcriptionnel des noyaux mères lors d’un cycle nucléaire donné influençait
l’activation de la transcription, au cycle suivant, dans les noyaux qui en dérivent
[7] (Figure 2A).
 | Figure 2 A. Schéma représentant la mémoire mitotique
entre deux cycles nucléaires consécutifs (nc 13 et nc14) de l’embryon de
drosophile. Les noyaux issus de noyaux mères dans lesquelles la
transcription génique est active (on, en vert) s’activent en moyenne
plus rapidement que ceux issus de noyaux mères dans lesquelles la
transcription génique est inactive (off, en rouge), au cycle nucléaire
suivant (nc14). B. Fonction de répartition
des temps d’activation au cycle nucléaire 14. Les valeurs des temps
d’activation transcriptionnelle des noyaux issus de noyaux mères
« actifs » sont représentées en vert, et celles des noyaux issus de
noyaux mères « inactifs » sont représentées en rouge. Les paramètres du
modèle mathématique sont estimés à partir de courbes ajustées à ces
valeurs expérimentales (courbes noires).
C. Représentation schématique des
états épigénétiques discrets nécessaires pour atteindre l’activation de
la transcription (état ON). La durée de chaque transition d’un état à
l’autre est fournie par le paramètre noté « b ».
D. Image de microscopie confocale
de la protéine Zelda étiquetée avec une GFP (par la technique
CRISPR/Cas9) montrant la présence de « microenvironnements nucléaires »
dans lesquels la protéine s’accumule.
E. Schéma représentant le rôle de Zelda
dans l’activation synchrone de la transcription de multiples gènes
zygotiques. ZLD : Zelda; GFP : green fluorescent
protein. |
Les cellules vont se diviser de nombreuses fois au cours du développement, et la
régulation génique des cellules mères doit être transmise aux cellules filles afin
de maintenir l’identité tissulaire. Cependant, à chaque division cellulaire
(mitose), la transcription s’arrête, les chromosomes se condensent, et la plupart
des facteurs de transcription se dissocient de la chromatine. Afin de transmettre
l’information d’identité cellulaire, certains mécanismes garantissent la
réactivation post-mitotique de la transcription de certains gènes d’identité dans
les cellules filles. Ce processus est appelé « bookmarking »
mitotique en référence aux facteurs qui restent associés à la chromatine durant la
mitose et servent de « marque-pages » [8]. Les facteurs de transcription « pionniers », outre leur
propriété de se lier aux nucléosomes, restent généralement associés aux chromosomes
mitotiques. La rétention mitotique de ces facteurs de transcription en fait des
candidats idéaux pour la transmission de l’identité cellulaire au cours des
divisions mitotiques. Sous l’effet de l’activation de ses enhancers, l’expression de
sna est très rapide, ce qui empêche d’analyser le rôle de Zelda
dans le tempo d’activation de sna. Nous avons donc utilisé la
version tronquée de l’enhancer distal de sna
(snaE) qui ne contient plus de sites de fixation de la protéine
Zelda, et lui avons ajouté, ou pas, des sites de fixation de Zelda. L’activation de
ce transgène est stochastique, mais toujours restreinte aux noyaux du mésoderme en
formation. Ce dispositif expérimental permet de suivre les temps d’activation
transcriptionnelle des noyaux filles provenant de noyaux mères
transcriptionnellement « actifs » ou « inactifs » (Figure 2B) : ces temps d’activation reflètent la
mémoire transcriptionnelle à travers la mitose [7]. Nous avons pu ainsi montrer que Zelda accélérait l’activation
transcriptionnelle des deux sous-populations de noyaux filles. Par une accélération
générale de la transcription, Zelda tend ainsi à diminuer le biais de mémoire
mitotique. Cependant ce biais de mémoire est récupéré lorsqu’on diminue l’expression
de ce facteur dans l’embryon, ce qui suggère l’implication d’autres facteurs dans le
processus de mémoire mitotique. Pour affiner notre compréhension du rôle biologique de Zelda dans l’activation du
génome zygotique, nous avons formalisé nos données en développant un modèle
mathématique, qui permet de mieux appréhender le nombre et la durée des étapes
nécessaires à l’activation de la transcription. Dans ce modèle, l’activation de la
transcription est atteinte après avoir parcouru une série de transitions aléatoires
entre états épigénétiques discrets (Figure
2C). Le modèle prédit que le type de distribution du temps
d’activation (loi exponentielle, loi gamma, etc.) dépend du nombre d’états discrets.
L’analyse des résultats expérimentaux en utilisant ce modèle indique qu’en moyenne,
les noyaux provenant de noyaux mères, dans lesquels la transcription est active,
doivent parcourir un nombre plus petit de transitions jusqu’à l’activation, et
suggère que Zelda agit principalement en diminuant la durée (et non le nombre) des
étapes de pré-initiation de la transcription, révélant ainsi son rôle accélérateur
dans l’embryon précoce de drosophile. |
Zelda se fixe transitoirement à la chromatine Compte tenu des caractéristiques des facteurs pionniers, nous nous attendions à ce
que Zelda joue un rôle dans la conservation de la mémoire transcriptionnelle durant
la mitose. Cependant, nos données génétiques et notre modélisation indiquent que
Zelda n’est pas le support de cette mémoire. Nous avons analysé la localisation de
Zelda dans des noyaux d’embryons vivants et avons montré que Zelda quitte le noyau à
chaque mitose, mais y retourne rapidement en fin d’anaphase. Afin de préciser les
propriétés dynamiques de cet activateur du génome, nous avons eu recours à des
expériences d’imagerie quantitative (fluorescence recovery after
photobleaching et fluorescence correlation
spectroscopy) sur embryon vivant. Nous avons montré que ce facteur de
transcription « pionnier » ne se liait à la chromatine que très transitoirement,
avec un temps de résidence estimé de l’ordre de quelques secondes. Cependant, ce
faible temps de résidence de la protéine Zelda sur la chromatine pourrait être
compensé par une augmentation locale de sa concentration. En effet, nous avons
montré que Zelda s’accumulait au sein de « microenvironnements » nucléaires
(hubs) (Figure
2D), ce qui a aussi été rapporté par une autre équipe en
utilisant une technique de microscopie à haute résolution (lattice light
sheet) permettant de suivre des particules uniques [4]. L’accumulation de Zelda dans ces
« microenvironnements » nucléaires pourrait favoriser une coopérativité entre
plusieurs facteurs de transcription [4] ou le
rapprochement de plusieurs segments d’ADN cibles (enhancers) pour
favoriser l’activation transcriptionnelle des gènes zygotiques (Figure 2E). Ces travaux de recherche [9]
ouvrent de nouvelles pistes pour mieux comprendre le rôle des facteurs de
transcription « pionniers » (e.g. Oct4, Pou5f3,
Sox2), l’activation des gènes et l’organisation nucléaire
durant la période d’activation du génome zygotique chez les vertébrés. Les gènes
codant des facteurs de transcription « pionniers », tels que ceux de la famille
Forkhead box (Fox), sont impliqués dans de nombreuses maladies
génétiques complexes telles que les cancers, la maladie de Parkinson ou les troubles
du spectre autistique [10].
De ce fait, la compréhension de l’activité spatio-temporelle de ces facteurs de
transcription « pionniers » permettrait de progresser dans la physiopathologie de
ces maladies dans le but d’identifier de nouvelles stratégies thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
Nous remercions Matthieu Dejean pour sa lecture critique du manuscrit. Ces
travaux ont été soutenus par l’ERC SyncDev et une subvention HFSP-CDA.
|
1. Tadros
W,
Lipshitz
HD, The
maternal-to-zygotic transition: a play in two acts .
Development.
2009;;136::3033.–42. 2. Hamm
DC,
Harrison
MM, Regulatory
principles governing the maternal-to-zygotic transition: insights from
Drosophila melanogaster .
Open Biol.
2018;;8::180183.. 3. Yamada
S,
Whitney
PH,
Huang
SK, et al. The
drosophila pioneer factor Zelda modulates the nuclear microenvironment of a
dorsal target enhancer to potentiate transcriptional output .
Curr Biol.
2019;;29::1387.–93. 4. Mir
M,
Stadler
MR,
Ortiz
SA, et al. Dynamic
multifactor hubs interact transiently with sites of active transcription in
Drosophila embryos .
Elife.
2018;; 7:( 10.7554/eLife.40497). 5. Mir
M,
Reime
A,
Haines
JE, et al.
Dense Bicoid hubs accentuate binding along the morphogen
gradient . Genes Dev .
2017;;31::1784.–94. 6. Bertrand
E,
Chartrand
P,
Schaefer
M, et al.,
Localization of ASH1 mRNA particles in living
yeast . Mol Cell .
1998;;2::437.–45. 7. Ferraro
T,
Esposito
E,
Mancini
L, et al.,
Transcriptional memory in the drosophila
embryo . Curr Biol .
2016;;26::212.–8. 8. Bellec
M,
Radulescu
O,
Lagha
M, Remembering the
past: mitotic bookmarking in a developing embryo .
Curr Opin Syst Biol.
2018;;11::41.–9. 9. Dufourt
J,
Trullo
A,
Hunter
J, et al. Temporal
control of gene expression by the pioneer factor Zelda through transient
interactions in hubs . Nat
Commun .
2018;;9::5194.. 10. Golson
ML,
Kaestner
KH, Fox
transcription factors: from development to disease .
Development.
2016;;143::4558.–70. |