| |
| Med Sci (Paris). 35(12): 1221–1223. doi: 10.1051/medsci/2019235.RsaI, un ARN régulateur aux multiples facettes, module
le métabolisme du pathogène opportuniste Staphylococcus
aureus Emma Desgranges,1 Delphine Bronesky,1* Anna Corvaglia,2 Patrice François,2 Carlos Caballero,3 Laura Prado,3 Alejandro Toledo-Arana,3 Inigo Lasa,4 Karen Moreau,5 François Vandenesch,5 Stefano Marzi,1 Pascale Romby,1 and Isabelle Caldelari1** 1Université de Strasbourg, CNRS, Architecture et réactivité
de l’ARN, UPR9002, F-67000Strasbourg,
France 2Laboratoire de recherche en génomique, département de
spécialités médicales, Genève hôpitaux universitaires, université de
Genève, Genève, Suisse 3Instituto de agrobiotecnología. CSIC-UPNA-GN,
31192-Mutilva,
Navarra,
Espagne 4Navarrabiomed, universidad pública de Navarra, departamento
de salud, IDISNA, Pamplona, Espagne 5CIRI, centre international de recherche en infectiologie,
Inserm U1111, université Lyon1, école normale supérieure de Lyon, CNRS
UMR5308, F-69008Lyon,
France MeSH keywords: |
Staphylococcus aureus est une bactérie commensale retrouvée chez environ
30 % des individus sains dont elle colonise la peau et la muqueuse nasale. Cependant,
c’est également une bactérie pathogène opportuniste responsable d’infections diverses
telles que orgelet, ostéomyélite, endocardite, ou encore septicémie en envahissant un
grand nombre de tissus et d’organes. Cette bactérie est capable de s’adapter à des
conditions hostiles et variées, telles que carence nutritive et stress osmotique,
oxydant, ou thermique, ainsi qu’à la réponse immunitaire de l’hôte, car elle produit une
grande diversité de facteurs de virulence. La synthèse de ces facteurs est finement
régulée par des protéines et des ARN régulateurs majoritairement non codants, souvent
désignés par l’abréviation sARN (dérivée de l’anglais, small RNA). Les
facteurs de transcription et les systèmes à deux composants contrôlent l’expression des
gènes impliqués non seulement dans le métabolisme, mais aussi dans la réponse au stress
et la virulence [1]. Par exemple,
la protéine du contrôle catabolique (carbon catabolite control protein
A, CcpA) a un rôle essentiel dans le choix de la source carbonée en
régulant le métabolisme central de la bactérie ainsi que la virulence [2, 3]. CcpA se fixe à une séquence promotrice spécifique appelée
cre (catabolite-responsive element), qui est très
conservée chez les bactéries à Gram positif [2].
Quant aux sARN, ils interagissent principalement avec leurs ARN messagers (ARNm) cibles.
L’hybridation peut conduire à la stabilisation/déstabilisation de l’ARNm ou à
l’activation/répression de sa traduction [4]. Nous avons montré que la transcription du sARN RsaI (RNA Staphylococcus aureus
I) est réprimée par CcpA en présence de glucose [5]. L’induction de la synthèse de RsaI signale que
la concentration en glucose diminue dans le milieu extracellulaire et que la croissance
des bactéries est ralentie. En interagissant avec ses ARNm cibles ou d’autres sARN, il
permet à la population bactérienne de modifier son métabolisme lorsque la source
carbonée primaire est consommée. |
RsaI est inhibé par CcpA en présence de glucose RsaI est un sARN de 144 nucléotides, très conservé chez les
staphylococcaceae, et qui comporte deux motifs riches en
guanosine ainsi qu’une longue région simple brin riche en pyrimidines. Un motif
cre de liaison à CcpA a été détecté dans la région promotrice
du gène codant RsaI. Ce sARN est exprimé dès l’entrée en phase stationnaire de
croissance des bactéries dans un milieu riche contenant 2 % de glucose [6]. Néanmoins, dans un milieu
sans glucose ou dans une souche délétée du gène ccpA, RsaI est
exprimé de manière constitutive, suggérant que CcpA est le principal répresseur de
RsaI en présence de glucose et que cette répression est levée après consommation du
glucose (en début de phase stationnaire). |
L’identification des cibles de RsaI révèle ses fonctions La technique MAPS (MS2-affinity purification coupled with RNA
sequencing) a été utilisée afin d’identifier l’ensemble des cibles ARN
directes de RsaI (Figure 1)
[7]. Cette technique
permet d’isoler les complexes formés entre RsaI et ses ARN associés in
vivo. RsaI a été au préalable fusionné à une étiquette MS2 placée en 5’
du sARN. Cette étiquette est spécifiquement reconnue par la protéine de capside du
phage MS2 (MS2-CP), qui est elle-même fixée sur une résine d’amylose pour retenir
spécifiquement les complexes contenant RsaI-MS2. En couplant à cette chromatographie
d’affinité une technique de séquençage à haut débit (Miseq,
Illumina), les ARN liés à RsaI-MS2 sont caractérisés.
L’interaction directe entre RsaI et ses cibles potentielles, ainsi que les
conséquences de son action sur la régulation du métabolisme de la bactérie ont été
ensuite validées par une combinaison d’approches in vivo et
in vitro. Ainsi, nous avons révélé l’existence de deux classes
de cibles pour RsaI: des ARNm impliqués dans l’importation du glucose
(glcU_2), le métabolisme des sucres (treB), la
détoxification des protéines (fn3K), et la formation des biofilms
(icaR), et des sARN possédant des séquences riches en cytosine,
complémentaires de la séquence du site de fixation du ribosome sur leurs ARNm cibles
(séquence de Shine-Dalgarno). Par ailleurs, le transcriptome différentiel indique
que RsaI inhiberait la voie des pentoses phosphate et, à l’inverse, activerait des
enzymes des voies de fermentation ainsi que d’autres protéines requises pour
résister au monoxyde d’azote (NO), un métabolite libéré par les cellules
endothéliales, les polynucléaires neutrophiles, et les macrophages de l’hôte.
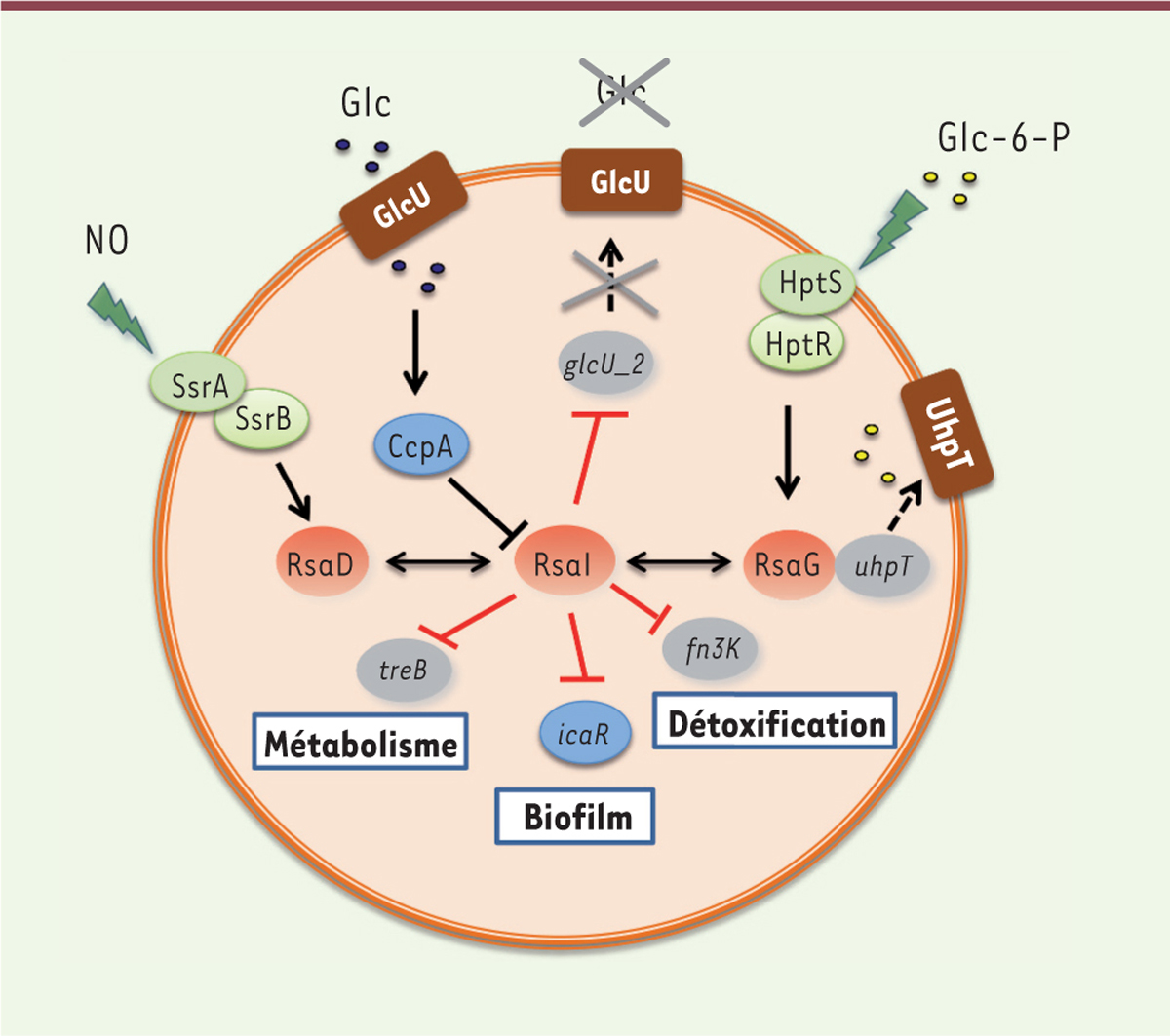
 | Figure 1. Représentation schématique des réseaux de régulation
centrés sur l’ARN régulateur RsaI. Les sARN sont
colorés en rouge, les facteurs de transcription en bleu, les systèmes à
deux composants en vert, les ARNm en gris, et les transporteurs en
marron. Les connecteurs noirs ou rouges indiquent respectivement un
contrôle transcriptionnel ou post-transcriptionnel. Les flèches
symbolisent une activation, tandis que les barres transversales
indiquent une inhibition. sARN: small RNA ; RsaI:
RNA Staphylococcus aureus I. |
|
RsaI contient deux domaines fonctionnels distincts La mutagenèse dirigée a permis de distinguer deux motifs fonctionnels de RsaI. Sa
région en simple brin riche en adénine et uridine interagit avec les séquences de
Shine-Dalgarno de ses ARNm cibles, inhibant ainsi leur traduction. C’est le cas pour
les transcrits codant le transporteur du glucose GlcU_2, le transporteur du
tréhalose TreB, et la fructosamine kinase Fn3K impliquée dans la déglycation des
protéines en présence de fortes concentrations de glucose (Figure 2A). Les deux
motifs riches en guanine interagissent quant à eux avec les motifs riches en
cytosine des sRNA RsaG et RsaD. RsaG est induit par la présence de
glucose-6-phosphate (G6P) dans le milieu extracellulaire sous le contrôle du système
à deux composants HptRS (hexose phosphate transport RS, R désignant
le régulateur et S le senseur), alors que RsaD répond au NO sous la
dépendance du système SsrAB (staphylococcal respiratory response
AB) (Figure 1). Etonnamment,
RsaG serait co-transcrit avec l’ARNm de uhpT (uptake of
hexose phosphate transporter) codant le transporteur du G6P
(Figure 1). Nous avons
montré que la fixation de RsaG n’affecte pas les fonctions régulatrices de RsaI,
telles que la répression traductionnelle de glcU_2 (Figures 1 et
2B). Il reste à déterminer si RsaI peut agir comme une « éponge à sARN
» afin d’empêcher leur action. |
RsaI module la synthèse de PIA-PNAG, un composant de la matrice des
biofilms S. aureus est la cause principale des infections nosocomiales chez
les patients opérés pour la pose d’une prothèse, où se développent facilement des
biofilms bactériens résistants au traitement antibiotique. L’opéron
icaADBC code les enzymes responsables de la synthèse de
l’exopolysaccharide PIA-PNAG (polysaccharide intercellular
adhesin-poly-N-acetylglucosamine), composant majeur de la matrice des
biofilms. Ces gènes sont réprimés par le facteur de transcription IcaR. L’ARNm
icaR a la particularité d’inhiber sa propre traduction en
induisant sa circularisation. En effet, une séquence riche en cytosine (anti-SD)
située dans la région 3’ non-traduite (3’UTR pour 3’ untranslated
region) s’hybride à la séquence SD pour empêcher la fixation du
ribosome et induire la dégradation de l’ARNm icaR par
l’endoribonucléase III (Figure
2C) [8].
L’ARNm icaR a été identifié comme une cible de RsaI par la
technique MAPS. Nous avons montré que RsaI interagit avec la 3’UTR de l’ARNm
icaR en aval de la séquence anti-SD (Figure 2C). Des expériences
permettant d’évaluer la production de PIA-PNAG indiquent que RsaI et la région 3’UTR
de l’ARNm icaR sont nécessaires pour inhiber le répresseur IcaR et
activer la synthèse de PIA-PNAG (Figure
2C).
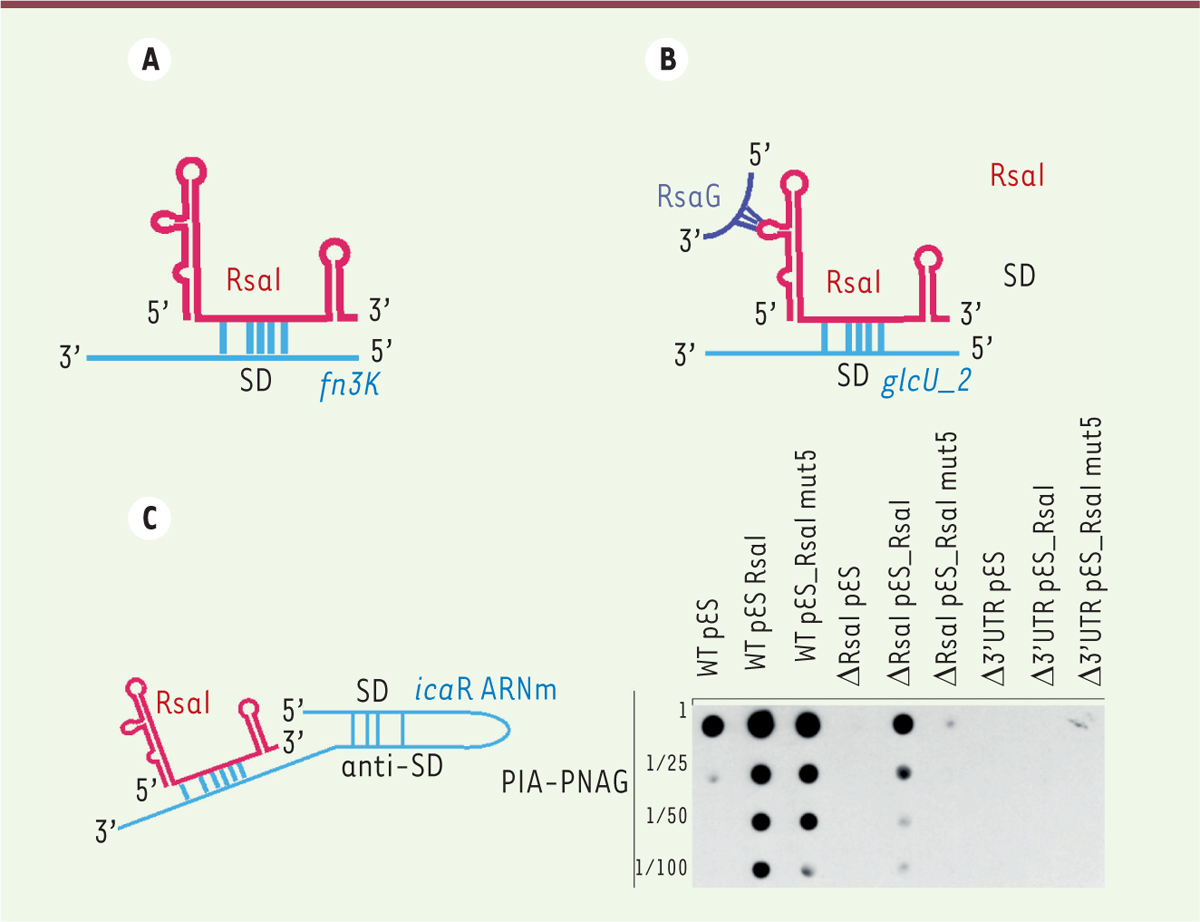
 | Figure 2.Divers mécanismes de régulation impliquant RsaI.
A. Schéma du complexe formé entre RsaI et l’ARNm
de fn3K. RsaI inhibe la traduction de cet ARNm en se
fixant à sa séquence de Shine-Dalgarno (SD).
B..Schéma du complexe formé entre
RsaI, l’ARNm de glcU_2, et RsaG. La liaison de RsaG
n’empêche pas RsaI de se fixer à son ARNm cible et d’y exercer son
contrôle. C. Effet de RsaI sur la synthèse
des exopolysaccharides PIA-PNAG in vivo. à gauche,
schéma représentant RsaI se liant à la région 3’ de l’ARNm
icaR. L’interaction entre les séquences
complémentaires SD et anti-SD est également indiquée. A droite, la
biosynthèse de PIA-PNAG a été quantifiée par la technique de dot
blot. Des échantillons ayant subi une série de dilutions
(jusqu’à 1:100) ont été déposés sur une membrane de nitrocellulose, et
la production de PIA-PNAG a été détectée avec des anticorps spécifiques
de cet exopolysaccharide dans la souche 132 de S.
aureus (WT, pour wild-type), la souche
délétée pour le gène rsaI (D rsaI), et
la souche portant une délétion de la région icaR 3’UTR
(Δ3’UTR). Ces trois souches ont été transformées soit avec un plasmide
pES (pES), soit avec le plasmide recombinant exprimant
rsaI (pES _RsaI) ou sa forme mutée
rsaI mut5 (pES _RsaI mut5), dont la
séquence d’interaction avec l’ARNm icaR a été inactivée
par substitution (d’après [ 5]). |
|
Ces résultats illustrent le lien étroit entre la régulation du métabolisme,
l’adaptation au stress, et la formation de biofilms bactériens chez S.
aureus, impliquant le facteur de transcription CcpA et le sARN RsaI, et
l’utilisation des sources carbonées. Cet ARN non-codant aux multiples facettes
pourrait être principalement produit au niveau des sites d’inflammation où la
concentration en glucose est faible (e.g., poumons, reins), et
intervenir dans la persistance des bactéries lorsque les conditions sont
défavorables à leur multiplication. Il reste encore beaucoup à apprendre sur le rôle
des sARN et sur leurs réseaux d’interactions dans le contrôle des voies métaboliques
essentielles à l’invasion, la survie, et la dormance de S. aureus.
Elucider ces mécanismes complexes de régulation permettra de mieux comprendre la
pathogénicité de S. aureus, et de développer de nouvelles
stratégies thérapeutiques contre les infections redoutables dont elle est parfois
responsable. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
1. Richardson
AR,
Somerville
GA,
Sonenshein
AL. Regulating the
intersection of metabolism and pathogenesis in Gram-positive
bacteria . Microbiol Spectr.
2015; ; 3:
:1.–27. 2. Seidl
K,
Stucki
M,
Ruegg
M, et al.
Staphylococcus aureus CcpA affects virulence determinant
production and antibiotic resistance .
Antimicrob Agents Chemother.
2006; ; 50:
:1183.–1194. 3. Li
C,
Sun
F,
Cho
H, et al.
CcpA mediates proline auxotrophy and is required for
Staphylococcus aureus pathogenesis . J
Bacteriol.
2010; ; 192:
:3883.–3892. 4. Wagner
EGH,
Romby
P. Small RNAs in
bacteria and archaea: who they are, what they do, and how they do
it . Adv Genet.
2015; ; 90:
:133.–208. 5. Bronesky
D,
Desgranges
E,
Corvaglia
A, et al.
A multifaceted small RNA modulates gene expression upon glucose
limitation in Staphylococcus aureus . EMBO
J.
2019; ; 38: :e99363.. 6. Geissmann
T,
Chevalier
C,
Cros
M-J, et al.
A search for small noncoding RNAs in Staphylococcus aureus
reveals a conserved sequence motif for regulation .
Nucleic Acids Res.
2009; ; 37:
:7239.–7257. 7. Lalaouna
D,
Desgranges
E,
Caldelari
I, et al.
MS2-affinity purification coupled with RNA sequencing approach in
the human pathogen Staphylococcus aureus .
Meth Enzymol.
2018; ; 612:
:393.–411. 8. Ruiz de los
Mozos
I,
Vergara-Irigaray
M,
Segura
V, et al.
Base pairing interaction between 5’- and 3’-UTRs controls icaR
mRNA translation in Staphylococcus aureus .
PLoS Genet.
2013; ; 9:
:e1004001.. |