| |
| Med Sci (Paris). 35(2): 106–109. doi: 10.1051/medsci/2019016.SUMO régule la capacité de TRIM5α à inhiber le
VIH-1 Sébastien Nisole,1* Juliette Fernandez,1 Ghizlane Maarifi,1 and Nathalie J. Arhel1** 1Institut de Recherche en Infectiologie de Montpellier (IRIM),
Université de Montpellier, CNRS UMR 9004, 1919, route de Mende, 34090Montpellier,
France MeSH keywords: Animaux, Antiviraux, Protéines de transport, Infections à VIH, VIH-1 (Virus de l'Immunodéficience Humaine de type 1), Humains, Protéine SUMO-1, Sumoylation, métabolisme, physiologie, génétique, immunologie |
Les facteurs de restriction rétrovirale Comme tous les virus, les rétrovirus, dont le virus de l’immunodéficience humaine de
type 1 (VIH-1), sont des « parasites » intracellulaires stricts. Le VIH-1 infecte
essentiellement les lymphocytes T CD4+, mais également les cellules
dendritiques et les macrophages. Suite à l’entrée du virus dans ses cellules-cibles,
la capside contenant l’ARN viral chemine à travers le cytoplasme jusqu’à la membrane
nucléaire où l’ARN viral est rétro-transcrit en ADN double-brin. De façon
concomitante, la capside subit un désassemblage contrôlé, permettant la
translocation du complexe de pré-intégration contenant l’ADN proviral à travers les
pores nucléaires afin qu’il s’intègre au génome de la cellule. Une fois intégré, la
transcription, l’épissage et la traduction des ARN viraux néo-synthétisés sont
assurés par la machinerie cellulaire et les nouvelles particules virales sont
assemblées et libérées à la surface des cellules. Parallèlement au détournement de
la machinerie cellulaire à son profit, le VIH-1 doit également éviter, ou
contrecarrer, un arsenal de défenses cellulaires mis en place pour limiter sa
réplication. Ces défenses sont assurées par des protéines cellulaires connues sous
le terme de facteurs de restriction, qui interfèrent avec de
multiples étapes de son cycle réplicatif [1]. APOBEC3G (apolipoprotein B mRNA editing enzyme
catalytic subunit 3G), TRIM5α (tripartite motif protein
5α), SAMHD1 (sterile alpha motif and histidine-aspartate
domain-containing protein 1), Mx2 (MX dynamin-like GTPase
2), et BST-2 (bone marrow stromal antigen 2), sont
quelques-uns des nombreux facteurs de restriction identifiés depuis une quinzaine
d’années, mais il est vraisemblable que d’autres facteurs de restriction anti-VIH
restent encore à découvrir [2] (→).
(→) Voir la Nouvelle de G. Chougui et al,
m/s n° 1, janvier 2019, page 9
TRIM5α est une protéine appartenant à la famille TRIM, qui regroupe des protéines
présentant une organisation commune, constituée de 3 domaines conservés comprenant
un domaine RING1,, un ou deux motifs B-Box2, et une région superhélice
(coiled-coil). TRIM5α possède, en plus de ces 3 domaines
canoniques, un domaine B30.23 à son extrémité
C-terminale, qui lui permet de reconnaître la capside virale après son entrée dans
le cytoplasme (Figure 1A). En
réponse à cette reconnaissance, la protéine s’auto-oligomérise autour de la capside
et entraîne son désassemblage précoce, inhibant ainsi la réplication du virus [3]. C’est chez le macaque
rhésus que cette activité antivirale de TRIM5α a été découverte en 2004 [4]. En effet, comme la plupart
des singes, les macaques résistent à l’infection par le VIH-1 grâce à l’expression
de cette protéine dans leurs cellules [5]. Les cellules humaines sont, quant à elles, incapables de
bloquer le VIH-1, mais la protéine TRIM5α humaine peut cependant inhiber certains
rétrovirus équins et murins [4].
 | Figure 1. Identification des sites de SUMOylation dans TRIM5a.
A. Représentation schématique de la protéine
TRIM5α. Le principal site de SUMOylation ainsi que les autres sites de
SUMOylation potentiels identifiés dans la protéine TRIM5α humaine sont
représentés. B..Principaux motifs
consensus de SUMOylation décrits dans la littérature. Ψ, acide-aminé
hydrophobe ; X, résidu quelconque ; pS, sérine phosphorylée. |
|
Régulation des facteurs de restriction Comme tout processus cellulaire, la restriction rétrovirale est un mécanisme
hautement régulé et ce, à de multiples niveaux. Cette régulation s’effectue
initialement au niveau transcriptionnel puisque la plupart des facteurs de
restriction ne sont pas ou peu transcrits constitutivement. Ils sont induits par les
interférons de type 1, des cytokines sécrétées par les cellules infectées qui
déclenchent une réponse antivirale dans les cellules avoisinantes afin de limiter la
propagation virale [5]. Un autre niveau de
régulation important est constitué par les modifications post-traductionnelles. La
plus fréquente de ces modifications, la phosphorylation, régule par exemple
l’activité antivirale de SAMHD1 [6]. L’autre principale modification post-traductionnelle est
l’ubiquitination, qui consiste en la fixation covalente d’une ou de plusieurs
protéines d’ubiquitine sur une protéine cible, ce qui conduit, en général, à sa
dégradation par le protéasome. C’est par ce mécanisme que le VIH-1 contrecarre la
restriction imposée par APOBEC3G ou BST-2 [1].
De nombreuses modifications apparentées à l’ubiquitination existent, comme la
SUMOylation, l’ISGylation4 ou la NEDDylation,
qui impliquent toutes la fixation covalente mais réversible d’une petite protéine à
une protéine acceptrice. La SUMOylation, par exemple, consiste en la conjugaison
d’une ou plusieurs protéines SUMO au niveau d’un résidu lysine d’une protéine cible.
Cette modification peut modifier la stabilité, la localisation sub-cellulaire ou
l’activité d’une protéine [7]. Tout comme l’ubiquitination, la SUMOylation fait intervenir 3 enzymes :
une enzyme activatrice E1 (hétérodimère SAE1/SAE2 [SUMO-1-activating enzyme
1/2]), une enzyme de conjugaison E2 (Ubc9
[ubiquitin-conjugating enzyme 9]) et l’une des nombreuses
E3-SUMO ligases. Le site consensus de SUMOylation est constitué du motif ΨKXE, dans
lequel Ψ est un acide-aminé hydrophobe (Ile, Leu ou Val), X un résidu quelconque, et
E un acide-aminé acide (Glu ou Asp). Outre ce motif principal, d’autres motifs
alternatifs ont été décrits, dont les principaux exemples sont représentés sur la
Figure 1B
[7]. |
SUMO régule l’activité et la localisation sub-cellulaire de TRIM5a Il était présumé depuis plusieurs années que l’activité de restriction de TRIM5α
pouvait être régulée par SUMO, mais la preuve de cette implication n’a été apportée
que récemment, et le mécanisme mis en jeu vient d’être élucidé [8-10]. En 2015, nous avons montré que la
surexpression de SUMO exacerbait l’activité anti-VIH de TRIM5α, tandis que
l’extinction de la machinerie de SUMOylation par ARN interférence l’inhibait,
suggérant ainsi que la SUMOylation de TRIM5α pourrait réguler son activité [8]. Nous avons pu confirmer que TRIM5α
constituait un substrat de SUMO, puisque nous sommes parvenus à détecter pour la
première fois des formes SUMOylées de TRIM5α dans des expériences de SUMOylation
in vitro ainsi que dans des cellules surexprimant TRIM5α, Ubc9
et SUMO. Nous avons initialement constaté que l’enzyme E3-SUMO ligase PIAS1
(protein inhibitor of activated STAT 1) pouvait catalyser la
SUMOylation de TRIM5α in vitro, en modifiant la lysine K10
localisée au sein d’un site consensus ΨKXE (VK10EE) de la protéine.
Cependant, la mutation de ce résidu K10 s’est révélée sans effet sur la capacité de
TRIM5α à inhiber l’infection par le VIH-1, remettant temporairement en question
l’implication de SUMO dans son activité de restriction. Récemment, nous avons à nouveau examiné cette implication sur la base d’une nouvelle
hypothèse : étant donné que PIAS1 est exclusivement nucléaire et qu’à l’inverse,
TRIM5α exerce son activité dans le cytoplasme, il paraissait peu vraisemblable que
cette E3-SUMO ligase soit responsable de la SUMOylation de TRIM5α in
vivo. Nous avons donc entrepris d’identifier la E3-SUMO ligase
responsable de la SUMOylation de TRIM5α in vivo, en limitant
l’étude aux enzymes dont la localisation était compatible avec celle de TRIM5α
[10]. Nous avons ainsi identifié RanBP2
(Ras-related nuclear protein-binding protein 2) comme étant la
E3-SUMO ligase qui modifie TRIM5α dans les cellules humaines. RanBP2, également
nommée Nup358, est une protéine de la famille des nucléoporines, qui forme des
filaments sur la face cytoplasmique des pores nucléaires et est notamment impliquée
dans le transport nucléo-cytoplasmique des protéines. Il est intéressant de noter
que RanBP2 est également la protéine qui permet l’ancrage des capsides de VIH-1 au
pore nucléaire, prérequis à la translocation des complexes de pré-intégration dans
le noyau [11]. RanBP2 est
associée aux protéines RanGAP (Ran GTPase-activating protein), Ubc9
et SUMO pour former un complexe multiprotéique impliqué dans la SUMOylation de
protéines dans le cytoplasme [12]. Nous avons pu montrer que TRIM5α est associée à ce complexe et que
RanBP2 est responsable de sa conjugaison à SUMO (Figure 2).
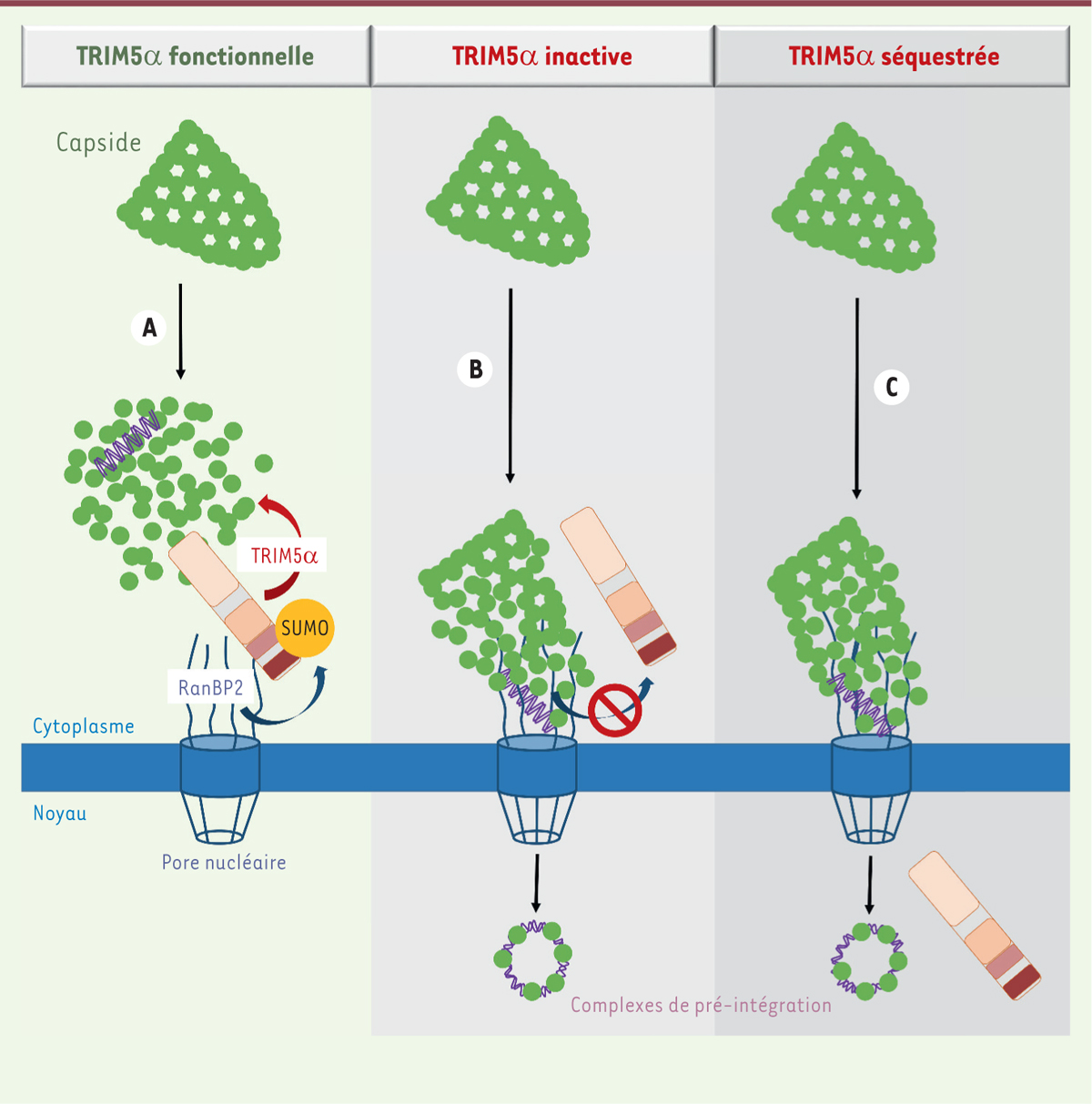
 | Figure 2. La SUMOylation de la protéine TRIM5a par RanBP2 régule son
activité antivirale. Suite à l’entrée du virus
de l’immunodéficience humaine de type 1 (VIH-1) dans le cytoplasme de
ses cellules-cibles, la capside virale chemine vers les pores
nucléaires. Le désassemblage contrôlé des capsides et la
rétro-transcription de l’ARN viral conduisent à la formation des
complexes de pré-intégration qui vont être transloqués dans le noyau.
A. La capside du VIH-1 subit un
désassemblage précoce par la protéine TRIM5α (tripartite motif
protein 5a) quand celle-ci est SUMOylée par RanBP2
(Ras-related nuclear protein -binding protein 2).
B. L’inhibition de la SUMOylation
de la protéine TRIM5α l’inactive et restaure ainsi la capacité du VIH-1
à réaliser son cycle de réplication.
C. Dans les cellules dendritiques, TRIM5α
est incapable de reconnaître les capsides dans le cytoplasme car elle
est déSUMOylée et séquestrée dans le noyau des cellules. |
Une fois l’enzyme E3 identifiée, il nous restait alors à déterminer quel résidu était
la cible de cette SUMOylation. En effet, PIAS1 n’étant pas l’enzyme responsable de
la SUMOylation de TRIM5α dans les cellules. Il paraissait donc vraisemblable qu’un
résidu autre que K10 en soit le substrat. Nous avons testé cette hypothèse et
découvert que la protéine TRIM5α mutée sur le résidu K10 était toujours SUMOylée
in vivo [10]. La
recherche d’autres sites potentiels de SUMOylation a permis d’identifier trois
autres lysines potentiellement SUMOylables : K84, K111 et K167 dans la protéine
humaine, et K85, K113 et K169 dans celle de macaque rhésus (Figure 1A) [10]. Dans nos expériences, la protéine TRIM5α humaine mutée sur
la lysine 84 fut la seule à ne plus être SUMOylée dans les cellules, démontrant que
le site EVK84LSP était le principal site de SUMOylation de la protéine
TRIM5α par RanBP2 (Figure 1A).
Ce site correspond à deux consensus différents puisqu’il constitue un motif
consensus inversé ainsi qu’un motif de SUMOylation phosphorylé (pSUM) caractérisé
par la présence d’une sérine phosphorylée (pSer) (Figure 1B) [7]. Ce mutant de TRIM5α est incapable d’inhiber l’infection virale,
démontrant ainsi que la SUMOylation de TRIM5α par RanBP2 sur le résidu K84 est
indispensable à son activité de restriction (Figure 2A). Ainsi, la mutation de ce résidu ou l’extinction
de l’expression de RanBP2 par ARN interférence empêche TRIM5α d’exercer son
activité, comme illustré dans la
Figure 2B
. Une autre observation intéressante a permis de mettre en évidence un niveau
supplémentaire de contrôle de l’activité antivirale de TRIM5α par la machinerie
SUMO. Nous avons en effet constaté que la SUMOylation de TRIM5α régulait non
seulement son activité, mais également sa localisation sub-cellulaire. Nous avons
ainsi pu montrer que la SUMOylation de TRIM5α lui permettait de transiter entre le
cytoplasme et le noyau [9].
Si TRIM5α est déSUMOylée dans le noyau, elle y est alors séquestrée et perd ainsi sa
capacité à bloquer les capsides virales présentes dans le cytoplasme [9] (Figure
2C). Cette séquestration nucléaire de TRIM5α peut être
provoquée artificiellement in vitro, mais elle peut également être
physiologique, notamment dans les cellules dendritiques. Dans ces cellules, la
déSUMOylation de TRIM5α entraîne sa séquestration dans des corpuscules nucléaires
appelés corps de Cajal, ce qui conduit à une absence de restriction dans ces
cellules [9, 13]. L’ensemble de nos travaux a donc permis de démontrer que SUMO constitue un régulateur
majeur des fonctions antivirales de TRIM5α. Il a permis de mettre en évidence le
rôle central des pores nucléaires et également la dualité de la protéine RanPB2,
bloquant l’infection par le VIH-1 dans les cellules, mais détournée par le virus
pour assurer sa réplication. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
Ce travail a été financé par l’Agence Nationale de Recherches sur le SIDA et les
hépatites virales (ANRS), le programme ATIP/Avenir et le Labex EpiGenMed, au titre
du programme « Investissements d’avenir » (ANR-10-LABX-12-01).
|
Footnotes |
1. Soliman
M,
Srikrishna
G,
Balagopal
A. Mechanisms of
HIV-1 control . Curr HIV/AIDS Rep.
2017; ; 14 :
:101.–109. 2. Chougui
G,
Martin
M,
Matkovic
R, et al.
Vpx n’a pas dit son dernier mot : « Hush ! » .
Med Sci (Paris).
2019; ; 35 :
:9.–12. 3. Stremlau
M,
Perron
M,
Lee
M, et al.
Specific recognition and accelerated uncoating of retroviral
capsids by the TRIM5alpha restriction factor . Proc
Natl Acad Sci USA.
2006; ; 103 :
:5514.–5519. 4. Stremlau
M,
Owens
CM,
Perron
MJ, et al.
The cytoplasmic body component TRIM5alpha restricts HIV-1
infection in old world monkeys . Nature.
2004; ; 427 :
:848.–853. 5. Bourke
NM,
Napoletano
S,
Bannan
C, et al.
Control of HIV infection by IFN-alpha: implications for latency
and a cure . Cell Mol Life Sci.
2018; ; 75 :
:775.–783. 6. Cribier
A,
Descours
B,
Valadao
AL, et al.
Phosphorylation of SAMHD1 by cyclin A2/CDK1 regulates its
restriction activity toward HIV-1 . Cell Rep.
2013; ; 3 :
:1036.–1043. 7. Flotho
A,
Melchior
F. Sumoylation: a
regulatory protein modification in health and disease .
Annu Rev Biochem.
2013; ; 82 :
:357.–385. 8. Dutrieux
J,
Portilho
DM,
Arhel
NJ, et al.
TRIM5alpha is a SUMO substrate .
Retrovirology.
2015; ; 12 : :28.. 9. Portilho
DM,
Fernandez
J,
Ringeard
M, et al.
Endogenous TRIM5alpha function Is regulated by SUMOylation and
nuclear sequestration for efficient innate sensing in dendritic
cells . Cell Rep.
2016; ; 14 :
:355.–369. 10. Maarifi
G,
Fernandez
J,
Portilho
DM, et al.
RanBP2 regulates the anti-retroviral activity of TRIM5α by
SUMOylation at a predicted phosphorylated SUMOylation motif .
Commun Biol.
2018; ; 1 : :193.. 11. Di Nunzio
F,
Danckaert
A,
Fricke
T, et al.
Human nucleoporins promote HIV-1 docking at the nuclear pore,
nuclear import and integration . PloS One.
2012; ; 7 : :e46037.. 12. Werner
A,
Flotho
A,
Melchior
F. The
RanBP2/RanGAP1*SUMO1/Ubc9 complex is a multisubunit SUMO E3
ligase . Mol Cell.
2012; ; 46 :
:287.–298. 13. Arhel
NJ,
Nisole
S,
Carthagena
L, et al.
Lack of endogenous TRIM5alpha-mediated restriction in rhesus
macaque dendritic cells . Blood.
2008; ; 112 :
:3772.–3776. |