| |
| Med Sci (Paris). 35(2): 113–115. doi: 10.1051/medsci/2019018.Des complexes protéiques impliqués dans la surveillance
de l’ARN nucléaire inhibent la transcription du VIH-1 Kader Salifou,1 Rosemary Kiernan,1** and Xavier Contreras1* 1Institut de génétique humaine, UMR 9002, CNRS, Université de
Montpellier, Gene regulation laboratory,
141, rue de la
Cardonille, 34396Montpellier,
France MeSH keywords: Exosome multienzyme ribonuclease complex, Régulation de l'expression des gènes viraux, VIH-1 (Virus de l'Immunodéficience Humaine de type 1), Humains, Complexes multiprotéiques, Maturation post-transcriptionnelle des ARN, Stabilité de l'ARN, ARN nucléaire, ARN viral, Facteurs de transcription, Transcription génétique, Latence virale, physiologie, génétique, métabolisme |
VIH-1 et latence, la principale barrière à l’éradication du virus La trithérapie anti-VIH-1 (virus de l’immunodéficience humaine de type 1) a atteint
une certaine maturité. Les combinaisons de molécules utilisées représentent une
approche thérapeutique très efficace pour supprimer la réplication du virus chez les
individus infectés et elle est de mieux en mieux tolérée par les patients.
Toutefois, malgré ces succès, la trithérapie ne permet pas la guérison [1] (→).
(→) Voir l’Éditorial de F. Barré-Sinoussi, m/s n° 6-7,
juin-juillet 2018, page 499
En effet, bien que la virémie soit réduite à un taux indétectable, l’arrêt de la
thérapie entraîne un rebond rapide de la réplication virale et une progression de la
maladie. Ce rebond s’explique par la persistance, chez les individus traités, de
virus compétents pour la réplication formant un réservoir de virus latents [2, 3] (→).
(→) Voir les Synthèses de C. Schwartz et al.,
m/s n° 2, février 2010, page 159 et de T. Cherrier
et al., m/s n° 3, mars 2010,
page 291
On peut définir les cellules infectées de façon latente comme des cellules contenant
un provirus compétent pour la réplication, mais dont le génome ne s’exprime pas
[4]. Le réservoir viral s’établit très tôt au cours de l’infection, avant la
séroconversion. La majeure partie du réservoir de VIH-1 est trouvée dans les
cellules mononucléées du sang (peripheral blood mononuclear cell,
PBMC) et notamment les lymphocytes T CD4+. Dans ces cellules, le VIH-1 a
pu s’intégrer, mais il est latent : il est transcriptionnellement inactif et ne se
réplique pas car il n’est plus dans un environnement permissif, ces cellules étant
ou devenant quiescentes. Les cellules contenant un virus latent sont rares (de
l’ordre de 1 pour un million de lymphocytes T CD4+). Cependant, le
réservoir de virus latent a une durée de vie très longue et surtout est insensible
aux trithérapies qui ne ciblent que le virus en cours de réplication. Des études ont défini de nombreux mécanismes impliqués dans l’inactivation de la
transcription du VIH latent. Notamment, le site d’intégration du virus et l’absence
de facteurs de transcription dans les cellules quiescentes contribuent fortement à
sa latence. Activer la transcription du virus latent afin de permettre son élimination est une
des stratégies thérapeutiques envisagées pour éradiquer le VIH. Il est donc crucial
de comprendre les mécanismes impliqués dans la répression de la transcription du VIH
lui permettant de rester latent dans les cellules qu’il a infectées. |
Les facteurs de l’exosome nucléaire répriment l’expression d’un gène rapporteur
sous contrôle du promoteur du VIH-1 Une des étapes principales de blocage de la transcription du VIH-1 dans les cellules
latentes est l’étape de transition entre initiation et élongation. L’ARN polymérase
entre en pause après la synthèse des premiers nucléotides de l’ARN messager viral
qui forment une structure en tige-boucle appelée TAR (trans-activation
response element). Nous avons précédemment montré l’implication de
l’exosome nucléaire dans la répression de l’expression du génome viral [5]. L’exosome nucléaire est un
complexe protéique jouant un rôle crucial dans la maturation des ARN (dont les ARN
ribosomiques) et l’élimination des ARN aberrants [6]. Nous avions en effet montré que l’inhibition
de l’expression de sa principale sous-unité catalytiques, RRP6 (ribosomal
RNA-processing protein 6), induisait une forte
augmentation de la transcription provenant d’un promoteur VIH-1. Cette inhibition
résultait de la dégradation de l’ARN viral naissant par RRP6. Plus récemment, dans un manuscrit paru dans PLOS Pathogens [7], nous avons caractérisé les
protéines associées à RRP6 et à son principal partenaire, MTR4 (exosome RNA
helicase). Nous avons pu montrer qu’il existe majoritairement deux
complexes de l’exosome nucléaire bien distincts, contenant chacun RRP6 et MTR4, et
caractérisés par la présence respective des protéines à doigts de zinc ZCCHC8
(zinc finger CCHC domain-containing protein 8) et ZFC3H1
(zinc finger C3H1-type containing). Des expériences
d’immunoprécipitation nous ont permis de montrer que ces protéines interagissaient
physiquement avec l’ARN polymérase 2. Par immunoprécipitation de chromatine (ChIP
[chromatin immunoprecipitation]) et ARN-ChIP, nous avons
également montré que ZFC3H1 et ZCCHC8 sont associés directement avec la chromatine
du promoteur du VIH, avec un pic d’intensité près du point +1 de transcription,
suggérant une implication dans les étapes précoces de la transcription du virus.
Enfin, il est important de souligner que seule la déplétion concomitante des deux
protéines a permis d’activer la transcription du promoteur VIH-1 à un niveau
comparable à celui des cellules n’exprimant plus RRP6. En revanche, l’extinction
unique de ZCCHC8 ou de ZFC3H1 n’a pas eu d’effet majeur. Il existerait donc une
compensation entre les deux protéines pour assurer la répression transcriptionnelle
du VIH-1. Ces résultats montrent donc l’existence de deux complexes de l’exosome caractérisés
respectivement par ZCCHC8 et ZFC3H1 qui répriment, indépendamment l’un de l’autre,
la transcription du VIH-1. L’interaction physique avec l’ARN polymérase ainsi que le
recrutement au niveau du point +1 de transcription suggèrent fortement que ces
complexes interviennent dans les étapes précoces de la transcription du virus. |
Les deux complexes majeurs de l’exosome nucléaire coopèrent pour réprimer le VIH
latent Pour étudier la latence du virus, nous avons utilisé deux modèles cellulaires
contenant un génome viral presque complet : des cellules J-Lat dérivées de la lignée
lymphocytaire Jurkat (J-Lat) ; et des cellules primaires (des lymphocytes T
CD4+ isolés d’individus sains) que nous avons infectées avec des
formes modifiées du VIH-1. Les cellules J-lat sont obtenues par la transduction de cellules Jurkat avec des
particules virales contenant un génome quasi-complet (uniquement dépourvu de la
protéine d’enveloppe et de la protéine Nef [negative regulatory
factor]) et qui codent également la GFP (green fluorescent
protein). L’expression de la GFP dans ces cellules dépend de la
réactivation transcriptionnelle du virus, comme cela se produit dans les cellules
exprimant le réservoir viral. Nous avons montré que la déplétion de MTR4 seule, ou
de ZFC3H1 et ZCCHC8 en association, entraînait l’activation de l’expression de la
GFP dans un nombre important de cellules. Couplée à un traitement des cellules par
la trichostatine A (TSA), un inhibiteur d’histones déacétylases utilisé couramment
pour induire l’activation transcriptionnelle dans ce système, la co-déplétion de
ZFC3H1 et ZCCHC8 entraîne une activation transcriptionnelle plus importante. Le second modèle de latence que nous avons utilisé a été décrit récemment. Il est
fondé sur l’utilisation de lymphocytes T CD4+ primaires infectés par un
vecteur VIH-1 modifié qui exprime deux protéines fluorescentes rapportrices, la GFP
et la mCherry (protéine fluorescente rouge monomérique) [8]. La GFP est exprimée sous le
contrôle du promoteur du VIH-1 et remplace les gènes codant Nef et la protéine
d’enveloppe. Elle permet donc de suivre l’expression du génome viral. La protéine
mCherry est sous le contrôle d’un promoteur constitutif
indépendant, EF1-alpha (human elongation factor-1 alpha) ; elle
permet de sélectionner les cellules infectées. Les cellules infectées latentes
n’expriment donc que la protéine mCherry, alors que les cellules
infectées de façon productive expriment les deux protéines fluorescentes
mCherry et GFP. Il est ainsi possible de les différencier par
une analyse en cytométrie de flux. Les cellules n’exprimant que la protéine
mCherry (donc latentes) ont ainsi été triées puis incubées en
présence de vecteurs lentiviraux exprimant des shRNA (short hairpin
RNA) qui inhibent l’expression de MTR4 ou celles de ZFC3H1 et ZCCHC8.
L’inhibition de l’expression de ces facteurs s’est accompagnée de l’apparition de
cellules doublement positives (pour la GFP et la protéine mCherry)
traduisant une activation de la transcription du génome viral dans une partie de la
population latente. L’ensemble de nos travaux montre donc l’existence de deux complexes distincts de
l’exosome nucléaire qui répriment la transcription du VIH-1 (Figure 1, partie gauche).
L’inhibition de ces complexes contribue à ré-activer le virus latent qui est la
barrière principale à l’éradication du VIH-1 (Figure 1, partie droite). Ces travaux s’inscrivent dans
l’optique d’une meilleure compréhension des mécanismes de latence pour ouvrir la
voie à de nouvelles pistes thérapeutiques qui pourraient conduire à l’éradication du
VIH-1 chez les patients infectés.
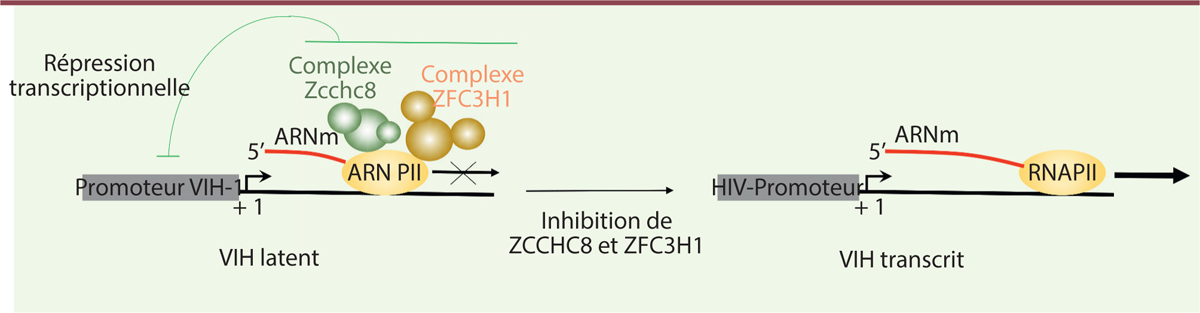
 | Figure 1. L’inhibition de ZCCHC8 et ZFC3H1 permet d’activer la
transcription du VIH-1 latent. Les deux
complexes de l’exosome nucléaire contenant respectivement ZFC3H1
(zinc finger CCHC domain-containing protein 8) et
ZCCHC8 (zinc finger C3H1-type containing) sont associés
avec l’ARN polymérase 2 (ARN PII) et répriment la transcription à partir
du promoteur du VIH-1 (virus de l’immunodéficience humaine de type 1).
L’inhibition de l’expression de ces facteurs induit une activation de la
transcription du virus latent. ARNm : ARN messagers. |
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les
données publiées dans cet article.
|
L’étude a été réalisée grâce aux financements suivants : ERC RNAMEDTGS, MSDAVENIR
HIDEINFLAME&SEQ, l’Agence Nationale de Recherches sur le Sida et les hépatites
virales (ANRS) et Sidaction pour RK.
|
1. Barré-Sinoussi
F.. L’infection
VIH/sida : l’histoire exemplaire d’une épidémie qui résiste .
Med Sci (Paris).
2018; ; 34 :
:499.–500. 2. Schwartz
C, Le
Douce
V,
Cherrier
T, et al.
Un virus tapi dans l’ombre : les bases moléculaires de la latence
du VIH-1. Partie I : la physiologie de la latence du VIH-1 .
Med Sci (Paris).
2010; ; 26 :
:159.–164. 3. Cherrier
T, Le
Douce
V,
Redel
L, et al.
Un virus tapi dans l’ombre : les bases moléculaires de la latence
du VIH-1. Partie II : la réactivation de la latence du VIH-1 et ses
implications thérapeutiques . Med Sci
(Paris).
2010; ; 26 :
:291.–296. 4. Mbonye
U,
Karn
J. The molecular
basis for human immunodeficiency virus latency . Annu
Rev Virol.
2017; ; 4 :
:261.–285. 5. Wagshcal
A,
Rousset
E,
Basavarajaiah
P, et al.
Microprocessor, Setx, Xrn2 and Rrp6 co-operate to induce
premature termination of transcription by RNAPII .
Cell.
2012; ; 150 :
:1147.–1157. 6. Lykke-Andersen
S,
Tomecki
R,
Jensenn
TH, et al.
The eukaryotic RNa exosome: same scaffold but variable catalytic
subunits . RNA Biol.
2011; ; 8 :
:61.–66. 7. Contreras
X,
Salifou
K,
Sanchez
G, et al.
Nuclear RNA surveillance complexes silence HIV-1
transcription . PLoS Pathog.
2018; ; 14 :
:e1006950.. 8. Chavez
L,
Calvanese
V,
Verdin
E. HIV latency is
established directly and early in both resting and activated primary CD4 T
cells . PLoS Pathog.
2015; ; 11 :
:e1004955.. |