| |
| Med Sci (Paris). 35(2): 187–190. doi: 10.1051/medsci/2019010.Le monde caché des clones mutants dans les tissus
normaux Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS ; CoReBio PACA,
case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09,
France MeSH keywords: Vieillissement, Clones cellulaires, Cellules épidermiques, Épithélium, Humains, Mutation, Cellules souches tumorales, États précancéreux, Tumeurs cutanées, Niche de cellules souches, Lumière du soleil, anatomopathologie, métabolisme, physiologie, génétique, étiologie, effets indésirables |
On s’accorde à penser que les cancers apparaissent lorsque, après avoir accumulé
plusieurs mutations somatiques successives, quelques cellules deviennent capables
d’échapper aux mécanismes de contrôle de la prolifération et, si elles ne sont pas
inhibées ou détruites par le système immunitaire, se multiplient pour donner naissance à
une tumeur [1]. Il devient dès lors essentiel d’analyser finement des tissus normaux afin de déterminer
s’ils comportent des clones de cellules portant déjà des mutations susceptibles de
faciliter leur évolution future vers un phénotype cancéreux. Cela pose évidemment des
problèmes techniques puisque l’on s’attend à ce que ces cellules mutantes soient très
minoritaires dans l’échantillon de tissu, et donc peu représentées dans l’ADN que l’on
en extrait. En dehors du cas plus favorable des cellules sanguines [2], les études les plus fouillées ont été réalisées
sur la peau [3] et, tout
récemment par la même équipe britannique, sur l’épithélium de l’œsophage [4]. L’étude de la peau avait mis en
évidence de nombreuses mutations (deux à six par mégabase d’ADN), touchant souvent des
gènes impliqués dans la cancérogenèse, bien qu’il s’agisse d’un tissu apparemment normal
et sans signe de cancer au niveau histologique. Mais on pouvait penser que le cas
examiné était particulier puisque cette peau avait été fortement exposée au soleil et
donc à la mutagenèse par les rayons ultra-violets (UV)1. Il était donc très intéressant de renouveler l’analyse sur un tissu non exposé, et c’est
ce que présente la publication parue fin novembre 2018 dans la revue
Science qui fait l’objet de cette chronique [4, 5]. |
Beaucoup de clones mutants, en relation avec l’âge La méthodologie de l’étude est proche de celle précédemment employée pour la peau
[3], avec quelques améliorations
(échantillons plus nombreux, de plus petite taille, séquençage plus approfondi,
etc.). Les auteurs ont travaillé sur des échantillons de tissu d’œsophage provenant
de neuf donneurs d’organe (décédés), ont isolé l’épithélium (qui ne présente aucune
anomalie à l’examen histologique) et ont prélevé pour analyse de petits fragments de
deux millimètres carrés, 844 en tout. Après extraction de l’ADN, 74 gènes liés au
cancer ont été séquencés à une redondance médiane élevée de 870, choisie pour
permettre la détection de séquences très minoritaires correspondant à de petits
clones de cellules portant une mutation donnée au sein de ce tissu. La limite de
détection dépend un peu du gène considéré (les taux d’erreurs de séquençage varient
selon la séquence) mais elle correspond à des fréquences (abondances relatives) se
situant entre 0,1 % et 1 %. Il s’avère que dans chacun des 844 échantillons, on
trouve de 5 à 25 mutations touchant ces 74 gènes, soit autant de clones de cellules
mutées représentant une fraction suffisante de la population cellulaire (dans ce
petit échantillon de 2 mm2) pour être détectables. Cela est illustré de
façon frappante dans la Figure 1, qui montre les clones mutés présents dans un
centimètre carré d’épithélium normal. Il s’agit d’une image synthétisée à partir des
données obtenues et qui tente de les représenter de manière intuitive : ce n’est pas
l’image réelle des clones trouvés dans 1 cm2 de peau, mais une manière
parlante et assez réaliste de présenter les résultats.
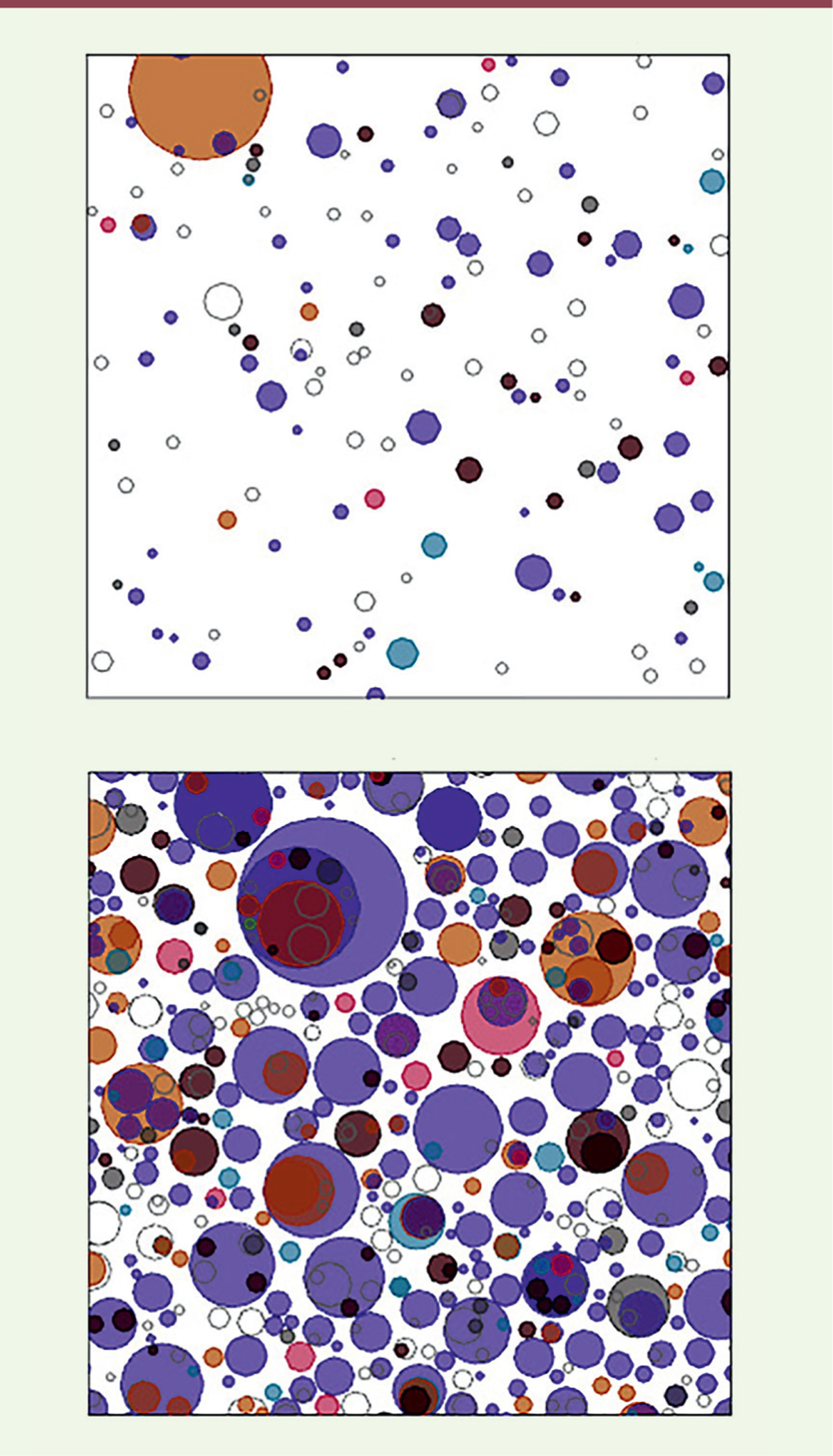
 | Figure 1.Nombre, taille et nature des clones de cellules mutantes présents dans 1
cm2 d’épithélium de l’oesophage. Cette figure contient beaucoup
d’informations et mérite d’être explicitée. Les clones détectés par
l’analyse de séquence sont figurés par des cercles dont la taille
correspond à la fréquence de la mutation trouvée (qui définit la
fraction de cellules portant cette mutation dans l’échantillon, donc
l’effectif du clone correspondant). Leur positionnement dans la figure
est aléatoire, sauf pour les sous-clones qui sont inclus dans les clones
auxquels ils sont apparentés (d’après les mutations qu’ils portent).
Enfin, la couleur de chaque cercle indique le gène muté auquel il
correspond – la couleur violette indique le gène NOTCH1
muté, la couleur bistre TP53. Le carré du haut se
rapporte à un échantillon provenant d’un homme jeune (20 à 23 ans) et
non-fumeur, celui du bas à un homme âgé de 44 à 47 ans (l’âge est
indiqué par un intervalle et non par une valeur précise pour mieux
préserver l’anonymat des donneurs) et gros fumeur. La différence est
flagrante et porte tant sur le nombre de clones de cellules mutées que
sur leur taille. On voit aussi que les mutations NOTCH1
(violet) et TP53 (bistre) sont dominantes (les autres
mutations sont repérées par des couleurs plus difficiles à distinguer)
(extrait partiel et remanié de la figure 3 de [ 4]). |
La comparaison des neuf échantillons de l’étude confirme une forte augmentation du
nombre de mutations avec l’âge mais aussi de nettes différences d’un individu à
l’autre ; l’effectif est insuffisant pour conclure sur un effet du tabac. En tous
cas, il est clair que ce tissu apparemment normal comporte un grand nombre de clones
cellulaires portant des mutations dans des gènes impliqués dans le cancer, clones
susceptibles de donner naissance à une tumeur macroscopique. Rappelons qu’ici on ne
répertorie que les mutations trouvées dans les 74 gènes étudiés : une extrapolation
à l’ensemble du génome, appuyée sur le séquençage intégral d’une vingtaine de clones
donne une fréquence globale de mutation de 0,2 à 0,8 par mégabase (voir plus loin).
Reste à examiner de plus près la nature de ces mutations. |
Des mutations driver, une sélection positive, pas de signature mutationnelle
nette Les cinq mutations les plus fréquentes dans ces cellules affectent, dans l’ordre, les
gènes NOTCH12,,
TP533,, NOTCH2,
FAT14, et NOTCH3.
Ces mutations étaient également dominantes dans la peau [3], elles ont en commun le fait d’inactiver des gènes impliqués
dans la différenciation vers les kératinocytes. Ces altérations orientent donc les
cellules qui les portent vers la voie alternative de prolifération, leur donnant
ainsi un avantage sélectif par rapport aux cellules non mutées au sein de
l’épithélium normal. Et l’analyse des mutations montre des traces très nettes de
sélection positive : seulement 2 % des mutations trouvées dans
NOTCH1 sont silencieuses, c’est-à-dire que presque 98 % des
mutations aboutissent à une protéine non fonctionnelle et donc abolissent la
fonction – favorisant la prolifération de la cellule et la formation d’un clone
mutant. De même, le gène TP53 présente une fréquence élevée de
mutations faux-sens ou tronquantes indiquant que ces altérations inactivantes sont
sélectionnées positivement. L’interprétation détaillée est un peu compliquée en ce
qui concerne NOTCH1 (qui s’avère plus souvent muté dans
l’épithélium normal que dans les tumeurs de l’œsophage), mais dans l’ensemble il est
clair que la plupart des clones ont été sélectionnés au sein de l’épithélium normal
en raison de la présence de mutations driver dans des gènes connus
pour être impliqués dans la cancérogenèse. Au-delà des 74 gènes examinés dans tous
les échantillons, les chercheurs ont choisi 21 spécimens dans lesquels un clone
était très largement dominant, au point de représenter plus de la moitié des
cellules. Ils ont alors effectué le séquençage intégral de l’ADN extrait, ce qui
permet d’accéder à l’examen exhaustif des mutations présentes. Le nombre total de
mutations est de l’ordre de 0,2 à 0,8 par mégabase, soit environ dix fois moins que
le chiffre trouvé pour la peau – mais cela correspond néanmoins à des centaines ou
milliers de mutations par cellule. On ne trouve guère de signature mutationnelle
nette (par exemple le jeu de mutations associées à l’usage du tabac n’est qu’à peine
perceptible) ; de même, les insertions, délétions et translocations souvent
présentes dans les cancers de l’œsophage ne sont guère visibles dans ces analyses.
La plupart de ces clones peuvent être considérés comme « pré-cancéreux », encore
proches de la normale, mais prêts à évoluer vers un phénotype tumoral moyennant
quelques mutations supplémentaires. |
Cet article, un peu difficile à suivre en raison de la masse de données et de leur
complexité, me semble revêtir une grande importance et a eu un large écho [5]. Il étend et généralise les résultats déjà
obtenus sur la peau [3], que l’on pouvait
considérer comme un cas particulier en raison de l’irradiation UV due au soleil. Il
montre que nous disposons aujourd’hui d’outils technologiques capables de détecter,
d’analyser et de quantifier des clones mutés ne représentant qu’un petit nombre de
cellules au sein d’un échantillon biologique, et que l’on peut étudier ces clones en
détail et les comparer aux échantillons provenant de tumeurs cancéreuses.
L’abondance de ces clones (en nombre de cellules, et en type d’altération) est
plutôt une mauvaise surprise : on ne s’attendait pas à trouver autant de cellules
mutantes dans des tissus normaux, mais c’est un fait et il vaut mieux en être
conscient si l’on cherche à développer des stratégies pour éviter les métastases,
qui sont responsables de l’essentiel de la mortalité due au cancer. En tous cas,
voici un nouveau domaine de recherche qui va certainement susciter de nombreux
travaux, et, espérons-le, des applications cliniques dans un futur assez proche. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1. Vogelstein
B,
Papadopoulos
N,
Velculescu
VE, et al.
Cancer genome landscapes .
Science.
2013; ; 339 :
:1546.–1558. 2. McKerrell
T,
Vassiliou
GS. Aging as a
driver of leukemogenesis . Sci Transl Med.
2015;; 7 : :306fs38.. 3. Martincorena
I,
Roshan
A,
Gerstung
M, et al.
Tumor evolution. High burden and pervasive positive selection of
somatic mutations in normal human skin .
Science.
2015; ; 348 :
:880.–886. 4. Martincorena
I,
Fowler
JC,
Wabik
A, et al.
Somatic mutant clones colonize the human oesophagus with
age . Science.
2018; ; 362 :
:911.–917. 5. Chanock
JJ. The paradox of
mutations and cancer . Science.
2018; ; 362 :
:893.–894. |