| |
| Med Sci (Paris). 36(1): 38–43. doi: 10.1051/medsci/2019267.L’ARN circulaire nous joue-t-il des tours
? Éric Lacazette,1 Leila Halidou Diallo,1 Florence Tatin,1 Barbara Garmy-Susini,1 and Anne-Catherine Prats1* 1UMR 1048-I2MC (Institut des maladies métaboliques et
cardiovasculaires), Inserm, Université de Toulouse,
UT3, Toulouse,
France |

Vignette (Photo © Inserm-Pascal Heitzer). Durant des décennies, la fonction de molécules régulatrices n’a été attribuée qu’aux
seules protéines. C’est seulement au début des années 1990 qu’est apparu un rôle pour
l’ARN comme régulateur des fonctions cellulaires avec la découverte de l’interférence
ARN et des ARN-guides. Deux familles de petits ARN non codants, les microARN (miARN) et
les piARN (Piwi-interacting RNA), ont ainsi été découvertes chez les
eucaryotes [33]
(→).
(→) Voir la Synthèse de L. Fressigné et M.J. Simard, m/s n° 2,
février 2018, page 137
L’importance des séquences non codantes s’est en fait imposée dans les années 2000 avec
le séquençage du génome humain qui a révélé l’existence de beaucoup moins de gènes
codants que ce à quoi on s’attendait : à peine plus de 20 000 ! Les séquences codantes
ne couvrent en effet que 1,5 % du génome humain, bien que 90 % de ces séquences d’ADN
soient transcrites en ARN. La majorité des transcrits sont donc des ARN non codants !
Les locus intergéniques sont en fait capables de produire de longs ARN non codants
(lncARN), d’une taille allant de 200 nucléotides (nt) à plus de 100 kilobases (kb).
Ceux-ci interviennent dans la régulation de l’expression des gènes par différents
mécanismes transcriptionnels et post-transcriptionnels [1]. Depuis bientôt 60 ans, nous savons que l’ARN joue un rôle absolument central dans la
transmission de l’information génétique [2]. L’actualité scientifique récente ne dément pas cette assertion puisque
l’ARN n’a pas encore dit son dernier mot ! On assiste en effet actuellement à la montée
en puissance d’une nouvelle famille d’ARN, les ARN circulaires ou circARN. Ces ARN ont
été identifiés il y a plus de 25 ans mais ils ont été considérés, alors, comme des
artefacts de la machinerie d’épissage, jusqu’à ce que des études récentes révèlent leur
implication fonctionnelle dans le contrôle de l’expression génique physiologique et
pathologique. C’est en 2012, à partir de données de transcriptomique à haut débit, que
Salzman et al. ont identifié 2 748 circARN potentiels chez l’homme,
ceux-ci résultant de l’assemblage de séquences d’exons non colinéaires [3]. Très rapidement, Memczak et
al., en 2013, ont identifié 2 000 circARN chez l’homme, 1 900 chez la
souris et 900 chez le nématode [4]. En éliminant les ARN linéaires, par l’action d’une exoribonuclease 3’, la
RNase R, incapable de dégrader ces ARN circulaires, Jeck et al. ont
réussi à enrichir les échantillons en circARN, ce qui a facilité leur identification.
Ils ont ainsi évalué à 14 % la proportion de gènes transcrits produisant des circARN
dans les fibroblastes humains [5]. En mars 2019, les progrès des technologies à grande échelle d’analyse des
ARN ont permis d’approfondir encore plus le paysage du transcriptome : Ji et
al. ont réalisé par séquençage d’ARN (RNASeq) des banques de transcriptomes
de l’homme, du macaque et de la souris, et ont dénombré respectivement chez ces trois
espèces 104 388, 96 675 et 82 321 circARN [6]. Cette analyse a également montré que les circARN produits à
partir de gènes orthologues entre les trois espèces, avaient un profil évolutif
spécifique, indiquant que cette nouvelle famille d’ARN possédait des fonctions
biologiques importantes pour les cellules. La production de ces ARN circulaires par les cellules eucaryotes apparaît ainsi
aujourd’hui comme un phénomène de grande ampleur, dont les conséquences sur la
régulation de l’expression génique et la physiopathologie ne peuvent plus être
négligées. |
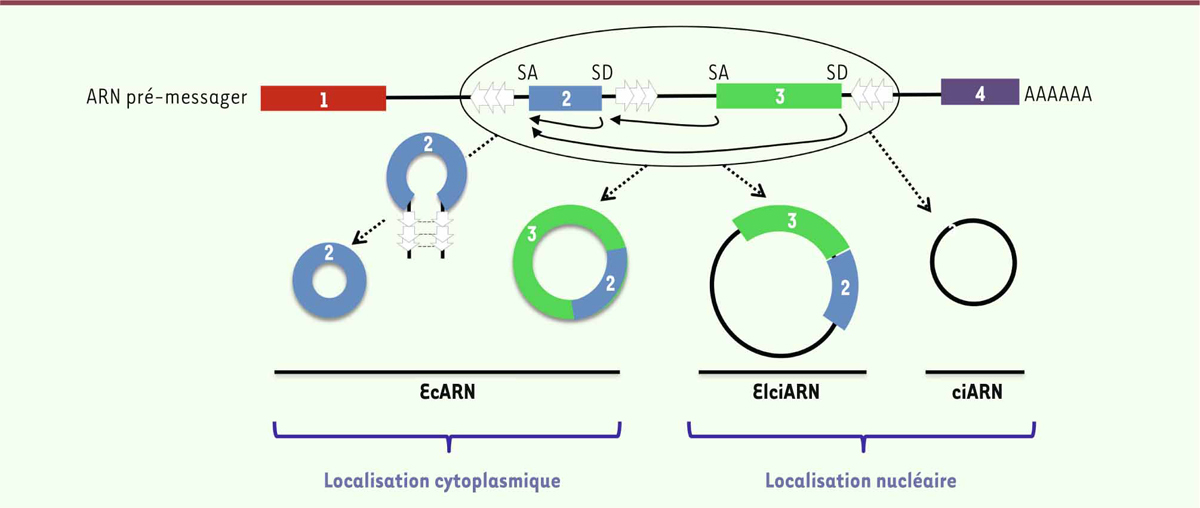
Quelle est l’origine des circARN ? Les circARN proviennent majoritairement de gènes codants. Ils sont produits par un
mécanisme d’épissage alternatif de l’ARN pré-messager, appelé rétro-épissage, dans
lequel le spliceosome lie un site d’épissage donneur à un site d’épissage accepteur
situé en amont (Figure 1)
[3]. Le rétro-épissage forme ainsi une
jonction spécifique pour chaque circARN, jonction qui est absente de l’ARNm linéaire
correspondant, et représente la « signature » de l’ARN circulaire. Le rétro-épissage
peut d’ailleurs entrer en compétition avec la maturation de l’ARNm linéaire. Ce
n’est pas un phénomène exceptionnel : les isoformes circulaires de centaines de
transcrits humains sont présentes dans les cellules à un niveau équivalent à celui
du transcrit linéaire correspondant [3]. Le
rétro-épissage est contrôlé par la présence de répétitions inverses complémentaires,
comme les séquences répétées Alu1, chez l’homme,
et de sites de liaisons de RBP (RNA-binding proteins) dans les
introns flanquant le site donneur et le site accepteur situé en amont (Figure 1). L’appariement des
séquences complémentaires, ou la formation de dimères de RBP, permet ainsi de
rapprocher les deux sites d’épissage et d’induire la formation de l’ARN circulaire.
Chez les primates, les séquences Alu constituent les séquences répétées les plus
abondantes et ont un rôle majeur dans la formation des ARN circulaires [5]. En ce qui concerne les RBP, on peut citer de
manière non exhaustive NF90/NF110, activateur du rétro-épissage qui stabilise la
structure appariée, et l’adénosine désaminase ADAR1 qui a un rôle inhibiteur en
déstabilisant l’appariement [7, 8]. Si aucune
donnée n’est encore disponible sur la régulation de cet épissage, on peut postuler
que l’édition de l’ARN joue un rôle important dans ce processus.
 | Figure 1. Formation des ARN circulaires par le mécanisme de rétro-épissage. Un ARN
pré-messager est schématisé. Les boîtes colorées représentent les exons
numérotés de 1 à 4, tandis que le trait noir représente les séquences
introniques. Les flèches au niveau des introns représentent des
séquences répétées de type Alu. Les ARN circulaires se forment par un
épissage entre un site donneur d’épissage situé en aval du site
accepteur d’épissage utilisé (alors que c’est l’inverse pour les ARN
linéaires). La présence de séquences répétées favorise ce processus par
hybridation de séquences complémentaires. La circularisation peut aussi
être générée par liaison puis dimérisation de RBP. Les EcARN sont
constitués de séquences exoniques et ont une localisation cytoplasmique,
tandis que les EIciARN et le ciARN sont respectivement constitués de
séquences exoniques et introniques pour les premiers et introniques pour
les seconds. Ces deux dernières catégories ont une localisation
nucléaire. SA : site accepteur d’épissage ; SD : site donneur d’épissage
; EcARN : exonic circRNA ; EIciARN :
exon-intron circRNA ; ciARN : circular
intronic RNA. |
|
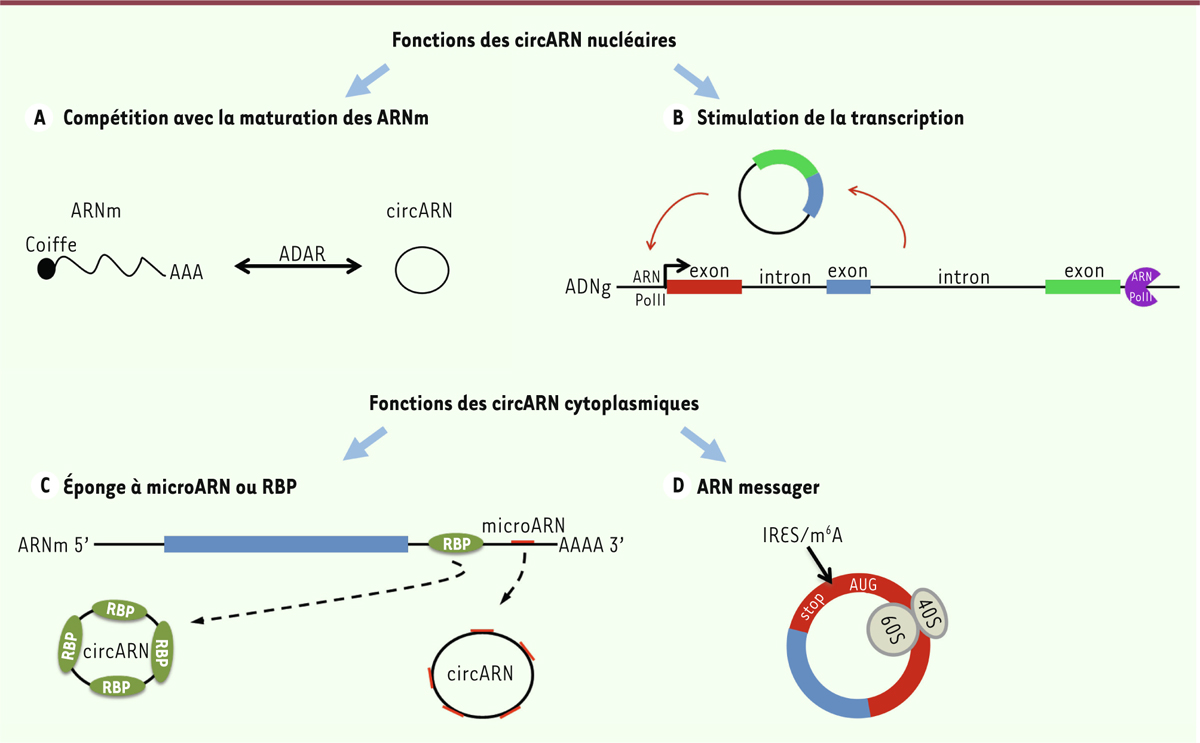
Les circARN, régulateurs de l’expression génique, sont-ils vraiment non codants
? Les circARN se subdivisent en trois catégories selon l’origine des séquences qu’ils
contiennent : les ciARN (circular intronic RNA) sont constitués de
séquences qui sont exclusivement d’origine intronique ; les EIciARN
(exon-intron circRNAs) possèdent, eux, des séquences d’origine
intronique et exonique (Figure
1). Ces deux catégories de circARN se localisent au niveau
nucléaire de la cellule. La troisième catégorie, les EcARN (exonic
circRNA), est prédominante. Ces circARN sont formés de séquences
exclusivement exoniques. Ils sont majoritairement localisés dans le cytoplasme. Ces
différences de localisation ont un impact sur la fonction de ces circARN. Les
circARN nucléaires modulent l’expression des gènes par au moins deux mécanismes
(Figure 2). Ils
peuvent, d’une part, entrer en compétition avec l’épissage de leur isoforme linéaire
(Figure 2A), et,
d’autre part, et c’est le cas des EIciARN, réguler la transcription de l’ARNm issu
de leur gène parental. Ce processus passe par une interaction du circARN avec l’ARN
polymérase II au niveau du promoteur du gène, ainsi qu’avec les snRNP (small
nuclear ribonucleoprotein) et hnRNP (heterogeneous nuclear
ribonucleoprotein) qui sont impliquées dans l’épissage de l’ARN
(Figure 2B) [9].
 | Figure 2. Fonctions des circARN. Les fonctions décrites pour les circARN nucléaires
(a-b) sont leurs capacités à interférer avec l’épissage de leur isoforme
linéaire (ce mécanisme pourrait en partie être contrôlé par la protéine
ADAR) et leur capacité à stimuler la transcription du gène dont ils sont
issus. Les fonctions décrites pour les circARN cytoplasmiques (c-d) sont
un rôle d’éponge à microARN ou RBP, ou une fonction d’ARN messager
lorsqu’ils possèdent un élément permettant l’initiation interne de la
traduction (IRES ou m6A) et un cadre de lecture autorisant la
synthèse d’un peptide ou d’une protéine. RBP : RNA-binding
protein ; IRES : internal ribosome entry
site ; m6A : N6-methyladenosine. |
Alors qu’au niveau nucléaire les circARN modulent essentiellement l’expression du
gène parental en cis, dans le cytoplasme, ils participent à des
mécanismes d’une toute autre ampleur. En effet, les EcARN (ARN circulaires
exoniques) peuvent jouer un rôle de compétiteur : ils séquestrent des miARN ou des
RBP qui se lient à des séquences qu’ils ont en commun avec l’ARN linéaire
correspondant (Figure 2C). Par
exemple, le circARN ciRS-7, qui cible plus de 70 sites présents sur le miARN miR-7,
possède une fonction d’éponge pour ce miARN (en le séquestrant) et
sa surexpression a un impact négatif sur le développement du système nerveux du
poisson zèbre [10]. L’ARN
circulaire circ-Foxo3 forme, lui, un complexe ternaire avec la cycline kinase CDK2
(cyclin-dependent kinase 2) et son co-facteur p21, deux
protéines impliquées dans la division cellulaire, ce qui entraîne une inhibition de
la progression dans le cycle cellulaire [11]. La stabilité importante des circARN leur procure un rôle
d’éponge beaucoup plus puissant que celui des ARN non codants linéaires, même s’ils
sont en quantités plus faibles. La grande stabilité des ARN circulaires a un impact sur leur quatrième fonction, en
passe de devenir leur fonction principale : un rôle codant (Figure 2D). Les EcARN comportent en
effet dans leur séquence, dans la plupart des cas, le codon initiateur de leur
isoforme linéaire, de même que des cadres de lectures ouverts. Leur association avec
les polysomes a ainsi fortement suggéré leur capacité d’être traduits, remettant en
cause leur qualification d’ARN non codants [12, 13]. |
Les circARN, une nouvelle classe d’ARN messagers Chez les eucaryotes, le mécanisme classique d’initiation de la traduction implique le
recrutement de la petite sous-unité du ribosome à l’extrémité 5’ coiffée de l’ARNm.
Kozak, en 1979, avait ainsi montré qu’un ARN circulaire ne pouvait être traduit,
faute de cette extrémité [14, 15]. Cette
règle du recrutement du ribosome à l’extrémité 5’ de l’ARNm s’est cependant avérée,
en 1988, ne pas être absolue. Il existe en effet chez de nombreux virus, mais aussi
dans des ARNm cellulaires, des sites d’entrée interne des ribosomes
(internal ribosome entry site, IRES) qui permettent une
initiation de la traduction au sein même de la séquence de l’ARN [16, 17]. En 1995, la traduction d’un ARN circulaire
synthétique a ainsi été effectivement démontrée [18] et plus récemment, il a été rapporté que la
présence d’un IRES dans des ARN circulaires, produits par rétro-épissage, permet
leur traduction [19]. La
preuve absolue de la traduction de circARN endogènes en protéines n’a cependant été
obtenue qu’en 2017, avec la détection par spectrométrie de masse, d’un peptide
correspondant à la région couvrant la jonction de rétro-épissage spécifique du
circMbl3, issu du locus muscleblind chez la drosophile, mettant
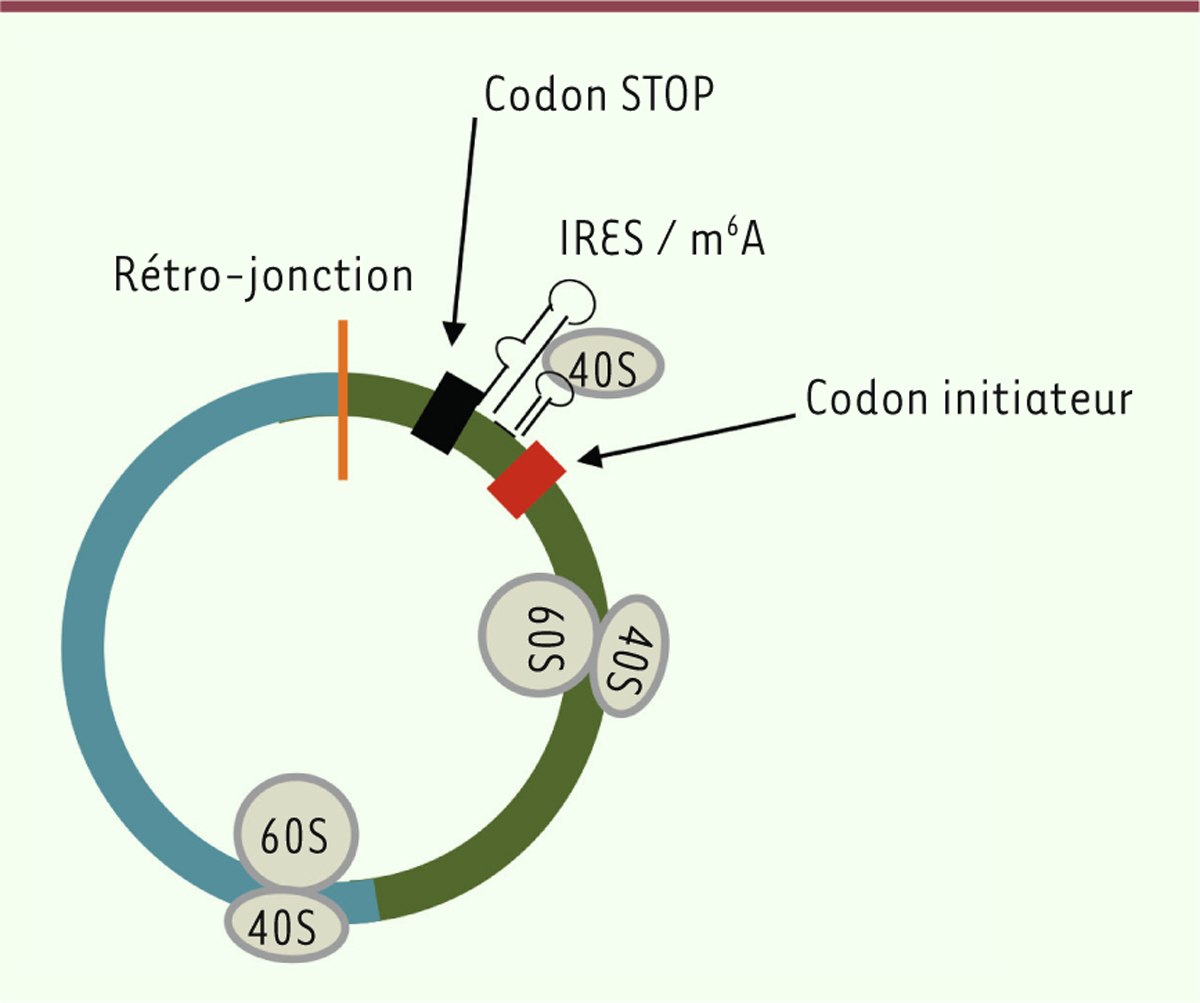
ainsi en évidence la synthèse d’une protéine à partir d’un circARN [12]. Pour ce circARN, le codon initiateur de la
traduction est identique à celui de l’ARNm linéaire muscleblind, et
le codon stop se situe quelques nucléotides en aval de la zone de rétro-jonction
(Figure 3) ; ces deux
caractéristiques semblent être fréquemment retrouvées pour les circARN qui sont
traduits. D’autres auteurs ont montré la traduction de l’ARN circulaire circZNF609,
exprimé durant la myogenèse et dont l’isoforme linéaire code un facteur de
transcription à doigt de zinc [20]. Comme pour circMbl3, le cadre de lecture de la protéine traduite
commence au même codon initiateur de la traduction que celui de l’ARNm linéaire et
le codon stop se trouve là aussi quelques nucléotides en aval de la jonction
d’épissage formant le circARN (Figure
3). Dans les deux cas, la traduction nécessite un IRES. Pour
circZNF609, l’activité de l’IRES dépend de l’épissage de l’ARN et est activée par un
choc thermique. Ce dernier point est particulièrement intéressant car il suggère que
les circARN, ou au moins certains d’entre eux, pourraient être traduits en
conditions de stress. Alors que les circARN identifiés présentent généralement des
IRES en 5’ du codon initiateur, l’ARN circulaire circSHPRH, produit par le gène
codant l’hélicase SHPRH (SNF2 histone-linker PHD RING helicase),
possède un IRES qui est situé en 3’ du codon initiateur et qui permet l’expression
d’une protéine de 146 kDa [21, 22]. Cette
particularité du circSHPRH provient de ce que la totalité de sa séquence est
codante. En effet, dans cet ARN circulaire, le codon initiateur de la traduction
chevauche deux codons stop répétés en tandem : UGAUGA.
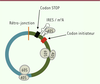 | Figure 3. Structure caractéristique d’un circARN traduit. Dans cet exemple, l’EcARN
est formé par l’assemblage de deux exons (vert, bleu). Le cadre de
lecture commence au codon initiateur [boîte rouge] en direction du codon
stop [boîte noire], en passant par la jonction entre les deux exons
(vert > bleu), puis la rétro-jonction qui a circularisé la molécule
(trait orange). Le codon stop se situe quelques nucléotides en aval de
la rétro-jonction. Les séquences permettant l’initiation de la
traduction (IRES/m6A) se trouvent généralement entre le codon
stop et le codon initiateur. Cette configuration est celle décrite pour
les circZNF609 et circMbl. La liaison interne de la sous-unité 40S du
ribosome à l’EcARN, puis la traduction par le ribosome 80S après
association des deux sous-unités 40S et 60S, sont schématisées. EcARN :
exonic circRNA ; IRES : internal ribosome
entry site ; m6A : N6-methyladenosine ;
Mbl : muscleblind. |
Si le mécanisme reposant sur des IRES paraît être le plus fréquemment impliqué pour
la traduction des circARN, un autre mécanisme permettant l’initiation de la
traduction indépendante de la coiffe a été découvert : il requiert la présence d’une
N6-méthyladénosine dans la région 5’ non traduite de l’ARNm
(m6A) [23].
Or, les ARN circulaires sont enrichis en motifs m6A et la spectrométrie
de masse a permis de démontrer que nombre d’entre eux sont traduits grâce à la
présence de courtes séquences contenant des m6A qui jouent le rôle d’IRES
[24]. |
La synthèse de protéines à partir des circARN, un enjeu biotechnologique Résistants à toutes les exoribonucléases, les ARNm circulaires présentent l’avantage
d’être beaucoup plus stables que leurs isoformes linéaires. Ils représentent donc un
outil de choix pour la production de protéines recombinantes. Une telle approche a
été suggérée par Wang et al. qui ont développé un minigène
rapporteur générant, par rétro-épissage, un circARN codant la GFP (green
fluorescent protein), circGFP [19]. De même, un vecteur viral adéno-associé (rAAV) exprimant un circGFP
contenant un IRES a montré son efficacité après injection chez la souris. Cette
expérience a également révélé une forte spécificité tissulaire de l’expression du
transgène, probablement liée à la paire d’introns et/ou à l’IRES utilisés [25]. Ces caractéristiques
pourraient être mises à profit afin de cibler l’expression d’un transgène dans un
tissu particulier. Wesselhoeft et al., en 2019, ont développé un système de production
de circARN par auto-épissage in vitro [26]. Ces circARN contenant un IRES en amont du
gène d’intérêt (gène codant la luciférase ou l’érythropoïétine humaine), ont été
utilisés pour transfecter des cellules, ainsi que pour des injections in
vivo chez la souris dans des nanoparticules lipidiques. Les auteurs se
sont en particulier intéressés aux mécanismes de reconnaissance des ARN exogènes par
les récepteurs de l’immunité innée, RIG-1 (retinoic-acid-inducible protein
1) ou TLR (Toll-like receptors), des processus
antiviraux développés par les cellules de mammifères à l’origine de réponses
immunitaires induites par les ARN linéaires. Contrairement aux isoformes linéaires
reconnues par ces récepteurs, les circARN échappent à ces senseurs. Leur traduction
en protéine n’est cependant effective que s’ils présentent un IRES, mais pas en
présence d’une méthyladénosine m6A (l’autre mécanisme permettant la
traduction indépendante de la coiffe) [26].
Ainsi, les ARN circulaires sont non seulement traduits car plus stables, mais ils
sont également moins immunogènes que les ARN linéaires. Ils représentent donc un
outil biotechnologique de choix et extrêmement prometteur pour l’expression de
protéines recombinantes in vitro et in vivo,
alliant production et absence de réponse de l’hôte. |
Les circARN, des acteurs impliqués dans différentes pathologies Le grand nombre d’ARN circulaires présents dans les cellules, ainsi que leurs
fonctions dans la régulation de l’expression génique, comme ARN non codants, ou dans
la production de protéines comme ARN messagers traduits, en font des acteurs majeurs
lors du développement de différentes pathologies. De nombreux circARN régulent la prolifération, la migration ou l’invasion cellulaire,
et agissent positivement ou négativement sur la progression des tumeurs, soit par le
mécanisme d’éponge à miARN ou RBP, soit lorsqu’ils sont traduits [27]. Le circARN 100269, par exemple, se
comporte comme un suppresseur de tumeur dans le cancer gastrique en séquestrant par
un mécanisme d’éponge à ARN, le microARN miR-630 [28]. L’ARN circ-FBXW7, quant à lui, présente la
capacité de supprimer les tumeurs en inhibant la prolifération des cellules
cancéreuses dans le glioblastome, en ayant, cette fois, une fonction d’ARNm qui sera
traduit grâce à la présence d’un IRES [29]. La protéine produite, FBXW7-185aa, présente une activité
antiproliférative en déstabilisant la protéine c-Myc. L’expression de circ-FBXW7 est
ainsi corrélée positivement à la survie des patients atteints de glioblastome. L’impact des circARN s’étend à des pathologies autres que le cancer. Parmi les
circARN identifiés à ce jour, plusieurs ont un rôle dans la myogenèse ou au niveau
du système nerveux [12, 20]. Le circ-ZNF609, mentionné plus haut, qui est traduit par
un mécanisme IRES-dépendant, est un activateur de la prolifération des myoblastes
régulé négativement lors de la myogenèse [20]. Il est retrouvé à des niveaux élevés dans des myoblastes portant la
mutation du gène codant la dystrophine, responsable de la myopathie de Duchenne,
suggérant son implication dans cette pathologie. Les circARN sont également
impliqués dans les pathologies neurodégénératives. Notamment, l’analyse à grande
échelle de l’expression des ARN circulaires dans un modèle murin de vieillissement
accéléré mimant la maladie d’Alzheimer a démontré la dérégulation de 235 circARN
[30]. Ils pourraient
avoir un rôle crucial dans la régulation des gènes responsables de la maladie
d’Alzheimer, par leur fonction d’éponge à miARN pour certains, ou de protéines, en
étant traduits pour d’autres. Les circARN apparaissent de plus en plus comme des
acteurs-clés du processus de vieillissement, non seulement du système nerveux, mais
de tissus comme le muscle et la peau [31]. Il a même été proposé que l’accumulation des circARN soit
un marqueur du vieillissement, si tant est que l’on considère ce processus comme une
maladie [32]. Cette famille
d’ARN ouvre ainsi des perspectives prometteuses en termes de cibles et de
biomarqueurs pour de nombreuses pathologies. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1.
Mattick
JS. The State of
Long Non-Coding RNA Biology . Noncoding RNA.
2018;; 4. 2.
Brenner
S
Jacob
F
Meselson
M
An unstable intermediate carrying information from genes to
ribosomes for protein synthesis . Nature.
1961; ; 190 :
:576.–581. 3.
Salzman
J
Gawad
C
Wang
PL
et al.
Circular RNAs are the predominant transcript isoform from
hundreds of human genes in diverse cell types . PLoS
One.
2012; ; 7 : :e30733.. 4.
Memczak
S
Jens
M
Elefsinioti
A
et al.
Circular RNAs are a large class of animal RNAs with regulatory
potency . Nature.
2013; ; 495 :
:333.–338. 5.
Jeck
WR
Sorrentino
JA
Wang
K
et al.
Circular RNAs are abundant, conserved, and associated with ALU
repeats . RNA.
2013; ; 19 :
:141.–157. 6.
Ji
P
Wu
W
Chen
S
et al.
Expanded Expression Landscape and Prioritization of Circular RNAs
in Mammals . Cell Rep.
2019; ; 26 : (3444–60)
:e5.. 7.
Li
LJ
Leng
RX
Fan
YG
et al.
Translation of noncoding RNAs: focus on lncRNAs, pri-miRNAs, and
circRNAs . Exp Cell Res.
2017; ; 361 :
:1.–8. 8.
Rybak-Wolf
A
Stottmeister
C
Glazar
P
et al.
Circular RNAs in the mammalian brain are highly abundant,
conserved, and dynamically expressed . Mol
Cell.
2015; ; 58 :
:870.–885. 9.
Li
Z
Huang
C
Bao
C
et al.
Exon-intron circular RNAs regulate transcription in the
nucleus . Nat Struct Mol Biol.
2015; ; 22 :
:256.–264. 10.
Hansen
TB
Jensen
TI
Clausen
BH
et al.
Natural RNA circles function as efficient microRNA
sponges . Nature.
2013; ; 495 :
:384.–388. 11.
Du
WW
Yang
W
Liu
E
et al.
Foxo3 circular RNA retards cell cycle progression via forming
ternary complexes with p21 and CDK2 . Nucleic Acids
Res.
2016; ; 44 :
:2846.–2858. 12.
Pamudurti
NR
Bartok
O
Jens
M
et al.
Translation of CircRNAs . Mol
Cell.
2017; ; 66 : (9–21)
:e7.. 13.
Ragan
C
Goodall
GJ
Shirokikh
NE
Preiss
T
Insights into the biogenesis and potential functions of exonic
circular RNA . Sci Rep.
2019; ; 9 : :2048.. 14.
Holcik
M
Sonenberg
N
Translational control in stress and apoptosis .
Nat Rev Mol Cell Biol.
2005; ; 6 :
:318.–327. 15.
Kozak
M.
Inability of circular mRNA to attach to eukaryotic
ribosomes . Nature.
1979; ; 280 :
:82.–85. 16.
Godet
AC
David
F
Hantelys
F
et al.
IRES trans-acting factors, key actors of the stress
response . Int J Mol Sci.
2019; ; 20 : 17.
Pelletier
J
Sonenberg
N
Internal initiation of translation of eukaryotic mRNA directed by
a sequence derived from poliovirus RNA .
Nature.
1988; ; 334 :
:320.–325. 18.
Chen
CY
Sarnow
P
Initiation of protein synthesis by the eukaryotic translational
apparatus on circular RNAs . Science.
1995; ; 268 :
:415.–417. 19.
Wang
Y
Wang
Z
Efficient backsplicing produces translatable circular
mRNAs . RNA.
2015; ; 21 :
:172.–179. 20.
Legnini
I, Di
Timoteo
G,
Rossi
F, et al.
Circ-ZNF609 is a circular RNA that can be translated and
functions in myogenesis . Mol Cell.
2017;; 66 :
:22.–37e9. 21.
Diallo
LH
Tatin
F
David
F
et al.
How are circRNAs translated by non-canonical initiation
mechanisms? . Biochimie.
2019; ; 164 :
:45.–52. 22.
Zhang
M
Huang
N
Yang
X
et al.
A novel protein encoded by the circular form of the SHPRH gene
suppresses glioma tumorigenesis . Oncogene.
2018; ; 37 :
:1805.–1814. 23.
Meyer
KD
Patil
DP
Zhou
J
et al.
5’ UTR m[ 6]A promotes
cap-independent translation . Cell.
2015; ; 163 :
:999.–1010. 24.
Yang
Y
Fan
X
Mao
M
et al.
Extensive translation of circular RNAs driven by N[ 6]-methyladenosine .
Cell Res.
2017; ; 27 :
:626.–641. 25.
Meganck
RM
Borchardt
EK
Castellanos Rivera
RM
et al.
Tissue-dependent expression and translation of circular RNAs with
recombinant AAV vectors in vivo . Mol Ther Nucleic
Acids.
2018; ; 13 :
:89.–98. 26.
Wesselhoeft
RA,
Kowalski
PS,
Parker-Hale
FC, et al.
RNA Circularization diminishes immunogenicity and can extend
translation duration in vivo . Mol Cell.
2019;; 74 :
:508.–20e4. 27.
Wu
J
Qi
X
Liu
L
et al.
Emerging epigenetic regulation of circular RNAs in human
cancer . Mol Ther Nucleic Acids.
2019; ; 16 :
:589.–596. 28.
Zhang
Y
Liu
H
Li
W
et al.
CircRNA_100269 is downregulated in gastric cancer and suppresses
tumor cell growth by targeting miR-630 . Aging
(Albany NY).
2017; ; 9 :
:1585.–1594. 29.
Yang
Y
Gao
X
Zhang
M
et al.
Novel role of FBXW7 circular RNA in repressing glioma
tumorigenesis . J Natl Cancer Inst.
2018; ; 110 : 30.
Zhang
S
Zhu
D
Li
H
et al.
Characterization of circRNA-associated-ceRNA networks in a
senescence-accelerated mouse prone 8 brain . Mol
Ther.
2017; ; 25 :
:2053.–2061. 31.
Cai
H
Li
Y
Niringiyumukiza
JD
et al.
Circular RNA involvement in aging: an emerging player with great
potential . Mech Ageing Dev.
2019; ; 178 :
:16.–24. 32.
Knupp
D
Miura
P
CircRNA accumulation: a new hallmark of aging? .
Mech Ageing Dev.
2018; ; 173 :
:71.–79. 33.
Lucile Fressigné
L,
Simard
MJ. La biogenèse des
ARN courts non codants chez les animaux . Med Sci
(Paris).
2018;; 34 :
:137.–44. |