| |
| Med Sci (Paris). 36(3): 206–209. doi: 10.1051/medsci/2020032.La réponse interféron Un grand pouvoir implique de grandes
responsabilités Ghizlane Maarifi,1 Nikaïa Smith,2 and Sébastien Nisole1* 1Institut de recherche en infectiologie de Montpellier
(IRIM), CNRS UMR9004, université de Montpellier,
1919 route de Mende,
34090Montpellier,
France 2Immunobiologie des cellules dendritiques, Inserm U1223,
Institut Pasteur, 25 rue
du Docteur Roux, 75015Paris,
France MeSH keywords: Immunité acquise, Maladies auto-immunes, Maladie chronique, Évolution de la maladie, Humains, Interféron de type I, NIMA-interacting peptidylprolyl isomerase, Transduction du signal, Protéines à motif tripartite, Maladies virales, effets des médicaments et substances chimiques, génétique, étiologie, immunologie, pharmacologie, physiologie, anatomopathologie |
Lorsque des cellules sont infectées par un virus, elles produisent des interférons
(IFN), de puissantes molécules antivirales qui protègent les cellules avoisinantes
de l’infection et permettent ainsi de limiter la propagation du virus dans
l’organisme. Il existe trois types d’IFN : I (IFN-a et IFN-b principalement), II
(IFN-g), et III (IFN-l). Les IFN de type I sont les principaux IFN produits au cours
d’une infection virale. Ils induisent, dans les cellules infectées et les cellules
avoisinantes, l’expression de centaines de gènes (interferon-stimulated
genes, ISG), ce qui va permettre l’établissement d’un état antiviral
[1]. La réponse IFN de type I débute par la reconnaissance des génomes viraux par des
détecteurs cellulaires connus sous le nom de pattern-recognition
receptors (PRR), parmi lesquels les toll-like
receptors (TLR), localisés à la surface des cellules ou dans les
endosomes, et les retinoic acid-inducible gene-I-like
(RIG-I-like) receptors (RLR), localisés dans le cytoplasme.
Cette reconnaissance déclenche des cascades de signalisation propres à chaque PRR,
mais qui aboutissent toutes à la phosphorylation du facteur de transcription IRF3
(interferon regulatory factor 3). Après sa translocation dans
le noyau cellulaire, IRF3 phosphorylé déclenche la transcription du gène codant
l’IFN-b [2] (Figure 1). La synthèse d’IFN-a
nécessite, quant à elle, l’expression du facteur de transcription IRF7, qui est
induite par l’IFN-b via une boucle autocrine ou paracrine.
Contrairement à l’IFN-b, codé par un gène unique, les différents sous-types d’IFN-a
induits par IRF7 sont codés par 13 gènes. Cette production séquentielle d’IFN
(IRF3/IFN-b, puis IRF7/IFN-a) permet une amplification de la réponse IFN [2] (Figure
1).
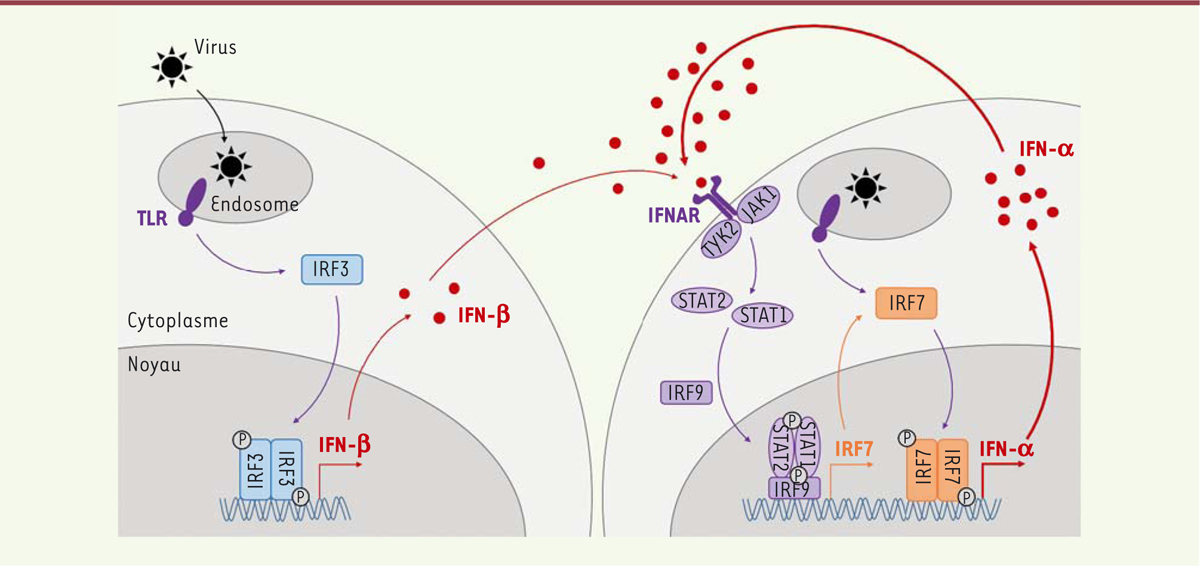
 | Figure 1. Les deux phases de la réponse IFN de type I. Suite à la détection d’un
virus par un PRR (ici, un TLR endosomal), une cascade de signalisation
aboutit à la phosphorylation d’IRF3, qui devient IRF3-P, ce qui
déclenche sa dimérisation et sa translocation dans le noyau, où il va
activer la transcription du gène codant l’IFN-b. Cette première phase
aboutit donc à la production et à la sécrétion d’IFN-b par la cellule
infectée. L’IFN-b va alors agir de façon autocrine ou paracrine en se
fixant à son récepteur (IFNAR), déclenchant ainsi la voie Jak/Stat.
L’activation de cette voie, qui implique les protéines STAT1, STAT2 et
IRF9, aboutit à la synthèse de centaines de protéines codées par les
interferon-stimulated genes (ISG), parmi lesquelles
IRF7. IRF7 va à son tour être phosphorylé en IRF7-P suite à l’activation
d’un PRR, se dimériser et, après translocation dans le noyau, induire
l’expression des gènes codant les IFN-a. Cette deuxième phase, qui
aboutit à la sécrétion d’IFN-a, permet l’amplification de la réponse
IFN. |
Tandis que la plupart des cellules du corps peuvent détecter les virus qui les
infectent et produire de faibles quantités d’IFN, les cellules dendritiques
plasmacytoïdes (plasmacytoid dendritic cells, pDC) sont
spécialisées dans cette fonction. En effet, contrairement aux autres cellules, les
pDC expriment IRF7 de façon constitutive, ce qui leur permet de sécréter rapidement
de grandes quantités d’IFN, libérées dans le sang et les tissus [3]. Cette capacité unique confère à ces
cellules sentinelles un rôle crucial dans la défense contre les virus, mais elle
impose également un système de contrôle rigoureux pour éviter à l’organisme les
dommages causés par une réponse IFN trop forte ou trop prolongée. |
Contrôle de la réponse IFN par les protéines TRIM Tandis que les voies de signalisation conduisant à la synthèse d’IFN reposent
essentiellement sur des cascades de phosphorylation, la régulation de ces voies fait
intervenir l’ubiquitination des protéines. Cette modification post-traductionnelle
consiste en l’association d’ubiquitine, une petite protéine de 76 acides aminés, à
des protéines-cibles, ce qui peut modifier leur stabilité, leur localisation
subcellulaire, ou leur activité [4]. La réaction d’ubiquitination fait intervenir trois enzymes (une
enzyme activatrice E1, une enzyme de conjugaison E2, et une ubiquitine-ligase E3)
qui interviennent séquentiellement pour lier de manière covalente l’ubiquitine à un
résidu lysine de la protéine-cible. L’ubiquitine possède elle-même sept résidus
lysine, et les enzymes d’ubiquitination des protéines peuvent donc coordonner la
formation de chaînes de poly-ubiquitines liées de façon covalente [4]. Parmi les nombreuses ubiquitine-ligases E3 présentes dans les cellules, les protéines
TRIM (tripartite motif) se sont récemment révélées particulièrement
importantes pour la régulation des voies de signalisation conduisant à la synthèse
d’IFN. Cette famille de protéines, très conservée au cours de l’évolution, comporte
75 membres chez l’homme. Ces protéines partagent une organisation tripartite, à
l’origine de leur nom, constituée d’un domaine RING (really interesting new
gene), d’un ou deux domaines B-box, et d’un domaine
coiled-coil. La plupart des protéines TRIM possèdent en plus un
ou plusieurs domaines C-terminaux de différents types. C’est le domaine RING qui
confère à ces protéines leur activité ubiquitine-ligase. Les protéines TRIM ont été
l’objet d’un regain d’intérêt à partir de 2004, année de la découverte de l’activité
antirétrovirale de TRIM5a, notamment contre le virus de l’immunodéficience humaine
(VIH)[5], même si
l’activité antivirale de TRIM22 et TRIM19, découverte auparavant, laissait déjà
présager qu’il s’agissait d’une vaste famille de facteurs antiviraux [6]. Les travaux ultérieurs ont
alors montré que l’implication des protéines TRIM dans la défense contre les
infections virales était en réalité plus large, puisque ces protéines peuvent
également réguler la réponse IFN. La première protéine TRIM immunomodulatrice
identifiée fut TRIM25. Cette protéine est en effet indispensable à l’activité
antivirale de RIG-I, un PRR cytoplasmique responsable de la détection des ARN
viraux, en induisant son ubiquitination [7]. Suite à cette découverte, de nombreuses autres protéines
TRIM ont été identifiées comme d’importants régulateurs des voies de signalisation
impliquées dans la défense antivirale innée, généralement via leur
capacité à ubiquitinyler leur cible [8]. Une étude a notamment montré qu’environ la moitié des 75 protéines
TRIM humaines étaient capables d’augmenter la réponse antivirale, notamment IFN,
lorsqu’elles étaient surexprimées dans une lignée cellulaire [9]. Partant de ce constat, nous avons entrepris d’étudier le rôle physiologique des
protéines TRIM dans les cellules humaines, notamment dans les pDC. Plutôt que de
surexprimer les protéines TRIM dans les pDC, nous avons pris le parti de diminuer
leur expression par la technique « d’interférence ARN » et d’évaluer les
conséquences de cette diminution sur la capacité de ces cellules à produire des IFN
en réponse à la détection d’un virus. L’étude a été faite sur des pDC purifiées à
partir de sang humain, mises en contact avec le VIH ou le virus de la grippe, deux
virus à ARN qui sont détectés par la molécule TLR7 présente de manière constitutive
dans les endosomes de ces cellules. Cette étude nous a permis d’identifier TRIM20,
22, 28, et 36 comme étant des inhibiteurs de la réponse IFN dans les pDC, tandis que
TRIM8 fut l’unique régulateur positif identifié [10]. TRIM8 s’est d’ailleurs avéré être un
acteur essentiel de l’activation des pDC, puisqu’en son absence, la production d’IFN
par ces cellules est quasiment inexistante, un résultat inattendu que nous avons
alors cherché à expliquer. |
Régulation d’IRF7 par TRIM8 et Pin1 Nous avons d’abord montré que TRIM8, dont la localisation est majoritairement
nucléaire dans les pDC, protégeait IRF7 de la dégradation après sa phosphorylation
et sa translocation dans le noyau. Etant donné que la grande majorité des protéines
TRIM contrôle la stabilité ou l’activité des protéines via leur
ubiquitination, nous avons suspecté l’implication de l’activité ubiquitine-ligase de
TRIM8. Cependant, le fait qu’une protéine TRIM8 dépourvue de son domaine RING
conserve sa capacité à stabiliser IRF7 phosphorylé (IRF7-P) a rapidement infirmé
cette hypothèse. Nous avons alors montré que TRIM8 protège IRF7-P de l’activité de
Pin1 (peptidyl-prolyl cis-trans isomerase, NIMA-interacting 1), une
enzyme qui induit sa dégradation [10]. Pin1
est une peptidyl-prolyl cis-trans isomérase (PPIase), c’est-à-dire une enzyme qui
catalyse l’isomérisation cis-trans de la liaison peptidique en amont d’un résidu
proline. Cette isomérisation modifie la conformation de la protéine-cible, altérant
ainsi sa stabilité, sa localisation sub-cellulaire, ou son activité. La
particularité de Pin1 par rapport aux autres PPIases est de reconnaitre
spécifiquement les résidus sérine ou thréonine phosphorylés qui sont suivis d’une
proline (motifs pSer/Pro et pThr/Pro). Puisque la plupart des voies de signalisation
mettent en jeu une cascade de phosphorylation de protéines sur des résidus sérine ou
thréonine, Pin1 joue le rôle d’interrupteur moléculaire de ces cascades et est ainsi
impliqué dans de très nombreuses fonctions cellulaires [11]. Parmi les très nombreux substrats de Pin1 identifiés figure le facteur de
transcription IRF3 [12].
Pin1 reconnait en effet IRF3 phosphorylé (IRF3-P) et catalyse son isomérisation. Le
changement de conformation ainsi induit conduit à l’ubiquitination d’IRF3 et à sa
dégradation par le protéasome. Pin1 est donc un inhibiteur de la réponse antivirale
dépendante d’IRF3 qui permet d’interrompre la synthèse d’IFN-b [12]. Deux protéines TRIM, TRIM21 et TRIM19,
augmentent la production d’IFN-b en empêchant la reconnaissance d’IRF3-P par Pin1
[13]. Tandis que TRIM21
empêche l’interaction entre IRF3-P et Pin1, TRIM19 agit en séquestrant Pin1 dans les
corps nucléaires PML (promyelocytic leukemia), l’empêchant ainsi
d’interagir avec sa cible [13, 14] (Figure 2A). Nous avons montré
qu’IRF7-P est également un substrat de Pin1, et que la synthèse d’IFN par les pDC
est donc fortement réprimée par cette enzyme [10]. Nos travaux lèvent le voile sur le système de contrôle de la
production d’IFN par les pDC, dont la régulation fine implique deux protéines
antagonistes : TRIM8, qui permet une production efficace d’IFN par ces cellules, et
Pin1, qui bloque cette production (Figure
2B). Cette découverte pourrait permettre d’élaborer de
nouvelles stratégies thérapeutiques contre des maladies liées à une surproduction
d’IFN, notamment les maladies virales chroniques et les maladies auto-immunes.
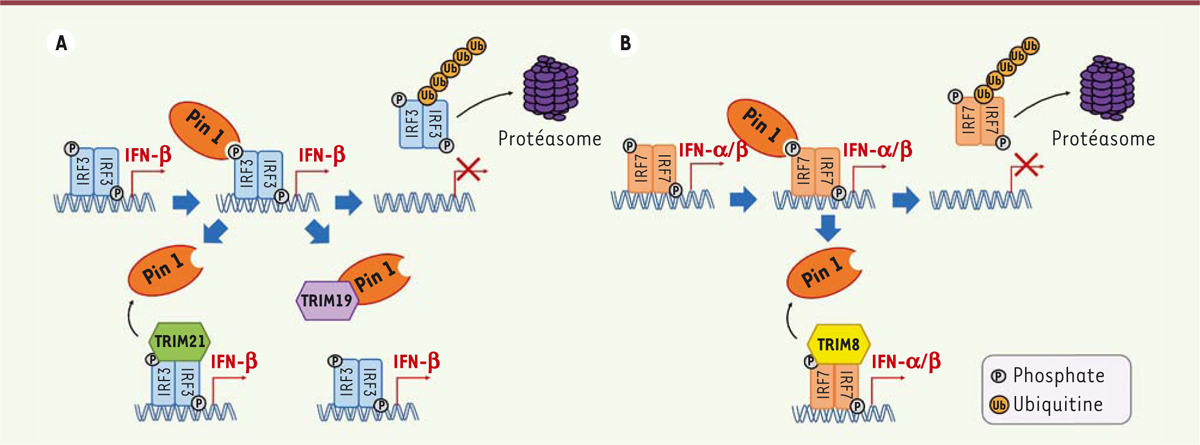
 | Figure 2. Régulation de la réponse IFN par Pin1 et les protéines TRIM.
A. Pin1 interagit avec IRF3-P et
modifie sa conformation, induisant ainsi son ubiquitination et son
adressage au protéasome. Les protéines TRIM21 et TRIM19 empêchent la
reconnaissance d’IRF3-P par Pin1, prolongeant ainsi la synthèse d’IFN-b.
B.IRF7-P est également un substrat
de Pin1. Dans les cellules dendritiques plasmacytoïdes (pDC), TRIM8
protège IRF7-P de l’activité enzymatique de Pin1, évitant sa dégradation
et empêchant ainsi l’arrêt de la transcription des gènes codant les
IFN-a et IFN-b. |
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
1.
Schneider
WM,
Chevillotte
MD,
Rice
CM.
Interferon-stimulated genes: a complex web of host
defenses . Annu Rev Immunol.
2014; ; 32 :
:513.–545. 2.
Kawai
T,
Akira
S. Innate immune
recognition of viral infection . Nat Immunol.
2006; ; 7 :
:131.–137. 3.
Swiecki
M,
Colonna
M. The multifaceted
biology of plasmacytoid dendritic cells . Nat Rev
Immunol.
2015; ; 15 :
:471.–485. 4.
Komander
D,
Rape
M. The ubiquitin
code . Annu Rev Biochem.
2012; ; 81 :
:203.–229. 5.
Stremlau
M,
Owens
CM,
Perron
MJ, et al.
The cytoplasmic body component TRIM5a restricts HIV-1 infection
in old world monkeys . Nature.
2004; ; 427 :
:848.–853. 6.
Nisole
S,
Stoye
JP,
Saib
A. TRIM family
proteins: retroviral restriction and antiviral defence .
Nat Rev Microbiol.
2005; ; 3 :
:799.–808. 7.
Gack
MU,
Shin
YC,
Joo
CH, et al.
TRIM25 RING-finger E3 ubiquitin ligase is essential for
RIG-I-mediated antiviral activity . Nature.
2007; ; 446 :
:916.–920. 8.
Rajsbaum
R,
Garcia-Sastre
A,
Versteeg
GA. TRIMmunity: the
roles of the TRIM E3-ubiquitin ligase family in innate antiviral
immunity . J Mol Biol.
2014; ; 426 :
:1265.–1284. 9.
Versteeg
GA,
Rajsbaum
R,
Sanchez-Aparicio
MT, et al.
The E3-ligase TRIM family of proteins regulates signaling
pathways triggered by innate immune pattern-recognition
receptors . Immunity.
2013; ; 38 :
:384.–398. 10.
Maarifi
G,
Smith
N,
Maillet
S, et al.
TRIM8 is required for virus-induced IFN response in human
plasmacytoid dendritic cells . Sci Adv.
2019;; 5 :
:eaax3511.. 11.
Liou
YC,
Zhou
XZ,
Lu
KP. Prolyl isomerase
Pin1 as a molecular switch to determine the fate of
phosphoproteins . Trends Biochem Sci.
2011; ; 36 :
:501.–514. 12.
Saitoh
T,
Tun-Kyi
A,
Ryo
A, et al.
Negative regulation of interferon-regulatory factor 3-dependent
innate antiviral response by the prolyl isomerase Pin1 .
Nat Immunol.
2006; ; 7 :
:598.–605. 13.
Yang
K,
Shi
HX,
Liu
XY, et al.
TRIM21 is essential to sustain IFN regulatory factor 3 activation
during antiviral response . J Immunol.
2009; ; 182 :
:3782.–3792. 14.
El Asmi
F,
Maroui
MA,
Dutrieux
J, et al.
Implication of PMLIV in both intrinsic and innate
immunity . PLoS Pathog.
2014; ; 10 :
:e1003975.. |