| |
| Med Sci (Paris). 36(3): 253–260. doi: 10.1051/medsci/2020022.Les rétrovirus endogènes Un rôle clé dans la programmation des lymphocytes T
CD4 Véronique Adoue1 and Olivier Joffre1* 1Centre de Physiopathologie Toulouse Purpan, Inserm
U1043 - BP
3028, 31024Toulouse Cedex 3,
France |

Les lymphocytes T CD4 sont des cellules immunitaires extrêmement efficaces pour protéger
l’organisme contre les dangers exogènes, tels que les virus, les bactéries ou les
parasites. De nombreuses données de la littérature montrent aussi que ces cellules ont
un rôle clé dans le processus d’immunosurveillance qui limite l’apparition et le
développement de tumeurs [1].
Leur manipulation est donc porteuse d’immenses espoirs en clinique humaine, notamment en
cancérologie et dans le domaine des maladies infectieuses. L’efficacité des lymphocytes
T CD4 dépend de leur capacité à adapter leur phénotype et leurs fonctions au type de
danger détecté par les cellules de l’immunité innée. Selon la nature de la menace, les
cellules présentes dans les tissus lésés émettent en effet des signaux moléculaires
différents qui engagent les lymphocytes T CD4 naïfs dans des lignages fonctionnellement
distincts appelés T helper (Th) [2, 3]. Nous
allons ici discuter des mécanismes moléculaires qui contrôlent l’activation, la
différenciation et la plasticité des lymphocytes T. Nous nous intéresserons tout d’abord
aux voies de régulation épigénétiques dépendant de la marque H3K9me3, qui correspond à
la triméthylation de l’histone H3 sur sa lysine 9, et aux éléments génomiques impliqués
dans ces processus. Nous envisagerons enfin comment les découvertes récentes pourraient
se traduire en avancées thérapeutiques dans un avenir proche. |
Activation et différenciation des lymphocytes T CD4 Les lymphocytes T CD4 sont donc des cellules extrêmement plastiques, capables
d’ajuster qualitativement et quantitativement leur programmation afin de délivrer
une réponse adaptée au danger détecté. Lors d’une infection par un agent pathogène
intracellulaire, l’IFN-g (interféron-g) et l’IL(interleukine)-12 produits notamment
par les cellules de l’immunité innée, coordonnent par exemple un programme
développemental qui aboutit à la différenciation des lymphocytes en cellules Th1,
notamment caractérisées par l’expression du facteur de transcription T-bet et par la
production d’IFN-g (Figure 1).
En revanche, si un helminthe (vers parasite intestinal) pénètre dans l’organisme,
les lymphocytes T CD4 spécifiques de ce pathogène se différencieront vers un
phénotype Th2. Ils exprimeront alors GATA-3 et produiront des quantités importantes
d’IL-4, d’IL-5 et d’IL-13.
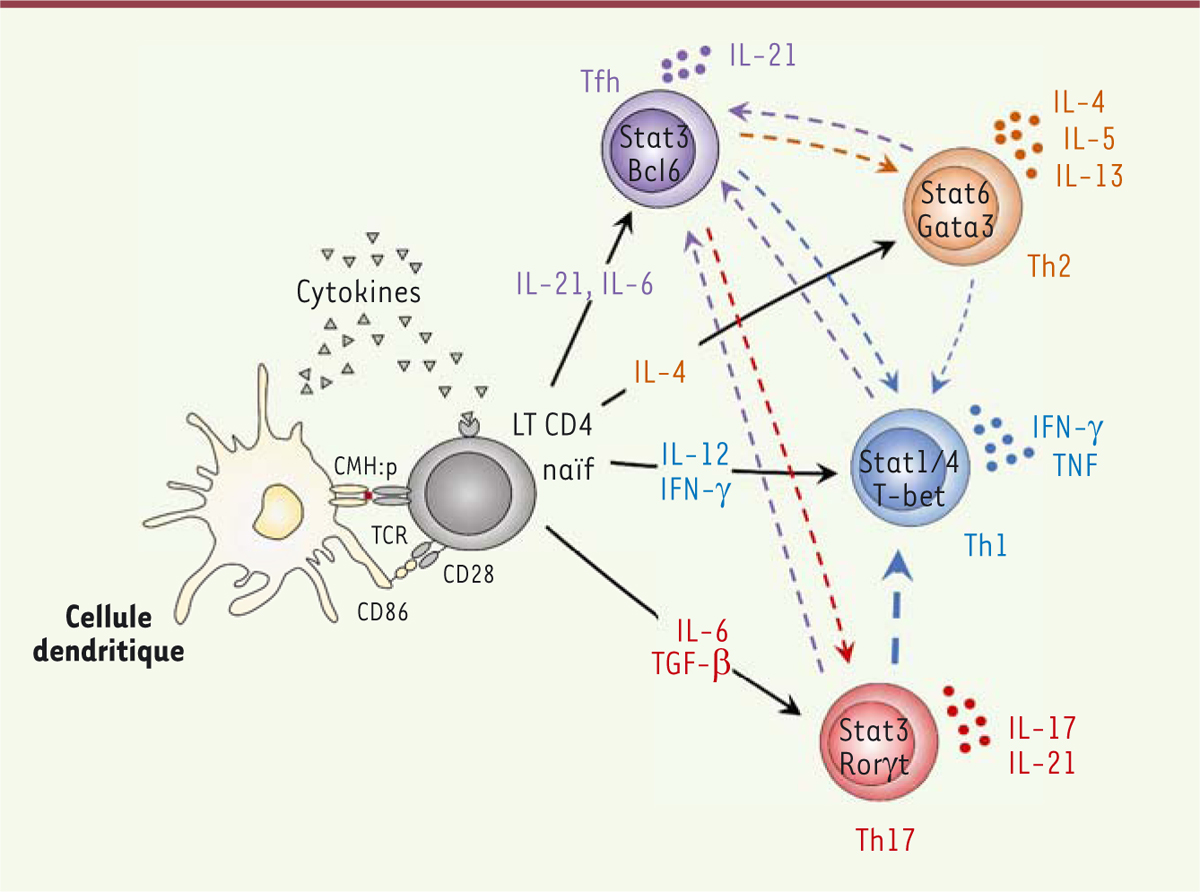
 | Figure 1.Hétérogénéité et plasticité du compartiment T CD4. L’intégration du
signal antigénique (CMH:p/TCR), du signal de co-stimulation (CD86/CD28)
et du signal polarisant (cytokines) permet au lymphocyte T CD4 naïf de
se différencier vers le phénotype T helper (Th) adapté au danger (seules
les principales populations de Th sont représentées). Lors de leur
différenciation, les lymphocytes T perdent en potentialité mais
conservent une certaine plasticité, variable selon les lignages. Ces
processus d’engagement, de détermination et de plasticité cellulaire
sont sous le contrôle de mécanismes de régulation épigénétique, à
l’interface entre l’environnement et le génome (adaptée de [ 3]). |
Quel que soit le lignage T helper considéré, la différenciation des
lymphocytes T CD4 obéit à un même schéma (Figure 1). Leur activation initiale nécessite l’intégration
de trois types de signaux [4,
5]. Le signal
antigénique, exposé sous la forme de complexes entre les molécules du CMH (complexe
majeur d’histocompatibilité) et des peptides provenant du pathogène à la surface des
cellules dendritiques (DC), garantit la spécificité de la réponse immunitaire. Il
est détecté par les lymphocytes T grâce à leur récepteur de l’antigène (T
Cell Receptor, TCR). Le deuxième signal, dit « de co-stimulation »,
implique l’interaction entre les molécules CD28 et CD80 ou CD86, exprimées
respectivement à la membrane des lymphocytes T et des DC. Il a notamment un rôle clé
dans la survie cellulaire et la régulation du métabolisme énergétique. Le troisième
signal prend souvent la forme de cytokines sécrétées par les cellules dendritiques
ou par des cellules tierces. Il est qualifié de polarisant car il instruit le choix
du lignage de la cellule. Ce sont donc les facteurs de transcription mobilisés en
aval des récepteurs des cytokines qui vont conférer la spécificité de programmation
à chaque lignage T helper. La plupart des récepteurs des cytokines impliqués dans la programmation des
lymphocytes T CD4 activent des évènements de signalisation qui aboutissent à
l’activation des protéines STAT (signal transducer and activator of
transcription)[6]. C’est notamment le cas des récepteurs de l’IL-12 et de l’IFN-g qui
coordonnent l’engagement des lymphocytes T naïfs dans le lignage Th1 en mobilisant
respectivement STAT-4 et STAT-1. Une fois activés par les tyrosines kinases JAK
(Janus kinase), ces facteurs de transcription vont rejoindre le
noyau afin d’induire l’expression du master regulator spécifique du
lignage (e.g. T-bet, pour le lignage Th1 ; GATA-3, pour le
phénotype Th2) et d’initier l’expression des cytokines effectrices [6]. Sous l’effet du master
regulator et des protéines STAT mobilisées par les cytokines sécrétées
par les lymphocytes T et les cellules du microenvironnement, un ensemble restreint
de facteurs de transcription se met en place. C’est ce réseau de régulateurs
transcriptionnels qui coordonne de façon dynamique le programme développemental qui
permettra in fine aux lymphocytes T d’acquérir des fonctions
effectrices propres au lignage, et adaptées à l’éradication du danger. Une fois différenciés, et selon le contexte physiopathologique, les lymphocytes T
helper vont devoir transmettre leur programmation à leur
descendance sur de multiples cycles de division, notamment lors de la phase
d’expansion clonale qui permet à une cellule spécifique d’un danger de générer des
millions de cellules filles identiques et de même spécificité. Conserver la
programmation établie lors de l’activation initiale est aussi nécessaire pour que
les lymphocytes T mémoires puissent garantir une protection efficace de l’organisme
à long terme, même dans un environnement cytokinique différent. En théorie, le
répertoire de facteurs de transcription mobilisé lors de l’étape d’activation par
les cellules dendritiques devrait être suffisant pour définir et maintenir
l’identité des lymphocytes T helper. Les boucles de retrocontrôle
positif qui se mettent en place permettent en effet à ces molécules d’être exprimées
de façon stable, à travers les cycles de division cellulaire. En réalité, l’activité
des facteurs de transcription est fortement conditionnée par le contexte nucléaire
dans lequel ils agissent. Leur recrutement sur les régions
cis-régulatrices des locus d’intérêt, tels que les promoteurs ou
les enhancers, s’accompagne d’un remodelage massif mais ciblé de la
chromatine. Cette réorganisation a un impact immédiat sur l’activité des autres
facteurs de transcription et conditionne, à plus long terme, la réponse de la
cellule aux signaux environnementaux. Ce remodelage épigénétique, initié par les
facteurs de transcription et orchestré par des acteurs moléculaires qui restent
encore largement à identifier, joue un rôle clé dans la biologie des lymphocytes T
CD4 [2, 7]. En rendant accessibles les locus spécifiques du lignage aux
facteurs trans, il permet, par exemple, une forte expression des
gènes d’intérêt tels que ceux qui codent les cytokines effectrices. À l’inverse, les
facteurs de transcription mobilisés vont aussi verrouiller des locus propres aux
phénotypes alternatifs. Ces mécanismes de répression génique ont un rôle causal dans
la définition de l’identité des lymphocytes T [7–10]. Ceci
explique pourquoi ces mécanismes de régulation épigénétique sont aujourd’hui l’objet
de beaucoup d’attention. Mieux les caractériser pourrait en effet permettre
d’identifier des molécules chromatiniennes sur lesquelles agir afin de stimuler la
génération de lymphocytes T mémoires dans un contexte vaccinal. Dans des situations
physiopathologiques dans lesquelles la plasticité des lymphocytes T est délétère
pour l’organisme, comme lorsque l’environnement tumoral désarme les lymphocytes Th1
censés éradiquer la tumeur, caractériser les voies de régulation épigénétique qui
contrôlent l’ouverture et la fermeture de la chromatine au niveau des gènes
spécifiques des cellules T helper devrait aussi permettre
d’identifier de nouvelles cibles thérapeutiques qui permettront de restaurer une
immunité protectrice. Pour des raisons fondamentales et dans un but thérapeutique,
il est donc aujourd’hui nécessaire de caractériser les voies moléculaires qui
contrôlent la biologie des lymphocytes T au niveau de la chromatine. |
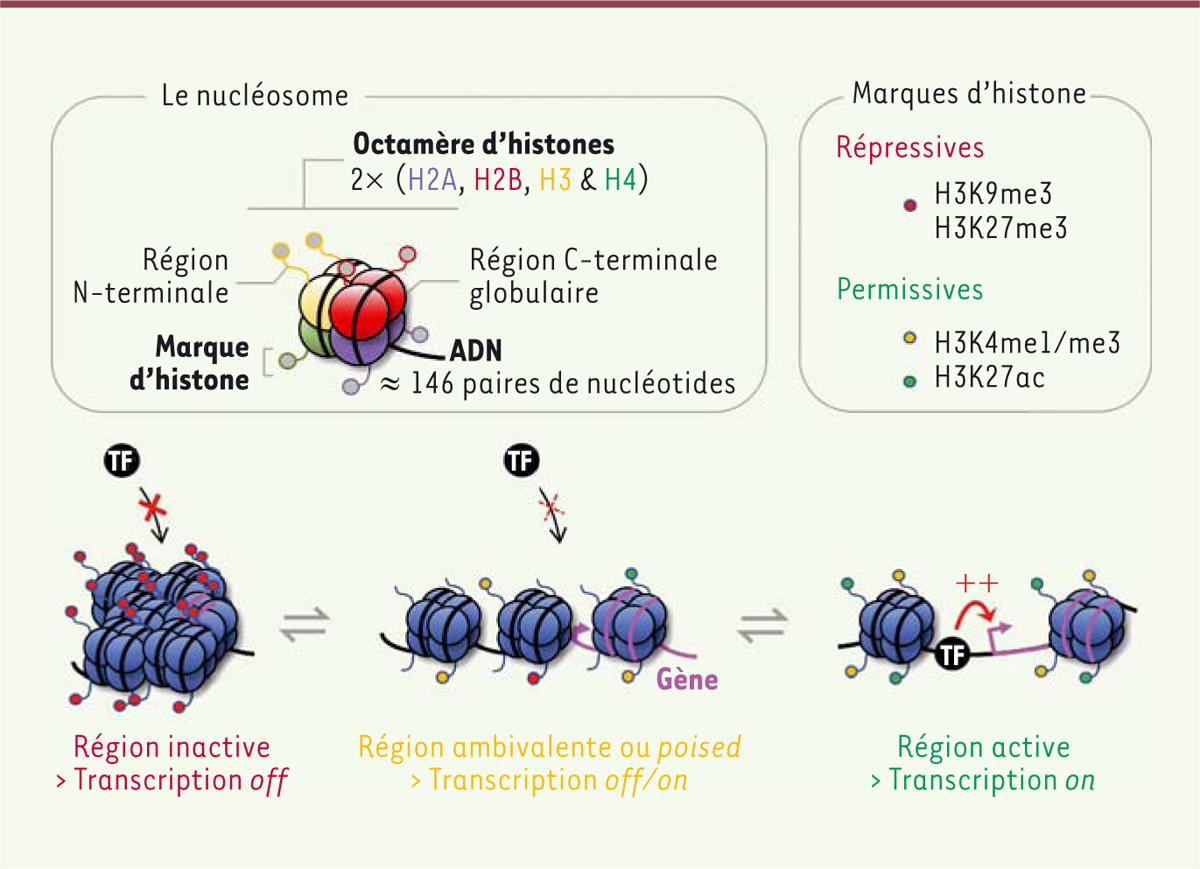
Régulation épigénétique de l’expression génique Le génome nucléaire humain, long d’environ deux mètres, ne se trouve jamais sous
forme d’ADN nu à l’intérieur des cellules. Il est organisé et compacté dans leurs
noyaux par de petites protéines basiques, les histones, qui ont une masse
moléculaire d’environ 15 kDa et qui sont phylogénétiquement très conservées.
L’ensemble ADN et histones forme la chromatine dont l’unité de base est le
nucléosome [11]
(→).
(→) Voir la Synthèse de A. Bertin et S. Mangenot, m/s n° 8-9,
août-septembre 2008, page 715
Deux copies des histones H2A, H2B, H3 et H4 s’associent pour en former le cœur
protéique autour duquel s’enroule le brin d’ADN sur une longueur d’environ 146
paires de nucléotides (Figure
2). Chez les eucaryotes, le nucléosome correspond au premier
niveau de compaction de l’information génétique nucléaire. À ce stade, la chromatine
prend la forme d’un collier de perles de onze nanomètres de diamètre, les
nucléosomes étant reliés entre eux par de l’ADN de liaison. C’est sous cette forme
de chromatine ouverte, appelée euchromatine, que se trouve les locus contenant les
gènes exprimés par chaque cellule (Figure
2). Les régions du génome contenant des gènes qui leur sont
inutiles, car appartenant par exemple à un autre lignage, sont compactées sous forme
de fibres pouvant atteindre plusieurs centaines de nanomètres, ce qui permet de
prévenir le recrutement de protéines trans-activatrices et
d’inhiber ainsi l’expression génique. Cette chromatine compacte, qui peut
éventuellement être déverrouillée, prend le nom d’hétérochromatine facultative.
Entre ces deux situations extrêmes, de nombreux locus existent sous une forme
ambivalente (poised), prêts à être mobilisés, ou au contraire
verrouillés, selon les informations environnementales qui seront reçues et intégrées
par la cellule (Figure 2).
C’est notamment le cas des gènes qui codent les cytokines et les master
regulators des différents lignages T helper dans les
lymphocytes T naïfs. Le gène Ifng, qui code l’IFN-g caractéristique
du lignage Th1, sera par exemple complètement ouvert et relâché si la cellule se
différencie dans ce lignage, ou, au contraire, compacté sous forme
d’hétérochromatine facultative si elle s’engage dans le lignage antagoniste Th2
[7–9,12].
 | Figure 2. La régulation de l’expression génique dépend de mécanismes épigénétiques
qui modifient la composition et la structure de la chromatine. Le
nucléosome, qui constitue l’unité de base de la chromatine, est
constitué d’un octamère d’histones autour duquel s’enroule le filament
d’ADN. Selon les informations intégrées par la cellule, et selon le
locus sur lequel elles sont présentes, ces
structures sont compactées afin de prévenir le recrutement des facteurs
de transcription (TF) et donc l’expression génique, ou, à l’inverse,
déplacées et relâchées pour permettre l’initiation ou l’amplification de
la transcription. Des voies épigénétiques complexes, impliquant
différentes modifications des régions amino-terminales des histones,
contrôlent ces processus. |
Les mécanismes de régulation qui contrôlent la compaction et le relâchement de la
chromatine, et donc l’expression des gènes, impliquent le dépôt sur l’ADN et les
histones de marques épigénétiques qui, bien que réversibles, sont transmissibles au
cours des divisions cellulaires. Ce marquage est catalysé par différents types
d’enzymes regroupées sous le nom de chromatin writers. À quelques
rares exceptions près, leur positionnement sur la chromatine n’a pas d’effet
biologique per se. Ces marques servent de point d’ancrage à des
chromatin readers qui permettent l’assemblage de complexes
protéiques multimériques qui, une fois échafaudés, modifient la composition, la
structure et la fonction de la chromatine. Selon la marque épigénétique, sa
localisation sur le génome, et le contexte chromatinien environnant, des complexes
différents sont assemblés. Ceci explique que certaines marques, qualifiées de «
permissives », sont associées à des effets positifs sur l’expression génique alors
que d’autres, présentées comme « répressives », sont impliquées dans la répression
de la transcription. Les marques épigénétiques ciblent les deux composantes
principales de la chromatine, l’ADN et les histones. La méthylation de l’ADN consiste en l’ajout d’un groupement méthyl sur le carbone 5
d’un résidu cytosine dans un contexte de di-nucléotide CpG, par une ADN
méthyltransférase (DNMT) [13]. Lorsqu’elle affecte des régions cis-régulatrices,
elle est de façon quasi-systématique associée à la répression de la transcription.
Les cytosines méthylées peuvent directement interférer avec le recrutement de
facteurs de transcription, servir de point d’ancrage à des
methyl-CpG-binding proteins (MBP) qui entrent en compétition
avec des molécules trans-activatrices, ou permettre le recrutement
de complexes multimériques qui vont catalyser la déposition et la propagation
d’autres marques répressives et la compaction de la chromatine [13]. La méthylation de l’ADN, lorsqu’elle est
présente sur le corps des gènes, a également été associée à des régions actives d’un
point de vue transcriptionnel et à l’épissage alternatif. Les modifications post-traductionnelles covalentes des histones sont des marques
épigénétiques qui jouent aussi un rôle majeur dans la régulation dynamique de
l’expression génique. Les quatre histones de cœur sont des protéines riches en
résidus lysine et arginine, des acides aminés basiques dont la charge positive
permet une interaction avec les groupements phosphate de l’ADN chargés négativement.
Elles possèdent un domaine carboxy-terminal globulaire très conservé contenant un
domaine histone fold qui permet leur hétérodimérisation. Leur
extrémité amino-terminale, plus variable et dépourvue de structure secondaire,
s’expose à la surface du nucléosome. C’est elle qui est la cible de la plupart des
modifications post-traductionnelles susceptibles de modifier la charge de l’histone,
les interactions protéine/ADN et protéine/protéine. Les acides aminés les plus
soumis à modifications, qui incluent l’acétylation, la méthylation, la
phosphorylation, la SUMOylation et l’ubiquitination, sont les résidus lysine,
arginine, sérine et thréonine. Au cours des dix dernières années, de nombreux
travaux ont notamment documenté un rôle clé pour l’acétylation et la méthylation des
lysines. L’acétylation des lysines par les histones acétyltransférases (HAT) concerne
principalement les résidus présents au niveau des extrémités amino-terminales des
histones H3 et H4. Elle est systématiquement associée à des locus dont la
transcription est active et elle a deux conséquences principales. Elle aboutit tout
d’abord à la neutralisation de la charge positive de l’acide aminé, ce qui favorise
le recrutement de la machinerie de transcription en diminuant l’interaction
électrostatique entre le filament d’ADN et les histones. L’acétylation des lysines
permet aussi le recrutement de protéines à bromodomaine (e.g. HAT, co-activateurs de
la transcription) qui auront un effet positif sur l’expression génique. Les conséquences de la méthylation des lysines sur les fonctions du génome sont
beaucoup moins stéréotypées. En effet, selon le résidu et l’histone ciblés, le
nombre de groupements méthyl ajoutés et la localisation de l’histone sur le génome,
la méthylation peut être associée à une régulation positive ou négative de
l’expression génique. La triméthylation de la lysine 4 de l’histone 3 (H3K4me3) est,
par exemple, retrouvée au niveau des promoteurs actifs où elle est impliquée dans
l’assemblage du complexe de pré-initiation de la transcription [14]. La monométhylation du même résidu
(H3K4me1) est une marque caractéristique non pas des promoteurs mais des
enhancers actifs (H3K27ac+) et
poised (H3K27ac-), c’est-à-dire en pause mais prêts
à être activés ou réprimés selon les signaux reçus par la cellule (Figure 2). Les
enhancers réprimés sont dépourvus d’H3K4me1, mais sont par
contre enrichis en H3K27me3, une marque catalysée par l’histone méthyltransférase
Ezh2 (enhancer of zeste homolog 2) qui fait partie du complexe
répresseur PRC2 (polycomb repressive complex 2). La marque H3K9me3 est elle-même extrêmement versatile [15]. Elle a été historiquement associée à la
formation de l’hétérochromatine constitutive [16, 17], c’est-à-dire aux régions péricentromériques et
télomériques du génome, pauvres en gènes codants, riches en éléments répétés, et
dont la chromatine est condensée dans toutes les cellules et à toutes les étapes du
cycle cellulaire. Elle a depuis été impliquée dans la régulation positive et
négative de l’expression génique. H3K9me3 permet notamment la répression de
promoteurs de gènes codant des régulateurs développementaux et le maintien de la
pluripotence des cellules souches embryonnaires [18]. Chez l’adulte, la répression de
l’expression génique dépendant de H3K9me3 dans l’euchromatine et l’hétérochromatine
facultative est également nécessaire pour définir et maintenir l’identité cellulaire
[8,9,19].
Cependant, H3K9me3 peut également être retrouvée sur le corps des gènes
transcriptionnellement actifs où elle serait impliquée dans l’élongation de la
transcription et l’épissage alternatif [20, 21]. Cette marque, extrêmement versatile, régule donc de nombreuses
fonctions du génome. Son rôle dans la biologie des lymphocytes T CD4 commence juste
à être révélé. |
H3K9me3 joue un rôle clé dans la différenciation et la stabilité des lymphocytes
T La marque H3K9me3 peut être déposée par différentes lysine méthyltransférases,
notamment celles de la famille SUV39H (histone-lysine
N-methyltransferase) qui inclue SUV39H1, SUV39H2 et SETDB1. Bien que
SUV39H1 et SUV39H2 aient initialement été associées à l’échafaudage de
l’hétérochromatine constitutive [17], des
travaux récents ont mis en évidence un rôle de SUV39H1 dans la régulation de
l’expression génique dans les lymphocytes T (Tableau I). Dans les lymphocytes Th2, la voie épigénétique
SUV39H1/H3K9me3 réprime le promoteur du gène Ifng et est ainsi
impliquée dans la stabilité du lignage Th2 [8,
22]
(→).
(→) Voir la Nouvelle de E. Zueva, m/s n° 12, décembre 2012,
page 1032
Tableau I.
| Molécule |
|
Effet biologique |
Référence |
| Nom |
Type |
|
|
|
| SETDB1 |
Lysine méthyltransférase |
Répression d’ERV cooptés en éléments
cis-régulateurs de gènes Th1Régulation du priming Th1 et de la
plasticité Th2 |
[9] |
|
| SUV39H1 |
Lysine méthyltransférase |
Répression du promoteur du gène
IfngRégulation de la plasticité des
cellules Th2 |
[8] |
|
| HP1a |
Chromatin reader
|
Répression du promoteur du gène
IfngRégulation de la plasticité des
cellules Th2 |
[8] |
|
| TRIM28 |
Co-répresseur transcriptionnel |
Régulation de la production d’IL-2 et de
TGF-b3Contrôle de la balance cellules Th17/celules T
régulatrices |
[39] |
|
| HP1g |
Chromatin reader
|
Répression du locus FoXP3
dépendante de la SUMO E3 ligase PIAS1Régulation du ratio
cellules T CD4 conventionnelles/régulatrices |
[40] |
|
| SETDB1 |
Lysine méthyltransférase |
Contrôle du développement intrathymique des
cellules TRépression du gène
Fcg RIIb dont la
signalisation interfère avec la transduction du signal du
complexe TCR |
[41] |
|
| SUV39H1 |
Lysine méthyltransférase |
Contrôle de l’expression d’un réseau de gènes
impliqués dans la programmation des cellules T CD8
mémoires.Contrôle des lignages T CD8 effecteurs
vs mémoires |
[23] |
|
| TRIM28 |
Co-répresseur transcriptionnel |
Régulation du développement intrathymique des
cellules TContrôle de la réponse des cellules T aux signaux
cytokiniques et antigéniques |
[42] |
|
| SETDB1 |
Lysine méthyltransférase |
Contrôle du développement intrathymique des
cellules TRépression du gène
Fcg RIIb dont la
signalisation réprime l’activation de ERK |
[43] |
|
| HP1b |
Chromatin reader
|
Régulation de l’expression du co-récepteur CD4 dans
les thymocytes en cours de développement et les cellules T
maturesRépression d’un silencer du gène CD4 |
[44] |
|
| HP1g |
Chromatin reader
|
Accumulation sur le corps du gène
Il2 dans les cellules T activéesRôle dans
la régulation positive de l’expression génique ? |
[20] |
|
| SETDB1 |
Lysine méthyltransférase |
Répression du locus Il17A
dépendante de l’axe OX40/NF-kB dans les cellules Th17 (mécanisme
de rétrocontrôle négatif) |
[26] |
|
| TRIM28 |
Co-répresseur transcriptionnel |
Régulation du réarrangement du locus
TCRaContrôle du développement des
lymphocytes Tab et des cellules NKT |
[45] |
Principaux travaux impliquant les voies épigénétiques dépendantes
d’H3K9me3 dans la biologie des lymphocytes T CD4. |
L’importance fonctionnelle de cette voie a également été décrite récemment dans la
différenciation des lymphocytes T CD8 effecteurs et mémoires [23]. Il a notamment été montré que SUV39H1 est nécessaire à la
répression des gènes associés à la mémoire immunologique dans les lymphocytes T CD8
activés qui s’engagent dans le lignage effecteur. Tout comme SUV39H1 et SUV39H2, SETDB1 (SET domain bifurcated histone lysine
methyltransferase 1) a aussi été impliquée dans l’assemblage de
l’hétérochromatine constitutive et dans la régulation dynamique de l’expression
génique dans différents types cellulaires incluant les cellules T [9,24-26] (Tableau I). Dans les lymphocytes
Th17, la voie de co-stimulation dépendante d’OX40 induit par exemple le recrutement
de SETDB1 sur le locus Il17 ce qui aboutit à sa répression [26]. Notre équipe a aussi récemment documenté
un rôle clé pour cette enzyme dans la biologie des lymphocytes Th1 et Th2, notamment
via la répression de rétrovirus endogènes (ERV) qui auraient,
au cours de l’évolution, été cooptés en modules cis-régulateurs de
gènes Th1 [9]. |
L’axe SETDB1/H3K9me3 contrôle l’identité des lymphocytes Th2 en réprimant des
rétrovirus endogènes cooptés en modules cis-régulateurs des gènes Th1 Les séquences dérivées de rétrovirus endogènes représentent près de 10 % du
patrimoine génétique de l’homme et de la souris. Elles correspondent à des vestiges
de génomes rétroviraux dont l’activité de transposition, potentiellement dangereuse,
a dû être réprimée par les cellules hôtes à l’aide de mécanismes épigénétiques
impliquant la méthylation de l’ADN et la marque H3K9me3 [27–29]. Au fur et à mesure de leur découverte, les ERV ont été
classés et nommés en fonction du rétrovirus exogène dont ils sont le plus proches.
Chez l’homme, ils ont ainsi été répartis en trois classes selon leur lien de parenté
avec les gammarétrovirus (classe I), les bêtarétrovirus (classe II) ou les
spumavirus (classe III). Il est également commun qu’une ou deux lettres ai(en)t été
ajoutée(s) au début de leur nom, pour indiquer l’espèce dans laquelle ils ont
initialement été identifiés. Les HERV et MuERV ont, par exemple, été respectivement
décrits pour la première fois chez l’homme et la souris. Le génome des ERV est
essentiellement constitué des gènes gag (protéine d’encapsidation), pol
(transcriptase inverse) et env (protéine d’enveloppe), qui codent des protéines
essentielles à la réplication virale. Leur expression est régulée par des séquences
LTR (long terminal repeats), généralement dupliquées aux extrémités
des séquences codantes et comprenant des régions initiatrices et régulatrices de la
transcription [30]. Les ERV, comme les autres éléments transposables, n’ont pas seulement représenté un
danger pour nos organismes. Ils ont également été de formidables moteurs de
l’évolution. Des études récentes suggèrent en effet que ces éléments génomiques ont
été détournés en modules cis-régulateurs pour le bénéfice de
l’hôte. Ils auraient notamment un rôle clé dans la régulation de réseaux géniques
impliqués dans la protection de l’organisme contre les agents pathogènes [9,30–34]. Au
cours de l’évolution, les gènes codant les protéines virales ont été excisés du
génome par recombinaison homologue entre les séquences LTR qui les flanquent. Les
LTR « solo » ainsi générés représentent aujourd’hui 90 % des séquences dérivées
d’ERV chez l’homme [30, 35]. Ce sont ces séquences, de par leur
capacité intrinsèque à recruter des facteurs de transcription, qui ont largement été
cooptées en éléments cis-régulateurs de l’expression des gènes. Il
est par exemple estimé que 20 % des sites de fixation pour de nombreux facteurs de
transcription tels que p53, OCT4, SOX2, et NANOG dérivent de LTR solo chez l’homme
comme chez la souris. La présence de sites d’épissage donneur, conservés dans
certaines classes d’ERV ou générés par mutation, a aussi probablement contribué à
leur cooptation en tant que promoteurs alternatifs. C’est le cas par exemple de la
séquence consensus de l’ERV MaLR (mammalian apparent
retrotransposon) qui contient un site d’épissage donneur à l’origine de
nombreux transcrits chimériques dans les oocytes de souris [36]. Comment expliquer que les séquences LTR aient ainsi pu échapper aux mécanismes de
répression épigénétique visant à réprimer les ERV ? Comme indiqué ci-dessus, les
données de la littérature obtenues au cours de la dernière décennie, notamment dans
les cellules souches embryonnaires, suggèrent que la répression des ERV dépend en
grande partie de mécanismes moléculaires impliquant la méthylation de l’ADN et la
déposition de la marque épigénétique H3K9me3 par la lysine méthyltransférase SETDB1.
Cette enzyme serait ciblée sur les ERV par des protéines à doigts de zinc et à
domaine KRAB, les « KRAB-ZFP » (krüppel-associated box domain zinc finger
proteins), le co-répresseur transcriptionnel TRIM28 (tripartite
motif-containing 28) servant d’intermédiaire moléculaire entre les deux
partenaires. Soulignons que la majorité des séquences consensus sur lesquelles se
fixent les KRAB-ZFP se situent dans les séquences internes des ERV [32]. Comme les LTR solo en sont
dépourvus, il est actuellement proposé que cela ait favorisé leur dérépression et
leur cooptation en élément enhancer ou promoteur des gènes situés à
proximité [35]. Le rôle des séquences dérivées d’ERV dans la régulation des réseaux géniques, ainsi
que les mécanismes épigénétiques qui contrôlent leur activité, commencent donc à
être caractérisés dans les cellules souches embryonnaires et les tissus adultes
[9,27,30–34,37]. Notre équipe vient notamment d’identifier le rôle d’un réseau d’ERV
dans la régulation des fonctions des lymphocytes T CD4 chez la souris [9]. Nous avons montré, à l’aide de tests de
différenciation in vitro et d’un protocole d’immunisation
in vivo, que les lymphocytes T CD4 naïfs déficients pour SETDB1
se différencient plus efficacement en cellules Th1 que leurs contreparties sauvages,
et que les cellules Th2 mutantes ne sont plus déterminées dans leur lignage. En
l’absence de la lysine méthyltransférase, les cellules Th2 sont en effet capables de
se transdifférencier vers un phénotype Th1 en présence d’un signal instructeur
spécifique de ce lignage. Des analyses transcriptomiques couplées à des expériences
de ChIP-Seq (chromatin immunoprecipitation followed by high-throughput
sequencing) et d’ATAC-seq (assay for transposase-accessible
chromatin with high-throughput sequencing) visant à cartographier la
distribution des marques H3K9me3, H3K4me1 et H3K27ac à travers le génome ainsi que
l’ouverture de la chromatine au niveau des locus spécifiques des lignages Th1 et
Th2, nous ont de plus permis de montrer que les dérégulations observées à l’échelle
cellulaire résultaient du déverrouillage d’un groupe restreint d’ERV normalement
réprimés dans les lymphocytes T CD4. Nos données indiquent en effet que la
déposition d’H3K9me3 par SETDB1 est nécessaire à la répression de séquences dérivées
d’ERV qui agissent comme des modules cis-régulateurs de gènes clés
du lignage Th1. Elles suggèrent de plus que la plupart des ERV qui régulent
l’activation du programme d’expression génique Th1 ne se comportent pas comme des
enhancers de gènes mais plutôt comme des modules régulant
l’activité d’enhancers adjacents. Dans les cellules Th2, SETDB1
serait donc ciblée sur des séquences d’ERV spécifiques pour y déposer la marque
répressive H3K9me3. La propagation de la marque jusqu’au enhancer
pro-Th1 adjacent aboutirait ensuite à son verrouillage et à la répression du
programme développemental associé. L’impact de l’interaction entre les voies de régulation épigénétiques impliquant
H3K9me3 et les séquences d’ERV se comportant comme des modules
cis-régulateurs dans la régulation des réseaux transcriptionnels
reste encore largement méconnu chez l’homme. Notre travail fait cependant écho à une
étude publiée en 2016 dans laquelle les auteurs ont montré que les mécanismes de
défense innés activés par l’IFN-g dans différents types de cellules humaines
dépendent de la mobilisation d’un panel d’ERV appartenant à la sous-famille MER41
[34]. Même si les éléments transposables
sont peu conservés entre l’homme et la souris, il semble donc que les mêmes
processus pourraient avoir été sélectionnés au cours de l’évolution pour contrôler
l’identité et les fonctions cellulaires, notamment celles nécessaires à la
protection de l’organisme. |
Voies épigénétiques dépendantes d’H3K9me3 : des cibles thérapeutiques
potentielles ? Le travail que nous venons de publier, associé aux données récentes de la
littérature, a donc permis d’identifier la molécule SETDB1 comme un régulateur
majeur de la biologie des lymphocytes T CD4. Cette enzyme est en effet nécessaire
pour réprimer le programme d’expression génique Th1 dans les lymphocytes T en cours
de différenciation. D’un point de vue physiopathologique, ces données suggèrent que
SETDB1 pourrait être nécessaire pour prévenir l’emballement des réponses
immunitaires de type 1, et donc l’apparition d’immunopathologies, et pour stabiliser
la programmation des cellules Th2. Les réponses Th1 jouant un rôle clé dans le rejet
des tumeurs [38] et dans la
protection de l’organisme contre les pathogènes intracellulaires, il pourrait être
bénéfique en clinique humaine de manipuler l’expression ou l’activité de SETDB1 afin
d’amplifier la réponse immunitaire chez des patients atteints de cancer ou
développant une réponse anti-infectieuse non protectrice. Chez des individus
souffrant de pathologies immunoinflammatoires de type Th2, interférer directement ou
indirectement avec l’activité de cette enzyme, et plus généralement avec la
déposition de la marque répressive H3K9me3 sur la chromatine, pourrait aussi être
bénéfique. Cela devrait en effet permettre de dévier la réponse immunitaire vers un
phénotype Th1, non pathogénique dans ce contexte. En accord avec cette hypothèse,
des travaux récents montrent que l’inhibition de SUV39H1 par la chaetocine permet de
diminuer la sévérité de la pathologie dans un modèle murin d’asthme allergique
[8]. Cette molécule est d’ailleurs déjà
utilisée en clinique humaine pour déstabiliser la programmation des cellules
tumorales. C’est aussi le cas d’inhibiteurs non spécifiques de SETDB1 tels que le
DZNep, la mithramycine A ou le paclitaxel. Dans un avenir proche, l’utilisation de
ces agents pharmacologiques pourrait donc permettre de rapidement traduire nos
observations en avancée thérapeutique pour l’homme. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
ANR - Contrats : EpiTreg (ANR-14- CE14-0021-01), ImmuneTrans (ANR-19-CE15-0022).
Région Occitanie NVEQ 2014 - Contrat : 14051291.
FRM - Contrat : AJE201212.
|
1.
Dunn
GP,
Bruce
AT,
Ikeda
H, et al.
Cancer immunoediting: from immunosurveillance to tumor
escape . Nat Immunol.
2002; ; 3 :
:991.–998. 2.
Kanno
Y,
Vahedi
G,
Hirahara
K, et al.
Transcriptional and epigenetic control of T helper cell
specification: molecular mechanisms underlying commitment and
plasticity . Annu Rev Immunol.
2012; ; 30 :
:707.–731. 3.
O’Shea
JJ,
Paul
WE. Mechanisms
underlying lineage commitment and plasticity of helper CD4+ T
cells . Science.
2010; ; 327 :
:1098.–1102. 4.
Curtsinger
JM,
Schmidt
CS,
Mondino
A, et al.
Inflammatory cytokines provide a third signal for activation of
naive CD4+ and CD8+ T cells . J
Immunol.
1999; ; 162 :
:3256.–3262. 5.
Harding
FA,
McArthur
JG,
Gross
JA, et al.
CD28-mediated signalling co-stimulates murine T cells and
prevents induction of anergy in T-cell clones .
Nature.
1992; ; 356 :
:607.–609. 6.
O’Shea
JJ,
Lahesmaa
R,
Vahedi
G, et al.
Genomic views of STAT function in CD4+ T helper cell
differentiation . Nat Rev Immunol.
2011; ; 11 :
:239.–250. 7.
Wilson
CB,
Rowell
E,
Sekimata
M. Epigenetic
control of T-helper-cell differentiation . Nat Rev
Immunol.
2009; ; 9 :
:91.–105. 8.
Allan
RS,
Zueva
E,
Cammas
F, et al.
An epigenetic silencing pathway controlling T helper 2 cell
lineage commitment . Nature.
2012; ; 487 :
:249.–253. 9.
Adoue
V,
Binet
B,
Malbec
A, et al.
The histone methyltransferase SETDB1 controls T helper cell
lineage integrity by repressing endogenous retroviruses .
Immunity.
2019; ; 50 :
:629.–644. 10.
Bird
JJ,
Brown
DR,
Mullen
AC, et al.
Helper T cell differentiation is controlled by the cell
cycle . Immunity.
1998; ; 9 :
:229.–237. 11.
Bertin
A,
Mangenot
S. Structure et
dynamique de la particule coeur de nucléosome . Med
Sci (Paris).
2008; ; 24 :
:715.–719. 12.
Grogan
JL,
Mohrs
M,
Harmon
B, et al.
Early transcription and silencing of cytokine genes underlie
polarization of T helper cell subsets .
Immunity.
2001; ; 14 :
:205.–215. 13.
Klose
RJ,
Bird
AP. Genomic DNA
methylation: the mark and its mediators . Trends
Biochem Sci.
2006; ; 31 :
:89.–97. 14.
Lauberth
SM,
Nakayama
T,
Wu
X, et al.
H3K4me3 interactions with TAF3 regulate preinitiation complex
assembly and selective gene activation .
Cell.
2013; ; 152 :
:1021.–1036. 15.
Mozzetta
C,
Boyarchuk
E,
Pontis
J,
Ait-Si-Ali
S. Sound of silence:
the properties and functions of repressive Lys
methyltransferases . Nat Rev Mol Cell Biol.
2015; ; 16 :
:499.–513. 16.
Lachner
M,
O’Carroll
D,
Rea
S, et al.
Methylation of histone H3 lysine 9 creates a binding site for HP1
proteins . Nature.
2001; ; 410 :
:116.–120. 17.
Peters
AH,
O’Carroll
D,
Scherthan
H, et al.
Loss of the Suv39h histone methyltransferases impairs mammalian
heterochromatin and genome stability . Cell.
2001; ; 107 :
:323.–337. 18.
Bilodeau
S,
Kagey
MH,
Frampton
GM, et al.
SetDB1 contributes to repression of genes encoding developmental
regulators and maintenance of ES cell state . Genes
Dev.
2009; ; 23 :
:2484.–2489. 19.
Liu
J,
Magri
L,
Zhang
F, et al.
Chromatin landscape defined by repressive histone methylation
during oligodendrocyte differentiation . J
Neurosci.
2015; ; 35 :
:352.–365. 20.
Vakoc
CR,
Mandat
SA,
Olenchock
BA,
Blobel
GA. Histone H3
lysine 9 methylation and HP1γ are associated with transcription elongation
through mammalian chromatin . Mol Cell.
2005; ; 19 :
:381.–391. 21.
Saint-André
V,
Batsché
E,
Rachez
C,
Muchardt
C. Histone H3 lysine
9 trimethylation and HP1γ favor inclusion of alternative
exons . Nat Struct Mol Biol.
2011; ; 18 :
:337.–344. 22.
Zueva
E,
Allan
RS,
Cammas
F, et al.
Contrôle épigénétique de la stabilité phénotypique et
fonctionnelle des lymphocytes Th2 par la voie Suv39h1/HP1a .
Med Sci (Paris).
2012; ; 28 :
:1032.–1034. 23.
Pace
L,
Goudot
C,
Zueva
E, et al.
The epigenetic control of stemness in CD8+ T cell fate
commitment . Science.
2018; ; 359 :
:177.–186. 24.
Loyola
A,
Tagami
H,
Bonaldi
T, et al.
The HP1α–CAF1–SetDB1-containing complex provides H3K9me1 for
Suv39-mediated K9me3 in pericentric heterochromatin .
EMBO Rep.
2009; ; 10 :
:769.–775. 25.
Schultz
DC,
Ayyanathan
K,
Negorev
D, et al.
SETDB1: a novel KAP-1-associated histone H3, lysine 9-specific
methyltransferase that contributes to HP1-mediated silencing of euchromatic
genes by KRAB zinc-finger proteins . Genes
Dev.
2002; ; 16 :
:919.–932. 26.
Xiao
X,
Shi
X,
Fan
Y, et al.
The costimulatory receptor OX40 inhibits interleukin-17
expression through activation of repressive chromatin remodeling
pathways . Immunity.
2016; ; 44 :
:1271.–1283. 27.
Bulut-Karslioglu
A, De La
Rosa-Velázquez
IA,
Ramirez
F, et al.
Suv39h-dependent H3K9me3 marks intact retrotransposons and
silences LINE elements in mouse embryonic stem cells .
Mol Cell.
2014; ; 55 :
:277.–290. 28.
Karimi
MM,
Goyal
P,
Maksakova
IA, et al.
DNA methylation and SETDB1/H3K9me3 regulate predominantly
distinct sets of genes, retroelements, and chimeric transcripts in
mESCs . Cell Stem Cell.
2011; ; 8 :
:676.–687. 29.
Rebollo
R,
Karimi
MM,
Bilenky
M, et al.
Retrotransposon-induced heterochromatin spreading in the mouse
revealed by insertional polymorphisms . PLOS
Genet.
2011; ; 7 :
:e1002301.. 30.
Thompson
PJ,
Macfarlan
TS,
Lorincz
MC. Long terminal
repeats: from parasitic elements to building blocks of the transcriptional
regulatory repertoire . Mol Cell.
2016; ; 62 :
:766.–776. 31.
Rowe
HM,
Kapopoulou
A,
Corsinotti
A, et al.
TRIM28 repression of retrotransposon-based enhancers is necessary
to preserve transcriptional dynamics in embryonic stem
cells . Genome Res.
2013; ; 23 :
:452.–461. 32.
Ecco
G,
Cassano
M,
Kauzlaric
A, et al.
Transposable elements and their KRAB-ZFP controllers regulate
gene expression in adult tissues . Dev Cell.
2016; ; 36 :
:611.–623. 33.
Chuong
EB,
Elde
NC,
Feschotte
C. Regulatory
activities of transposable elements: from conflicts to
benefits . Nat Rev Genet.
2017; ; 18 :
:71.–86. 34.
Chuong
EB,
Elde
NC,
Feschotte
C. Regulatory
evolution of innate immunity through co-option of endogenous
retroviruses . Science.
2016; ; 351 :
:1083.–1087. 35.
Friedli
M,
Trono
D. The developmental
control of transposable elements and the evolution of higher
species . Annu Rev Cell Dev Biol.
2015; ; 31 :
:429.–451. 36.
Peaston
AE,
Evsikov
AV,
Graber
JH, et al.
Retrotransposons regulate host genes in mouse oocytes and
preimplantation embryos . Dev Cell.
2004; ; 7 :
:597.–606. 37.
Sundaram
V,
Cheng
Y,
Ma
Z, et al.
Widespread contribution of transposable elements to the
innovation of gene regulatory networks . Genome
Res.
2014; ; 24 :
:1963.–1976. 38.
Fridman
WH,
Pagès
F,
Sautès-Fridman
C,
Galon
J. The immune
contexture in human tumours: impact on clinical outcome .
Nat Rev Cancer.
2012; ; 12 :
:298.–306. 39.
Chikuma
S,
Suita
N,
Okazaki
IM, et al.
TRIM28 prevents autoinflammatory T cell development in
vivo . Nat Immunol.
2012; ; 13 :
:596.–603. 40.
Liu
B,
Tahk
S,
Yee
KM, et al.
The ligase PIAS1 restricts natural regulatory T cell
differentiation by epigenetic repression .
Science.
2010; ; 330 :
:521.–525. 41.
Martin
FJ,
Xu
Y,
Lohmann
F, et al.
KMT1E-mediated chromatin modifications at the FcgRIIb promoter
regulate thymocyte development . Genes Immun.
2015; ; 16 :
:162.–169. 42.
Santoni de Sio
FR,
Barde
I,
Offner
S, et al.
KAP1 regulates gene networks controlling T-cell development and
responsiveness . FASEB J.
2012; ; 26 :
:4561.–4575. 43.
Takikita
S,
Muo
R,
Takai
T, et al.
A histone methyltransferase ESET is critical for T cell
development . J Immunol.
2016; ; 197 :
:2269.–2279. 44.
Taniuchi
I,
Sunshine
MJ,
Festenstein
R,
Littman
DR. Evidence for
distinct CD4 silencer functions at different stages of thymocyte
differentiation . Mol Cell.
2002; ; 10 :
:1083.–1096. 45.
Zhou
ZF,
Yu
J,
Chang
M, et al.
TRIM28 mediates chromatin modifications at the TCR enhancer and
regulates the development of T and natural killer T cells .
Proc Natl Acad Sci USA.
2012; ; 109 :
:20083.–20089. |