| |
| Med Sci (Paris). 36(4): 329–331. doi: 10.1051/medsci/2020054.Une mutation ponctuelle dans la protéine Rrp9 de la
particule U3 entraîne un découplage des clivages précoces de l’ARN
pré-ribosomique Mathieu Rederstorff1* 1Université de Lorraine, CNRS, IMoPA,
F-54000Nancy,
France MeSH keywords: Animaux, Endoribonucleases, Humains, Microscopie électronique, Mutation ponctuelle, Précurseurs des ARN, Maturation post-transcriptionnelle des ARN, Stabilité de l'ARN, Petites ribonucléoprotéines nucléolaires, Saccharomyces cerevisiae, Facteurs temps, métabolisme, physiologie, génétique, ultrastructure |
Chez les eucaryotes, la biogenèse des ribosomes est un mécanisme complexe prenant place
dans le nucléole [1]
(→).
(→) Voir la Synthèse de L. Tafforeau, m/s n° 6-7, juin-juillet
2015, page 622
L’assemblage et la maturation des sous-unités ribosomiques 40S et 60S y suivent deux
routes indépendantes. Ces voies débutent néanmoins par la transcription, par l’ARN
polymérase I, d’un ARN ribosomique (ARNr) précurseur commun, le pré-ARNr 35S. Des séries
de clivages endo- et exo-nucléolytiques ordonnés permettent ensuite de libérer, à partir
de ce précurseur, les futurs ARNr matures : l’ARNr 18S, de la petite sous-unité
ribosomique, d’une part, et les ARNr 5.8S et 28S, d’autre part. Ces derniers, associés à
l’ARNr 5S, transcrit de façon indépendante par l’ARN polymérase III, constitueront la
grande sous-unité ribosomique. Les clivages précoces du précurseur 35S ont lieu au sein
d’un macro-complexe ribonucléoprotéique, le small subunit
(SSU)-processome, ou pré-ribosome 90S, à l’origine de la petite
sous-unité ribosomique 40S. Dans cette nouvelle, nous présentons les travaux récents
portant sur le SSU-processome, réalisés notamment chez la levure Saccharomyces
cerevisiae. Compte tenu de la conservation entre espèces et de
l’universalité des processus de maturation des ARNr, ces résultats n’en revêtent pas
moins un caractère crucial pour la compréhension de ce mécanisme dans l’espèce
humaine. |
Microscopie électronique : un bond immense dans l’étude du
« processome ». Les premières observations d’unités transcriptionnelles actives sur l’ADN ribosomique
ont été possibles grâce aux expériences d’« étalements » de Miller (Miller
spreads), dans les années 1960 [2] (Figure
1, en bas). Ces images de microscopie électronique « en sapin de
Noël » révélaient la présence, sur les transcrits naissants, de boules terminales,
dont la nature ne fut déterminée, en partie, que beaucoup plus tard : ces boules
correspondent au processome en cours d’assemblage (Figure 1). Le processome final, ou pré-ribosome
90S, constitué, entre autres, de la petite particule ribonucléoprotéique nucléolaire
(small nucleolar ribonucleoprotein particle, snoRNP) U3,
comprend l’intégralité du pré-ARNr 35S.
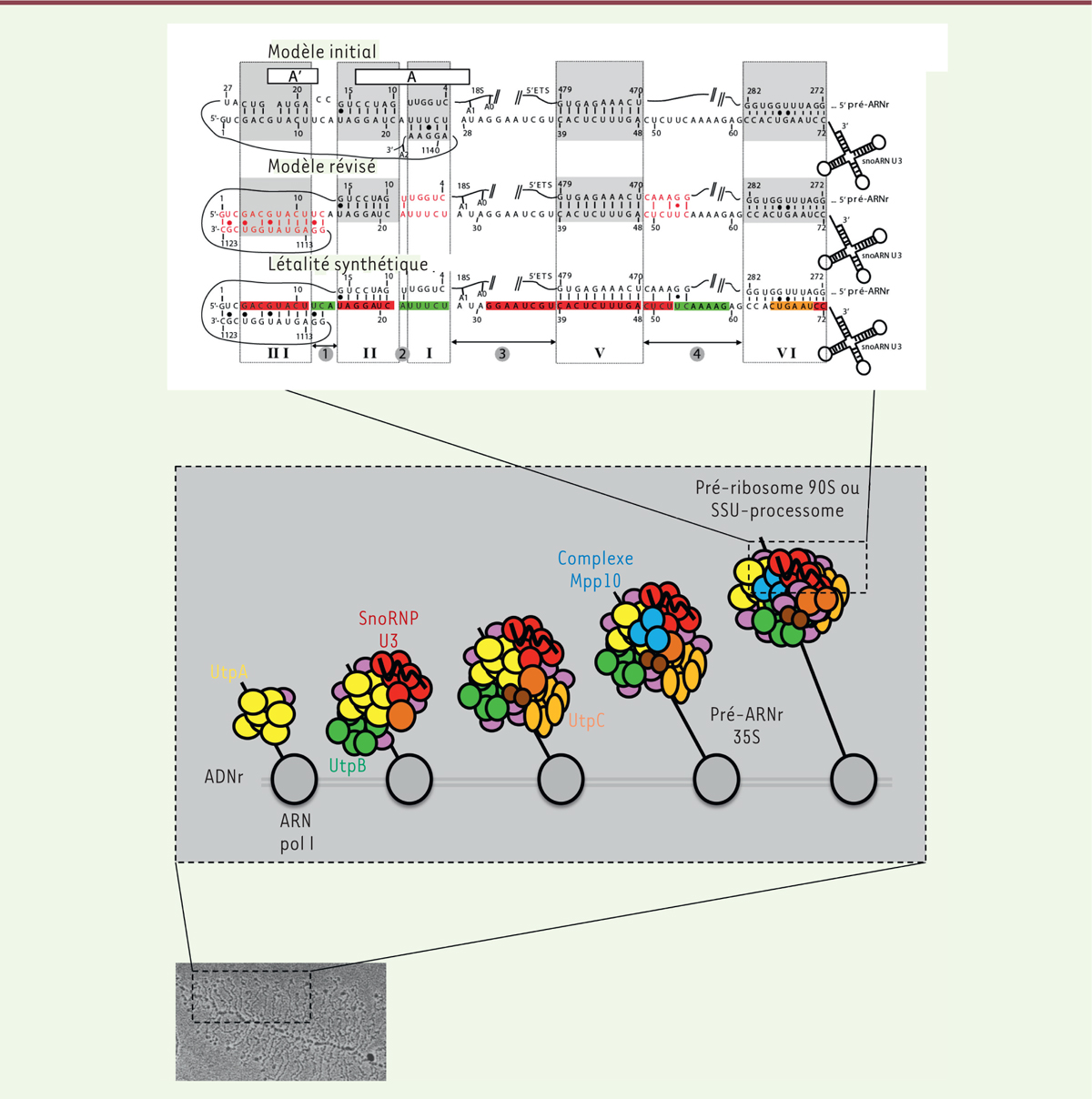
 | Figure 1.Assemblage séquentiel du « processome » et appariement
entre le pré-ARN ribosomique et le petit ARN nucléolaire
U3. (Bas) Visualisation d’un « étalement » de
Miller ( Miller spread) en microscopie électronique,
correspondant à une unité transcriptionnelle d’ADN ribosomique en cours
de transcription par l’ARN polymérase I (d’après [ 2]). Dans cette structure « en sapin de Noël », le
tronc correspond à l’ADN ribosomique, les branches aux transcrits 35S
naissants, de longueur croissante de gauche à droite en fonction de
l’avancement de la transcription, et les boules aux processomes en cours
d’assemblage. (Milieu) Représentation schématique des « boutons
terminaux » correspondant au processome en cours d’assemblage, sur les
transcrits naissants de longueurs croissantes. La chronologie d’arrivée
séquentielle des principaux sous-complexes du processome est illustrée
[ 5, 6]. (Haut)
Représentation de l’appariement entre le pré-ARN ribosomique et la
région 5’ fonctionnelle du petit ARN nucléolaire U3 (snoARN U3). Les
hélices intermoléculaires III, II, I, V et VI, ainsi que les segments
inter-hélices 1, 2, 3 et 4 sont représentés. Les régions 5’-ETS
( external transcribed spacer) et correspondant au
futur ARNr 18S, ainsi que les sites de coupure précoce A0, A1 et A2 sont
indiqués, tout comme les positions de certains nucléotides. Les
différences constatées entre le modèle initial et le modèle révisé sont
indiquées en rouge dans ce dernier [ 8, 9]. Enfin, le caractère synthétique
létal (rouge), synthétique malade (orange), ou l’absence d’effet
synthétique (vert) en présence du mutant de la protéine Rrp9 et des
mutations dans le snoARN U3 correspondant aux régions en question sont
illustrés dans le troisième appariement [ 10]. |
Ces dernières années, des progrès considérables ont été réalisés dans la
compréhension du mécanisme séquentiel d’assemblage du processome sur le pré-ARNr
naissant [3, 4] (Figure 1), et dans la détermination des structures
du complexe par cryo-microscopie électronique [4]. Plusieurs équipes ont notamment réussi à isoler, en utilisant des
méthodes de purification par affinité avec des transcrits de longueur croissante,
correspondant au long précurseur de l’ARNr 35S, les complexes s’associant de manière
ordonnée et séquentielle à ce précurseur [5,
6]. Une dernière étude a complété ces
travaux, et montré qu’en dépit de l’assemblage co-transcriptionnel des différents
constituants du processome sur le pré-ARNr 35S, les régions du pré-ARNr transcrites
en dernier (et non pas son extrémité 5’ terminale) étaient intégrées les premières
dans le processome [3]. Cela permet notamment
d’éviter l’assemblage de particules comportant un transcrit abortif ou dégradé. |
La génétique de la levure en complément de la microscopie électronique Le petit ARN nucléolaire (small nucleolar RNA, snoRNA) U3,
contrairement à la plupart des snoRNA à boîtes C/D [7] (→) n’est pas impliqué dans la
méthylation d’autres ARN. Autre particularité : il est associé à une protéine qui
lui est spécifique, la protéine Rrp9 chez la levure ou U3-55K chez l’homme. Enfin,
le snoRNA U3 possède, à son extrémité 5’, une extension qui n’existe pas chez les
autres membres de la famille des snoRNA à boîtes C/D, et qui permet son appariement
avec le pré-ARNr 35S (Figure
1, en haut). Au sein du processome, le snoRNP U3 est essentiel à la
réalisation des clivages précoces A0, A1 et A2 permettant de libérer, à partir du
précurseur 35S, l’ARNr 18S mature de la petite sous-unité ribosomique. En effet, sur
la base de comparaisons phylogéniques, de tests génétiques réalisés chez la levure,
ou encore de l’utilisation de sondes chimiques in vivo, la nature
de l’appariement entre le snoRNA U3 et le pré-ARNr 35S a pu être établie
(Figure 1, en haut, ancien
modèle). Cinq hélices intermoléculaires, séparées par quatre segments,
ont ainsi pu être identifiées, et nommées à partir de l’extrémité 5’ du snoRNA U3 :
hélices III, II, et I d’une part, appariées au futur ARNr 18S, et hélices V et VI
d’autre part, appariées à la partie 5’ ETS (external transcribed
spacer) du pré-ARNr. L’importance fonctionnelle des hélices V, VI et II
avait pu être montrée précédemment [8],
notamment grâce à des expériences de mutations compensatoires, mais ces expériences
n’avaient pas été concluantes pour les hélices I et III, et aucun test n’avait été
réalisé sur les segments inter-hélices. Récemment, l’obtention des structures du
processome par microscopie électronique a permis de confirmer l’existence des
hélices VI, V et II, mais pas celle de l’hélice I, et de proposer l’existence d’une
hélice III alternative (Figure 1, en
haut, modèle révisé) [9]. Cela
explique sans doute l’échec des précédentes tentatives d’étude des hélices I et
III.
(→) Voir la Synthèse de Y. Abel et al., m/s
n° 3, mars 2014, page 297
Néanmoins, une étude très récente a permis de faire la lumière sur certains aspects
de la formation des hélices intermoléculaires entre le snoRNA U3 et le pré-ARNr et
leur stabilisation. Cela a notamment été possible grâce à l’identification, chez la
levure S. cerevisiae, d’un mutant inédit de la protéine Rrp9, qui
entraîne un blocage et un découplage des coupures permettant de libérer l’ARNr 18S
[10]. En outre, un criblage synthétique
létal réalisé entre ce mutant et des mutants situés dans chaque hélice ou segment
dans la région 5’ fonctionnelle du snoRNA U3 a permis d’éclaircir le rôle de la
protéine Rrp9 dans la formation et la stabilisation de l’appariement avec le
pré-ARNr 35S, bien que le mécanisme mis en jeu soit probablement indirect
(Figure 1, en haut, létalité
synthétique) [10]. En effet,
cette étude a permis de préciser le réseau d’interactions de la protéine Rrp9 au
sein du processome, en identifiant de nouveaux partenaires de liaison de cette
protéine, Rrp36 et Sgd1. L’absence de structure tridimensionnelle connue pour ces
deux protéines ainsi que leur absence dans les structures de cryo-microscopie
électronique actuelles ont conduit à proposer un nouveau modèle localisant Rrp36 et
Sgd1 au voisinage de Rrp9, bien défini dans ces structures [10]. Ainsi, la puissance des techniques actuelles de cryo-microscopie électronique et la
précision des approches classiques de mutagenèse dirigée et de génétique chez la
levure ont permis de franchir une nouvelle étape dans la compréhension du mécanisme
d’assemblage de la petite sous-unité ribosomique. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
1.
Tafforeau
L
. Qu’en est-il de la biogenèse des ribosomes chez
l’homme ? . Med Sci (Paris).
2015; ; 31 :
:622.–628. 2.
Mougey
EB
,
O’Reilly
M
,
Osheim
Y
, et al.
The terminal balls characteristic of eukaryotic rRNA
transcription units in chromatin spreads are rRNA processing
complexes . Genes Dev.
1993; ; 7 :
:1609.–1619. 3.
Cheng
J
,
Baßler
J
,
Fischer
P
, et al.
Thermophile 90S pre-ribosome structures reveal the reverse order
of co-transcriptional 18S rRNA subdomain integration .
Mol Cell.
2019; ; 75 : (1256–69)
:e7.. 4.
Barandun
J
,
Hunziker
M
,
Klinge
S
. Assembly and structure of the SSU processome-a
nucleolar precursor of the small ribosomal subunit .
Curr Opin Struct Biol.
2018; ; 49 :
:85.–93. 5.
Chaker-Margot
M
,
Hunziker
M
,
Barandun
J
, et al.
Stage-specific assembly events of the 6-MDa small-subunit
processome initiate eukaryotic ribosome biogenesis .
Nat Struct Mol Biol.
2015; ; 22 :
:920.–923. 6.
Chen
W
,
Xie
Z
,
Yang
F
,
Ye
K
. Stepwise assembly of the earliest precursors of
large ribosomal subunits in yeast . Nucleic Acids
Res.
2017; ; 45 :
:6837.–6847. 7.
Abel
Y
,
Clerget
G
,
Bourguignon-Igel
V
, et al.
Les petits ARN nucléolaires nous surprennent encore
! . Med Sci (Paris).
2014; ; 30 :
:297.–302. 8.
Marmier-Gourrier
N
,
Cléry
A
,
Schlotter
F
, et al.
A second base pair interaction between U3 small nucleolar RNA and
the 5’-ETS region is required for early cleavage of the yeast pre-ribosomal
RNA . Nucleic Acids Res.
2011; ; 39 :
:9731.–9745. 9. Sun
Q,
Zhu
X,
Qi
J, et al.
Molecular architecture of the 90S small subunit
pre-ribosome . eLife.
2017;; 6. doi : 10.7554/eLife.22086.. 10. Clerget
G,
Bourguignon-Igel
V,
Marmier-Gourrier
N, et al.
Synergistic defects in pre-rRNA processing from mutations in the
U3-specific protein Rrp9 and U3 snoRNA . Nucleic
Acids Res.
2020;; 48 :
:3848.–68. |