| |
| Med Sci (Paris). 36(5): 504–514. doi: 10.1051/medsci/2020077.La levure modèle et outil… aussi pour la recherche
thérapeutique Stéphane Bach,1,2 Pierre Colas,1 and Marc Blondel3,4* 1Sorbonne Université, CNRS, UMR8227, Laboratoire de Biologie
Intégrative des Modèles Marins, Station Biologique de Roscoff,
place Georges Teissier,
29680Roscoff,
France 2Sorbonne Université, CNRS, FR2424, Plateforme de criblage KISSf,
Station Biologique de Roscoff, place Georges Teissier, 29680Roscoff,
France 3Univ Brest, Inserm, EFS, UMR1078, GGB, F-29200Brest,
France 4CHRU Brest, service de génétique clinique et de biologie de la
reproduction, F-29200Brest,
France |

Vignette (Brancolevure - Photo © Marie-Louise Jung). En 1991, se tenait à l’Institut Pasteur de Paris le premier colloque francophone « Levure
Modèle et Outil » (LMO) dont une des caractéristiques principales était de mêler, dans
un même congrès, ces deux utilisations de la levure : tant comme outil que comme modèle
biologique. Depuis, ces conférences sont organisées tous les deux à trois ans et se
tiennent maintenant en anglais, mais toujours avec ce même fil conducteur fondé sur sa
dualité d’utilisation, dont il est frappant de constater qu’elle est à l’origine même du
développement du modèle levure. En effet, les premières traces de l’utilisation
empirique par l’espèce humaine de la levure pour fermenter les sucres en alcool, ou
comme agent de levage pour la panification, remontent à plus de 5 000 ans. Une simple
visite dans la section égyptienne du musée du Louvre, dans laquelle des scènes de
brasserie ou encore des pains fossilisés sont exposés, suffit à s’en persuader.
Récemment les traces d’une brasserie de la période Natoufienne1 ont été découvertes en Israël, ce qui repousserait les premiers
signes de l’utilisation de la levure à plus de 13 000 ans [1]. Jusqu’aux travaux de Louis Pasteur, la théorie
de la génération spontanée, défendue principalement par des chimistes, prédominait.
Selon cette théorie, la fermentation était un processus chimique catalytique et, même si
différents naturalistes avaient observé au microscope et décrit la présence de
nombreuses levures associées à la fermentation alcoolique, l’hypothèse communément
admise était que ces levures n’étaient rien d’autre que des substances chimiques
complexes mais inertes, agissant comme des catalyseurs pour la conversion des sucres en
alcool. Si cette vision de la levure était partagée par les plus éminents chimistes de
l’époque, tels que l’allemand Justus von Liebig (1803-1873), dès 1837, une hypothèse
alternative, notamment défendue par Charles Cagniard-Latour (1777-1859), commençait déjà
à émerger : les levures seraient en fait des cellules vivantes se multipliant pendant le
processus de fermentation. Cette hypothèse reposait sur l’observation que la
fermentation commençait avec l’apparition des levures, progressait en parallèle de leur
multiplication et s’arrêtait en même temps que leur croissance [2]. C’est dans ce contexte que Louis Pasteur, alors
en poste à Lille où il avait été sollicité par des brasseurs locaux afin qu’il les aide
à comprendre et solutionner les difficultés régulières qu’ils rencontraient dans leurs
fermentations des sucres en alcool, commença à s’intéresser à cette question. En 1857,
il publia la première [3] d’une
série d’études qui non seulement confirmèrent que la levure correspondait effectivement
à un organisme vivant, mais également servirent de fondement à la microbiologie et à la
pasteurisation. Dès lors, il devint possible, sur des bases rationnelles et
scientifiques, d’optimiser le processus de fermentation, par exemple en éliminant les
bactéries contaminantes, mais aussi en sélectionnant et modifiant les souches de levure
elles-mêmes. L’utilisation de la levure comme modèle pouvait alors commencer à se
développer, sur la base même de son utilisation comme outil agroalimentaire. À ce stade, une précision sémantique est nécessaire. En effet, la plupart des
biologistes, cédant à l’abus de langage courant, nomment « levure » la levure de
boulanger (ou de brasseur) Saccharomyces cerevisiae. En réalité, il
existe de très nombreuses espèces de levures qui sont des champignons unicellulaires.
Citons par exemple une autre levure très utilisée comme organisme modèle (et aussi pour
la fermentation alcoolique, notamment en Afrique) : la levure de fission
Schizosaccharomyces pombe qui est ainsi parfois appelée « l’autre
levure ». La levure S. cerevisiae, tout comme l’autre levure S.
pombe, est un organisme unicellulaire eucaryote. Malgré sa simplicité (elle
ne contient qu’environ 6 200 gènes et l’épissage alternatif y est peu développé, ce qui
limite également son répertoire protéique [4]), elle n’en représente pas moins un prototype de cellule
eucaryote dans lequel sont présents la plupart des grands mécanismes régissant la vie
mais aussi la mort [5] des
cellules des eucaryotes supérieurs. Cet aspect essentiel, combiné à la grande facilité
de manipulation de cet organisme (la levure est un eucaryote aussi facile à manipuler
qu’une bactérie prototype telle qu’Escherichia coli), a conduit à son
essor comme l’un des organismes modèles eucaryotes majeurs. S.
cerevisiae fut ainsi le premier eucaryote dont le génome a été
intégralement séquencé et la divulgation de cette séquence, en 1996, a permis le
développement de la plupart des techniques globales (de « -omique ») largement utilisées
chez la levure, et dont la plupart des résultats sont accessibles librement2. La levure a ainsi permis des découvertes et
avancées majeures dans la compréhension du fonctionnement général de la cellule
eucaryote, comme en atteste la présence régulière de « levuristes » dans la liste des
récipiendaires des prix Nobel (en particulier de physiologie ou médecine) : citons, par
exemple, Roger Kornberg, prix Nobel de chimie 2006 pour ses travaux sur les bases
moléculaires de la transcription chez la levure [6] ; Paul Nurse et Lee Hartwell, co-récipiendaires du prix Nobel de
physiologie ou médecine 2001 pour la découverte des complexes CDK
(cyclin-dependent kinase)/cyclines, les régulateurs centraux du
cycle cellulaire eucaryote, respectivement chez S. pombe et S.
cerevisiae [7] ; ou,
plus récemment, Randy Schekman et Yoshinoro Ohsumi, tous deux lauréats du prix Nobel de
physiologie ou médecine, en 2013 pour le premier et 2016 pour le second, pour leurs
travaux séminaux, respectivement sur le trafic vésiculaire [8] et sur les mécanismes de l’autophagie [9]. Une autre illustration
spectaculaire de cette forte conservation de la levure à l’homme des mécanismes de
biologie cellulaire et de leurs acteurs clés provient de la tentative de remplacer de
façon systématique une partie (414) des quelques 2 000 gènes essentiels de levure, par
leurs homologues humains : près de la moitié de ces 414 gènes essentiels de levure
peuvent ainsi être « humanisés », et ce pourcentage est même encore plus important pour
certains complexes tels que le protéasome 26S pour lequel 21 des 28 sous-unités humaines
testées se sont avérées capables de remplacer leur équivalent chez la levure [10]. Suite à la publication, en
1996, du génome de la levure S. cerevisiae, il avait été estimé qu’au
moins 30 % des gènes humains impliqués dans des maladies présentaient un homologue
fonctionnel chez la levure [11]. Toutes ces observations ont rapidement conduit à considérer que la levure,
notamment S. cerevisiae, pouvait représenter à la fois un modèle et un
outil alternatifs et pertinents pour la recherche thérapeutique. |
La levure : un modèle pour la recherche thérapeutique La forte conservation des mécanismes cellulaires de la levure à l’espèce humaine,
ainsi que la capacité de nombreux gènes humains à complémenter la fonction de gènes
de levure homologues mutés, ont rapidement été exploitées. En effet, la
fonctionnalité, par rapport à l’allèle sauvage, de mutations suspectées être la
cause de différentes maladies humaines peut être déterminée en réalisant des tests
de complémentation dans des levures déficientes pour le gène correspondant
(Figure 1). Parmi les
premiers exemples, se trouve l’analyse de mutations du gène CBS
humain codant la cystathionine bêta-synthase et responsables d’une maladie
métabolique rare (l’homocystinurie, également facteur de risque pour des
athéroscléroses prématurées). Cette étude a été effectuée dans une souche de levure
dont le gène CYS4, l’homologue de CBS, est
inactivé [12]. Dans la
lignée de cette méthode, l’une des premières utilisations de la levure à des fins de
diagnostic médical est le test FASAY (functional analysis
of separated alleles in yeast) qui vise à déterminer le degré de
fonctionnalité des très nombreux allèles mutants du gène suppresseur de tumeur
P53 [13]. Une adaptation de ce test a récemment été développée afin de
déterminer la dominance éventuelle des allèles mutés de P53 les
plus couramment retrouvés dans les cancers (mutants hotspot), ainsi
que de certaines des isoformes principales de p53 et de deux protéines apparentées,
p63 et p73. Les résultats ont suggéré que la plupart de ces mutants et certaines de
ces isoformes étaient dominants, ce qui expliquerait le maintien d’hétérozygotie
observé pour le locus P53 chez de nombreux patients porteurs de ces
mutations hotspot [14]. La levure a également servi de modèle pour des aspects
inattendus de la biologie des eucaryotes supérieurs, tels que les prions ou le
vieillissement.
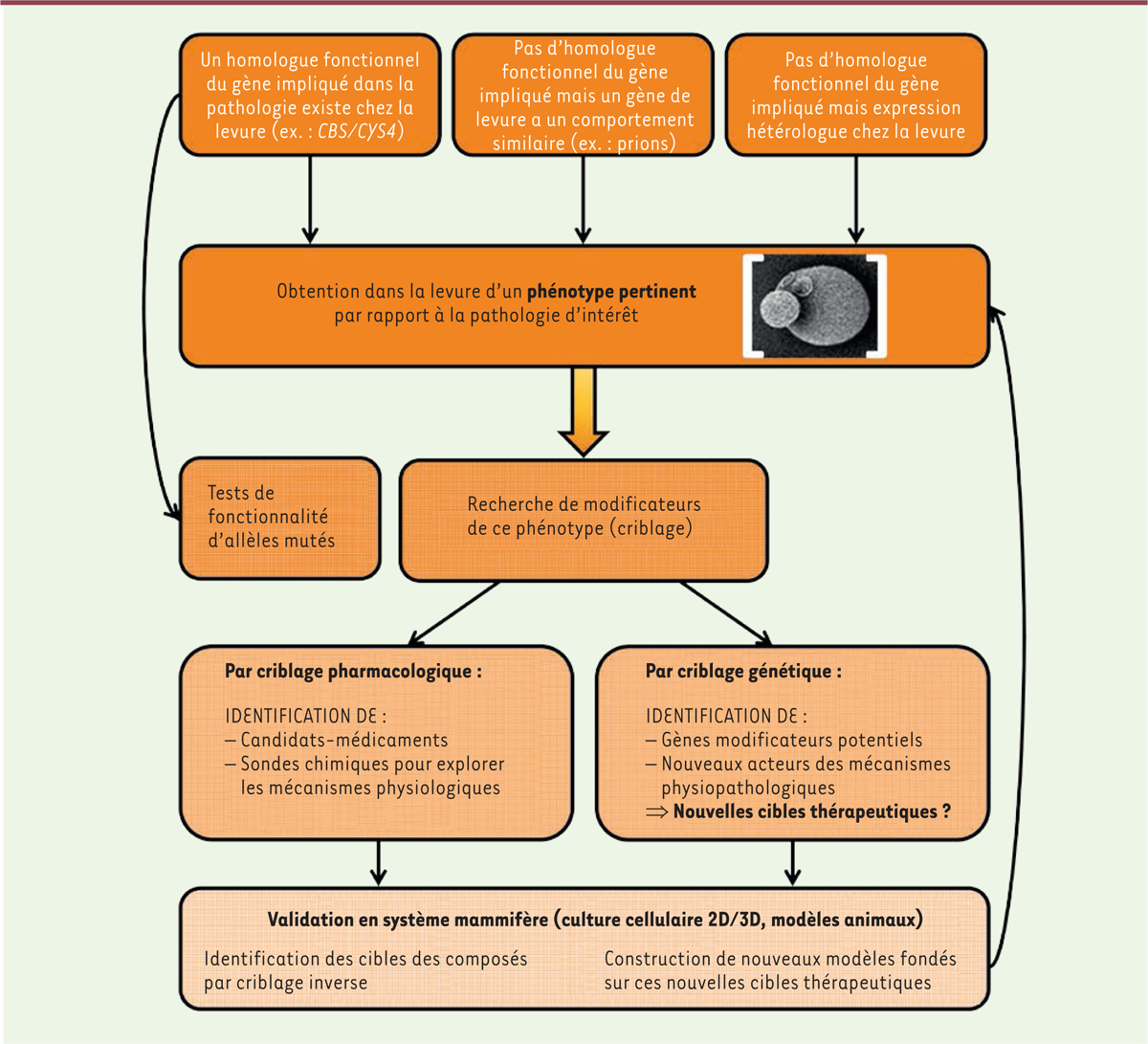
 | Figure 1. Vue d’ensemble de l’utilisation de la levure comme modèle
de maladies humaines. Le point crucial est
d’obtenir un modèle levure présentant un phénotype pertinent par rapport
à la pathologie d’intérêt. Des modificateurs de ce phénotype peuvent
ensuite être recherchés par des criblages tant pharmacologiques que
génétiques. |
Les prions ont, en fin de compte, été initialement découverts chez la levure, par des
expériences de génétique classique réalisées dans les années 1960-1970 (donc
longtemps avant la connaissance de la séquence des gènes et des protéines
concernées, et surtout bien avant la définition même du concept prion de protéine
infectieuse !), et astucieusement interprétées [15, 16]. Les résultats de ces expériences ont ensuite été revisités
par Reed Wickner qui, peu de temps après l’émergence du concept prion, suivant une
intuition brillante, a démontré que ces éléments « bizarres » d’hérédité
cytoplasmique non mendélienne chez la levure correspondaient en fait à des prions
[17]. Un nouveau champ
d’étude extrêmement actif fondé sur la levure pour étudier la biologie des prions et
élucider ses mystères, venait ainsi d’être créé. Les apports majeurs à mettre au
crédit de ces études ont été la découverte du rôle crucial de chaperons de protéines
dans le maintien et la propagation des prions [18], ou encore parmi les premières
démonstrations formelles de l’hypothèse « protéine seule » à la base du modèle prion
[19, 20]. S’agissant du vieillissement, il a
longtemps été perçu comme spécifique des organismes pluricellulaires avant qu’il ne
soit démontré que ce phénomène concernait également les microorganismes
unicellulaires, comme les bactéries ou la levure S. cerevisiae,
cette dernière présentant clairement des éléments génétiques et physiologiques de
contrôle du vieillissement communs avec les eucaryotes supérieurs, tels que
l’accumulation de cercles d’ADN ribosomique ou l’effet antivieillissement de la
restriction calorique [21]. Dans la lignée des tests d’analyse fonctionnelle, des modèles et des outils
chémobiologiques fondés sur l’utilisation de la levure (principalement S.
cerevisiae, mais aussi S. pombe) et permettant des
approches thérapeutiques innovantes, voient régulièrement le jour depuis une
vingtaine d’années. Ces approches reposent le plus souvent sur la mise au point d’un
modèle levure présentant un phénotype pertinent pour une pathologie considérée
(Figure 1). Pour ce
type de modélisation, différents cas de figure sont envisageables : du plus simple
et direct (existence d’un homologue fonctionnel), au plus artificiel (expression
hétérologue d’un gène humain ou de pathogène sans homologue chez la levure), en
passant par des situations intermédiaires (dans lesquelles un gène de levure sert de
prototype). Ce modèle peut alors servir pour différents criblages phénotypiques afin
d’identifier des modificateurs capables de supprimer ou d’exacerber le phénotype
préalablement obtenu. Ces modificateurs peuvent être n’importe quels objets
biologiques (gènes, anticorps, etc.) ou chimiques (petites molécules,
oligonucléotides, etc.) qui pourront représenter des pistes pour développer des
candidats-médicaments et/ou des outils originaux pour sonder des mécanismes
biologiques et physiopathologiques nouveaux ou mal connus. Dans le cas de criblages
pharmacologiques, puisqu’il s’agit de criblages phénotypiques, des criblages
« inverses » nécessiteront d’être réalisés, afin d’identifier les cibles cellulaires
des composés actifs isolés. Ils permettront non seulement d’appréhender le mécanisme
d’action de la « touche » (ou hit en anglais) obtenue, mais aussi
ses effets secondaires potentiels. Il existe de nombreuses façons de réaliser ces
criblages inverses, notamment par des méthodes biochimiques, telles que la
chromatographie d’affinité sur drogues immobilisées [22, 23]. Des méthodes génétiques utilisant la levure elle-même,
telles que les cribles d’haplo-insuffisance3,,
de surexpression ou encore de létalité synthétique4 (pour revue voir [24]), ainsi que le triple-hybride (voir plus loin), ont également été
développées (Figure 2). Ces
méthodes sont toutes automatisables et exploitent la puissance génétique du modèle
levure. Ainsi, outre le fait qu’elles offrent la possibilité d’identifier
d’éventuels candidats-médicaments, les approches de chémobiologie qui utilisent la
levure peuvent également permettre la découverte de leurs cibles cellulaires, et
donc potentiellement définir de nouvelles cibles thérapeutiques pour traiter les
maladies correspondantes.
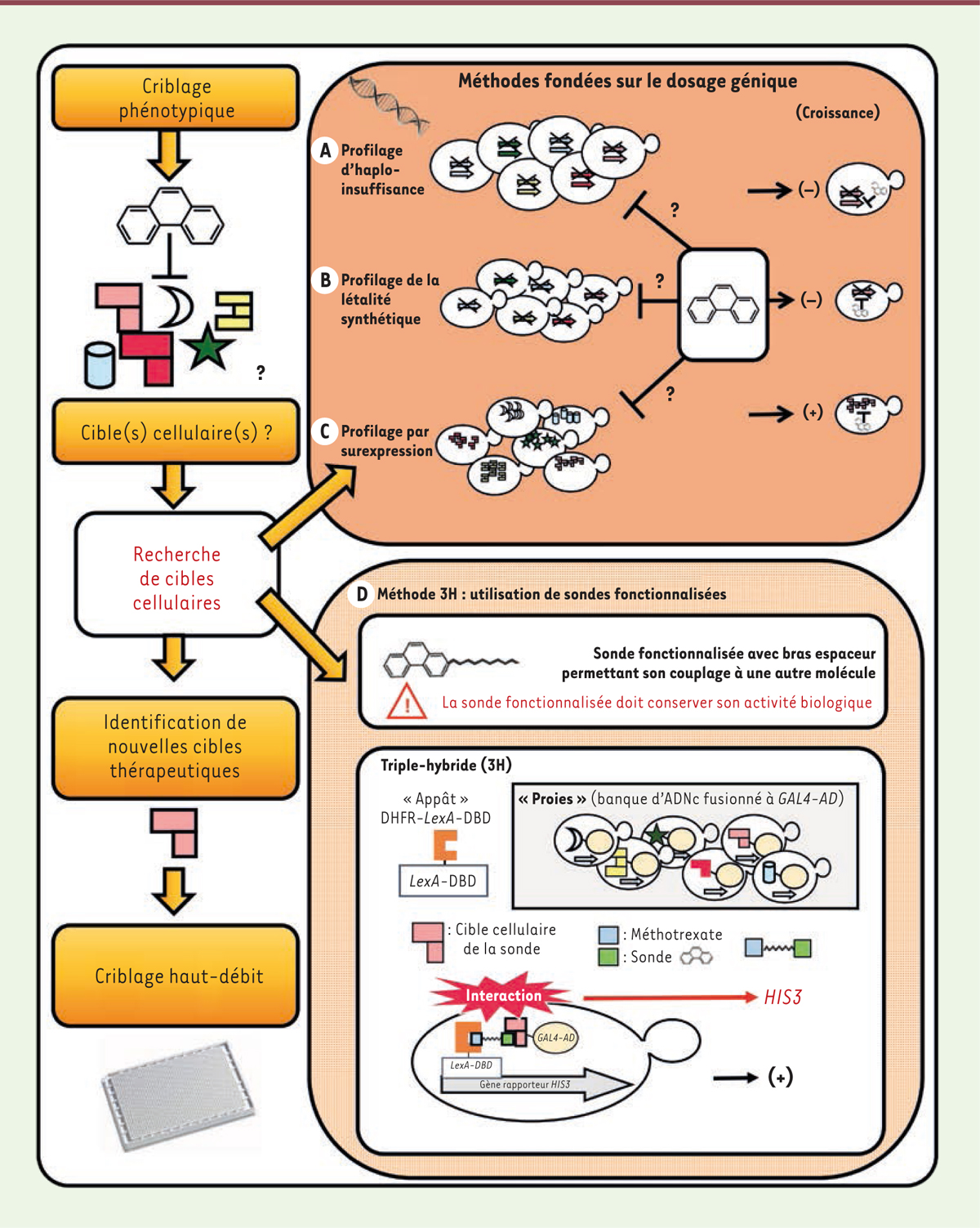
 | Figure 2. Méthodes fondées sur la levure d’identification des cibles
de composés (sondes) isolés dans des criblages pharmacologiques
phénotypiques. Quatre méthodes génétiques de
profilage de composés actifs sont présentées dans cette figure.
A. Profilage de souches diploïdes
haplo-insuffisantes chacune pour un des ~ 2 000 gènes de levure qui sont
essentiels (donc pour lesquelles le dosage génique pour le gène
considéré est d’environ 50 %). Cette méthode permet de repérer les gènes
essentiels dont l’haplo-insuffisance conduit à une hypersensibilité au
composé étudié, donc codant probablement des cibles de ce dernier.
B. Profilage de souches haploïdes
chacune délétée d’un des ~ 4 000 gènes de levure non-essentiels (donc
pour lesquelles le dosage génique pour le gène considéré est de 0 %).
Cette méthode permet l’identification de tous les gènes non-essentiels
dont l’inactivation totale conduit à une sensibilité accrue au composé
d’intérêt, donc potentiellement impliqués dans la résistance à ce
dernier. C. Profilage de souches de levure
surexprimant chacune un des ~ 6 000 gènes de levure (donc pour
lesquelles le dosage génique pour le gène considéré est >100 %).
Cette méthode permet d’identifier tous les gènes dont la surexpression
confère une résistance au composé étudié et donc codant des cibles
potentielles de ce dernier. Il est important de noter que ces trois
méthodes sont fondées sur la toxicité générale de la molécule considérée
pour la levure (puisque le paramètre mesuré est l’effet du composé sur
la croissance des souches de levures), et donc sur l’inférence que son
effet toxique implique les mêmes cibles cellulaires que sa capacité à
modifier le phénotype pathologique, ce qui n’est pas nécessairement le
cas. D. L’approche triple-hybride (3H) est
fondée sur la création d’une chimère entre la sonde étudiée et une autre
petite molécule dont la cible est connue (par exemple l’interaction
méthotrexate/dihydrofolate réductase, DHFR). Une étude
structure-activité préalable est nécessaire afin de contrôler si la
sonde fonctionnalisée est toujours biologiquement active. L’approche
triple-hybride est fondée sur l’utilisation de banques de levure
exprimant des fusions protéiques entre un domaine d’activation de la
transcription (AD) et les protéines cibles à tester d’une part, et,
d’autre part, la fusion entre la DHFR et un domaine de liaison à l’ADN
(DBD) d’un facteur de transcription. Les levures sont ensuite traitées
avec la sonde fonctionnalisée. Seules les levures exprimant la cible de
la sonde pourront se développer sur un milieu dépourvu d’histidine (+),
dans cet exemple de gène rapporteur. |
|
Parmi les tout premiers exemples de criblages pharmacologiques à visée thérapeutique
pour des maladies humaines s’appuyant sur la levure, se trouve une méthode de
criblage de molécules antiprion [25, 26].
L’hypothèse à l’origine de cette approche était que les mécanismes de
« prionisation » pouvaient être conservés de la levure à l’homme. Ce pari s’est
avéré validé puisque les petites molécules actives contre les prions de levure
[PSI+
] et [URE2] [61]
(→) dont cette méthode a permis l’identification, se sont par la
suite révélées actives contre le prion de mammifère in cellulo
[25] mais également in
vivo dans un modèle murin de maladies à prion [27, 28]. Ainsi, non seulement ce modèle fondé sur les deux prions
les plus étudiés de la levure permettait d’isoler des molécules actives contre le
prion de mammifère, mais il apportait en plus la preuve qu’au moins certains des
mécanismes de « prionisation » étaient conservés de la levure à l’homme. Ces
mécanismes conservés ont ensuite été identifiés par une approche de criblage inverse
qui a montré que le domaine V du grand ARN ribosomique (ARNr 25S chez la levure, 28S
chez l’homme) possédait une activité chaperon de protéine, ciblée par les molécules
antiprions isolées chez la levure, et qui est impliquée dans le maintien et la
propagation des prions, au moins chez la levure [23, 29].
(→) Voir la Synthèse de Cécile Voisset et Marc Blondel, m/s
n° 12, décembre 2014, page 1161
|
Autres maladies neurologiques Un certain nombre de modèles levure ont été développés pour d’autres maladies
neurodégénératives, telles que les maladies de Parkinson [30] ou de Huntington [31]. Dans ces deux exemples, respectivement
l’α-synucléine et la huntingtine mutée, deux protéines humaines dépourvues
d’homologues chez S. cerevisiae, ont été surexprimées chez la
levure dans laquelle elles s’agrègent et induisent une toxicité qui emprunte des
mécanismes similaires à celle observée dans les neurones de patients. Ces modèles
ont, là encore, permis d’identifier des molécules actives dans des modèles animaux
pour ces pathologies chroniques, validant une fois de plus ces approches de
chémobiologie utilisant la levure. Ils ont également permis de découvrir de
nouvelles voies, et donc de nouvelles cibles thérapeutiques, le plus souvent
insoupçonnées, pour combattre ces maladies pour lesquelles peu ou pas de traitements
efficaces n’ont encore été découverts. Ainsi, un criblage génétique réalisé en
utilisant un modèle levure de maladie de Huntington a identifié la kynurénine
2,3-monooxygénase, une enzyme conservée de la levure à l’homme, dont l’inactivation
contrecarre la toxicité de la huntingtine mutée, tant chez la levure que dans des
cellules humaines ou dans des modèles animaux [31]. Plus récemment, un modèle levure pour le retard mental lié à la
trisomie 21 a été créé. Ce modèle est fondé sur la surexpression du gène
CBS, qui fait partie, avec DYRK1a
(dual specificity tyrosine phosphorylation regulated kinase
1A)5, des principaux gènes du
chromosome 21 dont la surexpression, du fait de la triplication de ce chromosome,
serait responsable du déficit cognitif observé chez les patients trisomiques. Il a
permis d’identifier le disulfirame, un principe actif médicamenteux déjà administré
depuis des décennies pour le traitement de l’alcoolisme, qui s’est avéré capable de
supprimer le phénotype lié à la surexpression de CBS chez la
levure, mais également les troubles cognitifs de souris modèles pour la trisomie 21
[32], faisant de ce
composé un candidat à un repositionnement thérapeutique pour améliorer les troubles
cognitifs des patients trisomiques. |
Des modèles levures pour différentes maladies métaboliques d’origine génétique, en
particulier mitochondriales, ont également été développés avec succès chez
S. cerevisiae mais aussi chez S. pombe [33-35]. La levure S. cerevisiae
s’avère en effet particulièrement pertinente pour modéliser les maladies
mitochondriales liées à des mutations dans l’ADN de cet organite, en raison de la
possibilité de réaliser de la mutagénèse dirigée sur le génome mitochondrial du
champignon [36]. Des
approches de criblage inverse ont, là encore, permis d’identifier les cibles de
certaines molécules potentiellement actives contre ces maladies [37]. |
Infections virales et parasitaires La levure a également été très utilisée pour explorer le monde des virus et leur
impact sur les espèces animales et végétales (pour revue, voir [38]), en particulier dans le domaine
des cancers viro-induits, notamment liés à l’oncovirus d’Epstein-Barr (EBV) (pour
revue, voir [39]). Ainsi a
été développé un modèle levure reproduisant le mécanisme qui permet à l’EBV
d’échapper au système immunitaire [40, 41]. Ce
modèle a non seulement permis d’isoler, grâce à un criblage pharmacologique, des
molécules candidates permettant au système immunitaire de cibler les cancers liés à
ce virus [41], mais également, grâce à un
criblage génétique, des gènes de la cellule infectée impliqués dans la furtivité
d’EBV au système immunitaire [42]. De la sorte, a été identifiée la nucléoline, une protéine humaine
très conservée chez la levure, qui représente une nouvelle cible moléculaire
potentielle pour le traitement des cancers liés à l’EBV. Cette cible a elle-même
permis d’isoler de nouvelles molécules actives [42, 43]. Un autre type d’approche très fructueuse fait appel à l’expression de cibles
thérapeutiques (humaines ou parasitaires par exemple) dans une souche de levure
délétée du gène homologue et complémentée par le (ou les) gène(s) hétérologue(s).
Citons pour exemple l’utilisation d’une souche de levure délétée de son gène
AUR1 (aureobasidin resistance) codant
l’inositol phosphorylcéramide synthase (IPS) et complémentée par l’expression du
gène homologue LmjIPBCS des parasites du genre
Leishmania, responsables de la leishmaniose, ou, comme
contrôle, par le gène AUR1 lui-même. Des composés affectant
spécifiquement l’enzyme parasitaire, donc a priori moins
susceptible de présenter des effets secondaires chez les patients, ont ainsi été
isolés [44]. Une telle
approche avait déjà été utilisée pour rechercher des inhibiteurs affectant
spécifiquement l’une ou l’autre des kinases dépendant des cyclines, CDK1 ou CDK2,
humaines, en tirant profit du fait que chacune d’entre elle est capable de
complémenter l’absence de croissance d’une souche de levure délétée pour le gène
CDC28 qui code la principale CDK de la levure S.
cerevisiae [45]. Notons que de telles approches fondées sur la complémentation d’un
mutant de levure par l’un ou l’autre gène homologue de parasites ou humain peuvent
être multiplexées, de façon à identifier, en un seul criblage, des molécules
spécifiquement actives sur la cible d’intérêt, tout en ne présentant pas (ou peu)
d’activité sur un gène proche dont l’inactivation pourrait induire des effets
secondaires qui limiteraient l’application thérapeutique de tels composés. Tous ces exemples ne représentent qu’une fraction minime de toutes les utilisations
réalisées ou possibles de la levure comme modèle pour des maladies humaines. Ils
illustrent néanmoins la polyvalence de tels modèles et leur intérêt, dès lors qu’ils
présentent un phénotype pertinent par rapport à la pathologie étudiée : le fait de
pouvoir réaliser, le plus souvent en parallèle, des criblages pharmacologiques et
génétiques qui se nourrissent mutuellement. Il faut toutefois garder en tête les
limites de la levure qui ne représente qu’un modèle eucaryote simplifié (notamment
du fait de son unicellularité). Il est ainsi nécessaire de valider tous les
modificateurs de phénotypes identifiés dans des systèmes eucaryotes supérieurs, tels
que des cultures en deux ou trois dimensions de cellules de mammifères, ou dans des
modèles animaux, lorsque ceux-ci existent. Enfin, la possibilité de pouvoir
réaliser, relativement aisément, des criblages inverses (notamment par des approches
génétiques utilisant la levure) pour identifier les cibles des molécules actives,
représente un autre argument fort en faveur d’approches de chémobiologie intégrée
fondées sur la levure (Figure
2). |
La levure : un outil pour la recherche thérapeutique Si la levure offre des modèles pertinents pour de nombreuses pathologies humaines,
elle est également très utilisée en tant qu’outil au service de la recherche
thérapeutique. Elle est couramment exploitée pour la production de molécules
d’intérêt, que ce soient des protéines recombinantes ou différentes molécules
d’intérêt prophylactique ou thérapeutique, telles que des vaccins, l’hormone de
croissance humaine ou encore l’hydrocortisone. La production de cette dernière
molécule d’intérêt majeur pour l’industrie pharmaceutique mais très compliquée à
synthétiser, dans une souche de levure S. cerevisiae multi-modifiée
génétiquement et à partir d’un substrat peu onéreux (le glucose ou l’éthanol),
représente un magnifique et spectaculaire exemple de l’incroyable polyvalence de la
levure [46]. Dans le
domaine des vaccins, non seulement la levure est utilisée depuis longtemps pour la
production de protéines antigéniques, mais elle est également, depuis peu, utilisée
comme adjuvant du fait de la forte immunogénicité de certains sucres de sa paroi,
absents chez les mammifères. Certaines approches très récentes visent ainsi à
combiner les deux avantages de la levure (polyvalence pour la production de
protéines recombinantes antigéniques et immunogénicité inhérente de sa paroi) en
l’utilisant directement (sous forme vivante ou inactivée par chauffage) comme vaccin
pour un usage, pour le moment, uniquement vétérinaire (pour revue, voir [47]). Toutes ces approches
dans lesquelles la levure est utilisée comme « usine » de production de molécules
thérapeutiques sont d’autant plus facilitées que la levure appartient à la catégorie
des organismes inoffensifs, dits GRAS (generally recognized as
safe), un concept créé en 1958 par la FDA (food and drug
administration) américaine [48]. Enfin, notons que l’utilisation de la levure en tant
qu’outil a régulièrement été enrichie par les découvertes fondamentales obtenues
grâce à son utilisation en tant que modèle. C’est, par exemple, le cas pour la
production de protéines recombinantes hétérologues dont le rendement peut être
grandement amélioré par des mutations altérant le ratio entre les deux sous-unités
du ribosome [49]. |
La levure : un outil pour la recherche sur les interactions
protéine-protéine Terminons par l’un des principaux apports de la levure en tant qu’outil pour l’étude
des interactions protéine-protéine (IPP), notamment grâce aux méthodes dites de
double-hybride ou de bioluminescence. Les fondements de la méthode double-hybride
ont été établis en 1989 par la démonstration qu’une IPP pouvait être détectée en
co-exprimant dans la levure deux protéines chimériques (ou hybrides), obtenues en
fusionnant aux deux partenaires participant à l’interaction, des modules impliqués
dans la transcription génique : un domaine de liaison à l’ADN, pour « l’appât », et
un domaine activateur de transcription, pour « la proie ». L’interaction entre les
deux partenaires protéiques reconstitue alors un facteur de transcription actif,
capable de transcrire un gène rapporteur conférant à la levure un phénotype
facilement détectable [50].
La méthode double-hybride a rapidement connu un succès et des évolutions
considérables. Elle représente encore aujourd’hui une approche de premier choix pour
découvrir et caractériser des interactions protéiques. Il n’est pas exagéré de
considérer que le double-hybride compte parmi les méthodes ayant produit le plus
fort impact sur la biologie de l’ère génomique et post-génomique [51]. La méthode double-hybride
transcriptionnelle et ses nombreuses évolutions offrent de nombreuses applications à
la recherche thérapeutique (Figure
3) (pour revue, voir [52]). La découverte facilitée de partenaires d’interaction de
protéines, dont le dysfonctionnement provoque des pathologies, permet notamment
d’identifier de nouvelles cibles thérapeutiques potentielles, qu’il s’agisse du
partenaire de l’interaction ou de l’interaction elle-même. Des ligands protéiques
combinatoires (tels que des aptamères peptidiques), sélectionnés par double-hybride
pour leur capacité à reconnaître spécifiquement une protéine cible donnée,
représentent des outils de choix permettant de valider des cibles thérapeutiques.
Ils introduisent en effet des perturbations similaires à celles produites par des
petites molécules thérapeutiques, contrairement aux approches de génétique inverse
plus communément utilisées. Ces molécules combinatoires peuvent également guider la
découverte de petites molécules bioactives [53].
 | Figure 3. Le double-hybride (2H) et ses évolutions au service de la
recherche thérapeutique. A. Représentation
schématique du 2H classique qui permet de découvrir de nouvelles
interactions protéine-protéine (IPP) par criblages systématiques de
banques d’ADN complémentaire (ADNc) (DBD : domaine de
liaison à l’ADN ; AD : domaine d’activation de la
transcription). B. Le 2H permet
d’identifier des aptamères peptidiques (AP) interagissant spécifiquement
avec une protéine d’intérêt, qui permettent parfois de la valider en
tant que cible thérapeutique. C. Le 2H
permet également de rechercher des inhibiteurs d’IPP sur la base de leur
capacité à empêcher la formation du complexe 2H.
D. le triple-hybride (3H) permet
d’identifier des cibles potentielles d’un composé actif.
E. L’edgotypage,
qui est souvent basé sur le 2H, permet, au sein d’un réseau d’IPP,
l’identification des interactions (edges)
spécifiquement affectées par des mutations pathogènes. |
De nombreuses variantes de la méthode double-hybride transcriptionnelle ont permis le
développement d’essais de criblage à haut débit dans le but de découvrir des petites
molécules inhibitrices d’IPP. Ces variantes portent essentiellement sur la mise en
œuvre de gènes rapporteurs différents, codant, par exemple, des luciférases, qui
permettent de suivre les levures dans lesquelles a eu lieu l’interaction, ou des
protéines toxiques, ce qui permet une sélection positive des inhibiteurs d’IPP (pour
revue, voir [52]). Des méthodes de type
« triple-hybride » ont été développées afin d’identifier les cibles de petites
molécules bioactives (voir la méthode de criblage inverse présentée sur la
Figure 2
). Le principe consiste à coupler de façon covalente la molécule d’intérêt
à une autre molécule capable de reconnaître une protéine « appât ». Des banques
d’ADN complémentaires, exprimés comme « proies », sont alors criblées afin
d’identifier les cibles potentielles du composé exposé par l’« appât » [52]. L’approche double-hybride s’avère également très puissante pour caractériser des
formes mutantes de protéines associées à des pathologies humaines. Ainsi a récemment
été développé l’« edgotypage », une approche qui consiste à
déterminer la capacité d’une protéine mutée à interagir, ou non, avec l’ensemble des
partenaires d’interaction connus pour la forme sauvage de la protéine. La pertinence
de cette approche repose sur le fait que : 1) plus de la moitié des mutations
responsables de pathologies mendéliennes sont des mutations faux-sens (provoquant
des substitutions d’acides aminés), et une grande proportion d’autres types de
mutations peuvent permettre l’expression de formes protéiques tronquées ; 2)
différentes mutations affectant un gène donné peuvent produire des effets
pathologiques très différents. Une étude magistrale faisant appel à la méthode
double-hybride a été menée sur près de 3 000 formes mutantes faux-sens concernant
plus de 1 100 gènes associés à des pathologies mendéliennes variées. Les deux tiers
de ces mutations ont présenté un edgotype perturbé (c’est-à-dire
présentant des altérations d’interaction avec les partenaires de la forme sauvage),
de façon seulement partielle pour la moitié d’entre elles [54]. De telles analyses permettent ainsi de
mieux comprendre les effets biologiques de certaines mutations, ce qui pourrait
conduire à une meilleure prise en charge des patients concernés. L’utilisation du BRET (transfert d’énergie de bioluminescence par résonance) a
également été développée chez la levure pour y étudier des IPP. Le BRET dérive des
méthodes fondées sur le transfert de fluorescence (appelées FRET) initiées par
Theodor Förster en 1946. Il s’agit d’un phénomène de bioluminescence naturelle,
telle que celle produite par la pensée de mer (Renilla
reniformis)6. L’identification
puis le clonage des acteurs moléculaires à l’origine de cette luminescence (tels que
l’enzyme donneuse d’énergie, Renilla Luciférase, RLuc, et la Green
Fluorescent Protein, GFP) ont permis d’ouvrir le champ d’application du
BRET à l’étude des IPP. Dans cette méthode, l’utilisation d’une enzyme « donneuse »
d’énergie qui est transférée par résonnance à un fluorophore « accepteur »
spatialement proche (moins de 10 nm), évite les conséquences néfastes de
l’excitation du donneur qui est utilisée dans les méthodes FRET. Le donneur et
l’accepteur sont fusionnés aux deux protéines d’intérêt dont l’interaction peut
ainsi être détectée. C’est en 1999 que ce potentiel a, pour la première fois, été
exploité pour l’étude de protéines clés du rythme circadien chez la cyanobactérie
Synechococcus sp. (pour revue voir [55]). Le BRET sera mis en œuvre l’année
suivante dans des cellules de mammifères pour l’étude de récepteurs couplés aux
protéines G (RCPG) [55]. Le BRET a ensuite
été employé pour mettre en évidence des molécules d’intérêt thérapeutique par
criblage (par exemples des ligands de récepteurs de neuropeptides Y) [55]. C’est en 2006 que le BRET est utilisé pour
la première fois chez la levure pour caractériser l’oligomérisation du RCPG de la
phéromone a [56]. En tenant
compte des avantages qu’offre le champignon, le BRET a ensuite été utilisé pour le
criblage d’inhibiteurs d’IPP [57]. C’est principalement l’utilisation d’une levure mutante
erg6 (Delta(24)-sterol C-methyltransferase),
rendue hyper-perméable par altération de sa membrane due à un défaut d’ergostérol,
et le criblage effectué sur des complexes protéiques non préformés (la molécule à
tester est mise en contact avec la chimère protéique « acceptrice » avant
l’induction de l’expression de la chimère « donneuse ») qui ont contribué au succès
de cette approche. Le suivi de l’activité luciférase permet aussi d’évaluer la
cytotoxicité des molécules analysées. Plusieurs criblages ont été mis en œuvre afin
d’identifier des inhibiteurs de l’interaction CDK5/p25, une protéine kinase
impliquée notamment dans divers troubles neurodégénératifs [58, 59] et de nouveaux antibactériens, inhibiteurs de l’interaction
entre la sous-unité b’ et le facteur d’initiation s70 de l’ARN polymérase
bactérienne [60].
L’utilisation de nouveaux substrats de luciférase, de la NanoLuc® et de nouveaux
couples donneur/ accepteur (mutants de la luciférase et de l’accepteur, tels que la
mVenus ou encore la mTurquoise) permettront d’augmenter la sensibilité et l’usage du
BRET dans la levure pour y étudier de nouvelles IPP [57]. |
La levure est utilisée tant comme modèle que comme outil depuis de très nombreuses
années. Cette grande polyvalence a permis de multiples développements au service de
la recherche thérapeutique. Ainsi, longtemps après sa découverte et sa description
par Louis Pasteur, ces deux facettes de l’utilisation de la levure continuent de se
fertiliser mutuellement. La levure représente aujourd’hui un modèle précieux pour la
chémobiologie, en particulier pour les maladies rares et/ou pour lesquelles aucun
modèle mammifère n’existe encore. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données
publiées dans cet article.
|
Footnotes |
1.
Liu
L
,
Wang
J
,
Rosenberg
D
, et al.
Fermented beverage and food storage in 13,000 y-old stone mortars
at Raqefet Cave, Israel: investigating natufian ritual
feasting . J Archaeol Sci Rep.
2018; ; 21 :
:783.–793. 2.
Cagniard-Latour
C
. Mémoire sur la fermentation
vineuse . Ann Chimie Physique.
1838; ; 68 :
:206.–222. 3.
Pasteur
L
. Mémoire sur la fermentation
alcoolique . CR Acad Sci.
1857; ; 45 :
:1032.–1036. 4.
Goffeau
A
,
Barrell
BG
,
Bussey
H
, et al.
Life with 6000 genes . Science.
1996; ; 274 :
:63.–67. 5.
Carmona-Gutierrez
D
,
Bauer
MA
,
Zimmermann
A
, et al.
Guidelines and recommendations on yeast cell death
nomenclature . Microb Cell.
2018; ; 5 :
:4.–31. 6.
Kornberg
R
. The molecular basis of eukaryotic transcription
(Nobel lecture) . Angew Chem Int Ed Engl.
2007; ; 46 :
:6956.–6965. 7.
Nurse
PM
. Nobel lecture. Cyclin dependent kinases and cell
cycle control . Biosci Rep.
2002; ; 22 :
:487.–499. 8.
Schekman
R
. Charting the secretory pathway in a simple
eukaryote . Mol Biol Cell.
2010; ; 21 :
:3781.–3784. 9.
Ohsumi
Y
. Historical landmarks of autophagy
research . Cell Res.
2014; ; 24 :
:9.–23. 10.
Kachroo
AH
,
Laurent
JM
,
Yellman
CM
, et al.
Evolution. Systematic humanization of yeast genes reveals
conserved functions and genetic modularity .
Science.
2015; ; 348 :
:921.–925. 11.
Bassett
DE, Jr
Boguski
MS
,
Hieter
P
. Yeast genes and human disease .
Nature.
1996; ; 379 :
:589.–590. 12.
Kruger
WD
,
Cox
DR
. A yeast assay for functional detection of
mutations in the human cystathionine beta-synthase gene .
Hum Mol Genet.
1995; ; 4 :
:1155.–1161. 13.
Ishioka
C
,
Frebourg
T
,
Yan
YX
, et al.
Screening patients for heterozygous p53 mutations using a
functional assay in yeast . Nat Genet.
1993; ; 5 :
:124.–129. 14.
Billant
O
,
Leon
A
,
Le Guellec
S
, et al.
The dominant-negative interplay between p53, p63 and p73: a
family affair . Oncotarget.
2016; ; 7 :
:69549.–69564. 15.
Cox
BS
,
Tuite
MF
,
McLaughlin
CS
. The psi factor of yeast: a problem in
inheritance . Yeast.
1988; ; 4 :
:159.–178. 16.
Lacroute
F
. Non-Mendelian mutation allowing ureidosuccinic
acid uptake in yeast . J Bacteriol.
1971; ; 106 :
:519.–522. 17.
Wickner
RB
. [URE3] as an altered URE2 protein: evidence for a
prion analog in Saccharomyces cerevisiae .
Science.
1994; ; 264 :
:566.–569. 18.
Wickner
RB
,
Shewmaker
FP
,
Bateman
DA
, et al.
Yeast prions: structure, biology, and prion-handling
systems . Microbiol Mol Biol Rev.
2015; ; 79 :
:1.–17. 19.
King
CY
,
Diaz-Avalos
R
. Protein-only transmission of three yeast prion
strains . Nature.
2004; ; 428 :
:319.–323. 20.
Tanaka
M
,
Chien
P
,
Naber
N
, et al.
Conformational variations in an infectious protein determine
prion strain differences . Nature.
2004; ; 428 :
:323.–328. 21.
Longo
VD
,
Shadel
GS
,
Kaeberlein
M
,
Kennedy
B
. Replicative and chronological aging in
Saccharomyces cerevisiae . Cell Metab.
2012; ; 16 :
:18.–31. 22.
Guiffant
D
,
Tribouillard
D
,
Gug
F
, et al.
Identification of intracellular targets of small molecular weight
chemical compounds using affinity chromatography .
Biotechnol J.
2007; ; 2 :
:68.–75. 23.
Tribouillard-Tanvier
D
,
Dos Reis
S
,
Gug
F
, et al.
Protein folding activity of ribosomal RNA is a selective target
of two unrelated antiprion drugs . PLoS One.
2008; ; 3 : :e2174.. 24.
Hoon
S.
, St Onge
RP,
Giaever
G,
Nislow
C. Yeast chemical
genomics and drug discovery: an update . Trends
Pharmacol Sci.
2008; ; 29 :
:499.–504. 25.
Bach
S
,
Talarek
N
,
Andrieu
T
, et al.
Isolation of drugs active against mammalian prions using a
yeast-based screening assay . Nat Biotechnol.
2003; ; 21 :
:1075.–1081. 26.
Bach
S
,
Tribouillard
D
,
Talarek
N
, et al.
A yeast-based assay to isolate drugs active against mammalian
prions . Methods.
2006; ; 39 :
:72.–77. 27.
Oumata
N
,
Nguyen
PH
,
Beringue
V
, et al.
The toll-like receptor agonist imiquimod is active against
prions . PLoS One.
2013; ; 8 : :e72112.. 28.
Tribouillard-Tanvier
D
,
Beringue
V
,
Desban
N
, et al.
Antihypertensive drug guanabenz is active in vivo against both
yeast and mammalian prions . PLoS One.
2008; ; 3 : :e1981.. 29.
Blondel
M
,
Soubigou
F
,
Evrard
J
, et al.
Protein folding activity of the ribosome is involved in yeast
prion propagation . Sci Rep.
2016; ; 6 : :32117.. 30.
Cooper
AA
,
Gitler
AD
,
Cashikar
A
, et al.
Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron
loss in Parkinson’s models . Science.
2006; ; 313 :
:324.–328. 31.
Giorgini
F
,
Guidetti
P
,
Nguyen
Q
, et al.
A genomic screen in yeast implicates kynurenine 3-monooxygenase
as a therapeutic target for Huntington disease . Nat
Genet.
2005; ; 37 :
:526.–531. 32.
Marechal
D
,
Brault
V
,
Leon
A
, et al.
Cbs overdosage is necessary and sufficient to induce cognitive
phenotypes in mouse models of Down syndrome and interacts genetically with
Dyrk1a . Hum Mol Genet.
2019; ; 28 :
:1561.–1577. 33.
Couplan
E
,
Aiyar
RS
,
Kucharczyk
R
, et al.
A yeast-based assay identifies drugs active against human
mitochondrial disorders . Proc Natl Acad Sci
USA.
2011; ; 108 :
:11989.–11994. 34. Delerue
T,
Tribouillard-Tanvier
D,
Daloyau
M, et al.
A yeast-based screening assay identifies repurposed drugs that
suppress mitochondrial fusion and mtDNA maintenance defects .
Dis Model Mech.
2019; :12.. pii: dmm036558. doi: 10.1242/dmm.036558. 35.
Lasserre
JP
,
Dautant
A
,
Aiyar
RS
, et al.
Yeast as a system for modeling mitochondrial disease mechanisms
and discovering therapies . Dis Model Mech.
2015; ; 8 :
:509.–526. 36.
Bonnefoy
N
,
Fox
TD
. Genetic transformation of Saccharomyces
cerevisiae mitochondria . Methods Cell Biol.
2001; ; 65 :
:381.–396. 37.
Aiyar
RS
,
Bohnert
M
,
Duvezin-Caubet
S
, et al.
Mitochondrial protein sorting as a therapeutic target for ATP
synthase disorders . Nat Commun.
2014; ; 5 : :5585.. 38.
Zhao
RY
. Yeast for virus research. Microb .
Cell.
2017; ; 4 :
:311.–330. 39.
Lista
MJ
,
Voisset
C
,
Contesse
MA
, et al.
The long-lasting love affair between the budding yeast
Saccharomyces cerevisiae and the Epstein-Barr virus .
Biotechnol J.
2015; ; 10 :
:1670.–1681. 40.
Lista
MJ
,
Martins
RP
,
Angrand
G
, et al.
A yeast model for the mechanism of the Epstein-Barr virus immune
evasion identifies a new therapeutic target to interfere with the virus
stealthiness . Microb Cell.
2017; ; 4 :
:305.–307. 41.
Voisset
C
,
Daskalogianni
C
,
Contesse
MA
, et al.
A yeast-based assay identifies drugs that interfere with immune
evasion of the Epstein-Barr virus . Dis Model
Mech.
2014; ; 7 :
:435.–444. 42.
Lista
MJ
,
Martins
RP
,
Billant
O
, et al.
Nucleolin directly mediates Epstein-Barr virus immune evasion
through binding to G-quadruplexes of EBNA1 mRNA . Nat
Commun.
2017; ; 8 : :16043.. 43.
Reznichenko
O
,
Quillevere
A
,
Martins
RP
, et al.
Novel cationic bis(acylhydrazones) as modulators of Epstein-Barr
virus immune evasion acting through disruption of interaction between
nucleolin and G-quadruplexes of EBNA1 mRNA . Eur J
Med Chem.
2019; ; 178 :
:13.–29. 44.
Norcliffe
JL
,
Mina
JG
,
Alvarez
E
, et al.
Identifying inhibitors of the Leishmania inositol
phosphorylceramide synthase with antiprotozoal activity using a yeast-based
assay and ultra-high throughput screening platform .
Sci Rep.
2018; ; 8 : :3938.. 45.
Mayi
T
,
Facca
C
,
Anne
S
, et al.
Yeast as a model system to screen purine derivatives against
human CDK1 and CDK2 kinases . J Biotechnol.
2015; ; 195 :
:30.–36. 46.
Szczebara
FM
,
Chandelier
C
,
Villeret
C
, et al.
Total biosynthesis of hydrocortisone from a simple carbon source
in yeast . Nat Biotechnol.
2003; ; 21 :
:143.–149. 47.
Angrand
G,
Quillevere
A,
Loaec
N, et al.
Sneaking out for happy hour: yeast-based approaches to explore
and modulate immune response and immune evasion .
Genes (Basel).
2019; :10.. pii: E667. doi: 10.3390/genes10090667. 48.
Sewalt
V
,
Shanahan
S
,
Gregg
L
, et al.
The generally recognized as safe (GRAS) process for industrial
microbial enzymes . Industrial Biotechnology.
2016; ; 12 :
:295.–302. 49.
Bonander
N
,
Darby
RA
,
Grgic
L
, et al.
Altering the ribosomal subunit ratio in yeast maximizes
recombinant protein yield . Microb Cell Fact.
2009; ; 8 : :10.. 10.1186/1475-2859-8-10
50.
Fields
S
,
Song
O
. A novel genetic system to detect protein-protein
interactions . Nature.
1989; ; 340 :
:245.–246. 51.
Vidal
M
,
Fields
S
. The yeast two-hybrid assay: still finding
connections after 25 years . Nat Methods.
2014; ; 11 :
:1203.–1206. 52.
Hamdi
A
,
Colas
P
. Yeast two-hybrid methods and their applications
in drug discovery . Trends Pharmacol Sci.
2012; ; 33 :
:109.–118. 53.
Baines
IC
,
Colas
P
. Peptide aptamers as guides for small-molecule
drug discovery . Drug Discov Today.
2006; ; 11 :
:334.–341. 54.
Sahni
N
,
Yi
S
,
Taipale
M
, et al.
Widespread macromolecular interaction perturbations in human
genetic disorders . Cell.
2015; ; 161 :
:647.–660. 55.
Bacart
J
,
Corbel
C
,
Jockers
R
, et al.
The BRET technology and its application to screening
assays . Biotechnol J.
2008; ; 3 :
:311.–324. 56.
Gehret
AU
,
Bajaj
A
,
Naider
F
,
Dumont
ME
. Oligomerization of the yeast alpha-factor
receptor: implications for dominant negative effects of mutant
receptors . J Biol Chem.
2006; ; 281 :
:20698.–20714. 57.
Corbel
C
,
Sartini
S
,
Levati
E
, et al.
Screening for protein-protein interaction inhibitors using a
bioluminescence resonance energy transfer (BRET)-based assay in
yeast . SLAS Discov.
2017; ; 22 :
:751.–759. 58.
Corbel
C
,
Wang
Q
,
Bousserouel
H
, et al.
First BRET-based screening assay performed in budding yeast leads
to the discovery of CDK5/p25 interaction inhibitors .
Biotechnol J.
2011; ; 6 :
:860.–870. 59.
Corbel
C
,
Zhang
B
,
Le Parc
A
, et al.
Tamoxifen inhibits CDK5 kinase activity by interacting with
p35/p25 and modulates the pattern of tau phosphorylation .
Chem Biol.
2015; ; 22 :
:472.–482. 60.
Sartini
S
,
Levati
E
,
Maccesi
M
, et al.
New antimicrobials targeting bacterial RNA polymerase holoenzyme
assembly identified with an in vivo BRET-based discovery
platform . ACS Chem Biol.
2019; ; 14 :
:1727.–1736. 61.
Voisset
C
,
Blondel
M
. Chémobiologie à l’happy hour : la levure comme
modèle de criblage pharmacologique . Med Sci
(Paris).
2014; ; 30 :
:1161.–1168. |