Le cerveau humain est caractérisé par une complexité anatomique et une imbrication hiérarchique incomparables, du microcosme de la synapse à la macro-anatomie des réseaux neuronaux. Si nous comparons notre cerveau au globe terrestre et les neurones qui le peuplent aux habitants, le cerveau abrite dix fois plus d’habitants que notre monde, chacun d’entre eux passant, sans interruption, des centaines d’appels téléphoniques simultanément. Notre cerveau contient, dans un volume d’environ 1 500 cm3 seulement, environ 80 milliards de neurones divers, dont les prolongements ramifiés - dendrites et axones - mis bout à bout représentent une longueur de plusieurs centaines de milliers de kilomètres [1]. Il contient encore plus de cellules gliales, dont la diversité morphologique est comparable à celle des neurones [2] (Figure 1A, B).
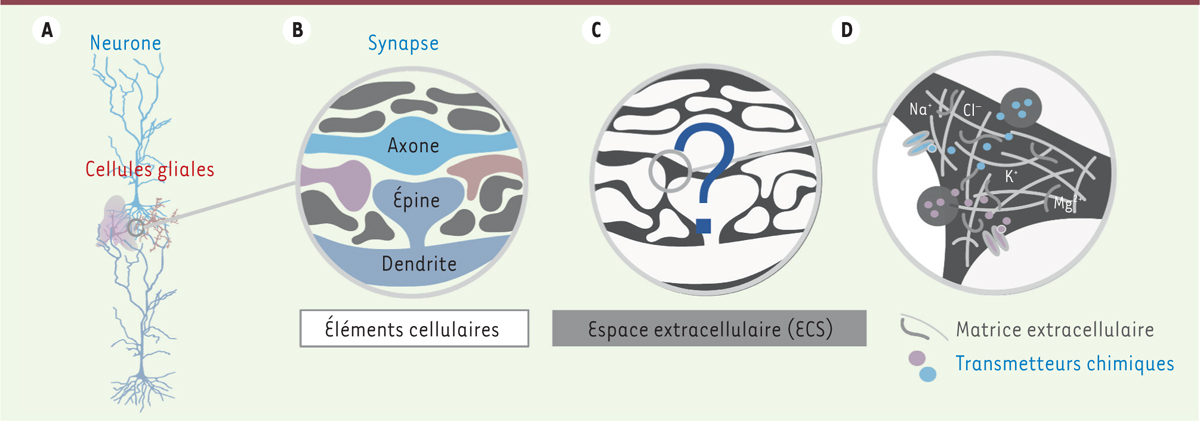
| Figure 1. Schémas représentant les neurones et les cellules gliales du
cerveau (A), leurs zones de contact, ou synapses (B), l’espace
extracellulaire (ECS) environnant (C), et la matrice extracellulaire
(D). Dans le cerveau, l’espace extracellulaire
entourant les neurones et les cellules gliales contient des ions, des
protéines et diverses autres molécules essentielles à leur
fonctionnement. |
Tous ces éléments cellulaires sont entourés par l’espace extracellulaire, qui occupe environ 20 % du volume cérébral. En tant que réservoir d’ions et voie de transport de substances actives, telles que les neurotransmetteurs, cet espace influence l’excitabilité électrique et le traitement des signaux par les neurones (Figure 1C, D). Étonnament, il a tendance à se dilater durant le sommeil, ce qui favorise notamment l’élimination des peptides β-amyloïdes, dont l’accumulation est impliquée dans la maladie d’Alzheimer [3]. Malgré sa contribution majeure au fonctionnement du cerveau, l’espace extracellulaire reste peu exploré, principalement en raison de l’absence des techniques permettant d’en obtenir des images détaillées dans des conditions physiologiques.
L’un des objectifs proclamés des neurosciences modernes est de déchiffrer intégralement l’architecture neuro-anatomique du cerveau, dans l’espoir que le plan complet des connexions neuronales (« connectome ») permettra de mieux comprendre le fonctionnement des circuits neuronaux qui sous-tendent les fonctions cérébrales supérieures telles que la pensée et les sentiments. Toutefois, les unités fonctionnelles de ce réseau, les synapses1, sont très petites, fragiles et dynamiques, et de surcroît difficile d’accès, et il est par ailleurs très difficile de cartographier l’enchevêtrement des structures neuro-cellulaires, sans oublier l’espace extracellulaire qui les entoure.
Pour relever ce défi technique, les chercheurs développent constamment de nouvelles méthodes d’imagerie (techniques de coloration, résolution spatiale en profondeur, traitement et analyse d’images, etc.). Jusqu’à présent, l’imagerie en tenseur de diffusion et la microscopie électronique ont été les principales techniques utilisées pour décrypter le connectome cérébral [4, 5]. Cependant, les deux méthodes présentent des inconvénients majeurs. Si la première, fondée sur l’imagerie par résonance magnétique, offre une profondeur de pénétration du tissu illimitée, elle a en revanche une très faible résolution spatiale (de l’ordre du millimètre), ce qui ne permet pas de visualiser les cellules individuelles, et encore moins d’analyser leur structure nanoscopique2. La microscopie électronique permet une analyse à cette échelle, mais elle est incompatible avec l’imagerie de cellules vivantes car elle exige que le tissu soit préalablement fixé et coupé en tranches très fines, ce qui induit des artefacts liés à la fixation, notamment un rétrécissement significatif de l’espace extracellulaire (Figure 2A).
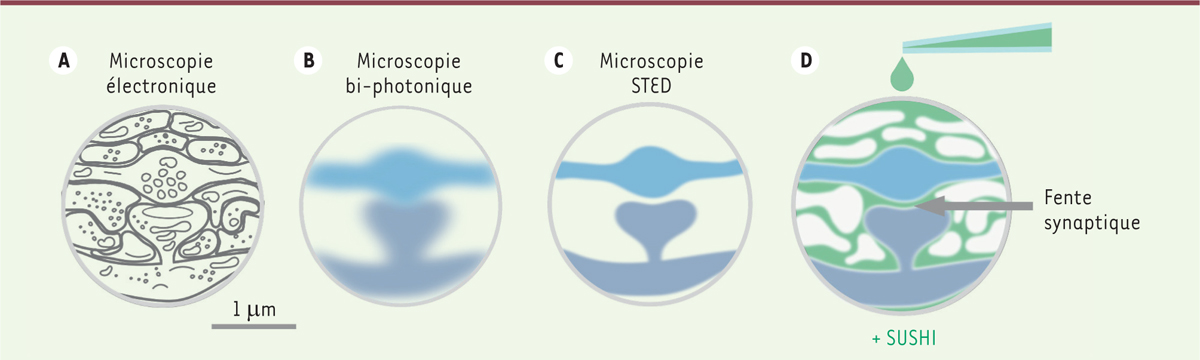
| Figure 2. La variante SUSHI de la technique de microscopie STED permet de
visualiser les détails de l’espace extracellulaire d’un cerveau
vivant. Illustrations schématiques d’un fragment de
tissu cérébral vu (A) par microscopie
électronique après fixation du tissu, (B) par
microscopie bi-photonique, (C) par microscopie
à super-résolution STED, ou (D) par la
variante SUSHI de celle-ci, qui révèle à la fois les structures cellulaires
et l’espace extracellulaire. |
La microscopie de fluorescence est une technique de microscopie optique qui permet d’observer les plus petits détails cellulaires dans des tissus vivants (Figure 2B, C). Son origine est assez ancienne, mais elle a été considérablement améliorée au cours des vingt dernières années, notamment avec le développement de la microscopie bi-photonique, la coloration génétiquement encodée avec des protéines fluorescentes (prix Nobel de chimie en 2008) et, plus récemment, l’imagerie de super-résolution (prix Nobel de chimie en 2014). Dans notre équipe de recherche, à l’université de Bordeaux, nous utilisons une microscopie de super-résolution fondée sur la technique de « déplétion par émission stimulée » (stimulated-emission-depletion ou STED), dont la résolution spatiale n’est plus limitée par la diffraction des ondes lumineuses. Ainsi, la technique STED permet de distinguer des objets tridimensionnels d’un volume inférieur à un attolitre (10-18 litre, soit 10-15 cm3) (Figure 2C). Récemment, nous avons développé des variantes de cette technique pour observer la dynamique des synapses dans l’hippocampe in vivo [6] et pour analyser la structure nanométrique des « synapses tripartites » formées par les structures neuronales et astrocytaires [7], qui étaient hors de portée des techniques classiques de microscopie optique [8]. En même temps, nous avons relevé le défi de visualiser l’espace extracellulaire, la « structure » du cerveau la plus mal explorée.
Au lieu de marquer des cellules individuellement, nous avons injecté un colorant fluorescent qui a diffusé dans tout l’espace extracellulaire, produisant ainsi des empreintes négatives (ombres) de toutes les cellules du tissu (Figure 2D). Nous avons nommé cette méthode super-resolution shadow imaging (SUSHI) [9]. Puisque l’espace intracellulaire est beaucoup plus vaste que l’espace extracellulaire, cette coloration fournit des images très contrastées, tout en évitant d’introduire des substances étrangères dans les cellules (Figure 3A). La méthode SUSHI permet donc, pour la première fois, d’obtenir une vue panoramique de tissu cérébral vivant avec un très haut niveau de détail. Nous avons d’abord utilisé cette technique pour observer des tranches de cerveau, car elles offrent un accès optique optimal avec un minimum d’interférences. Mais la technique peut également être utilisée pour obtenir des images du cerveau intact d’une souris vivante à travers une « fenêtre crânienne », le colorant étant injecté par voie intra-ventriculaire ou intra-orbitale afin de traverser la barrière hémato-encéphalique.
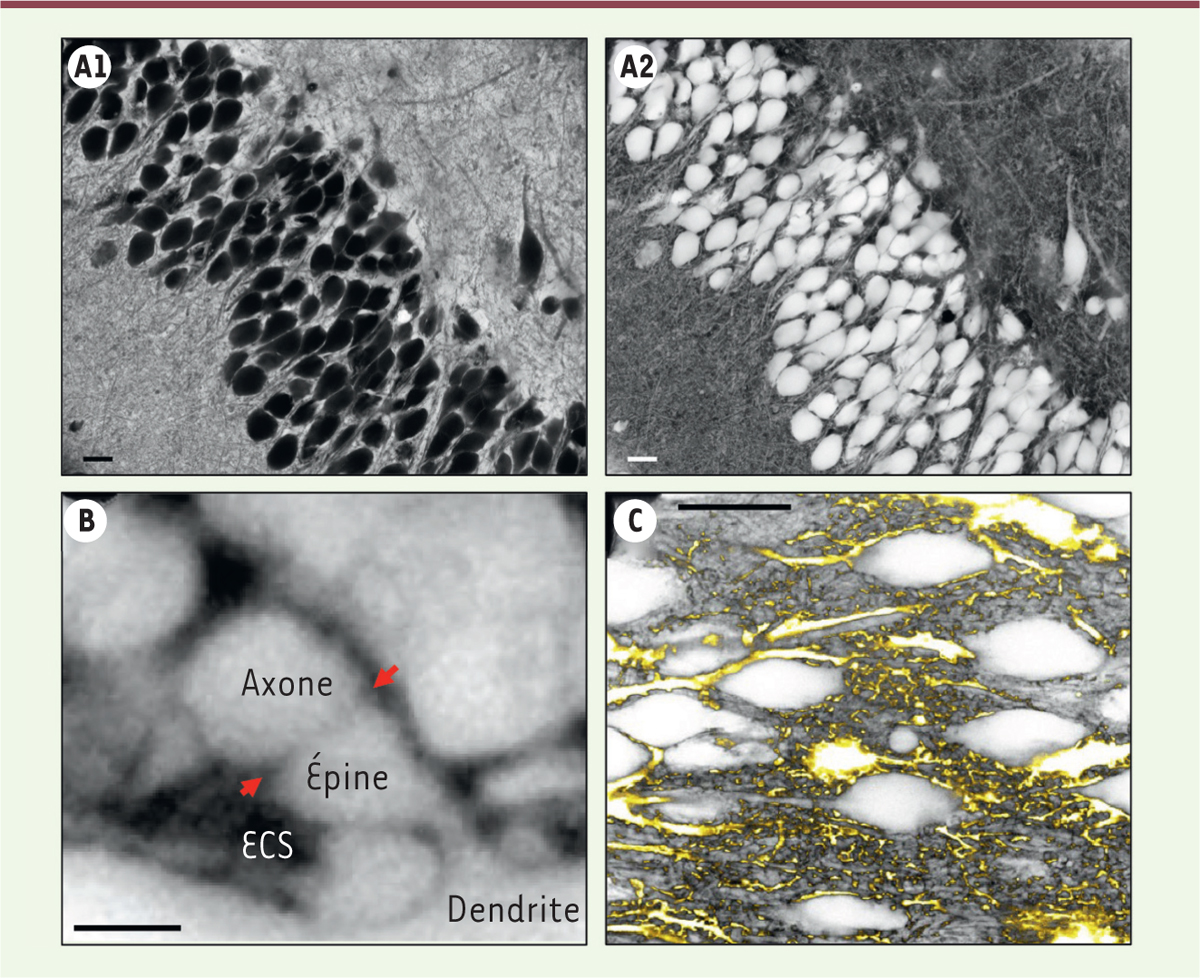
| Figure 3. Image d’un fragment du tissu cérébral vivant, obtenue par la
technique SUSHI. (A) Organisation micro-anatomique
du tissu avant (A1) et après (A2) inversion du contraste de l’image. (B) À plus fort grossissement de l’image, des
caractéristiques nanoscopiques comme les fentes synaptiques (flèches rouges)
deviennent visibles entre les structures pré-synaptiques (axone) et
post-synaptiques (tronc et épines dendritiques), entourées par l’espace
extracellulaire (ECS). Barre d’échelle : 1 mm. (C) La technique SUSHI peut aussi être combinée
avec un marquage « positif » classique (en jaune) des cellules gliales
(astrocytes) pour visualiser le contexte anatomique. Barre d’échelle : 20
mm. |
Jusqu’à récemment, les neuroscientifiques ne pouvaient examiner l’espace extracellulaire du cerveau qu’au prix d’importantes limitations (faible résolution spatiale ou fixation du tissu produisant des artefacts d’imagerie). La levée de ce verrou technologique ouvre la voie à l’étude de questions fondamentales concernant le rôle de cet espace dans le fonctionnement cérébral. Comment évolue l’espace extracellulaire en fonction de l’activité cérébrale (e.g., lors de l’apprentissage, du sommeil, etc.) à l’échelle cellulaire ou synaptique ? Ces changements sont-ils régulés au niveau local ou global ? Quels en sont les mécanismes et les conséquences fonctionnelles ? Comment sont-ils perturbés dans des conditions pathologiques ?
La technique SUSHI permet d’obtenir des images de la structure non seulement de l’espace extracellulaire, mais aussi de l’ensemble des cellules d’un tissu vivant de manière non biaisée, ce qui représente un progrès considérable pour l’étude du connectome cérébral. Elle permet de distinguer individuellement les cellules cérébrales et d’en différencier les types par la morphologie, tout en révélant les structures neuronales les plus fines telles que les fentes synaptiques, d’une largeur d’environ 20 nm (Figure 3B). Cela représente une prouesse en microscopie optique puisque, jusqu’à présent, ces structures ne pouvaient être visualisées que par la microscopie électronique. En effet, la fente synaptique apparaît en microscopie électronique comme une bande claire entre deux structures sombres (les terminaisons neuronales pré- et post-synaptiques), ce qui constitue des conditions de contraste favorables, tandis que dans le cas contraire de deux cellules fluorescentes claires entourant une fente sombre, qui caractérise la situation en microscopie optique, la fente synaptique est indétectable. Enfin, il est possible de combiner la technique SUSHI avec une coloration « positive » classique des cellules, ce qui permet d’analyser l’interface entre la cellule colorée positivement et son environnement. En particulier, il est possible de voir comment les cellules gliales sont « incorporées » dans le tissu neuronal environnant (Figure 3C).
L’imagerie par la technique SUSHI non seulement permet de mener des recherches fondamentales sur le cerveau, mais convient également à la recherche clinique et à la démarche diagnostique sur différents types de tissus et d’organes (tissu tumoral, organes du système immunitaire, etc.) [10]. Elle permet notamment de visualiser des cellules qu’il est difficile ou impossible de marquer « positivement » sans perturber le fonctionnement cellulaire. Ainsi, il est désormais possible de visualiser comment des cellules en train de migrer, comme les macrophages ou les cellules cancéreuses, se déplacent et se repèrent au sein de leur milieu environnant, ce qui est d’une grande importance pour comprendre les processus inflammatoires ou la formation des métastases cancéreuses.