Théories explicatives de la dyslexie
2007
| ANALYSE |
19-
Facteurs génétiques
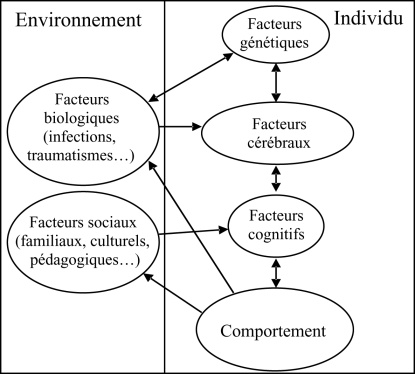
Ce chapitre analyse les données relatives aux facteurs génétiques des troubles spécifiques des apprentissages en s'appuyant sur les données d'épidémiologie génétique (études familiales et études de jumeaux) et sur les progrès de la biologie moléculaire qui ont permis la mise en évidence de gènes susceptibles d'être impliqués dans ces troubles, essentiellement dans la dyslexie. Ces troubles sont à la fois sous l'influence de facteurs génétiques et environnementaux (figure 19.1 ).
).
 ).
).Études familiales
L'hypothèse de la nature familiale de la dyslexie est évoquée depuis longtemps (Stephenson, 1907 ; Hallgren, 1950
; Hallgren, 1950 ) et un faisceau de présomptions rassemblées depuis une vingtaine d'années fait en effet penser que la dyslexie possède une origine génétique (DeFries et coll., 1987
) et un faisceau de présomptions rassemblées depuis une vingtaine d'années fait en effet penser que la dyslexie possède une origine génétique (DeFries et coll., 1987 ).
).
 ; Hallgren, 1950
; Hallgren, 1950 ) et un faisceau de présomptions rassemblées depuis une vingtaine d'années fait en effet penser que la dyslexie possède une origine génétique (DeFries et coll., 1987
) et un faisceau de présomptions rassemblées depuis une vingtaine d'années fait en effet penser que la dyslexie possède une origine génétique (DeFries et coll., 1987 ).
).Le meilleur indice d'une composante familiale est l'augmentation du risque chez les apparentés d'un sujet atteint. Il s'agit d'un risque relatif comparant le risque des apparentés d'individus sains au risque du trouble chez les apparentés d'individus malades. On utilise en général les antécédents familiaux du premier degré (parents, frères et sœurs). Mais ce sont les études de jumeaux qui permettent d'apprécier le poids des facteurs génétiques par rapport aux facteurs environnementaux par le calcul de l'héritabilité. Ceci ne signifie pas pour autant une relation causale directe. On peut supposer qu’une modification de l’environnement de deux vrais jumeaux, pendant leur développement intra-utérin ou lors de la période périnatale puisse entraîner un effet identique après la naissance, par une réponse similaire à une modification de l’environnement. Ces hypothèses mériteraient des travaux dans le domaine de la dyslexie.
Troubles du langage oral
La présence de plusieurs sujets atteints de troubles du langage dans une même famille n'est pas une éventualité rare, traduisant à la fois l'influence de l'environnement et de la génétique. Plusieurs études familiales ont montré que les apparentés du premier degré d'un sujet atteint de troubles du langage avaient un risque relatif deux à sept fois supérieur à celui de la population générale de développer des difficultés du même ordre (Brzustowicz, 1998 ). De même, dans une méta-analyse, Stromswold (2001
). De même, dans une méta-analyse, Stromswold (2001 ) montre que la prévalence des troubles du langage varie de 24 à 78 % (moyenne 46 %) dans les familles où il y a déjà un sujet atteint, alors qu'elle ne varie qu'entre 3 à 46 % (moyenne 18 %) dans les groupes témoins des différentes études.
) montre que la prévalence des troubles du langage varie de 24 à 78 % (moyenne 46 %) dans les familles où il y a déjà un sujet atteint, alors qu'elle ne varie qu'entre 3 à 46 % (moyenne 18 %) dans les groupes témoins des différentes études.
 ). De même, dans une méta-analyse, Stromswold (2001
). De même, dans une méta-analyse, Stromswold (2001 ) montre que la prévalence des troubles du langage varie de 24 à 78 % (moyenne 46 %) dans les familles où il y a déjà un sujet atteint, alors qu'elle ne varie qu'entre 3 à 46 % (moyenne 18 %) dans les groupes témoins des différentes études.
) montre que la prévalence des troubles du langage varie de 24 à 78 % (moyenne 46 %) dans les familles où il y a déjà un sujet atteint, alors qu'elle ne varie qu'entre 3 à 46 % (moyenne 18 %) dans les groupes témoins des différentes études.Troubles du langage écrit
Les troubles du langage écrit apparaissent aussi très fréquemment dans les familles à risque (Flax et coll., 2003 ). Une question importante qui se pose cependant est celle de savoir si les familles à risque de trouble du langage écrit sont aussi celles qui sont le plus à risque de trouble du langage oral et réciproquement. À première vue, les études concernant les facteurs de risque semblent soutenir l'idée d'un continuum entre le trouble du langage oral et le trouble du langage écrit. Elles montrent que les enfants manifestant des troubles du langage oral présentent très fréquemment aussi un trouble de lecture. Cependant, cette tendance est moins évidente que cela apparaît, pour deux raisons. D'abord, les parents atteints ont été souvent identifiés sur la base de tests en langage écrit mais pas en langage oral. Nous ne pouvons donc pas exclure la possibilité que certains d'entre eux avaient aussi des troubles du langage oral. Deuxièmement, bien que le développement verbal soit souvent retardé chez les enfants à risque de troubles du langage écrit, ils ont été rarement identifiés comme ayant un retard de langage oral. Par exemple, seulement un enfant dans l'étude de Snowling et coll. (2003
). Une question importante qui se pose cependant est celle de savoir si les familles à risque de trouble du langage écrit sont aussi celles qui sont le plus à risque de trouble du langage oral et réciproquement. À première vue, les études concernant les facteurs de risque semblent soutenir l'idée d'un continuum entre le trouble du langage oral et le trouble du langage écrit. Elles montrent que les enfants manifestant des troubles du langage oral présentent très fréquemment aussi un trouble de lecture. Cependant, cette tendance est moins évidente que cela apparaît, pour deux raisons. D'abord, les parents atteints ont été souvent identifiés sur la base de tests en langage écrit mais pas en langage oral. Nous ne pouvons donc pas exclure la possibilité que certains d'entre eux avaient aussi des troubles du langage oral. Deuxièmement, bien que le développement verbal soit souvent retardé chez les enfants à risque de troubles du langage écrit, ils ont été rarement identifiés comme ayant un retard de langage oral. Par exemple, seulement un enfant dans l'étude de Snowling et coll. (2003 ) avait répondu aux critères diagnostiques de trouble spécifique du langage oral.
) avait répondu aux critères diagnostiques de trouble spécifique du langage oral.
 ). Une question importante qui se pose cependant est celle de savoir si les familles à risque de trouble du langage écrit sont aussi celles qui sont le plus à risque de trouble du langage oral et réciproquement. À première vue, les études concernant les facteurs de risque semblent soutenir l'idée d'un continuum entre le trouble du langage oral et le trouble du langage écrit. Elles montrent que les enfants manifestant des troubles du langage oral présentent très fréquemment aussi un trouble de lecture. Cependant, cette tendance est moins évidente que cela apparaît, pour deux raisons. D'abord, les parents atteints ont été souvent identifiés sur la base de tests en langage écrit mais pas en langage oral. Nous ne pouvons donc pas exclure la possibilité que certains d'entre eux avaient aussi des troubles du langage oral. Deuxièmement, bien que le développement verbal soit souvent retardé chez les enfants à risque de troubles du langage écrit, ils ont été rarement identifiés comme ayant un retard de langage oral. Par exemple, seulement un enfant dans l'étude de Snowling et coll. (2003
). Une question importante qui se pose cependant est celle de savoir si les familles à risque de trouble du langage écrit sont aussi celles qui sont le plus à risque de trouble du langage oral et réciproquement. À première vue, les études concernant les facteurs de risque semblent soutenir l'idée d'un continuum entre le trouble du langage oral et le trouble du langage écrit. Elles montrent que les enfants manifestant des troubles du langage oral présentent très fréquemment aussi un trouble de lecture. Cependant, cette tendance est moins évidente que cela apparaît, pour deux raisons. D'abord, les parents atteints ont été souvent identifiés sur la base de tests en langage écrit mais pas en langage oral. Nous ne pouvons donc pas exclure la possibilité que certains d'entre eux avaient aussi des troubles du langage oral. Deuxièmement, bien que le développement verbal soit souvent retardé chez les enfants à risque de troubles du langage écrit, ils ont été rarement identifiés comme ayant un retard de langage oral. Par exemple, seulement un enfant dans l'étude de Snowling et coll. (2003 ) avait répondu aux critères diagnostiques de trouble spécifique du langage oral.
) avait répondu aux critères diagnostiques de trouble spécifique du langage oral.Les études familiales mettent en évidence l'augmentation du risque relatif pour les apparentés d'un cas de dyslexie de développer des troubles du langage oral et écrit. L'agrégation familiale des cas de dyslexie a été remarquée il y a déjà bien longtemps (Stephenson, 1907 ; Hallgren, 1950
; Hallgren, 1950 ) : si un enfant est dyslexique, il existe un risque d'environ 40 à 50 % qu'un de ses frères et sœurs le soit, et qu'un des deux parents l'ait été (Vogler et coll., 1985
) : si un enfant est dyslexique, il existe un risque d'environ 40 à 50 % qu'un de ses frères et sœurs le soit, et qu'un des deux parents l'ait été (Vogler et coll., 1985 ; Wolff et Melngailis, 1994
; Wolff et Melngailis, 1994 ) (figure 19.2
) (figure 19.2 ). Cela n'exclut pas l'observation par ailleurs de cas sporadiques, dans la mesure où les mêmes dysfonctionnements cérébraux et cognitifs peuvent survenir de novo. Pour la dyslexie, le risque de retrouver le trouble chez un apparenté de l'individu atteint est de 8 à 10 fois plus élevé que ce même risque dans la parenté d'un individu non atteint.
). Cela n'exclut pas l'observation par ailleurs de cas sporadiques, dans la mesure où les mêmes dysfonctionnements cérébraux et cognitifs peuvent survenir de novo. Pour la dyslexie, le risque de retrouver le trouble chez un apparenté de l'individu atteint est de 8 à 10 fois plus élevé que ce même risque dans la parenté d'un individu non atteint.
 ; Hallgren, 1950
; Hallgren, 1950 ) : si un enfant est dyslexique, il existe un risque d'environ 40 à 50 % qu'un de ses frères et sœurs le soit, et qu'un des deux parents l'ait été (Vogler et coll., 1985
) : si un enfant est dyslexique, il existe un risque d'environ 40 à 50 % qu'un de ses frères et sœurs le soit, et qu'un des deux parents l'ait été (Vogler et coll., 1985 ; Wolff et Melngailis, 1994
; Wolff et Melngailis, 1994 ) (figure 19.2
) (figure 19.2 ). Cela n'exclut pas l'observation par ailleurs de cas sporadiques, dans la mesure où les mêmes dysfonctionnements cérébraux et cognitifs peuvent survenir de novo. Pour la dyslexie, le risque de retrouver le trouble chez un apparenté de l'individu atteint est de 8 à 10 fois plus élevé que ce même risque dans la parenté d'un individu non atteint.
). Cela n'exclut pas l'observation par ailleurs de cas sporadiques, dans la mesure où les mêmes dysfonctionnements cérébraux et cognitifs peuvent survenir de novo. Pour la dyslexie, le risque de retrouver le trouble chez un apparenté de l'individu atteint est de 8 à 10 fois plus élevé que ce même risque dans la parenté d'un individu non atteint.Cependant, l'agrégation familiale suggère, mais ne prouve pas l'origine génétique. En effet, les familles partagent non seulement une partie de leurs gènes, mais également un certain environnement. On peut imaginer que des parents qui ne lisent pas constituent, pour leurs enfants, un environnement peu favorable à l'apprentissage de la lecture.
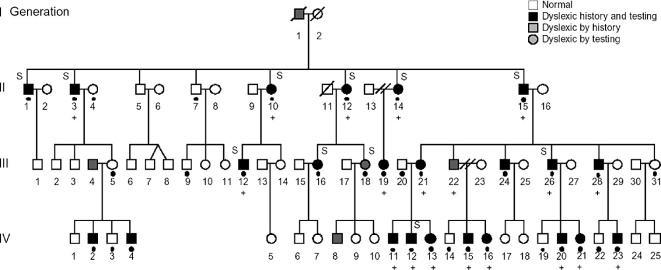
 | Figure 19.2 Exemple d'une famille comportant des cas multiples de dyslexie sur quatre générations (Nopola-Hemmi et coll., 2002 ) ) |
Études de jumeaux
Pour estimer le poids des facteurs génétiques par rapport aux facteurs environnementaux, on se reporte généralement aux études de jumeaux monozygotes (MZ) et dizygotes (DZ). Ces études de jumeaux ont précisément comme objectif de calculer l'héritabilité, c'est-à-dire le pourcentage de la variance expliquée par les facteurs génétiques. On mesure pour cela la concordance des pathologies chez les deux types de jumeaux. On observe que lorsqu'un jumeau monozygote est dyslexique, la probabilité que l'autre le soit également est d'environ 70 %. En revanche, la probabilité n'est plus que de 45 % pour les jumeaux dizygotes (Plomin et coll., 1994 ; Stromswold, 2001
; Stromswold, 2001 ). Comme on peut penser que les jumeaux monozygotes partagent entre eux les mêmes facteurs environnementaux que les jumeaux dizygotes, la différence de concordance s'explique donc par le fait que les jumeaux monozygotes sont similaires génétiquement à 100 % alors que les jumeaux dizygotes ne le sont qu'à 50 % (pour les gènes qui varient) ; les données de concordance permettent ainsi de calculer l'héritabilité.
). Comme on peut penser que les jumeaux monozygotes partagent entre eux les mêmes facteurs environnementaux que les jumeaux dizygotes, la différence de concordance s'explique donc par le fait que les jumeaux monozygotes sont similaires génétiquement à 100 % alors que les jumeaux dizygotes ne le sont qu'à 50 % (pour les gènes qui varient) ; les données de concordance permettent ainsi de calculer l'héritabilité.
 ; Stromswold, 2001
; Stromswold, 2001 ). Comme on peut penser que les jumeaux monozygotes partagent entre eux les mêmes facteurs environnementaux que les jumeaux dizygotes, la différence de concordance s'explique donc par le fait que les jumeaux monozygotes sont similaires génétiquement à 100 % alors que les jumeaux dizygotes ne le sont qu'à 50 % (pour les gènes qui varient) ; les données de concordance permettent ainsi de calculer l'héritabilité.
). Comme on peut penser que les jumeaux monozygotes partagent entre eux les mêmes facteurs environnementaux que les jumeaux dizygotes, la différence de concordance s'explique donc par le fait que les jumeaux monozygotes sont similaires génétiquement à 100 % alors que les jumeaux dizygotes ne le sont qu'à 50 % (pour les gènes qui varient) ; les données de concordance permettent ainsi de calculer l'héritabilité.L'héritabilité de la dyslexie est comprise entre 50 et 65 %. Ces données sont issues, en particulier, des deux plus grandes études sur les jumeaux : l'étude du Colorado (DeFries et coll., 1987 ; Olson et coll., 1989
; Olson et coll., 1989 ; Castles et coll., 1999
; Castles et coll., 1999 ) et l'étude de Londres (Stevenson et coll., 1987
) et l'étude de Londres (Stevenson et coll., 1987 ).
).
 ; Olson et coll., 1989
; Olson et coll., 1989 ; Castles et coll., 1999
; Castles et coll., 1999 ) et l'étude de Londres (Stevenson et coll., 1987
) et l'étude de Londres (Stevenson et coll., 1987 ).
).Des données similaires ont été collectées concernant les difficultés en mathématiques, aboutissant à une héritabilité d'environ 50 % (Knopik et deFries 1999a et b
et b ; Alarcon et coll. 1997
; Alarcon et coll. 1997 ; Oliver et coll., 2004
; Oliver et coll., 2004 ).
).
 et b
et b ; Alarcon et coll. 1997
; Alarcon et coll. 1997 ; Oliver et coll., 2004
; Oliver et coll., 2004 ).
).Les études de jumeaux permettent également de distinguer les facteurs partagés (facteurs d'environnement familial) et non partagés (environnement social, scolaire...). Dans le cas de la dyslexie, les facteurs environnementaux non partagés sont prédominants par rapport aux facteurs environnementaux partagés.
Toutes ces données permettent d'établir qu'il y a bien une contribution génétique à la dyslexie n’identifient pas les factures génétiques et n’expliquent pas leur mode d’action.
Études de liaison dans les troubles des apprentissages
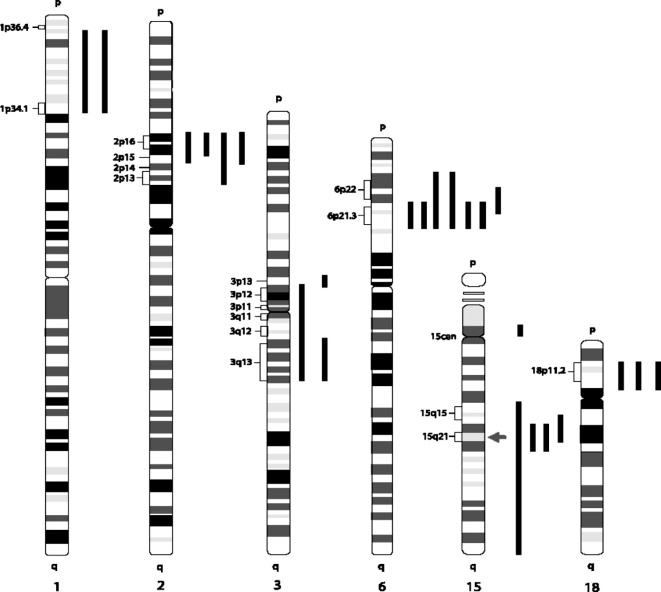
Les analyses de liaison génétique ont montré plusieurs régions chromosomiques significativement liées aux troubles des apprentissages (c'est-à-dire des régions chromosomiques transmises par les parents significativement plus souvent aux enfants avec troubles qu'aux enfants sans trouble). Le lien entre la dyslexie et les régions sur les chromosomes 1, 2, 3, 6, 15 et 18 a été rapporté par des équipes indépendantes dans plusieurs études (tableau 19.I ).
).
 ).
).Tableau 19.I Régions chromosomiques impliquées dans les troubles des apprentissages
|
Références
|
Loci
|
Nombre de familles
(nombre d'individus) |
Pays
|
Phénotype
|
|---|---|---|---|---|
|
1p34-36
|
9
|
États-Unis
|
Apprentissage global
|
|
|
8 (165)
|
États-Unis
|
Lecture de pseudo-mots
|
||
|
2p15-p16
|
1 (36)
|
Norvège
|
Apprentissage global
|
|
|
89 (195)
|
Grande-Bretagne
| |||
|
119 (180)
|
États-Unis
|
Langage écrit
|
||
|
96 (877)
|
Canada
|
Lecture de pseudo-mots
|
||
|
3p12-q13
|
1 (74)
|
Finlande
|
Apprentissage global
|
|
|
89 (195)
|
Grande-Bretagne
| |||
|
119 (180)
|
États-Unis
|
Lecture
|
||
|
6p21.3
|
9 (358)
|
États-Unis
| ||
|
6 (94)
|
États-Unis
|
Conscience phonologique
|
||
|
9
|
États-Unis
|
Conscience phonologique
|
||
|
82 (181)
|
Grande-Bretagne
| |||
|
7 (180)
|
États-Unis
| |||
|
8 (195)
|
États-Unis
Grande-Bretagne |
Lecture
|
||
|
15q21
|
9 (84)
|
États-Unis
|
Apprentissage global
|
|
|
6 (94)
|
États-Unis
|
Lecture de mots
|
||
|
8 (171)
|
États-Unis
|
Lecture de mots
|
||
|
7 (67)
|
Allemagne
|
Dysorthographie
|
||
|
7 (67)
|
Allemagne
|
Dysorthographie
|
||
|
101 (146)
| ||||
|
18p11.2
|
173 (338)
|
Grande-Bretagne
|
Apprentissage global
|
|
|
119 (180)
|
Grande-Bretagne
États-Unis |
Lecture
|
Les chromosomes impliqués dans la dyslexie ainsi que les régions qui ont été retrouvées par plusieurs études de liaison sont représentés dans la figure 19.3 .
.
 .
.La multiplicité des sites chromosomiques impliqués suggère que les troubles spécifiques des apprentissages sont, dans la plupart des cas, des déficits ayant une composante génétique complexe dans laquelle plusieurs gènes sont impliqués.
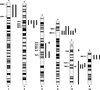 | Figure 19.3 Six chromosomes dont des régions sont liées à la dyslexie (d'après Grigorenko et coll., 2003 ) ) |
Identification de gènes
Les régions chromosomiques liées indiquent les régions du génome au sein desquelles se trouvent très probablement des gènes associés à la dyslexie. Néanmoins, ces régions peuvent abriter des centaines de gènes, qui sont catalogués mais qui pour la plupart n'ont jamais été étudiés. Il y a donc un fossé énorme entre l'identification d'une région liée, et l'identification au sein de cette région, d'un gène associé. Les progrès de la biologie moléculaire permettent de réduire de jour en jour ce fossé. À ce jour, 4 gènes candidats ont été identifiés comme pouvant être associés à une susceptibilité génétique à la dyslexie.
Gène DYX1C1
Des chercheurs finlandais ont identifié le premier gène associé à la dyslexie dans l'une des 6 régions chromosomiques suspectes. Il s'agit du gène DYX1C1 de la région q21 du chromosome 15, qui a été identifié grâce à une translocation au sein d'une famille (Taipale et coll., 2003 ).
).
 ).
).La protéine exprimée par ce gène est trouvée dans divers tissus incluant les neurones de la zone corticale et les cellules gliales. Elle intervient dans les interactions protéine-protéine.
Les auteurs finlandais ont analysé la variation des séquences de DYX1C1 dans de larges populations finlandaises. Deux changements rares sont identifiés : en position – 3 une adénine remplace une guanine, et en position 1 249 une thymine remplace une guanine. Ces changements sont trouvés dans des cas de dyslexie et chez des témoins sains, mais néanmoins plus fréquemment chez les cas. Quelques sujets sont porteurs d'un chromosome présentant à la fois les deux changements. Les deux polymorphismes peuvent potentiellement affecter la fonction du gène de deux façons différentes. La première mutation peut moduler l'expression du gène, et la deuxième qui créé un codon stop dans la région codante est responsable d'une protéine tronquée.
Cependant, six études longitudinales indépendantes menées au Royaume-Uni, aux États-Unis, au Canada et en Italie trouvent bien les allèles -3A et 1 249T présents dans toutes leurs populations mais ne retrouvent pas de lien entre ces polymorphismes et les problèmes de lecture (Scerri et coll., 2004 ; Wigg et coll., 2004
; Wigg et coll., 2004 ; Bellini et coll., 2005
; Bellini et coll., 2005 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Marino et coll., 2005
; Marino et coll., 2005 ; Meng et coll., 2005a
; Meng et coll., 2005a ). Il paraît peu probable que la population finlandaise, bien que présentant une plus grande homogénéité génétique que les autres populations, puisse être la seule à être susceptible aux effets fonctionnels de ces allèles.
). Il paraît peu probable que la population finlandaise, bien que présentant une plus grande homogénéité génétique que les autres populations, puisse être la seule à être susceptible aux effets fonctionnels de ces allèles.
 ; Wigg et coll., 2004
; Wigg et coll., 2004 ; Bellini et coll., 2005
; Bellini et coll., 2005 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Marino et coll., 2005
; Marino et coll., 2005 ; Meng et coll., 2005a
; Meng et coll., 2005a ). Il paraît peu probable que la population finlandaise, bien que présentant une plus grande homogénéité génétique que les autres populations, puisse être la seule à être susceptible aux effets fonctionnels de ces allèles.
). Il paraît peu probable que la population finlandaise, bien que présentant une plus grande homogénéité génétique que les autres populations, puisse être la seule à être susceptible aux effets fonctionnels de ces allèles.Plusieurs explications sont suggérées pour rendre compte de ces différences entre l'étude finlandaise et les essais de réplications : implication d'un gène situé à proximité de DYX1C1 pouvant être transmis en même temps que les allèles ; les associations mises en évidence dans l'étude finlandaise pourraient être de faux positifs pour des raisons méthodologiques (Scerri et coll., 2004 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Meng et coll., 2005a
; Meng et coll., 2005a ) ; le gène DYX1C1 pourrait ne pas correspondre aux gènes impliqués dans les analyses de liaison des premières études DYX1 (Scerri et coll., 2004
) ; le gène DYX1C1 pourrait ne pas correspondre aux gènes impliqués dans les analyses de liaison des premières études DYX1 (Scerri et coll., 2004 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Meng et coll., 2005a
; Meng et coll., 2005a ). Ainsi, les chercheurs continuent de rechercher dans la région 15q15-21 les gènes qui pourraient influencer la susceptibilité à la dyslexie.
). Ainsi, les chercheurs continuent de rechercher dans la région 15q15-21 les gènes qui pourraient influencer la susceptibilité à la dyslexie.
 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Meng et coll., 2005a
; Meng et coll., 2005a ) ; le gène DYX1C1 pourrait ne pas correspondre aux gènes impliqués dans les analyses de liaison des premières études DYX1 (Scerri et coll., 2004
) ; le gène DYX1C1 pourrait ne pas correspondre aux gènes impliqués dans les analyses de liaison des premières études DYX1 (Scerri et coll., 2004 ; Cope et coll., 2005a
; Cope et coll., 2005a ; Meng et coll., 2005a
; Meng et coll., 2005a ). Ainsi, les chercheurs continuent de rechercher dans la région 15q15-21 les gènes qui pourraient influencer la susceptibilité à la dyslexie.
). Ainsi, les chercheurs continuent de rechercher dans la région 15q15-21 les gènes qui pourraient influencer la susceptibilité à la dyslexie.La fonction du gène DYX1C1 a été étudiée par des neurobiologistes qui ont démontré que le gène était impliqué dans la migration neuronale (Wang et coll., 2006 ). Des coupes de cerveaux de rats ont été faites à un stade de maturation du cerveau correspondant au 4e ou 5e mois de gestation chez l'humain, c'est-à-dire au moment où s'effectue la migration neuronale. Lorsque le gène DYX1C1 est invalidé chez le rat au cours du développement cérébral (par une technique dite « d'ARN interfèrent »), la phase de migration des neurones de leur position péri-ventriculaire à leur situation corticale finale ne se fait pas correctement. En outre, cette migration anormale induit parfois des ectopies à la surface du cortex de la souris. Les chercheurs ont donc découvert un gène qui, potentiellement, pourrait contribuer à une explication des anomalies de migration neuronale observées dans le cerveau des dyslexiques comme l'avaient montré Galaburda et coll. (1985
). Des coupes de cerveaux de rats ont été faites à un stade de maturation du cerveau correspondant au 4e ou 5e mois de gestation chez l'humain, c'est-à-dire au moment où s'effectue la migration neuronale. Lorsque le gène DYX1C1 est invalidé chez le rat au cours du développement cérébral (par une technique dite « d'ARN interfèrent »), la phase de migration des neurones de leur position péri-ventriculaire à leur situation corticale finale ne se fait pas correctement. En outre, cette migration anormale induit parfois des ectopies à la surface du cortex de la souris. Les chercheurs ont donc découvert un gène qui, potentiellement, pourrait contribuer à une explication des anomalies de migration neuronale observées dans le cerveau des dyslexiques comme l'avaient montré Galaburda et coll. (1985 ). Ces données fonctionnelles renforcent la pertinence du gène DYX1C1 par rapport à la dyslexie. Mais il est probable que les véritables allèles de susceptibilité n'ont pas encore été découverts, ce qui peut expliquer l'incapacité des différentes équipes à répliquer l'association initialement proposée (Fisher et Francks, 2006
). Ces données fonctionnelles renforcent la pertinence du gène DYX1C1 par rapport à la dyslexie. Mais il est probable que les véritables allèles de susceptibilité n'ont pas encore été découverts, ce qui peut expliquer l'incapacité des différentes équipes à répliquer l'association initialement proposée (Fisher et Francks, 2006 ; Galaburda et coll., 2006
; Galaburda et coll., 2006 ).
).
 ). Des coupes de cerveaux de rats ont été faites à un stade de maturation du cerveau correspondant au 4e ou 5e mois de gestation chez l'humain, c'est-à-dire au moment où s'effectue la migration neuronale. Lorsque le gène DYX1C1 est invalidé chez le rat au cours du développement cérébral (par une technique dite « d'ARN interfèrent »), la phase de migration des neurones de leur position péri-ventriculaire à leur situation corticale finale ne se fait pas correctement. En outre, cette migration anormale induit parfois des ectopies à la surface du cortex de la souris. Les chercheurs ont donc découvert un gène qui, potentiellement, pourrait contribuer à une explication des anomalies de migration neuronale observées dans le cerveau des dyslexiques comme l'avaient montré Galaburda et coll. (1985
). Des coupes de cerveaux de rats ont été faites à un stade de maturation du cerveau correspondant au 4e ou 5e mois de gestation chez l'humain, c'est-à-dire au moment où s'effectue la migration neuronale. Lorsque le gène DYX1C1 est invalidé chez le rat au cours du développement cérébral (par une technique dite « d'ARN interfèrent »), la phase de migration des neurones de leur position péri-ventriculaire à leur situation corticale finale ne se fait pas correctement. En outre, cette migration anormale induit parfois des ectopies à la surface du cortex de la souris. Les chercheurs ont donc découvert un gène qui, potentiellement, pourrait contribuer à une explication des anomalies de migration neuronale observées dans le cerveau des dyslexiques comme l'avaient montré Galaburda et coll. (1985 ). Ces données fonctionnelles renforcent la pertinence du gène DYX1C1 par rapport à la dyslexie. Mais il est probable que les véritables allèles de susceptibilité n'ont pas encore été découverts, ce qui peut expliquer l'incapacité des différentes équipes à répliquer l'association initialement proposée (Fisher et Francks, 2006
). Ces données fonctionnelles renforcent la pertinence du gène DYX1C1 par rapport à la dyslexie. Mais il est probable que les véritables allèles de susceptibilité n'ont pas encore été découverts, ce qui peut expliquer l'incapacité des différentes équipes à répliquer l'association initialement proposée (Fisher et Francks, 2006 ; Galaburda et coll., 2006
; Galaburda et coll., 2006 ).
).Gènes KIAA0319 et DCDC2
La liaison au bras court du chromosome 6 (Smith et coll., 1991 ; Cardon et coll., 1994
; Cardon et coll., 1994 et 1995
et 1995 ) représente le résultat le mieux répliqué dans la littérature sur la dyslexie (Bisgaard et coll., 1987
) représente le résultat le mieux répliqué dans la littérature sur la dyslexie (Bisgaard et coll., 1987 ; Fisher et coll., 1999
; Fisher et coll., 1999 ; Gayán et coll., 1999
; Gayán et coll., 1999 ; Grigorenko et coll., 2000
; Grigorenko et coll., 2000 ; Fisher et coll., 2002
; Fisher et coll., 2002 ; Kaplan et coll., 2002
; Kaplan et coll., 2002 ; Turic et coll., 2003
; Turic et coll., 2003 ). Les études convergent sur la partie 6p23-21.3 nommée DYX2 mais cette région de consensus contient plusieurs centaines de gènes.
). Les études convergent sur la partie 6p23-21.3 nommée DYX2 mais cette région de consensus contient plusieurs centaines de gènes.
 ; Cardon et coll., 1994
; Cardon et coll., 1994 et 1995
et 1995 ) représente le résultat le mieux répliqué dans la littérature sur la dyslexie (Bisgaard et coll., 1987
) représente le résultat le mieux répliqué dans la littérature sur la dyslexie (Bisgaard et coll., 1987 ; Fisher et coll., 1999
; Fisher et coll., 1999 ; Gayán et coll., 1999
; Gayán et coll., 1999 ; Grigorenko et coll., 2000
; Grigorenko et coll., 2000 ; Fisher et coll., 2002
; Fisher et coll., 2002 ; Kaplan et coll., 2002
; Kaplan et coll., 2002 ; Turic et coll., 2003
; Turic et coll., 2003 ). Les études convergent sur la partie 6p23-21.3 nommée DYX2 mais cette région de consensus contient plusieurs centaines de gènes.
). Les études convergent sur la partie 6p23-21.3 nommée DYX2 mais cette région de consensus contient plusieurs centaines de gènes.Francks et coll. (2004 ) ont identifié une région 6p22 où de multiples polymorphismes montrent des associations avec des déficits de lecture. Ces associations ont été retrouvées dans un grand nombre de familles dyslexiques au Berkshire (Royaume-Uni) et au Colorado (États-Unis). La région d'intérêt contient 3 gènes. Le gène THEM2 code pour une enzyme du métabolisme ; TTRAP code pour une protéine impliquée dans des processus cellulaires fondamentaux tels que la mort cellulaire programmée et la réponse immune. Ces deux gènes sont largement exprimés. Quant au gène KIAA0319, il est exprimé principalement dans le tissu nerveux (Londin et coll., 2003
) ont identifié une région 6p22 où de multiples polymorphismes montrent des associations avec des déficits de lecture. Ces associations ont été retrouvées dans un grand nombre de familles dyslexiques au Berkshire (Royaume-Uni) et au Colorado (États-Unis). La région d'intérêt contient 3 gènes. Le gène THEM2 code pour une enzyme du métabolisme ; TTRAP code pour une protéine impliquée dans des processus cellulaires fondamentaux tels que la mort cellulaire programmée et la réponse immune. Ces deux gènes sont largement exprimés. Quant au gène KIAA0319, il est exprimé principalement dans le tissu nerveux (Londin et coll., 2003 ) et code pour une protéine qui est impliquée dans l'adhésion et les interactions entre neurones adjacents.
) et code pour une protéine qui est impliquée dans l'adhésion et les interactions entre neurones adjacents.
 ) ont identifié une région 6p22 où de multiples polymorphismes montrent des associations avec des déficits de lecture. Ces associations ont été retrouvées dans un grand nombre de familles dyslexiques au Berkshire (Royaume-Uni) et au Colorado (États-Unis). La région d'intérêt contient 3 gènes. Le gène THEM2 code pour une enzyme du métabolisme ; TTRAP code pour une protéine impliquée dans des processus cellulaires fondamentaux tels que la mort cellulaire programmée et la réponse immune. Ces deux gènes sont largement exprimés. Quant au gène KIAA0319, il est exprimé principalement dans le tissu nerveux (Londin et coll., 2003
) ont identifié une région 6p22 où de multiples polymorphismes montrent des associations avec des déficits de lecture. Ces associations ont été retrouvées dans un grand nombre de familles dyslexiques au Berkshire (Royaume-Uni) et au Colorado (États-Unis). La région d'intérêt contient 3 gènes. Le gène THEM2 code pour une enzyme du métabolisme ; TTRAP code pour une protéine impliquée dans des processus cellulaires fondamentaux tels que la mort cellulaire programmée et la réponse immune. Ces deux gènes sont largement exprimés. Quant au gène KIAA0319, il est exprimé principalement dans le tissu nerveux (Londin et coll., 2003 ) et code pour une protéine qui est impliquée dans l'adhésion et les interactions entre neurones adjacents.
) et code pour une protéine qui est impliquée dans l'adhésion et les interactions entre neurones adjacents.Cope et coll. (2005b ) rapportent également, dans une étude cas-témoins indépendante de Cardiff (Royaume-Uni), une association avec le groupe KIAA0319-THEM2-TTRAP, qu'ils ont confirmé par un test de déséquilibre de transmission. Dans leur étude, deux polymorphismes de KIAA0319 apparaissent plus particulièrement associés avec la dyslexie. Dans un cas, il s'agit du remplacement d'une alanine par une thréonine, mais ce changement est également relativement fréquent dans les populations non affectées et en conséquence ne peut pas représenter l'haplotype à risque associé à la dyslexie. Les études fonctionnelles récentes indiquent que les chromosomes qui portent l'haplotype potentiellement à risque donnent une expression réduite de KIAA0319, mais des niveaux normaux pour THEM2/TTRAP (Paracchini et coll., 2006
) rapportent également, dans une étude cas-témoins indépendante de Cardiff (Royaume-Uni), une association avec le groupe KIAA0319-THEM2-TTRAP, qu'ils ont confirmé par un test de déséquilibre de transmission. Dans leur étude, deux polymorphismes de KIAA0319 apparaissent plus particulièrement associés avec la dyslexie. Dans un cas, il s'agit du remplacement d'une alanine par une thréonine, mais ce changement est également relativement fréquent dans les populations non affectées et en conséquence ne peut pas représenter l'haplotype à risque associé à la dyslexie. Les études fonctionnelles récentes indiquent que les chromosomes qui portent l'haplotype potentiellement à risque donnent une expression réduite de KIAA0319, mais des niveaux normaux pour THEM2/TTRAP (Paracchini et coll., 2006 ). On ne sait pas quelle séquence des variants confère cette différence dans la régulation.
). On ne sait pas quelle séquence des variants confère cette différence dans la régulation.
 ) rapportent également, dans une étude cas-témoins indépendante de Cardiff (Royaume-Uni), une association avec le groupe KIAA0319-THEM2-TTRAP, qu'ils ont confirmé par un test de déséquilibre de transmission. Dans leur étude, deux polymorphismes de KIAA0319 apparaissent plus particulièrement associés avec la dyslexie. Dans un cas, il s'agit du remplacement d'une alanine par une thréonine, mais ce changement est également relativement fréquent dans les populations non affectées et en conséquence ne peut pas représenter l'haplotype à risque associé à la dyslexie. Les études fonctionnelles récentes indiquent que les chromosomes qui portent l'haplotype potentiellement à risque donnent une expression réduite de KIAA0319, mais des niveaux normaux pour THEM2/TTRAP (Paracchini et coll., 2006
) rapportent également, dans une étude cas-témoins indépendante de Cardiff (Royaume-Uni), une association avec le groupe KIAA0319-THEM2-TTRAP, qu'ils ont confirmé par un test de déséquilibre de transmission. Dans leur étude, deux polymorphismes de KIAA0319 apparaissent plus particulièrement associés avec la dyslexie. Dans un cas, il s'agit du remplacement d'une alanine par une thréonine, mais ce changement est également relativement fréquent dans les populations non affectées et en conséquence ne peut pas représenter l'haplotype à risque associé à la dyslexie. Les études fonctionnelles récentes indiquent que les chromosomes qui portent l'haplotype potentiellement à risque donnent une expression réduite de KIAA0319, mais des niveaux normaux pour THEM2/TTRAP (Paracchini et coll., 2006 ). On ne sait pas quelle séquence des variants confère cette différence dans la régulation.
). On ne sait pas quelle séquence des variants confère cette différence dans la régulation.Meng et coll. (2005b ) ont étudié les mêmes familles du Colorado que celles incluses dans l'étude de Francks et coll. (2004
) ont étudié les mêmes familles du Colorado que celles incluses dans l'étude de Francks et coll. (2004 ), mais ils ont ciblé leur recherche sur un gène différent : DCDC2. Dans ce gène, 2 500 nucléotides d'une région intronique sont absents chez un petit nombre de personnes dyslexiques et correspondent à une délétion contenant un microsatellite ou STRs (Short Tandem Repeats). Le nombre de copies de STRs varie dans différentes populations rendant compte de différents allèles. Le gène DCDC2 est exprimé dans de nombreux tissus incluant le cerveau (Schumacher et coll., 2006
), mais ils ont ciblé leur recherche sur un gène différent : DCDC2. Dans ce gène, 2 500 nucléotides d'une région intronique sont absents chez un petit nombre de personnes dyslexiques et correspondent à une délétion contenant un microsatellite ou STRs (Short Tandem Repeats). Le nombre de copies de STRs varie dans différentes populations rendant compte de différents allèles. Le gène DCDC2 est exprimé dans de nombreux tissus incluant le cerveau (Schumacher et coll., 2006 ). Le produit de ce gène ressemble à une protéine cytoplasmique (DCX) qui est impliquée dans un déficit de migration neuronale retrouvé dans deux troubles sévères : la lissencéphalie et le syndrome du double cortex.
). Le produit de ce gène ressemble à une protéine cytoplasmique (DCX) qui est impliquée dans un déficit de migration neuronale retrouvé dans deux troubles sévères : la lissencéphalie et le syndrome du double cortex.
 ) ont étudié les mêmes familles du Colorado que celles incluses dans l'étude de Francks et coll. (2004
) ont étudié les mêmes familles du Colorado que celles incluses dans l'étude de Francks et coll. (2004 ), mais ils ont ciblé leur recherche sur un gène différent : DCDC2. Dans ce gène, 2 500 nucléotides d'une région intronique sont absents chez un petit nombre de personnes dyslexiques et correspondent à une délétion contenant un microsatellite ou STRs (Short Tandem Repeats). Le nombre de copies de STRs varie dans différentes populations rendant compte de différents allèles. Le gène DCDC2 est exprimé dans de nombreux tissus incluant le cerveau (Schumacher et coll., 2006
), mais ils ont ciblé leur recherche sur un gène différent : DCDC2. Dans ce gène, 2 500 nucléotides d'une région intronique sont absents chez un petit nombre de personnes dyslexiques et correspondent à une délétion contenant un microsatellite ou STRs (Short Tandem Repeats). Le nombre de copies de STRs varie dans différentes populations rendant compte de différents allèles. Le gène DCDC2 est exprimé dans de nombreux tissus incluant le cerveau (Schumacher et coll., 2006 ). Le produit de ce gène ressemble à une protéine cytoplasmique (DCX) qui est impliquée dans un déficit de migration neuronale retrouvé dans deux troubles sévères : la lissencéphalie et le syndrome du double cortex.
). Le produit de ce gène ressemble à une protéine cytoplasmique (DCX) qui est impliquée dans un déficit de migration neuronale retrouvé dans deux troubles sévères : la lissencéphalie et le syndrome du double cortex.Dans une étude allemande, Schumacher et coll. (2006 ) impliquent également le gène DCDC2 dans le déficit de lecture d'après les résultats de tests de déséquilibre de transmission. L'association est plus significative chez les individus affectés par une dyslexie sévère. Cependant, aucun allèle à risque fonctionnel n'est identifié et la délétion STR n'a pas été étudiée. Les études pour évaluer si la délétion/variation STR est associée à des changements dans l'expression de DCDC2 restent à faire.
) impliquent également le gène DCDC2 dans le déficit de lecture d'après les résultats de tests de déséquilibre de transmission. L'association est plus significative chez les individus affectés par une dyslexie sévère. Cependant, aucun allèle à risque fonctionnel n'est identifié et la délétion STR n'a pas été étudiée. Les études pour évaluer si la délétion/variation STR est associée à des changements dans l'expression de DCDC2 restent à faire.
 ) impliquent également le gène DCDC2 dans le déficit de lecture d'après les résultats de tests de déséquilibre de transmission. L'association est plus significative chez les individus affectés par une dyslexie sévère. Cependant, aucun allèle à risque fonctionnel n'est identifié et la délétion STR n'a pas été étudiée. Les études pour évaluer si la délétion/variation STR est associée à des changements dans l'expression de DCDC2 restent à faire.
) impliquent également le gène DCDC2 dans le déficit de lecture d'après les résultats de tests de déséquilibre de transmission. L'association est plus significative chez les individus affectés par une dyslexie sévère. Cependant, aucun allèle à risque fonctionnel n'est identifié et la délétion STR n'a pas été étudiée. Les études pour évaluer si la délétion/variation STR est associée à des changements dans l'expression de DCDC2 restent à faire.Bien que ces données soient encourageantes, elles ne prouvent pas une relation causale avec un trouble de la lecture. La démonstration directe que la fonction des gènes KIAA0319 ou DCDC2 est modifiée chez les dyslexiques n'est pas faite. Il est probable que les deux gènes contribuent au risque mais leur influence relative varie selon les échantillons de populations étudiés.
Enfin, des études fonctionnelles par ARN interférent chez le rat montrent que les gènes DCDC2 et KIAA0319 sont eux aussi tous deux nécessaires pour la migration neuronale (Meng et coll., 2005b ; Paracchini et coll., 2006
; Paracchini et coll., 2006 ).
).
 ; Paracchini et coll., 2006
; Paracchini et coll., 2006 ).
).Gène ROBO1
Une liaison avec la région DYX5 du chromosome 3 a été trouvée dans une famille finlandaise sur 4 générations (27 des 74 membres sont diagnostiqués avec une dyslexie, cf. figure 19.2 ) suggérant une transmission particulière d'un gène à effet dominant (Nopola-Hemmi et coll., 2001
) suggérant une transmission particulière d'un gène à effet dominant (Nopola-Hemmi et coll., 2001 ). L'étude de telles familles peut constituer une aide dans l'identification de gènes de susceptibilité. Une région du chromosome 3 (3p12-q13) était partagée (héritée du même chromosome fondateur) par 19 des 21 sujets affectés étudiés dans la famille présentant cette liaison à DYX5. Par ailleurs, une translocation entre les chromosomes 3 et 8 a été identifiée chez un sujet dyslexique indépendant (Hannula-Jouppi et coll., 2005
). L'étude de telles familles peut constituer une aide dans l'identification de gènes de susceptibilité. Une région du chromosome 3 (3p12-q13) était partagée (héritée du même chromosome fondateur) par 19 des 21 sujets affectés étudiés dans la famille présentant cette liaison à DYX5. Par ailleurs, une translocation entre les chromosomes 3 et 8 a été identifiée chez un sujet dyslexique indépendant (Hannula-Jouppi et coll., 2005 ). Ces auteurs ont mis en évidence que le point de translocation du chromosome 3, dans ce cas unique, présentait une modification dans un intron du gène ROBO1.
). Ces auteurs ont mis en évidence que le point de translocation du chromosome 3, dans ce cas unique, présentait une modification dans un intron du gène ROBO1.
 ) suggérant une transmission particulière d'un gène à effet dominant (Nopola-Hemmi et coll., 2001
) suggérant une transmission particulière d'un gène à effet dominant (Nopola-Hemmi et coll., 2001 ). L'étude de telles familles peut constituer une aide dans l'identification de gènes de susceptibilité. Une région du chromosome 3 (3p12-q13) était partagée (héritée du même chromosome fondateur) par 19 des 21 sujets affectés étudiés dans la famille présentant cette liaison à DYX5. Par ailleurs, une translocation entre les chromosomes 3 et 8 a été identifiée chez un sujet dyslexique indépendant (Hannula-Jouppi et coll., 2005
). L'étude de telles familles peut constituer une aide dans l'identification de gènes de susceptibilité. Une région du chromosome 3 (3p12-q13) était partagée (héritée du même chromosome fondateur) par 19 des 21 sujets affectés étudiés dans la famille présentant cette liaison à DYX5. Par ailleurs, une translocation entre les chromosomes 3 et 8 a été identifiée chez un sujet dyslexique indépendant (Hannula-Jouppi et coll., 2005 ). Ces auteurs ont mis en évidence que le point de translocation du chromosome 3, dans ce cas unique, présentait une modification dans un intron du gène ROBO1.
). Ces auteurs ont mis en évidence que le point de translocation du chromosome 3, dans ce cas unique, présentait une modification dans un intron du gène ROBO1.Il s'agit d'un gène qui code pour un récepteur transmembranaire impliqué dans la transduction du signal qui aide à réguler le guidage des axones, notamment d'un hémisphère à l'autre. ROBO1 se situe en 3p12 dans la région concernée. La recherche de gènes dans cette famille finlandaise a montré que les personnes affectées portent une combinaison inhabituelle de polymorphismes. L'un des variants identifiés présente une modification de la structure de la protéine ROBO1 par insertion/délétion d'un résidu d'acide aspartique. Mais ce changement est également retrouvé chez les sujets non atteints et ne peut donc pas être considéré à lui seul comme causal (Hannula-Jouppi et coll., 2005 ). Par ailleurs, il a été retrouvé une diminution de l'expression de ROBO1 chez certains individus porteurs de l'haplotype à risque, mais cela n'a pas été vérifié chez tous les membres de la famille portant l'haplotype à risque.
). Par ailleurs, il a été retrouvé une diminution de l'expression de ROBO1 chez certains individus porteurs de l'haplotype à risque, mais cela n'a pas été vérifié chez tous les membres de la famille portant l'haplotype à risque.
 ). Par ailleurs, il a été retrouvé une diminution de l'expression de ROBO1 chez certains individus porteurs de l'haplotype à risque, mais cela n'a pas été vérifié chez tous les membres de la famille portant l'haplotype à risque.
). Par ailleurs, il a été retrouvé une diminution de l'expression de ROBO1 chez certains individus porteurs de l'haplotype à risque, mais cela n'a pas été vérifié chez tous les membres de la famille portant l'haplotype à risque.En conclusion,
la recherche sur la génétique de la dyslexie est à la fois riche de succès et d'incertitudes. Tout d'abord, le poids des facteurs génétiques, amplement illustré par les études de familialité et de jumeaux, ne fait désormais plus aucun doute (nonobstant l'hétérogénéité de la population dyslexique). Les études de liaison ont mis en évidence une dizaine de régions chromosomiques liées, dont six ont été confirmées par plusieurs études indépendantes. Au sein de ces six régions, quatre gènes candidats ont été proposés. Deux d'entre eux (DYX1C1, ROBO1) présentent des mutations chez des cas ou familles rares, mais on ne sait pas si des allèles de ces gènes augmentent le risque de formes plus courantes de dyslexie. Pour les deux autres (KIAA0319 et DCDC2), des haplotypes de susceptibilité au sein de grandes populations ont été proposés. Ces haplotypes restent néanmoins à confirmer. Hormis les cas rares de mutation radicale invalidant la fonction de la protéine (DYX1C1 dans une famille finlandaise), il semble que les allèles de susceptibilité produisent leurs effets par une altération de l'expression de la protéine (certains haplotypes sont d'ailleurs situés dans des régions régulatrices). Néanmoins, les données d'expression restent fragmentaires et demandent à être confirmées. Le résultat le plus remarquable sur les propriétés fonctionnelles de ces gènes provient des études sur l'animal en cours de développement. Ces études montrent que les quatre gènes candidats pour la dyslexie sont impliqués dans la migration des neurones au cours du développement cérébral, trois dans la migration radiaire des corps cellulaires vers le cortex, et un (ROBO1) dans la migration des axones et des dendrites. Il paraît peu probable qu'une telle convergence de fonctions soit due au hasard. De plus, ces résultats permettent d'établir un lien entre les gènes associés à la dyslexie et les anomalies cérébrales qui ont été observées dans les cerveaux de certaines personnes dyslexiques (Galaburda et coll., 1985 ; Galaburda et coll., 2006
; Galaburda et coll., 2006 ).
).
 ; Galaburda et coll., 2006
; Galaburda et coll., 2006 ).
).Il est important de souligner que, si l'usage en génétique veut qu'on désigne ces gènes comme des « gènes de la dyslexie », il s'agit là d'un abus de langage, en fait d'un raccourci pour désigner des « gènes dont certains allèles augmentent le risque de dyslexie ». Il va de soi qu'aucun de ces gènes n'est spécifique à la dyslexie, et qu'il ne s'agit pas non plus de gènes de la lecture, ni même du langage oral. Comme presque tous nos gènes, ceux-ci existent dans des formes voisines chez les autres mammifères, voire même chez la drosophile (ROBO1). Ces gènes exercent, chez tous ces animaux, des fonctions multiples, et sont d'ailleurs exprimés dans de nombreux organes différents au cours du développement et de la vie. Ce qui les relie plus spécifiquement à la dyslexie, c'est le fait d'être aussi impliqués dans un stade particulier du développement cérébral, et notamment dans la mise en place de certaines aires cérébrales qui seront bien plus tard recrutées par l'apprentissage de la lecture1
.
En ce qui concerne les autres troubles spécifiques des apprentissages, la recherche en génétique en est encore à ses balbutiements. On ne sait de la dyscalculie que la forte héritabilité des difficultés en mathématiques. En ce qui concerne la dysorthographie, les études génétiques ne l'ont en fait pas vraiment distinguée de la dyslexie, beaucoup d'études utilisant des mesures d'orthographe aussi bien que de lecture pour définir le phénotype. En particulier, du fait de la transparence de l'orthographe, les études allemandes utilisent typiquement l'orthographe comme le principal phénotype révélateur de la dyslexie. C'est ainsi qu'on peut considérer que le gène DCDC2 est en fait associé à la dysorthographie dans l'étude de Schumacher et coll. (2006 ). L'association du même gène par l'étude américaine a, elle, été obtenue sur la base d'un score composite combinant lecture et orthographe. Pour l'instant, aucune étude n'a recherché des facteurs génétiques qui pourraient distinguer dyslexie de dysorthographie.
). L'association du même gène par l'étude américaine a, elle, été obtenue sur la base d'un score composite combinant lecture et orthographe. Pour l'instant, aucune étude n'a recherché des facteurs génétiques qui pourraient distinguer dyslexie de dysorthographie.
 ). L'association du même gène par l'étude américaine a, elle, été obtenue sur la base d'un score composite combinant lecture et orthographe. Pour l'instant, aucune étude n'a recherché des facteurs génétiques qui pourraient distinguer dyslexie de dysorthographie.
). L'association du même gène par l'étude américaine a, elle, été obtenue sur la base d'un score composite combinant lecture et orthographe. Pour l'instant, aucune étude n'a recherché des facteurs génétiques qui pourraient distinguer dyslexie de dysorthographie.Enfin, il est important de prendre en compte l'hétérogénéité qui se cache derrière les généralités énoncées ci-dessus. Celle-ci se situe à tous les niveaux :
• au niveau des causes primaires, tous les cas de dyslexie ne sont pas nécessairement d'origine génétique. Il est fort probable que dans un certain nombre de cas, la cause primaire puisse résider dans des facteurs non génétiques, à savoir typiquement d'autres facteurs biologiques, notamment ceux entraînant des anomalies cérébrales à la naissance (Daigneault et Braun, 2002 ) ;
) ;
 ) ;
) ;
• au niveau des causes génétiques, il existe également une grande hétérogénéité. Dans certains cas rares, il semble qu'une mutation d'un gène unique soit suffisante pour provoquer la dyslexie. L'identité du gène responsable peut varier d'un cas à l'autre. Dans la plupart des cas, les personnes dyslexiques ne semblent pas porteuses d'une mutation rare, mais plutôt d'allèles de susceptibilité. Ces allèles sont fréquents au sein la population normale, et ne constituent pas en soit une cause suffisante de la dyslexie. Chaque allèle de susceptibilité augmente un peu le risque de dyslexie, le trouble ne se révélant que lors de combinaisons particulièrement défavorables d'allèles de susceptibilité, et/ou d'interactions de ces allèles avec des facteurs non génétiques augmentant également le risque. On est là dans la logique des maladies génétiques complexes ;
• au niveau des causes non biologiques, il existe un grand nombre de facteurs environnementaux (biochimiques, traumatiques, linguistiques, socioculturels, pédagogiques) qui peuvent moduler l'expression des facteurs génétiques, positivement ou négativement.
Chaque individu dyslexique possède donc son propre profil cognitif et ses propres particularités cérébrales, qui sont le résultat de la combinaison spécifique de facteurs génétiques dont il est porteur, et des facteurs non génétiques auxquels il a été exposé.
Bibliographie
[1] alarcon m, defries jc, light jg, pennington bf. A twin study of mathematics disability.
Journal of Learning Disabilities. 1997;
30:617- 623

[2] andrews s. Communicating a new gene vital for speech and language.
Clinical Genetics. 2002;
61:97- 100

[3] bellini g, bravaccio c, calamoneri f, donatella cocuzza m, fiorillo p. No evidence for association between dyslexia and DYX1C1 functional variants in a group of children and adolescents from Southern Italy.
J Mol Neurosci. 2005;
27:311- 314

[4] bisgaard ml, eiberg h, moller n, niebuhr e, mohr j. Dyslexia and chromosome 15 heteromorphism: negative lod score in a Danish material.
Clin Genet. 1987;
32:118- 119

[5] brzustowicz lm. Molecular genetic approaches to the study of language.
Hum Biol. 1998;
70:325- 345

[6] cardon lr, smith sd, fulker dw, kimberling wj, pennington bf, defries jc. Quantitative trait locus for reading disability on chromosome 6.
Science. 1994;
266:276- 279

[7] cardon lr, smith sd, fulker dw, kimberling wj, pennington bf, defries jc. Quantitative trait locus for reading disability: correction.
Science. 1995;
268:1553

[8] castles a, datta h, gayán j, olson rk. Varieties of developmental reading disorder: genetic and environmental influences.
Journal of Experimental Child Psychology. 1999;
72:73- 94

[9] cope na, hill g, van den bree m, harold d, moskvina v. No support for association between dyslexia susceptibility 1 candidate 1 and developmental dyslexia.
Mol Psychiatry. 2005a;
10:237- 238

[10] cope n, harold d, hill g, moskvina v, stevenson j, holmans p. Strong evidence that KIAA0319 on chromosome 6p is a susceptibility gene for developmental dyslexia.
Am J Hum Genet. 2005b;
76:581- 591

[11] daigneault s, braun cm. Pure Severe Dyslexia After a Perinatal Focal Lesion: Evidence of a Specific Module for Acquisition of Reading.
J Dev Behav Pediatr. 2002;
23:256- 265

[12] defries jc, fulker dw, labuda mc. Evidence for a genetic aetiology in reading disability of twins.
Nature. 1987;
329:537- 539

[13] fagerheim t, raeymaekers p, tonnessen fe, pedersen m, tranebjaerg l, lubs ha. A new gene (DYX3) for dyslexia is located on chromosome 2.
J Med Genet. 1999;
36:664- 669

[14] fisher se, francks c. Genes, cognition and dyslexia: learning to read the genome.
Trends in Cognitive Sciences. 2006;
10:250- 257

[15] fisher se, marlow aj, lamb j, maestrini e, williams df. A quantitative-trait locus on chromosome 6p influences different aspects of developmental dyslexia.
Am J Hum Genet. 1999;
64:146- 156

[16] fisher se, francks c, marlow aj, macphie il, newbury df. Independent genome-wide scans identify a chromosome 18 quantitative-trait locus influencing dyslexia.
Nat Genet. 2002;
30:86- 91

[17] flax jf, realpe-bonilla t, hirsch ls, brzustowicz lm, bartlett cw, tallal p. Specific language impairment in families: evidence for co-occurrence with reading impairments.
J Speech Lang Hear Res. 2003;
46:530- 543

[18] francks c, paracchini s, smith sd, richardson aj, scerri ts. A 77-kilobase region of chromosome 6p22.2 is associated with dyslexia in families from the United Kingdom and from the United States.
Am J Hum Genet. 2004;
75:1046- 1058

[19] galaburda am, sherman gf, rosen gd, aboitiz f, geschwind n. Developmental dyslexia: four consecutive patients with cortical anomalies.
Ann Neurol. 1985;
18:222- 233

[20] galaburda am, loturco j, ramus f, fitch rh, rosen gd. From genes to behavior in developmental dyslexia.
Nature Neuroscience. 2006;
9:1213- 1217

[21] gayán j, smith sd, cherny ss, cardon lr, fulker dw, brower am. Quantitative-trait locus for specific language and reading deficits on chromosome 6p.
Am J Hum Genet. 1999;
64:157- 164

[22] grigorenko el, wood fb, meyer ms, hart la, speed wc. Susceptibility loci for distinct components of developmental dyslexia on chromosomes 6 and 15.
Am J Hum Genet. 1997;
60:27- 39

[23] grigorenko el, wood fb, meyer ms, pauls dl. Chromosome 6p influences on different dyslexia-related cognitive processes: further confirmation.
Am J Hum Genet. 2000;
66:715- 723

[24] grigorenko el, wood fb, meyer ms, pauls je, hart la, pauls dl. Linkage studies suggest a possible locus for developmental dyslexia on chromosome 1p.
Am J Med Genet. 2001;
105:120- 129

[25] grigorenko el, wood fb, golovyan l, meyer m, romano c, pauls d. Continuing the search for dyslexia genes on 6p.
Am J Med Genet. 2003;
118B:89- 98

[26] hallgren b. Congenital word-blindness. A clinical and genetic study.
Acta Psychiatrica et Neurologica. 1950;
suppl 65:1- 287

[27] hannula-jouppi k, kaminen-ahola n, taipale m, eklund r, nopola-hemmi j. The axon guidance receptor gene ROBO1 is a candidate gene for developmental dyslexia.
Plos Genetics. 2005;
1:e50

[28] kaplan de, gayan j, ahn j, won tw, pauls d. Evidence for linkage and association with reading disability on 6p21.3-22.
Am J Hum Genet. 2002;
70:1287- 1298

[29] knopik vs, defries jc. Etiology of covariation between reading and mathematics performance: A twin study.
Twin Research. 1999a;
2:226- 234

[30] knopik vs, defries jc. Reading and mathematics performance in twin pairs with and without reading difficulties.
Behavior Genetics. 1999b;
29:360- 361

[31] londin er, meng h, gruen jr. A transcription map of the 6p22.3 reading disability locus identifying candidate genes.
BMC Genomics. 2003;
4:25

[32] marino c, giorda r, luisa lorusso m, vanzin l, salandi n. A family-based association study does not support DYX1C1 on 15q21.3 as a candidate gene in developmental dyslexia.
Eur J Hum Genet. 2005;
13:491- 499

[33] meng h, hager k, held m, page gp, olson rk. TDT-association analysis of EKN1 and dyslexia in a Colorado twin cohort.
Hum Genet. 2005a;
118:87- 90

[34] meng h, smith sd, hager k, held m, liu j, olson rk. DCDC2 is associated with reading disability and modulates neuronal development in the brain.
Proc Natl Acad Sci USA. 2005;
102:17053- 17058 – Erratum in: Proc Natl Acad Sci USA 2005b, 102 : 18763

[35] morris dw, robinson l, turic d, duke m, webb v. Family-based association mapping provides evidence for a gene for reading disability on chromosome 15q.
Hum Mol Genet. 2000;
9:843- 848

[36] nopola-hemmi j, myllyluoma b, haltia t, taipale m, ollikainen v. A dominant gene for developmental dyslexia on chromosome 3.
J Med Genet. 2001;
38:658- 664

[37] nopola-hemmi j, myllyluoma b, voutilainen a, leinonen s, kere j, ahonen t. Familial dyslexia: neurocognitive and genetic correlation in a large Finnish family.
Developmental Medicine and Child Neurology. 2002;
44:580- 586

[38] nothen mm, schulte-körne g, grimm t, cichon s, vogt ir. Genetic linkage analysis with dyslexia: evidence for linkage of spelling disability to chromosome 15.
Eur Child Adolesc Psychiatry. 1999;
8:56- 59

[39] oliver b, harlaar n, thomas meh, kovas y, walker so. A twin study of teacher-reported mathematics performance and low performance in 7-year-olds.
Journal of Educational Psychology. 2004;
96:504- 517

[40] olson rk, gillis jj, rack jp, fulker dw. Specific deficits in component reading and language skills: genetic and environmental influences.
Journal of Learning Disabilities. 1989;
22:339- 348

[41] paracchini s, thomas a, castro s, lai c, paramasivam m. The chromosome 6p22 haplotype associated with dyslexia reduces the expression of KIAA0319, a novel gene involved in neuronal migration.
Hum Mol Genet. 2006;
15:1659- 1666

[42] petryshen tl, kaplan bj, hughes ml, tzenova j, field ll. Supportive evidence for the DYX3 dyslexia susceptibility gene in Canadian families.
J Med Genet. 2002;
39:125- 126

[43] plomin r. The Emanuel Miller Memorial Lecture 1993. Genetic research and identification of environmental influences.
J Child Psychol Psychiatry. 1994;
35:817- 834

[44] rabin m, wen xl, hepburn m, lubs ha, feldman e, duara r. Suggestive linkage of developmental dyslexia to chromosome 1p34-p36.
Lancet. 1993;
342:178

[45] ramus f. Neurobiology of dyslexia: a reinterpretation of the data.
Trends Neurosci. 2004;
27:720- 726

[46] scerri ts, fisher se, francks c, macphie il, paracchini s. Putative functional alleles of DYX1C1 are not associated with dyslexia susceptibility in a large sample of sibling pairs from the UK.
J Med Genet. 2004;
41:853- 857

[47] schulte-körne g, grimm t, nothen mm, muller-myhsok b, cichon s. Evidence for linkage of spelling disability to chromosome 15.
Am J Hum Genet. 1998;
63:279- 282

[48] schumacher j, anthoni h, dahdouh f, könig ir, hillmer am. A strong genetic evidence for DCDC2 as a susceptibility gene for dyslexia.
Am J Hum Genet. 2006;
78:52- 62

[49] smith sd, kimberling wj, pennington bf, lubs ha. Specific reading disability: identification of an inherited form through linkage analysis.
Science. 1983;
219:1345- 1347

[50] smith sd, kimberling wj, pennington bf. Screening for multiple genes influencing dyslexia.
Reading and Writing. 1991;
3:285- 298

[51] snowling mj, gallagher a, frith u. Family risk of dyslexia is continuous: individual differences in the precursors of reading skill.
Child Dev. 2003;
74:358- 373

[52] stephenson s. Six cases of congenital word-blindness affecting three generations of one family.
Ophthalmoscope. 1907;
5:482- 484

[53] stevenson j, graham p, fredman g, mcloughlin v. A twin study of genetic influences on reading and spelling ability and disability.
Journal of Child Psychology and Psychiatry. 1987;
28:229- 247

[54] stromswold k. The heritability of language: A review and metaanalysis of twin, adoption, and linkage studies.
Language. 2001;
77:647- 723

[55] taipale m, kaminen n, nopola-hemmi j, haltia t, myllyluoma b. A candidate gene for developmental dyslexia encodes a nuclear tetratricopeptide repeat domain protein dynamically regulated in brain.
Proc Natl Acad Sci USA. 2003;
100:11553- 11558

[56] turic d, robinson l, duke m, morris dw, webb v. Linkage disequilibrium mapping provides further evidence of a gene for reading disability on chromosome 6p21.3-22.
Mol Psychiatry. 2003;
8:176- 185

[57] vogler gp, defries jc, decker sn. Family history as an indicator of risk for reading disability.
J Learn Disabil. 1985;
18:419- 421

[58] wang y, paramasivam m, thomas a, bai j, kaminen-ahola n. DYX1C1 functions in neuronal migration in developing neocortex.
Neuroscience. 2006;
143:515- 522

[59] wigg kg, couto jm, feng y, anderson b, cate-carter td. Support for EKN1 as the susceptibility locus for dyslexia on 15q21.
Mol Psychiatry. 2004;
9:1111- 1121

[60] wolff ph, melngailis i. Family patterns of developmental dyslexia: Clinical findings.
American Journal of Medical Genetics. 1994;
54:122- 131

→ Aller vers SYNTHESE