| |
| Med Sci (Paris). 36(8-9): 717–724. doi: 10.1051/medsci/2020126.La présentation sur ribosome Évolution et sélection acellulaire de banques moléculaires Priscillia Lagoutte1* 1Univ Lyon, CNRS, Laboratoire de biologie tissulaire et ingénierie thérapeutique, LBTI, UMR 5305
.
7 passage du Vercors
,
F-69637
,
Lyon
,
France |
En octobre 2018, le prix Nobel de chimie a été décernée à Frances H. Arnold, Georges P. Smith et Sir Greg P. Winter pour leur travaux sur l’ingénierie protéique qui a conduit au développement des méthodes d’évolution moléculaire dirigée des protéines [
1
] (
→
).
(→) Voir le Repères de P. Minard, m/s n°2, février 2019, page 169
L’exposition sur phage (en anglais,
phage displa
y), mise au point par Georges Smith en 1985, a représenté une innovation majeure en introduisant les méthodes combinatoires en biologie, appliquées aux peptides, et qui, quelques années plus tard, a été transposée aux protéines, en particulier aux anticorps. Cette innovation a entraîné une vraie révolution dans l’industrie pharmaceutique [
2
]. La technique du
phage display
consiste à utiliser comme outils de présentation des bactériophages (ou phages, des virus infectant les bactéries) qui expriment à leur surface des peptides ou des protéines issus d’une banque
(
Figure 1
)
. Un très grand nombre de variants protéiques (des millions à plusieurs centaines de millions) peuvent être exposés à la surface des phages, chacun étant représentatif d’un variant de la banque protéique/peptidique. Des cycles itératifs de sélection permettent d'obtenir des phages exprimant des peptides possédant de nouvelles propriétés de ligand vis-à-vis d’une cible choisie. Si, initialement, cette méthode a été développée pour identifier des peptides se liant à la cible, elle a vite été adaptée aux protéines, ce qui a permis de sélectionner, parmi des banques de fragments d’anticorps exprimés par les phages, ceux qui étaient spécifiques d’un antigène cible choisi. Au fil du temps, cette méthode a été utilisée par un nombre grandissant de laboratoires et a rendu possible le criblage et la sélection de peptides, de protéines, d’échafaudages moléculaires, afin d’améliorer leur affinité, leur spécificité, leur activité catalytique ou même leur stabilité, en fonction de la cible choisie [
3
-
5
]. Aujourd’hui, l’exposition sur phage reste la méthode de référence pour la sélection de ligands. Cependant, le
phage display
présente quelques limites. La première est la taille des banques criblées (c’est-à-dire le nombre réel de séquences que l’on peut tester simultanément) qui est limitée par les efficacités de transformation bactérienne lors de la création de la banque de phages
1,
. La seconde limitation est le phénomène d’avidité lié à la possible présentation sous forme de multiples copies d’un ligand protéique sur un même phage
2,
, ce qui biaise la sélection. La dernière limitation du
phage display
est le biais d’expression. En effet, l’expression d’un variant protéique à la surface d’un phage peut être toxique et empêcher son assemblage correct conduisant alors à sa contre-sélection ; à l’inverse, certains variants sont plus facilement exprimés, en raison des codons utilisés par la bactérie. Ils seront alors sélectionnés préférentiellement. C’est ainsi que ces dernières années, des méthodes de présentation alternatives ont été développées. C’est le cas notamment de l’exposition sur ribosome (en anglais,
ribosome display
), une méthode fondée sur la formation d’un complexe ternaire ARN-ribosome-protéine en milieu acellulaire. Le
ribosome display
est, de nos jours, devenu l’une des méthodes d’exposition les plus utilisées. Il présente de nombreux avantages par rapport aux méthodes de présentations «
in vivo »
3
comme le
phage display
.
 | Figure 1.
Cycle de sélection du phage display.
La banque de phages est criblée et sélectionnée contre une cible d’intérêt (1 et 2). Après plusieurs lavages (3), seul les phages capables d’interagir sont retenus. Ces derniers sont élués (4) et utilisés pour infecter un tapis bactérien (5). Les phages produits et amplifiés (6) peuvent alors être utilisés pour un nouveau cycle de sélection (7).
|
|
Mise en œuvre du ribosome display
La présentation sur ribosome (ou
ribosome display
) a été développée en 1994 par Mattheakis
et al.
[
6
]. Elle se fonde sur la formation d’un lien physique entre l’information phénotypique (la protéine) et l’information génotypique (l’ARN). Un complexe entre l’ARN et la protéine se forme grâce au ribosome (ribonucléoprotéine ou RNP). L’exposition sur ribosome et le succès des cycles de sélection reposent sur la formation d’un complexe ternaire stable et la fonctionnalité des variants protéiques qui sont exposés à la surface du ribosome.
Le ribosome est une machinerie moléculaire dont la fonction est de synthétiser les protéines à partir de l’ARN messager correspondant. Dans le cas de la synthèse protéique, les triplets de nucléotides sont traduits en acides aminés. En fin de traduction, le ribosome rencontre un triplet de nucléotides correspondant à un codon STOP (TAA, TGA, TAG), ce qui conduit à sa dissociation à la fois de l’ARN et de la protéine nouvellement synthétisée
(
Figure 2A
)
. Dans le cas du
ribosome display
, le système a été modifié pour que le complexe ternaire ribonucléoprotéine formé entre l’ARN messager (ARNm), le ribosome et la protéine nouvellement synthétisée
(
Figure 2B
)
ne puisse pas se dissocier. L’information génétique (l’ARN) reste alors liée à la protéine correspondante qui se replie à l’extérieur du ribosome. Le succès de la présentation de protéines sur ribosome repose donc sur trois éléments : 1) la formation des complexes ternaires ribonucléoprotéiques, 2) la stabilité des complexes ternaires et 3) le repliement correct des protéines exposées à la surface des ribosomes.
 | Figure 2.
Mécanismes de traduction par le ribosome (A) dans le cas de la synthèse d’une protéine et (B) dans le cas du ribosome display.
|
La formation du complexe ternaire ARN-ribosome-protéine nécessite l’absence d’un codon « stop » sur la séquence nucléotidique codant la protéine à synthétiser. En effet, cette absence de codon « stop » ne permet pas la fixation du facteur de terminaison de la traduction et empêche donc la dissociation des deux sous-unités du ribosome
(
Figure 2B
)
. Cependant, dans ces conditions, le temps de demi-vie du complexe ternaire est court et, à terme, les deux sous-unités ribosomales peuvent se dissocier. Pour renforcer la stabilité du complexe, une séquence d’arrêt du ribosome est fusionnée à la séquence codante de la protéine à synthétiser. Une séquence en particulier est décrite dans le cas du
ribosome display
: la séquence SecM. La protéine SecM est impliquée dans la régulation de la traduction du gène
secA
d’
Escherichia coli
, qui intervient dans la voie de la sécrétion de la bactérie. Au niveau de la région C-terminale de la protéine SecM, se trouve un motif de dix-sept acides aminés
150
FSTPVWISQAQGIRAGP
166
, qui comporte notamment une proline en position 166, signal d’arrêt de l’élongation [
7
]. Au moment de la traduction de la proline, la conformation des sites A (site de liaison de l’aminoacyl-ARNt) et P (site de liaison du peptidyl-ARNt) du ribosome est modifiée, ce qui induit de nouvelles interactions entre le ribosome, l’ARN et la protéine. Ainsi, la fusion de la séquence SecM à la fin de la séquence de la protéine à synthétiser n’interfère pas avec sa traduction, mais empêche le ribosome de se dissocier de la protéine et de son ARN codant
(
Figure 2B
)
. L’ARNm, sensible à l’action des RNases, est fragile. Il est donc important de préserver son intégrité pour pouvoir identifier le variant interagissant avec la cible. Pour éviter la dégradation de l’ARN, des boucles sont ajoutées en 5’ et 3’ de la séquence nucléique. Toutefois, la formation du complexe ternaire et sa stabilité ne suffisent pas à l’obtention de variants protéiques fonctionnels liés à la présence du ribosome. En fait, il est nécessaire que la protéine nouvellement synthétisée se replie entièrement et correctement à l’extérieur du ribosome. Ainsi, le dernier élément indispensable pour le bon fonctionnemment du
ribosome display
est l’ajout d’une séquence «
linker
» (ou lien peptidique) codant une protéine non structurée, en aval de la séquence protéique
(
Figure 3A
)
. Grâce à ce
linker
fusionné à la protéine, celle-ci est projetée à l’extérieur du ribosome, où elle peut se replier. Des portions de séquences de la protéine P3 du phage M13 (riche en sérine et glycine), de la protéine D du phage λ, de l’hélice de Tol A ou de la protéine tonB d’
E. coli
, riche en proline, ont été décrites comme des
linker
efficaces dans le cadre du
ribosome display
[
8
].
 | Figure 3.
Principe du ribosome display. A.
Séquence nucléotidique utilisée dans le cadre du
ribosome display
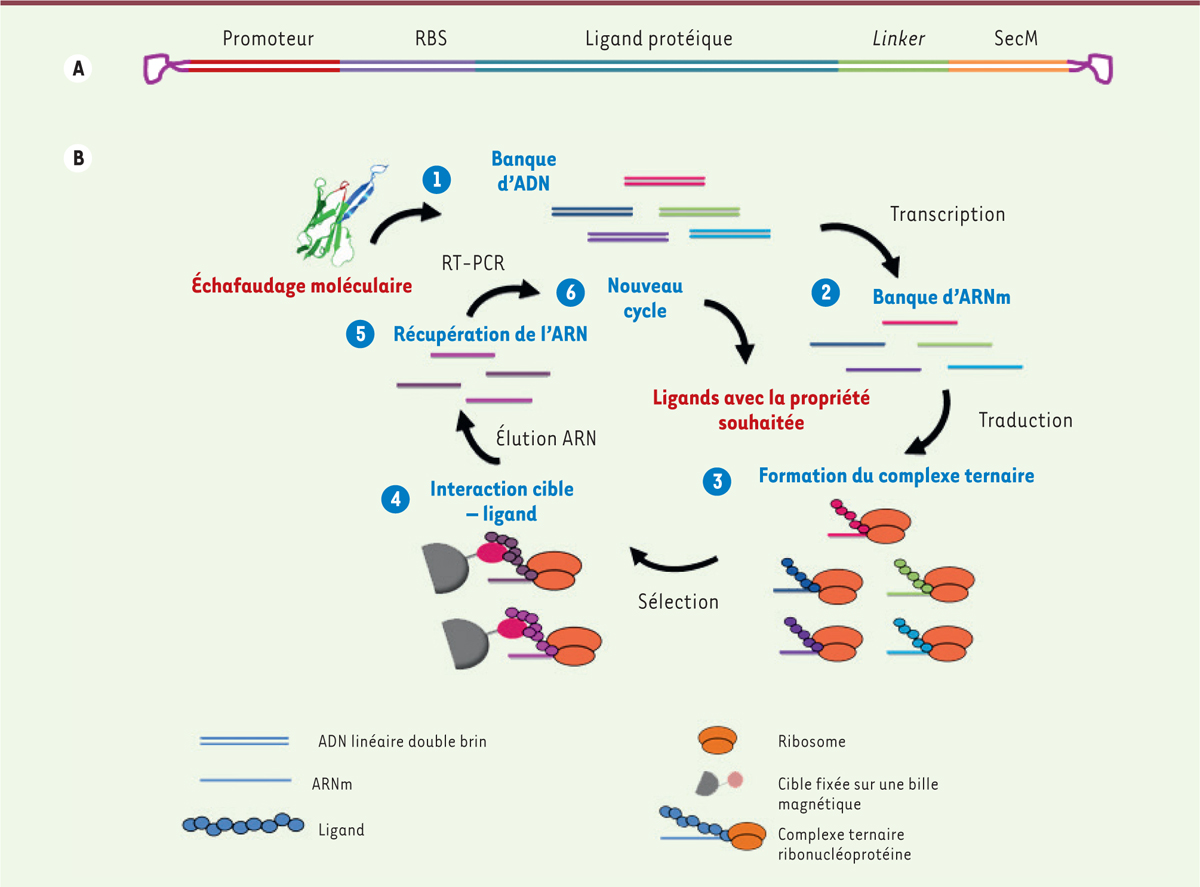
pour permettre la formation et la stabilité du complexe ternaire. La séquence contient un promoteur, un site de fixation des ribosomes (RBS), les séquences codant les ligands protéiques, un linker et enfin une séquence d’arrêt du ribosome (SecM).
B..
Cycle de sélection du
ribosome display
. La banque d’ADN est transcrite en ARNm (1 et 2) qui est alors traduit pour permettre la formation des complexes ternaires ARNm-ribosome-protéine (3). Les complexes ternaires sont ensuite sélectionnés contre une cible d’intérêt (4). L’ARNm des complexes retenus contre la cible est ensuite récupéré (4) et rétro-transcrit en ADN (5) pour un nouveau cycle de sélection (6).
|
|
Cycle de sélection par exposition sur ribosome
La sélection de protéines ou de peptides par exposition sur ribosome s’effectue en plusieurs étapes [
8
,
9
]. La première consiste à générer une banque d’ADN la plus diversifiée possible. L’ADN contient un promoteur, un site de fixation au ribosome (RBS) et la séquence nucléotidique de la banque de variants fusionnée en 3’ aux séquences codant le
linker
suivi de la séquence SecM d’arrêt du ribosome
(
Figure 3A
)
. La préparation de la banque d’ADN est réalisée par l’assemblage de fragments obtenus par PCR (
polymerase chain reaction
) à partir de séquences d’ADN
(
Figure 3B
, étape 1)
. La banque d’ADN ainsi obtenue est ensuite transcrite et traduite en système acellulaire
(
Figure 3B
, étapes 2 et 3)
en utilisant des lysats cellulaires eucaryotes (réticulocytes de lapins, cellules humaines, germes de blé, cellules d’insectes, etc.) ou procaryotes (
E. coli
S30), ou des milieux cellulaires reconstitués (
PURExpress
). Les complexes ternaires formés dans le milieu acellulaire sont ensuite criblés et sélectionnés par interaction vis-à-vis d’une cible d’intérêt
(
Figure 3B
, étape 4)
. Les sélections peuvent être réalisées, en fonction de conditions définies par l’expérimentateur, sur des cibles en solution, ou préalablement immobilisées sur des surfaces, comme des plaques immuno-adsorbantes, des puces ou encore des billes magnétiques. Contrairement aux méthodes d’exposition sur cellules (ou sur phages), des conditions de sélections beaucoup plus drastiques (utilisation de réactifs toxiques pour les cellules, pH ou températures extrêmes, etc.) peuvent être utilisées en milieu acellulaire. Ce processus de sélection permet de ne retenir que les variants protéiques possédant la fonction souhaitée. Le complexe ternaire ainsi sélectionné permettra d’identifier l’ARNm correspondant toujours associé
(
Figure 3B
, étape 5)
. Deux méthodes de récupération de la séquence peuvent être utilisées. La première repose sur l’addition d’EDTA (acide éthylènediamine tétraacétique) qui chélate les ions divalents nécessaires à la stabilité du ribosome. Le ribosome se dissocie alors libérant l’ARNm qui peut être amplifié sous forme d’ADNc par transcription inverse (RT-PCR)
(
Figure 3B
, étape 6)
. La seconde méthode est spécifique des milieux acellulaires eucaryotes. Dans ce cas, la RT-PCR
in situ
est réalisée directement sur l’ARNm encore associé au complexe. Dans les deux cas, l’ADN obtenu après rétro-transcription à la fin du cycle de sélection est utilisé soit pour un nouveau cycle de sélection, soit pour être cloné et séquencé et ainsi conduire à l’identification du ou des variants protéiques enrichis.
Plusieurs cycles de
ribosome display
peuvent être réalisés de manière itérative en augmentant les contraintes afin de sélectionner un ligand protéique capable d’interagir avec sa cible avec une forte affinité ou dont la stabilité, ou l’activité catalytique, ont été améliorées.
|
Application et utilisation du ribosome display |
Sélection et évolution d’anticorps
L’une des premières utilisations de l’exposition sur ribosome a été de cribler une banque de fragments d’anticorps du type scFv
4
[
10
,
31
] (
→
).
(→) Voir le numéro thématique
Anticorps monoclonaux en thérapeutique
,
m/s
n° 12, décembre 2019, pages 915-1232
Depuis, le
ribosome display
a été utilisé fréquemment pour la sélection, l’évolution et même la maturation des anticorps. Une étude de 2016 a par exemple révélé la puissance du
ribosome display
couplé aux puces protéiques pour cribler à moyen débit plusieurs fragments d’anticorps contre une vingtaine d’antigènes différents [
11
]. Plus récemment, une étude a permis la génération d’un anticorps neutralisant contre le virus Zika [
12
].
Les fragments d’anticorps du type
nanobody
(ou VHH), dérivant de domaines variables des anticorps de camélidés, sont de plus en plus utilisés et plusieurs plateformes académiques ou sociétés privées (
Hybrigenics
,
Ablynx
) ont fondé leur développement d’outils sur cet échafaudage moléculaire. En 2019, un premier nanobody, le caplacizumab, qui cible le facteur de Willebrand (vWF) dans le cas de la maladie de Moschcowitz (ou purpura thrombotique thrombocytopénique)
5
, a ainsi été approuvé par la FDA (
Food and Drug Administration
) et mis sur le marché [
13
]. Actuellement, des banques de VHH synthétiques, d’une taille supérieure à 10
12
molécules, sont développées pour être utilisées pour la sélection de
nanobodies
par
ribosome display
. Elles permettent d’obtenir en quelques semaines des nanobodies contre une cible d’intérêt sans nécessiter les étapes d’immunisation des Camélidés [
14
].
|
Sélection de ligands protéiques
Les anticorps sont des protéines qui, par leur plasticité moléculaire et les possibilités d’ingénierie qu’elles offrent, représentent des outils de choix en recherche, dans le domaine du diagnostic et en thérapeutique. Cependant, certaines caractéristiques intrinsèques des anticorps (chaîne polypeptidique, présence de ponts disulfures, difficultés de production, etc.) ont conduit la communauté scientifique à se tourner vers des alternatives de type ligand protéique. Les ligands protéiques sont des échafaudages moléculaires protéiques dont la structure présente des positions hautement flexibles et qui tolèrent des modifications. La génération de banques de variants protéiques de ces échafaudages moléculaires permet d’obtenir des ligands dont les propriétés natives ont été modifiées et de les faire évoluer pour répondre à de nouvelles fonctions. Ainsi, des ligands dérivant de domaines répétés [
15
], de domaines protéiques d’interaction [
16
], ou encore de protéines entières [
17
], ont servi d’échafaudage moléculaire pour générer des banques de grande diversité. Pas moins d’une vingtaine de ligands protéiques ont aujourd’hui été décrits
(
Tableau I
)
. Des sociétés comme
Molecular Partners
,
Affibody
ou
Affilogic
utilisent le
ribosome display
pour obtenir des ligands à des fins diagnostiques, thérapeutiques ou à destination de la recherche fondamentale. Ainsi, plusieurs DARPins (
designed ankyrin repeat proteins
)
6
générés par la plateforme de
Molecular Partners
sont en phases II et III d’essais cliniques en oncologie, en ophtalmologie ou en immuno-oncologie. C’est le cas notamment de la molécule Abicipar qui traite la dégénérescence maculaire néovasculaire liée à l’âge [
18
,
19
]. Les résultats concluants des essais de phase III ont conduit à la demande de commercialisation du produit auprès de la FDA pour une mise sur le marché en 2020. La société
Affibody
commercialise aujourd’hui plusieurs Affibodies qui, de la même façon que les anticorps, peuvent être utilisés pour la recherche pour la purification ou la détection de protéines en imagerie ou en protéomique [
20
,
21
].
Affilogic
, l’une des seules compagnies de biotechnologie française active dans ce domaine, utilise la technologie
ribosome display
pour la sélection de ses molécules propriétaires : les affitines/nanofitines. Les nanofitines, dérivant de protéines hyperstables capables d’interagir avec l’ADN (Sso7d et Sac7d de
Sulfolobus solfataricus
, Aho7d d’
Acidianus hospitalis
), ont aujourd’hui été sélectionnées contre une soixantaine de cibles pour des applications en recherche, diagnostique et thérapeutique. À titre d’exemple, des nanofitines ont été utilisées pour le suivi de tumeurs, en utilisant la tomographie par émission de positons (TEP) [
22
].
Tableau I.
| Échafaudage moléculaire protéique |
Ossature protéique parentale |
|
ABD (
albumine-binding domaine
)
|
Albumine |
| Alpharep |
Hélice artificielle issue des domaines HEAT
a
|
| Adhiron |
Protéine phytostatine |
| Adnectine |
Domaine 10 de la fibronectine humaine III |
| Affibodies |
Domaine Z de la protéine A de
Staphyloccocus aureus
|
| Affiline |
γ-β cristalline |
| Affiline |
Ubiquitine |
| Affimère |
Inhibiteur de la cystéine protéase Stéfine-A |
| Affitine/ Nanofitine |
Sso7d, Sac7d, Aho7d (Protéines se liant à l’ADN) |
| Anticaline |
Lipocaline |
| Antidine |
Monomère d’avidine |
| Armadillo |
Motif armadillo trouvé dans la β-caténine |
| Atrimère |
Lectine de type C |
| Avimère |
Domaine A des récepteurs LDL (lipoprotéines de faible densité) |
| β-Hairpin |
Épingle à cheveux |
| Darpins |
Domaine ankyrine |
| Gp2 |
Protéine 2 du phage T7 |
| Kringle |
Domaine de la protéine plasmine |
| Pronectine |
14
e
domaine extracellulaire de la fibronectine III
|
| Pyrazinamidase |
Enzyme pyrazynamidase |
| Knottine |
Miniprotéine à nœud cystéine |
| Domaine Kunitz |
Inhibiteur des protéases à sérine |
| Repebodies |
Domaine répété riche en leucine issu des récepteurs des lymphocytes |
Exemples de ligands protéiques utilisés pour le criblage par ribosome display.
|
|
Sélection de banques de peptides
Le
ribosome display
a été utilisé pour le criblage de banque peptidiques à des fins diagnostiques et thérapeutiques. À titre d’exemple, un peptide 12-mer (de 12 acides aminés) interagissant avec une protéine d’enveloppe du virus de l’hépatite C a été obtenu par
ribosome display
après 13 cycles de sélection de la banque peptidique sur un complexe présentant la protéine virale. Grâce à son interaction avec le virus, ce peptide peut être utilisé pour détecter précocement l’infection, mais également en thérapeutique, par sa capacité à inhiber l’infectivité du virus [
23
]. Ce type de criblage a également permis d’identifier les épitopes reconnus par des anticorps. Dans ce cas, il a été démontré que deux ou trois cycles de sélection par
ribosome display
suffisent pour identifier un épitope à partir de banques de peptides comptant de 10
8
à 10
11
variants [
24
].
|
Le
ribosome display
a été appliqué dans le but de faire évoluer des protéines afin de leur conférer de nouvelles propriétés, que ce soit en termes de stabilité, de spécificité ou d’affinité. Une banque de variants de la transcriptase inverse M-MuLV (
Moloney murine leukemia virus
) du virus Moloney de la leucémie murine, a ainsi été générée dans le but d’identifier un mutant de la protéine qui soit le plus actif. Dans cette étude, les réactions de
ribosome display
ont été compartimentalisées par émulsion (une méthode visant à individualiser chaque élément d’une population grâce à une émulsion eau-huile), de telle sorte qu’un seul variant de la banque soit représenté par compartiment. Dans chaque compartiment, l’activité transcriptase inverse de la protéine mutée sélectionnée est testée en vérifiant sa capacité de synthèse d’ADNc à partir d’un ARNm. Par cette méthode, des variants thermostables et fonctionnels de la protéine ont été identifiés et isolés [
25
]. Un autre exemple d’évolution des protéines par
ribosome display
est la sélection de variants enzymatiques fonctionnels. Plusieurs mutants de la β-lactamase TEM-1, une enzyme impliquée dans la résistance des bactéries aux antibiotiques, ont ainsi été criblés par
ribosome display
vis-à-vis d’un inhibiteur spécifique de l’enzyme bactérienne (BLIP, pour
beta-lactamase-inhibitory protein
). Seul un variant ayant conservé son activité enzymatique, et ayant donc échappé à l’inhibition par BLIP, a été sélectionné, validant l’approche d’évolution et de sélection des enzymes par
ribosome display
[
26
].
|
La puissance et les limites du ribosome display
Le
ribosome display
connaît depuis plusieurs années une attention particulière de la communauté scientifique. Cette méthode de sélection ne nécessite pas le passage par une amplification
in cellulo
(les bactéries pour les phages)
,
ce qui réduit considérablement le temps de sélection et augmente la taille des banques qu’il est possible de cribler par rapport à d’autres approches, comme le
phage display
. Cependant, la présence dans le cycle de sélection d’une étape d’amplification par PCR peut être à l’origine de l’introduction de mutations dans l’ARNm et conduire à une maturation d’affinité ou de spécificité
in vitro
. Le
ribosome display
reste une méthode difficile à mettre en œuvre, notamment en raison de la manipulation des ARNm, qui sont fragiles, et peu de laboratoires maîtrisent cette technique encore maintenant. Des progrès récents devraient permettre d’utiliser pleinement la puissance du
ribosome display
et de pallier ces inconvénients. Ainsi, des optimisations sont réalisées afin de maximiser la stabilité du complexe ternaire. La séquence SecM a ainsi été améliorée et de nouveaux systèmes d’expression acellulaire, dépourvus de nucléases, ont été développés [
27
,
28
]. L’avènement de la biologie synthétique et de nouvelles technologies pour la génération de banques donnent désormais toute sa puissance au
ribosome display
. En effet, les oligonucléotides synthétisés par bloc de trinucléotides remplacent les oligonucléotides traditionnels pour l’assemblage de fragments d’ADN dégénérés. La synthèse de banques à partir des trinucléotides phosphoramidites présente l’avantage de pouvoir contrôler le ratio des différents acides aminés à chaque position de la banque, d’éviter la présence de codons « stop » prématurés et de permettre l’obtention de banques de très grandes diversités [
29
]. La diversité de ces banques est également élargie avec la possibilité d’ajouter des acides aminés non naturels sur lesquels des groupements fonctionnels non canoniques ont été ajoutés. Ces acides aminés peuvent être directement introduits dans la banque après une reprogrammation du code génétique. En d’autres termes, des quadruplets ou des triplets de nucléotides codant pour un STOP peuvent correspondre à un acide aminé non standard. Au moment de la traduction, ces acides aminés sont ajoutés à la séquence protéique. Cela est d’autant plus facile à mettre en œuvre qu’on utilise un milieu acellulaire recombinant. En effet, ces systèmes utilisent des composés purifiés dans lesquels il est facile d’introduire ou d’éliminer des composants. Ainsi, des acides aminés non naturels, synthétiques, peuvent être ajoutés dans le milieu acellulaire, de même que des facteurs de terminaison peuvent être supprimés dans le cadre du
ribosome display
. Pour finir, afin d’utiliser au maximum les capacités d’exploration du
ribosome display
, celui-ci a été couplé à des méthodes de séquençage haut débit ou à des puces protéiques [
11
,
30
]. Les puces protéiques permettront en parallèle le criblage et la sélection de variants vis-à-vis de plusieurs cibles. Le séquençage haut débit permettra, quant à lui, de multiplexer l’identification des ligands sélectionnés et faire ressortir des ligands rares. En effet, pour un même séquençage, il sera possible d’identifier en parallèle, des ligands issus de différentes sélections, selon la profondeur de séquençage (plusieurs milliers). Les outils bio-informatiques mis en œuvre pour l’analyse des données de séquençage aideront à l’identification de motifs d’interactions ou à déterminer des positions conservées importantes pour la stabilité ou l’affinité du ligand. Le
ribosome display
permet donc en quelques semaines le criblage de banques de grande taille (plusieurs ordres de grandeurs au-dessus du
phage display
) et la sélection de variants protéiques dont l’affinité contre une cible, la stabilité dans un tampon ou encore l’activité catalytique sont améliorées.
|
Ce travail bénéficie d’un financement de l’ANR (projet CardiActiv, ANR-17-CE14-0033-01).
L’auteure déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Je tiens à remercier Sandrine Vadon-Le Goff pour sa relecture critique et pour ses conseils.
|
Footnotes |
1.
Minard
P.
L’évolution dirigée des protéines. Med Sci (Paris).
2019;
;
35
:
:169.
–
175
.
2.
Smith
GP
.
Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface.
.
Science.
1985;
;
228
:
:1315.
–
1317
.
3.
Lowman
HB
,
Wells
JA
.
Affinity maturation of human growth hormone by monovalent phage display.
.
J Mol Biol.
1993;
;
234
:
:564.
–
578
.
4.
Clackson
T
,
Wells
JA
.
In vitro selection from protein and peptide libraries.
.
Trends Biotechnol.
1994;
;
12
:
:173.
–
184
.
5.
Ledsgaard
L
,
Kilstrup
M
,
Karatt-Vellatt
A
, et al.
Basics of antibody phage display technology.
.
Toxins (Basel).
.
2018
;
:10.
.
6.
Mattheakis
LC
,
Bhatt
RR
,
Dower
WJ
.
An in vitro polysome display system for identifying ligands from very large peptide libraries.
.
Proc Natl Acad Sci USA.
1994;
;
91
:
:9022.
–
9026
.
7.
Tsai
A
,
Kornberg
G
,
Johansson
M
,
et al.
The dynamics of SecM-induced translational stalling.
.
Cell Rep.
2014;
;
7
:
:1521.
–
1533
.
8.
Zahnd
C
,
Amstutz
P
,
Plückthun
A
.
Ribosome display: selecting and evolving proteins in vitro that specifically bind to a target.
.
Nat Methods.
2007;
;
4
:
:269.
–
279
.
9.
Li
R
,
Kang
G
,
Hu
M
,
et al.
Ribosome display: a potent display technology used for selecting and evolving specific binders with desired properties.
.
Mol Biotechnol.
2018;
;
61
:
:60.
–
71
.
10.
Hanes
J
,
Plückthun
A
.
In vitro selection and evolution of functional proteins by using ribosome display.
.
Proc Natl Acad Sci USA.
1997;
;
94
:
:4937.
–
4942
.
11.
Cong
C
,
Yu
X
,
He
Y
,
et al.
Cell-free ribosome display and selection of antibodies on arrayed antigens.
.
Proteomics.
2016;
;
16
:
:1291.
–
1296
.
12.
Kunamneni
A
,
Ye
C
,
Bradfute
SB
,
et al.
Ribosome display for the rapid generation of high-affinity Zika-neutralizing single-chain antibodies.
.
PLoS One.
2018;
;
13
:
:e0205743.
.
13.
Morrison
C.
.
Nanobody approval gives domain antibodies a boost.
.
Nat Rev Drug Discov.
2019;
;
18
:
:485.
–
487
.
14.
Ferrari
D
,
Garrapa
V
,
Locatelli
M
, et al.
A novel nanobody scaffold optimized for bacterial expression and suitable for the construction of ribosome display libraries.
.
Mol Biotechnol.
2020;
;
62
:
:43.
–
55
.
15.
Steiner
D
,
Forrer
P
,
Plückthun
A
.
Efficient selection of DARPins with sub-nanomolar affinities using SRP phage display.
.
J Mol Biol.
2008;
;
382
:
:1211.
–
1227
.
16.
Koide
A
,
Gilbreth
RN
,
Esaki
K
,
et al.
High-affinity single-domain binding proteins with a binary-code interface.
.
Proc Natl Acad Sci USA.
2007;
;
104
:
:6632.
–
6637
.
17.
Kalichuk
V
,
Kambarev
S
,
Béhar
G
, et al.
Affitins: ribosome display for selection of Aho7c-based affinity proteins.
.
Methods Mol Biol.
2020;
;
2070
:
:19.
–
41
.
18.
Kunimoto
D
,
Ohji
M
,
Maturi
RK
,
et al.
Evaluation of abicipar pegol (an anti-VEGF DARPin therapeutic) in patients with neovascular age-related macular degeneration: studies in Japan and the United States.
.
Ophthalmic Surg Lasers Imaging Retina.
2019;
;
50
:
:e10.
–
e22
.
19.
Moisseiev
E
,
Loewenstein
A
.
Abicipar pegol: a novel anti-VEGF therapy with a long duration of action.
.
Eye.
2019
;
:1.
–
2
.
20.
Grönwall
C
,
Sjöberg
A
,
Ramström
M
,
et al.
Affibody-mediated transferrin depletion for proteomics applications.
.
Biotechnol J.
2007;
;
2
:
:1389.
–
1398
.
21.
Lee
SB
,
Hassan
M
,
Fisher
R
,
et al.
Affibody molecules for in vivo characterization of HER2-positive tumors by near-infrared imaging.
.
Clin Cancer Res.
2008;
;
14
:
:3840.
–
3849
.
22.
Goux
M
,
Becker
G
,
Gorré
H
,
et al.
Nanofitin as a new molecular-imaging agent for the diagnosis of epidermal growth factor receptor over-expressing tumors.
.
Bioconjug Chem.
2017;
;
28
:
:2361.
–
2371
.
23.
Chen
F
,
Zhao
Y
,
Liu
M
,
et al.
Functional selection of hepatitis C virus envelope E2-binding peptide ligands by using ribosome display.
.
Antimicrob Agents Chemother.
2010;
;
54
:
:3355.
–
3364
.
24.
Osada
E
,
Shimizu
Y
,
Akbar
BK
,
et al.
Epitope mapping using ribosome display in a reconstituted cell-free protein synthesis system.
.
J Biochem.
2009;
;
145
:
:693.
–
700
.
25.
Skirgaila
R
,
Pudzaitis
V
,
Paliksa
S
,
et al.
Compartmentalization of destabilized enzyme-mRNA-ribosome complexes generated by ribosome display: a novel tool for the directed evolution of enzymes.
.
Protein Eng Des Sel.
2013;
;
26
:
:453.
–
461
.
26.
Amstutz
P
,
Pelletier
JN
,
Guggisberg
A
,
et al.
In vitro selection for catalytic activity with ribosome display.
.
J Am Chem Soc.
2002;
;
124
:
:9396.
–
9403
.
27.
Goldman
DH
,
Kaiser
CM
,
Milin
A
,
et al.
Mechanical force releases nascent chain-mediated ribosome arrest in vitro and in vivo.
.
Science.
2015;
;
348
:
:457.
–
460
.
28.
Ohashi
H
,
Kanamori
T
,
Osada
E
,
et al.
Peptide screening using PURE ribosome display.
.
Methods Mol Biol.
2012
;
:251.
–
259
.
29.
Suchsland
R
,
Appel
B
,
Müller
S
.
Preparation of trinucleotide phosphoramidites as synthons for the synthesis of gene libraries.
.
Beilstein J Org Chem.
2018;
;
14
:
:397.
–
406
.
30.
Lagoutte
P
,
Lugari
A
,
Elie
C
,
et al.
Combination of ribosome display and next generation sequencing as a powerful method for identification of affibody binders against β-lactamase CTX-M15.
.
N Biotechnol.
2019;
;
50
:
:60.
–
69
.
31.
Beck
A
,
Pèlegrin
A
.
Watier H (coordinateurs).
.
Anticorps monoclonaux en thérapeutique. Med Sci (Paris).
2019;
;
35
:
:915.
–
1232
.
|