L’autophagie est un mode de dégradation de molécules endogènes par les lysosomes, activé en réponse à diverses situations de stress et essentiel à l’homéostasie cellulaire et tissulaire [ 1 , 13 ] ( → )
L’autophagie se caractérise par la séquestration de matériel cytoplasmique dans un organelle à double membrane : l’autophagosome. La formation de l’autophagosome fait intervenir une quinzaine de protéines ATG ( autophagy-related ) conservées chez les eucaryotes, et qui s’organisent en complexes moléculaires avec d’autres protéines [ 2 ]. Comme dans de nombreuses autres situations de remodelage membranaire, la nucléation de l’autophagosome mobilise le lipide membranaire phosphatidylinositol-3-phosphate (PI3P) afin de faciliter la mise en place des échafaudages moléculaires nécessaires aux étapes précoces de sa formation. Les membranes contenant le PI3P participent ainsi au recrutement local du système de conjugaison ATG5-ATG12-ATG16L1 et de son partenaire WIPI2 ( WD repeat domain phosphoinositide-interacting protein 2 ), une protéine qui se lie au PI3P, indispensable aux étapes précoces de l’autophagie et à la lipidation de la protéine cytosolique LC3 ( microtubule-associated protein 1A/1B-light chain 3 , ATG8 chez la levure), marqueur essentiel des autophagosomes [ 3 ]. En situation de stress nutritionnel (un inducteur fort de l’autophagie), cette synthèse de PI3P est assurée par le complexe PI3-kinase de classe III (PI3KC3), principalement composé des protéines VPS15 ( vacuolar protein sorting 15 ), bécline1/ATG6, ATG14L, NRBF2 ( nuclear receptor-binding factor 2 ) et de la kinase lipidique VPS34.
(→) Voir le numéro thématique Autophagie , m/s n° 3, mars 2017, pages 213-340
Le PI3P est également présent sur des vésicules situées à la base du cil primaire des cellules. Le cil primaire est composé d’un corps basal, formé autour du centriole mère, et d’un axonème, dont la structure est organisée par un faisceau de microtubules acétylés [ 4 , 5 ]. L’axonème contient dans sa membrane de nombreux récepteurs qui activent différentes voies de signalisation (Hedgehog, PDGF [ platelet-derived growth factor ], Wnt) en réponse à certaines stimulations chimiques (hormones, facteurs de croissance), des canaux calciques, des transporteurs (glucose), et des mécano-senseurs qui répondent notamment à des forces de cisaillement ou de compression.
Le dialogue entre la machinerie autophagique et le cil primaire a été mis en évidence dans différents types de cellules ciliées, dont les cellules épithéliales rénales, cultivées en l’absence de sérum dans le milieu de culture [ 6 ]. Les forces de cisaillement induisent également le recrutement d’une partie de la machinerie autophagique à la base du cil [ 7 - 9 ]. Cette réponse autophagique « ciliaire » des cellules épithéliales permet leur adaptation aux conditions environnementales induites par les forces de cisaillement, notamment par la modification de leur taille [ 9 ] et la reprogrammation métabolique [ 8 ]. Les résultats de nombreuses études indiquent que la kinase lipidique PI3KC2α, une source alternative de PI3P chez les eucaryotes, participe directement à la ciliogenèse et au maintien des fonctions et de la spécificité du cil primaire [ 10 ]. Dans une étude récente, nous avons montré que les forces de cisaillement ont ainsi un effet direct sur la stabilité et sur la localisation intracellulaire de PI3KC2α dans les cellules ciliées : en effet, sous l’effet de ce stress mécanique et de façon concomitante à la réponse autophagique, PI3KC2α se relocalise à la base du cil primaire et sa synthèse est augmentée [ 11 ].
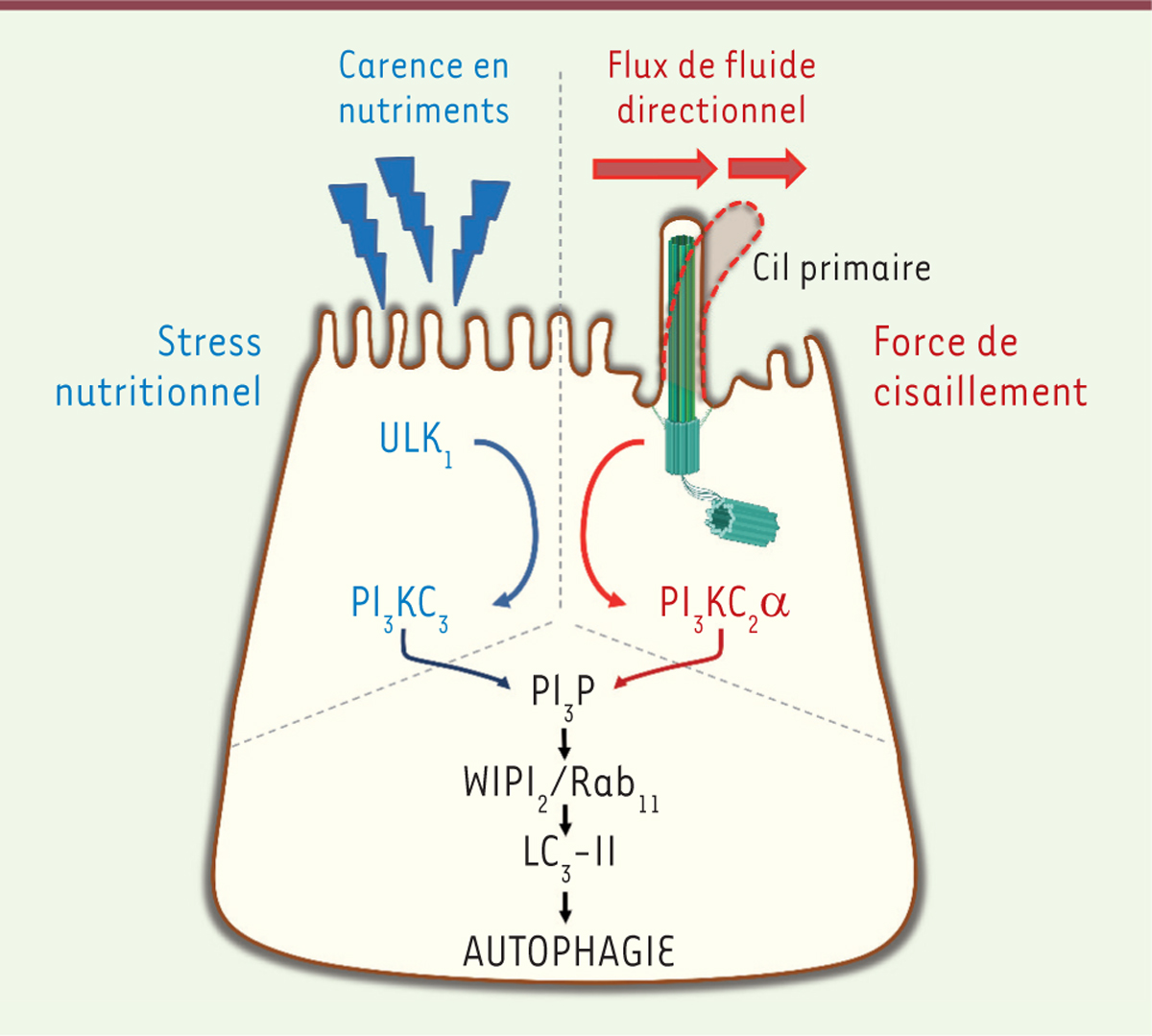
La synthèse d’un pool de PI3P spécifique associé au cil primaire lors de la réponse au stress mécanique induisant une réponse autophagique, elle-même associée au PI3P, suggère une possible implication de la kinase PI3KC2α dans le dialogue entre le cil primaire et la machinerie autophagique. Cette hypothèse est étayée par une précédente étude montrant que la protéine bécline1/ATG6, partenaire de liaison de la kinase VPS34 dans le complexe PIK3C3 lors de la réponse autophagique à un stress nutritionnel, n’est pas recrutée à la base du cil en situation de stress mécanique, à la différence d’un certain nombre d’autres protéines ATG [ 6 ]. Ce constat suggérait que le complexe PI3KC3, dont le rôle est central dans la réponse autophagique canonique, pourrait ne pas être requis lors de l’induction de l’autophagie dépendante du cil primaire en réponse aux forces de cisaillement. Ainsi, l’invalidation du gène codant VPS34 (l’enzyme lipidique du complexe PI3KC3), comme celle du gène codant la bécline1 (son partenaire de liaison), n’a pas d’effet sur la réponse autophagique et la production du pool de PI3P associées au stress mécanique. À l’inverse, l’invalidation du gène codant PI3KC2α, qui n’a pas d’effet sur la réponse autophagique à un stress nutritionnel, inhibe l’apparition du pool de PI3P associé au cil primaire dans les cellules soumises à des forces de cisaillement. En conséquence, la réponse autophagique observée dans cette situation, ainsi que la modification subséquente de la taille cellulaire ne sont plus observées en l’absence de PI3KC2α [ 11 ]. Les résultats de ces expériences indiquent que la réponse autophagique des cellules ciliées aux forces de cisaillement est dépendante de PI3KC2α, et indépendante de PI3KC3. La présence de la protéine WIPI2 au cil primaire suggère cependant que les évènements moléculaires autophagiques en aval de la synthèse de PI3P sont identiques à ceux observés lors de l’autophagie induite par le stress nutritionnel. Cette hypothèse est notamment étayée par l’implication de la protéine Rab11a (une petite GTPase régulant le trafic et la dynamique membranaire des endosomes de recyclage) dans les étapes précoces de l’assemblage de l’autophagosome, via les protéines WIPI2 et ATG16L1, et en liaison avec la présence de PI3P [ 12 ]. Nous avons montré que les protéines Rab11a, WIPI2 et ATG16L1 sont présentes à la base du cil primaire en réponse aux forces de cisaillement. De plus, ces protéines se localisent sur des structures membranaires contenant le PI3P [ 11 ]. La différence entre les réponses autophagiques induites par chacune des deux situations se situe donc bien dans les premières étapes de la formation de l’autophagosome. Cette assertion est corroborée par le fait que les protéines du complexe ULK1 ( Unc-51-like autophagy activating kinase 1 ), qui est à l’origine de la formation de l’autophagosome lors de la carence nutritionnelle, ne sont pas mobilisées lors de la réponse autophagique au stress mécanique ( Figure 1 ) .
| Figure 1.
Mobilisation des lipide-kinases PI3KC3 ou PI3KC2
a
selon le type de stress cellulaire.
Lors de la réponse autophagique à une carence en nutriments (stress nutritionnel), le complexe enzymatique PI3KC3 est mobilisé en aval de la kinase ULK1 (
Unc-51-like autophagy activating kinase 1
) pour la production d’un
pool
de PI3P (phosphatidyl-inositol-3-phosphate). En revanche, lors d’un stress mécanique induit par un flux de liquide directionnel activant le cil primaire, c’est la kinase PI3KC2α qui est responsable d’une production de PI3P sur des vésicules présentes à la base du cil, production qui déclenche à son tour la réponse autophagique induite par les forces de cisaillement. La machinerie en lien avec le PI3P lors de la réponse autophagique (WIPI2 [
WD repeat domain phosphoinositide-interacting protein 2
], Rab11, puis LC3 [
microtubule-associated protein 1A/1B-light chain 3
]) est, quant à elle, identique dans les deux situations.
|
Enfin, nous avons pu constater un défaut de régulation du volume cellulaire dans les cellules des tubules proximaux de reins provenant de souris mutantes Pik3c2a +/- , qui synthétisent une quantité réduite de PI3KC2α. Cette régulation étant directement dépendante de l’axe cil primaire/autophagie en conditions – physiologiques – de flux d’urine primitive à la surface apicale des cellules épithéliales rénales [ 9 ], ce résultat témoigne de l’importance de PI3KC2α dans la réponse autophagique induite par le flux de liquide biologique à la surface des cellules ciliées. Les résultats que nous avons obtenus [ 11 ] nous ont donc conduits à proposer que le pool de PI3P synthétisé par la PI3KC2α est mobilisé spécialement lors d’une situation de stress induit par les forces de cisaillement sur les cellules ciliées ( Figure 1 ) , notamment dans le tubule rénal, où le flux d’urine primitive participe à l’adaptation physiologique de l’épithélium. Dans ce cadre, le complexe PI3KC3 n’est pas requis, sauf à participer à la réponse autophagique en situation de stress nutritionnel.
Ces résultats mettent en lumière la complexité des processus conduisant à la régulation des lipide-kinases mobilisables pour la synthèse de PI3P (principalement PI3KC3 et PI3KC2α) selon le contexte physiologique, et soulèvent la question du rôle de la kinase PI3KC2α dans le contrôle de la machinerie autophagique dans différentes situations de stress. Les fonctions de cette enzyme ne sont en effet pas restreintes aux cellules rénales en réponse aux forces de cisaillement. Elle joue également un rôle important dans différents processus tels que l’angiogenèse et l’adaptation des cellules endothéliales au flux sanguin, ou encore dans la dégranulation des mastocytes. L’implication de la machinerie autophagique dans le contrôle des fonctions cellulaires de la PI3KC2α et de la localisation de son produit de synthèse, le PI3P, est encore mal connue, de même que la régulation de son activité enzymatique au cours de l’autophagie. Ces questions ouvrent de nouvelles voies de recherche pour mieux comprendre le rôle des PI3-kinases et l’adaptation de la réponse autophagique aux diverses situations de stress auxquelles la cellule doit faire face.