| |
| Med Sci (Paris). 37(3): 300–303. doi: 10.1051/medsci/2021008.Les gènes du visage Chroniques génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS ; CoReBio PACA, case 901,Parc scientifique de Luminy
,
13288Marseille Cedex 09
,
France MeSH keywords: Face, Génome, Humains, anatomie et histologie |
J’ai consacré une chronique récente [
1
] (
→
) aux tentatives d’établir le « portrait-robot » d’une personne à partir de l’analyse de son ADN – et souligné que si l’on arrive à peu près à définir ainsi la couleur des yeux, des cheveux et la teinte de la peau, la prédiction de la forme du visage restait peu performante et reposait surtout sur le rattachement à un groupe d’ascendance (« groupe ethnique » selon une terminologie courante mais assez impropre), donnant une idée de l’allure générale du visage. Cela, alors que la ressemblance précise et durable entre jumeaux homozygotes indique qu’il y a bien, pour l’essentiel, détermination génétique. Un des enjeux majeurs pour de telles études est d’arriver à définir de manière objective et quantitative la forme d’un visage. On peut bien sûr isoler une caractéristique, par exemple la taille du nez, et en faire le critère d’une étude d’association pangénomique (GWAS pour
genome-wide association study
), comme cela a été effectué récemment [
2
] en utilisant les données de l’entreprise
23andMe
. On identifie ainsi une douzaine de locus de manière significative, mais on n’accède pas pour autant à une analyse de l’ensemble du visage. Des travaux récents, menés pour l’essentiel par les équipes de Mark Shriver (Université de Pennsylvanie, États-Unis) et de Peter Claes (Université de Louvain, Belgique) ont permis des progrès très significatifs et sont appliqués, non pas (encore ?) à la prédiction de visages, mais à l’identification objective et détaillés des locus gouvernant leur forme [
3
,
4
].
(
→
) Voir la Chronique génomique de B. Jordan,
m/s
n° 8-9, août-septembre 2020, page 813
|
Les 63 éléments d’un visage et leurs corrélats génomiques
La méthodologie, principalement détaillée dans le premier article publié par
Nature Genetics
en 2018 [
3
], ne définit pas
a priori
les éléments constitutifs du visage. Elle part des images stéréographiques digitales des visages des participants (tous européens) qui sont représentés par un maillage de 10 000 points. Après une étape de normalisation (pour corriger les différences de taille, d’âge, de corpulence), ces jeux de 10 000 points sont comparés pour établir lesquels varient de manière corrélée : c’est une étape de classement hiérarchique (
hierarchical clustering
) comme on en pratique souvent dans les études d’expression de gènes. Il s’agit là d’un classement non dirigé, c’est-à-dire sans catégories définies
a priori
. Ce sont les données elles-mêmes qui définissent la manière dont elles sont associées. On voit dans la
Figure 1
, la manière dont les étapes successives décomposent les éléments du visage, depuis le segment 1 (le visage entier) jusqu’à des éléments de petite taille, au cinquième niveau du classement. On a alors une description de la forme d’un visage par un ensemble de 63 segments (1 + 2 + 4 + 8 + 16 + 32) dont le choix résulte des données elles-mêmes, et non d’une décision
a priori
plus ou moins arbitraire. On pourra dès lors utiliser chacun de ces segments pour effectuer une étude d’association génome entier (GWAS) visant à identifier les gènes influant sur sa forme
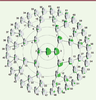 | Figure 1.
Définition des éléments constitutifs d’un visage par classement hiérarchique des coordonnées, effectué sur un ensemble de 2 329 visages.
La décomposition sépare chaque segment d’un niveau en deux segments pour le niveau suivant (image modifiée de la figure 1 de [
3
]).
|
Reste encore à définir, pour chaque segment, la variable dont on va chercher l’association avec des locus au cours de l’analyse GWAS. On n’a pas ici une variable binaire, comme la distinction « atteint/indemne » utilisée en recherche médicale, ou une estimation « grand nez/petit nez », comme précédemment indiqué [
2
]. En fait, et sans entrer dans les détails, les auteurs effectuent pour chaque segment, une analyse de la variation (au sein de l’échantillon) entre les points qui le constituent. Ils l’expriment en termes de composantes principales, et utilisent les composantes majeures comme variables dans l’analyse GWAS. Ils procèdent ainsi à 63 analyses GWAS, portant dans un premier temps sur une cohorte de 2 329 personnes (
discovery sample
), révélant ainsi 38 locus qui sont testés ensuite dans une deuxième cohorte (
replication sample
) comptant 1 719 participants. Quinze locus au total sont confirmés et statistiquement significatifs
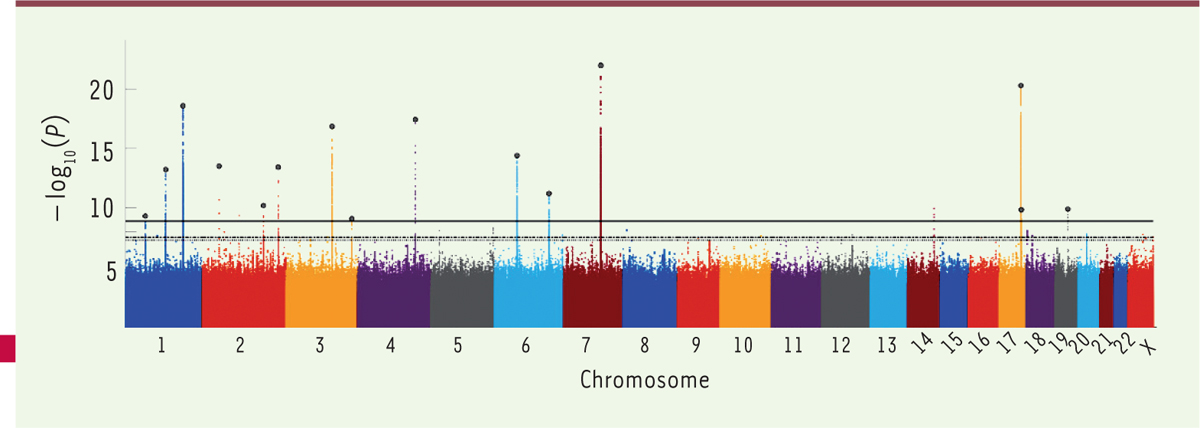
(Figure 2)
. Comme on pouvait s’y attendre, ces locus influencent pour la plupart plusieurs segments : par exemple, le locus majeur situé en 7q21.3 joue sur une dizaine de segments concernant le bas du visage – c’est-à-dire qu’il est retrouvé dans une dizaine de GWAS pratiqués pour ces segments. Sur ces 15 locus, 4 sont complètement nouveaux et 11 avaient déjà été impliqués de manière plus ou moins précise dans la forme du visage. Rappelons que la population étudiée ici est exclusivement européenne.
 | Figure 2.
Manhattan plot montrant les positions de 15 locus (il y a deux pics en 17q24.3) repérés par les 63 GWAS pratiquées pour les segments de visage
(
Figure 1
)
. La ligne horizontale la plus haute représente le seuil de significativité sur l’ensemble de l’étude (extrait partiel et remanié de la figure 3 de [
3
]).
|
L’analyse fonctionnelle est compliquée par le fait que (comme souvent dans les GWAS) la majorité des locus repérés se situe en dehors des gènes proprement dit ; leur effet doit donc porter sur la régulation de l’expression et non sur la séquence codante. Les auteurs utilisent alors la base de données GREAT
1
qui prédit les fonctions de régions de régulation en cis [
5
], et trouvent que ces locus sont associés au développement craniofacial (différenciation des chondrocytes
2,
, développement des os du visage et fente labio-palatine (« bec de lièvre »), suggérant que ces locus influent sur le développement embryonnaire des régions faciales. Une analyse supplémentaire, envisageant les amplificateurs (
enhancers
)
3,
connus, montre que la plupart de ces locus sont situés au voisinage d’
enhancers
actifs dans les cellules de la crête neurale
4
. Au total, cette étude montre que le processus objectif de décomposition du visage en segments permet des études GWAS rigoureuses, confirme des locus déjà connus ou suspectés, et en ajoute de nouveaux dont les caractéristiques sont compatibles avec leur rôle supposé. Bien entendu, cela ne nous dévoile pas le mécanisme précis par lequel le gène ou l’amplificateur présent en 7q24.3 définit la forme du menton.
|
Encore plus fort : 203 locus !
Deux ans plus tard, les deux équipes récidivent avec un article, encore dans
Nature Genetics
[
4
], qui porte cette fois sur 8 246 personnes (toujours européennes) et met en évidence 203 locus dont 120 répondent aux critères les plus stricts. Le principe est le même : représentation de chaque visage par 7 160 coordonnées, classement hiérarchique regroupant ces points en 63 segments, puis analyse GWAS pour chaque segment sur la population étudiée. Ici, le premier échantillon comprend 4 680 personnes provenant des États-Unis (mais d’ascendance européenne), et l’échantillon de réplication est composé de 3 566 citoyens du Royaume-Uni. Le résultat est montré sur la
Figure 3
, un
Miami plot
5
figurant en haut, l’analyse de la première population et, en bas, celle de la deuxième. On voit que les deux images sont très semblables, même si un regard attentif peut déceler quelques différences.
 | Figure 3.
Miami plot des résultats de GWAS sur l’échantillon de population nord-américain (en haut) et britannique (en bas).
Les différences sont minimes (bonne réplication) (extrait de la figure 1 du supplément de [
4
]).
|
Parmi les 203 locus repérés, 150 avaient déjà été impliqués dans la morphologie faciale, ou dans des affections touchant au visage, et 53 sont totalement nouveaux. De plus, les locus repérés ici le sont dans le cadre d’une étude des variations normales du visage, en dehors de toute pathologie. Une analyse plus détaillée de chaque locus permet d’identifier les gènes situés à proximité et aussi de visualiser les segments faciaux influencés par ce locus. On en voit un exemple dans la
Figure 4
, qui montre une image détaillée de la région 14q23.1 et indique les zones du visage concernées par ce locus. Dans cette région se trouve le gène
DACT1 (dapper, antagonist of beta-catenin, 1)
, un des « nouveaux gènes » repérés par cette étude. Ce gène est connu comme antagoniste de la voie de signalisation
Wnt
(
Wingless Integration site
), qui est impliquée dans le développement cranio-facial ; il joue aussi un rôle inhibiteur pour le début de la migration des cellules de la crête neurale, ce qui renforce l’hypothèse d’un rôle dans le développement facial [
6
] (
→
).
 | Figure 4.
À gauche
, zoom sur la position du chromosome 14 révélée par plusieurs analyses GWAS concernant principalement les segments 52, 54 et 55 (voir
Figure 1
). La référence du Snip le plus significatif est indiquée, et les couleurs montrent le degré de déséquilibre de liaison (r
2
) entre celui-ci et chacun des autres snip de la zone. La position sur le chromosome 14 est indiquée en bas.
À droite
, effet du changement de l’allèle mineur à l’allèle majeur de ce locus sur la forme du visage : en bleu dépression, en rouge protrusion (extrait partiel et modifié de la figure 9 de la section « données supplémentaires » de [4]). Snip :
single nucleotide polymorphism
, SNP ou snip.
|
(→) Voir la Nouvelle de G. Couly et E. Dupin,
m/s
n° 6-7, juin-juillet 2019, page 510
D’une manière générale, les Snip
6
présentant les plus forts signaux d’association avec un segment donné concernent principalement la région du nez et celle du front et des yeux : il s’agit là des zones les plus directement influencées par la structure osseuse sous-jacente. L’association est nettement moins forte pour la lèvre supérieure, et intermédiaire pour les joues et le bas du visage. De plus, certains des gènes présents dans les régions identifiées sont impliqués dans le développement des membres et dans ses variations pathologiques, indiquant une architecture génétique partagée entre le visage et les membres.
|
Les auteurs ne s’arrêtent pas à la mise en évidence des locus, et commencent à explorer les contributions de plusieurs locus à un même segment, celles d’un seul locus à plusieurs segments, ainsi que les interactions entre deux ou plusieurs locus qui peuvent indiquer des interactions biologiques entre les produits correspondants. En raffinant ces analyses, ils montrent que, même en l’absence de possibilités d’études fonctionnelles directes (par mutation, délétion [
knock-out
], etc.), un examen poussé de ces données, combiné avec l’exploitation de bases de données spécialisées, permet de progresser dans la compréhension de l’architecture polygénique de structures morphologiques complexes. Pour en revenir à la question du « portrait-robot », il est concevable que ces travaux permettent, à terme, de définir la forme d’un visage à partir d’une analyse d’ADN, mais nous en sommes encore loin. Il reste encore beaucoup de chemin – notons aussi que cette étude porte exclusivement sur des populations européennes – mais la définition objective de la forme d’un visage et la pratique d’analyses GWAS sur les segments ainsi définis représentent un progrès essentiel dans cette direction.
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B
.
ADN et portrait-robot : où en est-on ?
Med Sci (Paris).
2020;
;
36
:
:813.
–
6
.
2.
Pickrell
JK
,
Berisa
T
,
Liu
JZ
,
et al.
Detection and interpretation of shared genetic influences on 42 human traits.
.
Nat Genet.
2016;
;
48
:
:709.
–
717
.
3.
Claes
P
,
Roosenboom
J
,
White
JD
,
et al.
Genome-wide mapping of global-to-local genetic effects on human facial shape.
.
Nat Genet.
2018;
;
50
:
:414.
–
423
.
4.
White
JD
,
Indencleef
K
,
Naqvi
S
, et al.
Insights into the genetic architecture of the human face.
.
Nat Genet.
2020
Dec 7. doi:
10.1038/s41588-020-00741-7
.
5.
McLean
C. Y.
,
Bristor
D
,
Hiller
M
,
et al.
GREAT improves functional interpretation of cisregulatory regions.
.
Nat Biotechnol.
2010;
;
28
:
:495.
–
501
.
6.
Couly
G
,
Dupin
E
.
La crête neurale embryonnaire construit notre identité faciale : le visage des vertébrés.
.
Med Sci (Paris).
2019;
;
35
:
:510.
–
511
.
|