| |
| Med Sci (Paris). 38(4): 374–380. doi: 10.1051/medsci/2022041.L’alphabet génétique élargi Une déviation explorée par la nature chez une famille de bactériophages Pierre-Alexandre Kaminski1* 1Institut Pasteur, Université de Paris, CNRS UMR2001, Biologie des bactéries pathogènes à Gram-positif
,
F-75015
,
Paris
,
France |
Vignette (© Pierre-Alexandre Kaminski).
Chez la plupart des organismes, l’information génétique est codée par quatre bases : adénine (A), guanine (G), cytosine (C) et thymine (T). Cependant, en plus de ces bases canoniques, des bases modifiées sont également retrouvées en grand nombre dans l’ARN, reflétant les fonctions très diverses que les molécules d’ARN remplissent à l’intérieur des cellules. L’ADN contient également des bases modifiées, mais en nombre plus restreint. Nous ne citerons ici que les bases méthylées : m
4
dC, m
5
dC, m
6
dA, hm
5
dU/hmU, et hm
5
dC/hmC
1
, auxquelles nous pouvons ajouter les bases épigénétiques m
5
C, hm
5
C, f
5
C et ca
5
C, identifiées dans l’ADN des organismes supérieurs [
1
].
Chez les procaryotes, la méthylation de l’ADN sur le C-5 (carbone 5) ou le N-4 (azote 4) de la cytosine (m
5
C, m
4
C) et sur le N-6 (azote 6) de l’adénine (m
6
A) joue aussi un rôle important car ces modifications permettent de discriminer l’ADN endogène bactérien de l’ADN exogène (du bactériophage ayant infecté la bactérie) et contribuent à préserver l’intégrité génétique de la bactérie. En effet, dans les systèmes de restriction-modification (RM)
2,
, l’ADN de l’hôte est méthylé par une enzyme, l’ADN méthyltransférase, ce qui le protège de la digestion par l’endonucléase de restriction correspondante. L’ADN du phage envahissant n’est lui pas méthylé et est donc clivé. La méthylation des bases de l’ADN est également impliquée dans l’initiation de la réplication et l’expression des gènes qui, pour la bactérie
Caulobacter crescentus
comme pour d’autres a protéobactéries
3
, dépend du cycle cellulaire [
2
].
|
Les modifications de bases des bactériophages Les bactériophages contiennent des bases méthylées, mais aussi d’autres bases modifiées qui ne se retrouvent dans aucun autre organisme. Ces bases modifiées jouent un rôle dans les stratégies de résistance des bactériophages aux systèmes de restriction bactériens, les systèmes qui permettent aux bactéries de couper l’ADN des virus qui les infectent en fonction de séquences déterminées (et que l’on utilise en biologie sous le terme « enzymes de restriction »). Chez les bactériophages, ces modifications de bases peuvent être pré-réplicatives. Dans ce cas, il s’agit de modifications enzymatiques touchant directement les désoxynucléotides monophosphates (dNMP), les précurseurs de l’ADN. Les dNMP ainsi modifiés enzymatiquement sont ensuite convertis en désoxynucléotides triphosphate (dNTP) modifiés puis incorporés dans l’ADN du bactériophage par une ADN polymérase. Certains bactériophages ayant incorporé dans leur ADN de tels nucléotides modifiés enzymatiquement peuvent les modifier une seconde fois, on parle alors de modification post-réplicative ou d’« hypermodification ». Des modifications touchant les purines (A, G) et les pyrimidines (C, T, U [uracile]) ont été identifiées dans l’ADN des bactériophages, mais ce sont les pyrimidines qui présentent la plus grande diversité.
L’uracile, le 5-hydroxyméthyluracile (hm
5
U), le 5-hydroxyméthyldéoxyuracil (hm
5
dU), l’α-glutamylthymine, l’α-putrescinylthymine, et le 5-dihydroxypentyluracil remplacent ainsi la thymine dans l’ADN du bactériophage SP10 infectant
Bacillus
et du bactériophage FW-14 qui infecte les bactéries du genre
Delftia.
De même, la 5-(2-aminoéthoxy)méthyluridine (
N
e
O
m
5
dU) et la 5-(2-aminoéthyl)uridine (
N
e
5
dU) sont présentes dans l’ADN des bactériophages ViI and M6 [
3
]. Dans les génomes des bactériophages T qui infectent
Escherichia coli,
la 5-méthylcytosine (m
5
C), la 5-hydroxycytosine, la 5-hydroxyméthylcytosine (hm
5
C) et la glucosylhydroxyméthyl cytosine (glucosyl-m
5
C) remplacent la totalité des cytosines [
4
]. Certaines voies de biosynthèses de ces bases ont été décrites. C’est le cas du dU, du hm
5
dU, du
N
e
O
m
5
dU, du
N
e
5
dU, de la hm
5
C et de la glucosyl-m
5
C [
3
,
4
].
La glucosyl-5-hydroxyméthylation de l’ADN constitue un moyen efficace pour le bactériophage d’échapper à l’action de la plupart des systèmes restriction-modification bactériens. Cette modification de l’ADN (retrouvée chez le bactériophage T4) interfère avec les systèmes CRISPR-Cas de type I-E, communément trouvés dans les entérobactéries, et les systèmes CRISPR-Cas de type II-A, dont le représentant le plus connu est CRISPR-Cas9, en diminuant l’affinité des complexes Cascade
4,
et Cas9-crRNA (CRISPR-ARN)
5
pour l’ADN phagique [
5
].
En ce qui concerne les purines, huit modifications ont été décrites jusqu’à présent : la N-carbamoyl-méthyladénine dans l’ADN du bactériophage Mu [
6
], la déoxyarchaéosine dans l’ADN du bactériophage 9d [
7
], la N6-méthyladénine dans l’ADN du bactériophage lambda, la 7-cyano-7-déazaguanine (chez le bactériophage CAjan) [
8
], tous infectant
E. coli,
la 7-méthylguanine chez le bactériophage infectant
Shigella sonnei
[
9
,
10
], la désoxyinosine et la 2′-désoxy-7-amido-7-déazaguanosine (chez le bactériophage infectant Campylobacter) [
11
] et la 2-aminoadénine (ou 2,6-diaminopurine) chez le phage S-2L infectant la cyanobactéries
Synechococcus
[
12
,
13
]. Ces bases sont présentes en proportions variables selon les génomes, pouvant atteindre 100 %, soit un remplacement total de la base canonique correspondante, ce qui est le cas de la 2-aminoadénine qui se substitue à l’adénine dans l’ADN du bactériophage S-2L [
4
,
12
].
Initialement, la 2-aminoadénine, ou base Z, avait été synthétisée en laboratoire et utilisée comme analogue de l’adénine, à la fin des années 1990. Il s’agissait alors d’étudier les propriétés structurales des acides nucléiques. La liaison entre la 2-aminoadénine et la thymine, remplaçant la paire adénine-thymine et apportant une liaison entre bases supplémentaire, s’est alors avérée affecter la flexibilité locale de la molécule d’ADN, la rendant plus rigide [
14
], ce qui empêche son interaction avec les protéines de courbure de l’hélice. De plus, les petites molécules, telles que les antibiotiques ou les anticancéreux, n’accèdent plus à l’ADN ainsi modifié, les empêchant d’agir, montrant ainsi l’importance de cette modification dans la structure de l’ADN [
15
,
16
]. La 2-aminoadénine a ensuite été identifiée chez des bactériophages, dont le cyanophage S-2L, indiquant ainsi que sa présence dans l’ADN phagique est compatible avec un bon fonctionnement des enzymes nécessaires à la réplication de l’ADN, y compris les ADN polymérases.
|
PurZ et les bactériophages S-2L et PhiVC8
Le cyanophage S-2L a été isolé à partir d’échantillons d’eau prélevés à proximité de Leningrad (aujourd’hui Saint Pétersbourg) en 1976. Ce bactériophage possède une capside icosaédrique de 56 nm de diamètre et une queue flexible non contractile de 120 nm de longueur. Il infecte et lyse un nombre restreint de cyanobactéries du genre
Synechococcus
:
S. sp.
698,
Synechococcus elongatus
58 et
S. elongatus
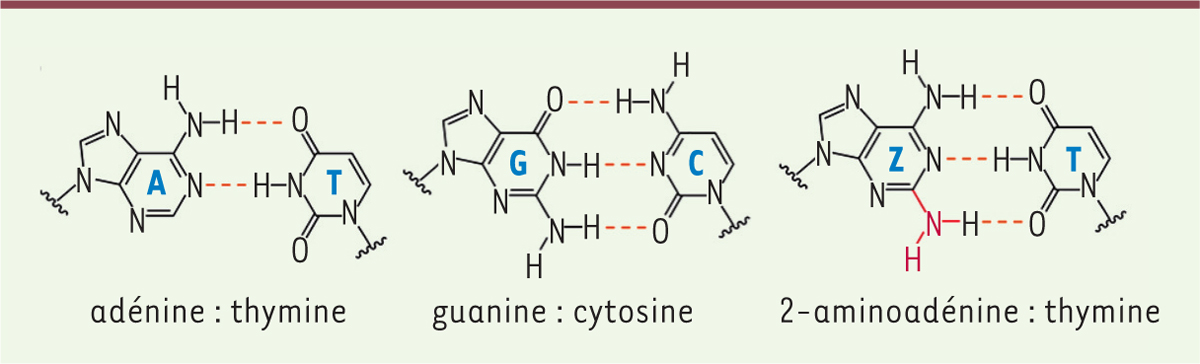
6907. L’analyse de la composition de son ADN a montré que l’adénine en était absente et totalement remplacée par la 2-aminoadénine. La présence de cette base stabilise la structure secondaire de l’ADN car elle forme, grâce à son groupement amine, une troisième liaison hydrogène avec la thymine
(
Figure 1
)
. La 2-aminoadénine confère ainsi au bactériophage une résistance aux enzymes de restriction bactériennes qui reconnaissent des séquences comprenant une adénine : elles ne reconnaissent plus les séquences phagiques contenant la 2-aminoadénine et ne peuvent les couper [
17
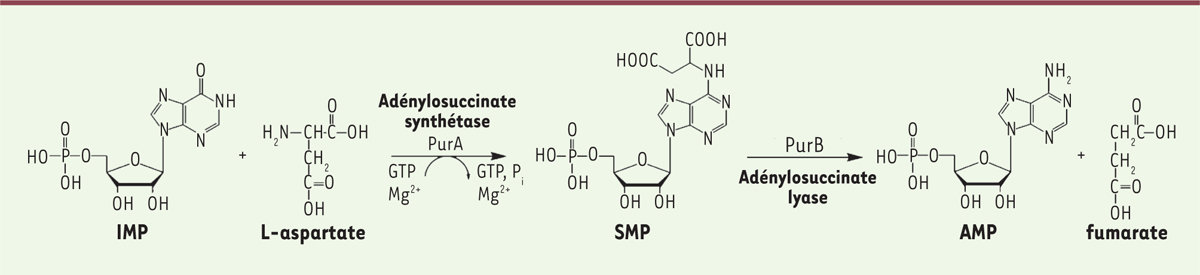
]. L’interférence de la 2-aminoadénine avec les systèmes CRISPR-Cas n’a toutefois pas été établie. Le génome de S-2L a été séquencé, à l’initiative de Philippe Marlière (alors à l’Institut Pasteur), en collaboration avec le Genoscope (GenBank AX955019.1). Son analyse a révélé la présence d’un gène codant une protéine présentant des similitudes avec l’adénylosuccinate synthétase (ou PurA). Chez tous les êtres vivants, PurA catalyse la conversion de l’inosine 5’-monophosphate (IMP) en adénylosuccinate (SMP) en présence de GTP, de magnésium (Mg
2+
) et de L-aspartate. Cette conversion est la première étape de la biosynthèse
de novo
de l’adénosine 5’-monophosphate (AMP) qui sera incorporée dans la séquence d’ADN
(
Figure 2
)
[
18
].
 | Figure 1.
Appariements adénine:thymine, guanine:cytosine et 2,6-diaminopurine (2-aminoadénine):thymine.
|
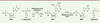 | Figure 2.
Réaction catalysée par l’adénylosuccinate synthétase.
IMP : inosine 5’-monophosphate ; SMP : adénylosuccinate ; AMP : adénosine 5’-monophosphate.
|
La protéine identifiée chez S-2L, l’adénylosuccinate synthétase-
like
protéine, nommée PurZ, pouvait ainsi être la première enzyme de la voie de biosynthèse de la 2-aminoadénine, cette modification étant donc pré-réplicative. En effet, si la 2-aminoadénine était formée après la réplication (modification post-réplicative), une rupture des liaisons hydrogène entre bases et une ouverture de la double hélice seraient nécessaires, ce qui semble peu probable au niveau énergétique.
La comparaison de la séquence du gène codant PurZ avec les séquences référencées dans les banques de données a permis d’identifier des gènes codant des protéines qui présentaient une identité d’environ 40 % avec PurZ dans les génomes de différents bactériophages : PhiVC8, J2, JSF33, VP5, QH et JF15 ; tous ces bactériophages étaient des bactériophages lytiques pour la bactérie
Vibrio cholerae
, responsable du choléra.
PhiVC8 a été isolé à partir d’un échantillon d’eau. Ce bactériophage appartient à l’ordre des
Caudovirales
et à la famille des
Podoviridae.
Il possède une capside hexagonale et une courte queue [
19
]. Son génome est un ADN linéaire double brin de 39 422 paires de bases (pb), qui contient un gène codant PurZ. Il possède une ADN polymérase appartenant à la famille de l’ADN polymérase PolA, ou PolI. La présence du gène codant PurZ et de l’ADN polymérase suggère la présence de la 2-aminoadénine dans l’ADN de PhiVC8, dont la biosynthèse est assurée par le phage, et suggère qu’une ADN polymérase dédiée permet son incorporation.
Les adénylosuccinate synthétases-
like
PurZ de S-2L et de PhiVC8 ne sont cependant pas des équivalents fonctionnels des adénylosuccinate synthétases PurA des archébactéries (archées), des bactéries ou des eucaryotes. Leur expression dans des bactéries dépourvues du gène codant PurA (mutant ∆
purA
d’
E. coli
) ne permet en effet pas de restaurer leur auxotrophie, contrairement aux gènes codant PurA de l’archébactérie hyperthermophile
Pyrococcus
sp. strain ST700 [
20
], du parasite
Plasmodium falciparum
[
21
] ou même de l’homme [
22
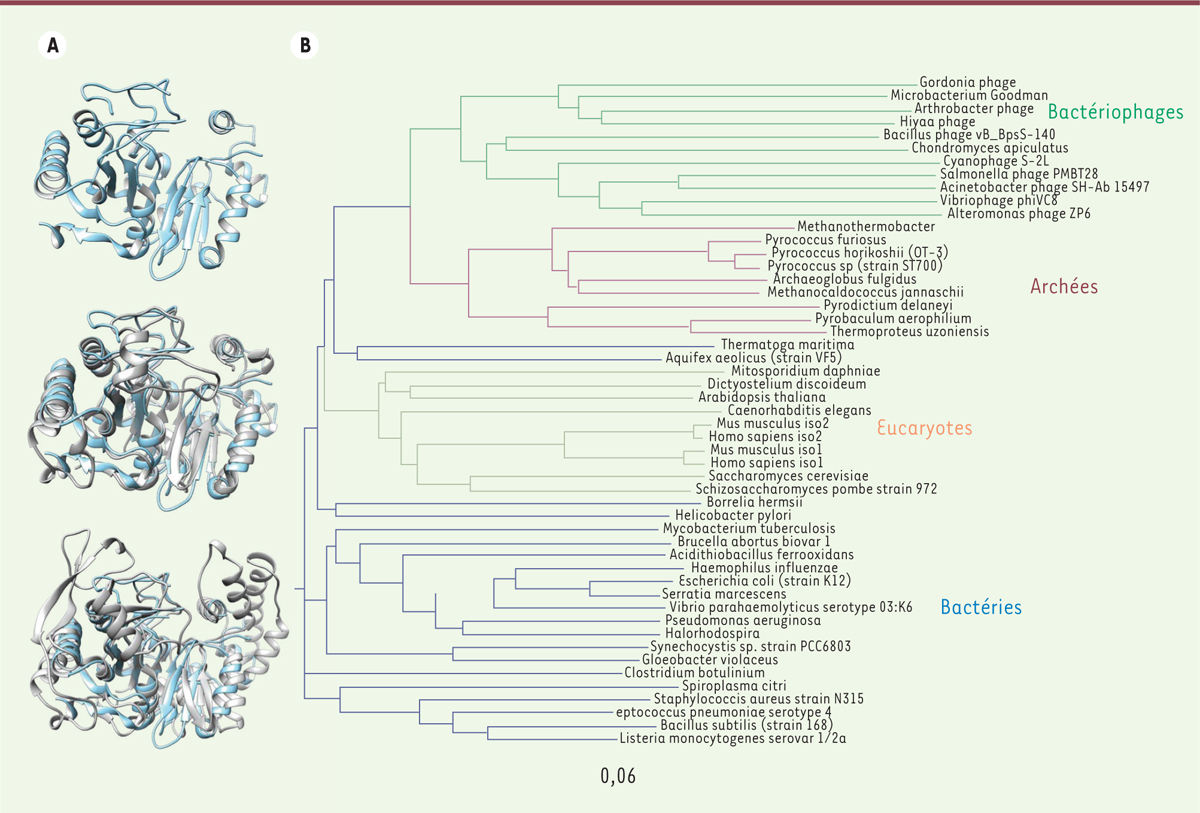
]. La cristallisation de PurZ du bactériophage PhiVC8, sous sa forme apo (inactive), révèle une structure en 3 dimensions (3D) de la protéine qui est identique à celle de PurA de l’archée Pyrococcus horikoshii
(
Figure 3A
)
. Cette similarité structurale entre PurA d’archées et PurZ de bactériophages est retrouvée lorsqu’on examine la phylogénie des adénylosuccinate synthétases. On distingue en effet clairement une branche phylogénique qui regroupe les PurA d’archées et les PurZ de bactériophages, distincte de celle regroupant les PurA des eucaryotes et des bactéries
(
Figure 3B
)
. Il est donc tentant de penser que les PurZ des bactériophages auraient une origine ancienne et que la 2-aminoadénine a été utilisée comme support de l’information génétique au moins depuis la divergence des bactéries infectées par des bactériophages : les actinobactéries, les protéobactéries et les cyanobactéries.
 | Figure 3.
PhiVC8 PurZ ressemble au PurA des archébactéries. A.
Structures de PhiVC8 PurZ apo (en bleu, en haut), de PhiVC8 PurZ superposée à PurA de
P. horikoshii
(en gris, au milieu), et de PhiVC8 PurZ superposée à PurA d’
Escherichia coli
(en gris, en bas).
B.
Arbre phylogénique de quelques adénylosuccinate synthétases représentatives des différents groupes. Les branches des bactériophages sont indiquées en vert, celles des archae en brun, celles des eucaryotes en jaune et celles des bactéries en bleu.
|
Bien que de structure 3D similaire, PurA de
P. horikoshii
et PurZ de PhiVC8 n’étant pas des équivalents fonctionnels, il semblait donc que PurZ de PhiVC8 et PurA de P. horikoshii devaient catalyser la même réaction, mais qu’ils impliquaient des substrats différents. En présence de désoxyguanosine 5’-monophosphate (dGMP), d’adénosine 5’-triphosphate (ATP) et de L-aspartate comme donneur de groupement amine, PurZ (de S-2L ou de PhiVC8) catalyse la formation de N6-succino-2-amino-2’ désoxyadénylate (dSMP), un composé jamais décrit jusqu’à présent. PurZ, qui a été appelé pour cette raison N6-succino-2-amino-2’ désoxyadénylate synthase, a été identifié dans 60 isolats de bactériophages, dont 13 provenant de contigs
6,
de bactériophages dans les métagénomes, suggérant que la base Z (la 2-aminoadénine) est plus largement répandue que ce qui avait été imaginé [
20
].
Les structures de PurZ sous sa forme active, en complexe avec ses différents ligands, ont permis d’identifier les déterminants de la spécificité de substrat. En particulier, la sérine 14 et l’isoleucine 235 de PurZ sont impliquées dans sa spécificité pour le dGMP. Cependant, aucun résidu n’a pu être identifié comme étant spécifique du dGMP par rapport au GMP, ou de l’ATP par rapport au GTP [
23
].
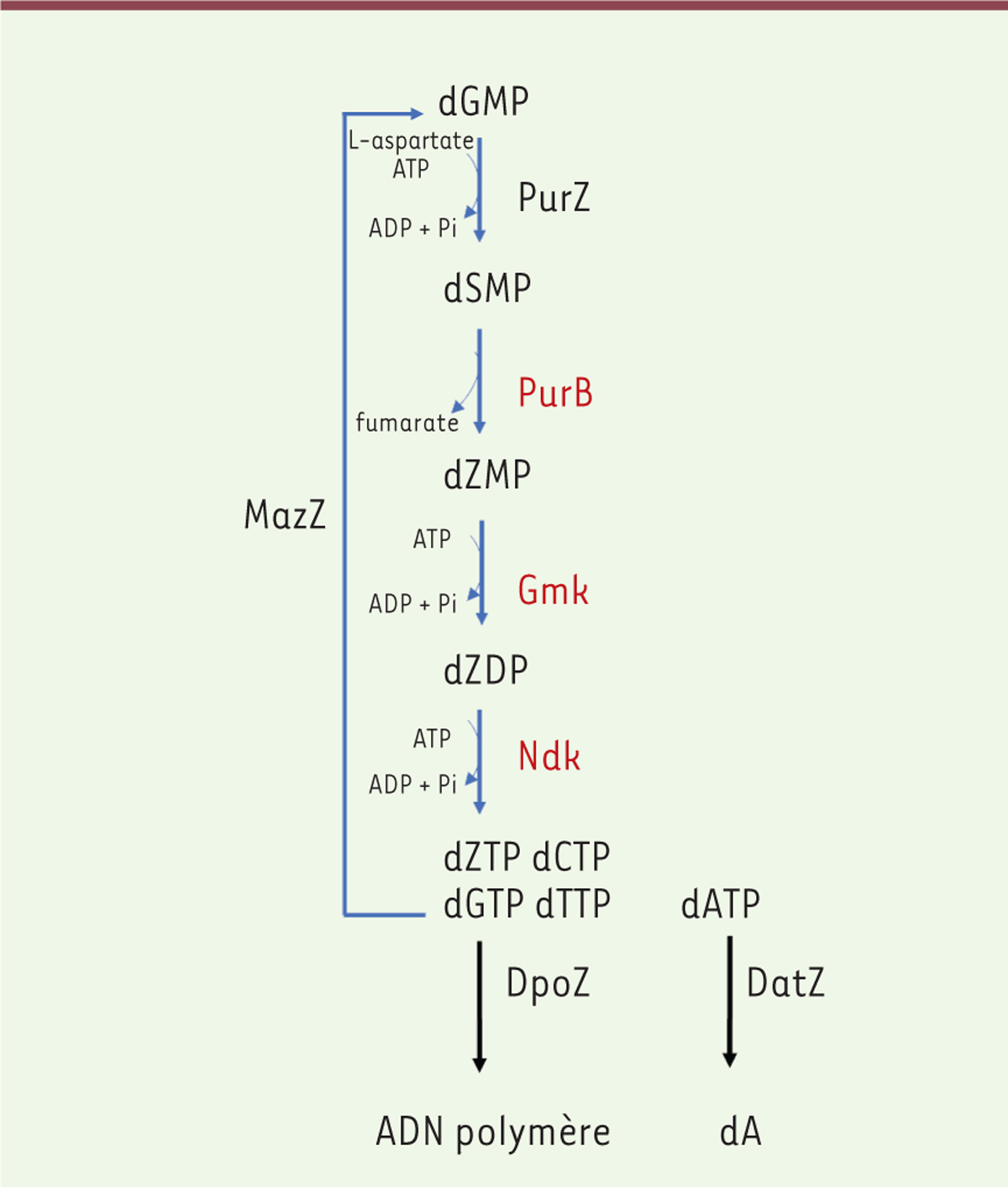
Le génome de PhiVC8 ne contenant pas de gène codant une lyase
7
capable d’hydrolyser le N6-succino-2-amino-2’ désoxyadénylate en 2-amino-2’ désoxyadénylate (dZMP) et fumarate, ni de gène codant une kinase capable de phosphoryler le dZMP, ces activités devaient donc être codées par
V. cholerae
, la bactérie hôte infectée par PhiVC8.
Nous avons cloné, exprimé et purifié les différentes molécules de
V. cholerae
susceptibles de participer à la synthèse de la 2-aminoadénine : l’adénylosuccinate lyase (PurB), la guanylate kinase (Gmk) et la nucléoside diphosphate kinase (Ndk). PurB convertit le dSMP en dZMP ; Gmk et Ndk permettent d’obtenir respectivement les formes diphosphate, dZDP, puis triphosphate, dZTP, du dZMP [
23
,
24
]. Le dZTP ainsi produit est un substrat de l’ADN polymérase I. Pezo
et al.
ont montré par ailleurs que l’affinité de l’ADN polymérase de PhiVC8 (DpoZ) pour le dZTP était supérieure à celle pour le dATP, favorisant ainsi l’incorporation de dZTP dans l’ADN du bactériophage [
25
]. La voie de biosynthèse du dZTP et son incorporation dans l’ADN sont donc désormais connues
(
Figure 4
)
. Comment le dZTP est préférentiellement « choisi » est donc compris. Mais alors, comment le dATP, et donc l’adénine, peuvent-ils être exclus du processus et donc de l’ADN ? Cette élimination du dATP se fait à deux niveaux : avant la réplication, par l’action d’une triphosphohydrolase spécifique du dATP (DatZ) [
20
,
22
] et, après la réplication, par l’activité 3’ exonucléase de l’ADN polymérase DpoZ présente chez la plupart des bactériophages, à l’exception de S-2L [
25
]. L’origine du dGMP, substrat de la N6-succino-2-amino-2’ désoxyadénylate, a été trouvée en caractérisant l’activité de l’enzyme MazZ, une diphosphohydrolase spécifique du dGTP [
20
,
23
]
(
Figure 4
)
.
 | Figure 4.
Biosynthèse de la 2,6-diaminopurine (2-aminoadénine) et exclusion de l’adénine.
dGMP : désoxyguanosine 5’-monophosphate ; dSMP : N6-succino-2-amino-2’ désoxyadénylate ; dZMP : 2-amino-2’ désoxyadénylate ; dZDP : 2-amino-2’ désoxyadénosine 5’-diphosphate ; dZTP : 2-amino-2’ désoxyadénosine 5’-triphosphate ; dATP : 2’ désoxyadénosine 5’-triphosphate ; dCTP : 2’ désoxycytidine 5’-triphosphate ; dGTP : 2’ désoxyguanosine 5’-triphosphate ; dTTP : 2’ désoxythymidine 5’-triphosphate; dA : désoxyadénosine ; ATP : adénosine 5’-triphosphate ; ADP : adénosine 5’-diphosphate ; PurZ : N6-succino-2-amino-2’ désoxyadenylate synthase ; PurB : adénylosuccinate lyase ; Gmk : guanylate kinase ; Ndk : nucléoside diphosphate kinase ; DpoZ : ADN polymérase Z ; DatZ : désoxyadénosine 5’-triphosphohydrolase ; MazZ : désoxyguanosine 5’-diphosphohydrolase.
|
La présence de la 2-aminoadénine n’est pas limitée aux bactériophages S-2L et PhiVC8. PurZ est en effet également présent chez de nombreux bactériophages infectant les bactéries à Gram-négatif, comme l’
Acinetobacter
phage SH-Ab 15497(MG674163), le
Salmonella
phage PMBT 28 (MG641885), l’
Alteromonas
phage ZP6 (MK203850), mais aussi chez des bactériophages lytiques pour des bactéries à Gram-positif, tels l’
Arthrobacter
phage Wayne (KU160672), le
Gordonia
phage Ghobes (KX557278) ou encore le
Streptomyces
phage Hiyaa (MK279841)
(
Figure 5
)
. La relation entre l’expression de PurZ par le bactériophage et la présence de la 2-aminoadénine dans son génome apparaît rationnelle puisque cette base est détectée dans les génomes des
Acinetobacter
phages SH-Ab 15497, Wayne et Ghobes [
24
,
25
] qui expriment PurZ. La plupart des bactériophages possèdent une ADN polymérase DpoZ, à l’exception de S-2L et du
Bacillus
phage vB_BpsS-140, qui codent une sous-unité alpha de l’ADN polymérase III.
 | Figure 5.
Contexte génomique autour de PurZ chez différents bactériophages.
PurZ : N6-succino-2-amino-2’ désoxyadénylate synthase ; PurB : adénylosuccinate lyase ; DatZ : désoxyadénosine 5’-triphosphohydrolase ; MazZ : désoxyguanosine 5’-diphosphohydrolase ; DNA polI : ADN polymérase I ; Prim pol : primase polymérase ; hel : hélicase ; ssap : protéine liant l’ADN simple brin ; VRR-NUC : nucléase ; MarR : régulateur transcriptionnel ; DNA bp : protéine se liant à l’ADN ; dUTPase : désoxyuridine triphosphatase ; adk : adénylate kinase ; RecA-L-exo RecA-like : exonucléase ; RecB-L-exo RecB-like : exonucléase ; DEADH : hélicase de la famille DEAD ; RecA : recombinase A ; sig70 RNA pol : facteur sigma 70 de l’ARN polymérase ; DNA polIII a subunit : sous-unité a de l’ADN polymérase III.
|
Comme pour PhiVC8, les ADN polymérases DpoZ des phages Wayne et Ghobes ont une préférence pour le dZTP par rapport au dATP, et cela, que l’ADN matrice contienne la base Z ou non pour la DpoZ de l’
Acinetobacter
phage SH-Ab 15497 [
25
].
L’exclusion du dATP par DatZ est conservée chez les bactériophages infectant les bactéries Gram-négatif. Chez les bactériophages infectant les bactéries Gram-positif, cette action d’exclusion doit vraisemblablement être réalisée par une enzyme appartenant à la famille des désoxyuridine 5’-triphosphate nucléotidohydrolases (dUTPase). Par contre, la dGTP diphosphohydrolase MazZ est retrouvée sous deux formes différentes chez les bactériophages spécifiques de bactéries Gram-négatif, mais pas chez ceux infectant les bactéries Gram-positif pour lesquelles une autre enzyme, qui reste à identifier, doit intervenir. Notons que le
Gordonia
phage Ghobes et le
Streptomyces
phage Hiyaa possèdent une adénylosuccinate lyase qui leur est propre et qui pourrait être spécifique du dZMP. Chez le
Bacillus
phage vB_BpsS-140, le dZMP pourrait être phosphorylé en dZDP par une adénylate kinase. Chaque bactériophage exprime donc, en plus de PurZ, des gènes différents pour synthétiser le dXTP et éliminer le dATP. Ceux-ci proviennent probablement de transferts horizontaux entre bactériophages.
La voie de biosynthèse de la 2-aminoadénine a été validée
in vivo
chez
V. cholerae
[
23
] mais aussi chez
E. coli,
en exprimant dans la bactérie PurZ, PurZ et MazZ, PurZ et DatZ ou PurZ, DatZ et MazZ. Si l’expression de PurZ seul permet de détourner le métabolisme d’
E. coli
et d’incorporer la 2-aminoadénine en faible proportion dans l’ADN plasmidique (0,6 %) et chromosomique (0,1 %), l’expression conjointe de PurZ, DatZ et MazZ permet d’obtenir des taux de substitution supérieurs, de 4 % dans l’ADN plasmidique et de 12 % dans l’ADN chromosomique [
27
]. Mais au-delà de ce pourcentage, l’incorporation de la 2-aminoadénine devient toxique pour la bactérie. L’obtention d’un génome autre que celui d’un bactériophage, où l’adénine est systématiquement remplacée par la 2-aminoadénine, constitue donc un défi, mais cela devrait permettre de comprendre comment un organisme s’adapte à une modification chimique d’un composant de son alphabet génétique.
|
L’ensemble des travaux que nous avons décrits présente une déviation naturelle de l’ADN qui n’a pas de précédent. Il s’agit d’une illustration de ce que la nature a exploré, mais ne représente certainement qu’une des possibilités pour le stockage de l’information génétique [
28
]. Un nombre considérable de travaux ont été réalisés afin d’obtenir de tels supports avec, par exemple, des analogues de pyrimidines, des analogues de purines, des espèces de pyrimidine auto-agrégeantes qui pourraient avoir une pertinence prébiotique [
29
], des sucres différents du ribose et du désoxyribose, tels le glycérol, le thréose ou même un anhydrohexitol, et, enfin, des modifications de la liaison phosphate [
30
]. Récemment, des systèmes génétiques comportant des bases hydrophobes [
31
], ou construits à partir de huit nucléotides qui forment quatre paires orthogonales ont été synthétisés [
32
]. Ces systèmes, qui sont répliqués et transcrits en ARN, élargissent ainsi le champ des structures moléculaires susceptibles de favoriser la vie, y compris d’autres formes de vie, comme celles que l’on pourrait trouver dans le cosmos.
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Carell
T
,
Brandmayr
C
,
Hienzsch
A
,
et al.
Structure and Function of Noncanonical Nucleobases.
.
Angew Chem Int Ed.
2012;
;
51
:
:7110.
–
7131
.
2.
Adhikari
S
,
Curtis
PD
.
DNA methyltransferases and epigenetic regulation in bacteria.
.
FEMS Microbiol Rev.
2016;
;
40
:
:575.
–
591
.
3.
Lee
Y-J
,
Dai
N
,
Walsh
SE
,
et al.
Identification and biosynthesis of thymidine hypermodifications in the genomic DNA of widespread bacterial viruses.
.
Proc Natl Acad Sci USA.
2018;
;
115
:
:E3116.
–
E3125
.
4.
Weigele
P
,
Raleigh
EA
.
Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses.
.
Chem Rev.
2016;
;
116
:
:12655.
–
12687
.
5.
Vlot
M
,
Houkes
J
,
Lochs
SJA
,
et al.
Bacteriophage DNA glucosylation impairs target DNA binding by type I and II but not by type V CRISPR-Cas effector complexes.
.
Nucleic Acids Res.
2018;
;
46
:
:873.
–
885
.
6.
Swinton
D
,
Hattman
S
,
Crain
PF
,
et al.
Purification and characterization of the unusual deoxynucleoside, alpha-N-(9-beta-D-2’-deoxyribofuranosylpurin-6-yl)glycinamide, specified by the phage Mu modification function.
.
Proc Natl Acad Sci USA.
1983;
;
80
:
:7400.
–
7404
.
7.
Thiaville
JJ
,
Kellner
SM
,
Yuan
Y
,
et al.
Novel genomic island modifies DNA with 7-deazaguanine derivatives.
.
Proc Natl Acad Sci USA.
2016;
;
113
:
:E1452.
–
E1459
.
8.
Hutinet
G
,
Lee
Y
,
Crécy-Lagard
V de
,
et al.
Hypermodified DNA in Viruses of E. coli and Salmonella.
.
EcoSal Plus.
2021;
;
9
:
:eESP00282019.
.
9.
Nikolskaya
II
,
Lopatina
NG
,
Debov
SS
.
Methylated guanine derivative as a minor base in the DNA of phage DDVI Shigella disenteriae.
.
Biochim Biophys Acta.
1976;
;
435
:
:206.
–
210
.
10.
Dunn
DB
,
Smith
JD
.
The occurrence of 6-methylaminopurine in deoxyribonucleic acids.
.
Biochem J.
1958;
;
68
:
:627.
–
636
.
11.
Crippen
CS
,
Lee
Y-J
,
Hutinet
G
,
et al.
Deoxyinosine and 7-Deaza-2-Deoxyguanosine as Carriers of Genetic Information in the DNA of Campylobacter Viruses.
.
J Virol.
2019;
;
93
: :
:e01111.
–
19
.
12.
Kirnos
MD
,
Khudyakov
IY
,
Alexandrushkina
NI
,
et al.
2-aminoadenine is an adenine substituting for a base in S-2L cyanophage DNA.
.
Nature.
1977;
;
270
:
:369.
–
370
.
13.
Khudyakov
IY
,
Kirnos
MD
,
Alexandrushkina
NI
,
et al.
Cyanophage S-2L contains DNA with 2,6-diaminopurine substituted for adenine.
.
Virology.
1978;
;
88
:
:8.
–
18
.
14.
Salerno
D
,
Marrano
CA
,
Cassina
V
,
et al.
Nanomechanics of negatively supercoiled diaminopurine-substituted DNA.
.
Nucleic Acids Res.
2021;
;
49
:
:11778.
–
86
.
15.
Bailly
C
,
Waring
MJ
.
The use of diaminopurine to investigate structural properties of nucleic acids and molecular recognition between ligands and DNA.
.
Nucleic Acids Res.
1998;
;
26
:
:4309.
–
4314
.
16.
Bailly
C
,
Suh
D
,
Waring
MJ
,
et al.
Binding of daunomycin to diaminopurine- and/or inosine-substituted DNA.
.
Biochemistry.
1998;
;
37
:
:1033.
–
1045
.
17.
Szekeres
M
,
Matveyev
AV
.
Cleavage and sequence recognition of 2,6-diaminopurine-containing DNA by site-specific endonucleases.
.
FEBS Lett.
1987;
;
222
:
:89.
–
94
.
18.
Honzatko
RB
,
Fromm
HJ
.
Structure-function studies of adenylosuccinate synthetase from Escherichia coli.
.
Arch Biochem Biophys.
1999;
;
370
:
:1.
–
8
.
19.
Solís-Sánchez
A
,
Hernández-Chiñas
U
,
Navarro-Ocaña
A
,
et al.
Genetic characterization of ØVC8 lytic phage for Vibrio cholerae O1.
.
Virol J.
2016;
;
13
:
:47.
.
20.
Bouyoub
A
,
Barbier
G
,
Forterre
P
,
et al.
The adenylosuccinate synthetase from the hyperthermophilic archaeon Pyrococcus species displays unusual structural features.
.
J Mol Biol.
1996;
;
261
:
:144.
–
154
.
21.
Jayalakshmi
R
,
Sumathy
K
,
Balaram
H
.
Purification and Characterization of Recombinant Plasmodium falciparum Adenylosuccinate Synthetase Expressed in Escherichia coli.
.
Protein Expr Purif.
2002;
;
25
:
:65.
–
72
.
22.
Powell
SM
,
Zalkin
H
,
Dixon
JE
.
Cloning and characterization of the cDNA encoding human adenylosuccinate synthetase.
.
FEBS Lett.
1992;
;
303
:
:4.
–
10
.
23.
Sleiman
D
,
Garcia
PS
,
Lagune
M
,
et al.
A third purine biosynthetic pathway encoded by aminoadenine-based viral DNA genomes.
.
Science.
2021;
;
372
:
:516.
–
20
.
24.
Zhou
Y
,
Xu
X
,
Wei
Y
,
et al.
A widespread pathway for substitution of adenine by diaminopurine in phage genomes.
.
Science.
2021;
;
372
:
:512.
–
6
.
25.
Pezo
V
,
Jaziri
F
,
Bourguignon
P-Y
,
et al.
Noncanonical DNA polymerization by aminoadenine-based siphoviruses.
.
Science.
2021;
;
372
:
:520.
–
4
.
26.
Czernecki
D
,
Legrand
P
,
Tekpinar
M
,
et al.
How cyanophage S-2L rejects adenine and incorporates 2-aminoadenine to saturate hydrogen bonding in its DNA.
.
Nat Commun.
2021;
;
12
:
:2420.
.
27.
Czernecki
D
,
Bonhomme
F
,
Kaminski
PA
,
et al.
Characterization of a triad of genes in cyanophage S-2L sufficient to replace adenine by 2-aminoadenine in bacterial DNA.
.
Nat Commun.
2021;
;
12
:
:4710.
.
28.
Cleaves
HJ
,
Butch
C
,
Burger
PB
,
et al.
One Among Millions: The Chemical Space of Nucleic Acid-Like Molecules.
.
J Chem Inf Model.
2019;
;
59
:
:4266.
–
4277
.
29.
Cafferty
BJ
,
Fialho
DM
,
Khanam
J
,
et al.
Spontaneous formation and base pairing of plausible prebiotic nucleotides in water.
.
Nat Commun.
2016;
;
7
:
:11328.
.
30.
Eremeeva
E
,
Herdewijn
P
.
Non canonical genetic material.
.
Curr Opin Biotechnol.
2019;
;
57
:
:25.
–
33
.
31.
Zhang
Y
,
Ptacin
JL
,
Fischer
EC
,
et al.
A semi-synthetic organism that stores and retrieves increased genetic information.
.
Nature.
2017;
;
551
:
:644.
–
647
.
32.
Hoshika
S
,
Leal
NA
,
Kim
M-J
,
et al.
Hachimoji DNA and RNA: A genetic system with eight building blocks.
.
Science.
2019;
;
363
:
:884.
–
887
.
|